2. 广东顺德创新设计研究院,佛山 528000;
3. 广东省食品质量安全重点实验室 农业部农产品贮藏保鲜质量安全风险评价实验室 华南农业大学食品学院,广州 510642
2. Guangdong Shunde Innovative Design Institute, Foshan 528000;
3. Guangdong Provincial Key Laboratory of Food Quality and Safety, Key Risk Assessment Laboratory of Agricultural Product Preservation, Ministry of Agriculture, College of Food Sciences, South China Agricultural University, Guangzhou 510642
随着分子生物学的快速发展,基因组水平研究已经成为热点。无论是基因组学分析还是基因文库的建立都需要提取出血液中的全基因组DNA[1]。而提取得到的DNA的浓度、纯度以及一级结构的完整性会影响到后续分子生物学的研究。传统的核酸提取方法大多利用离心、沉淀和色谱等方法,这些方法普遍存在操作时间长、过程复杂和用到有毒试剂等缺点[2]。故建立快速、高效、稳定且安全的DNA提取方法是分子生物学研究迫切需要的。
表面修饰的磁性纳米粒子作为新型的功能材料,具有磁响应性和生物安全性[3]。在生物医药方面已得到广泛应用。磁珠法提取核酸是利用磁性纳米粒子与核酸结合,在外磁场的作用下达到分离纯化核酸的目的[4]。
本研究通过对自制的硅羟基磁珠提取全基因组DNA的影响因素考察。优化Fe3O4@SiO2纳米粒子提取全血DNA过程,采用核酸提取浓度、OD260/280及OD260/230三参数进行评价,探讨磁珠量、裂解液、缓冲液及洗脱温度对DNA提取效果的影响。同时,本试剂盒与商业化试剂盒进行性能比对,旨为快速提取基因组DNA方法提供最优方案。
1 材料与方法 1.1 材料新鲜冷冻的血液样品保存于-80℃超低温冰箱;自制试剂盒;商业化磁珠法基因组DNA提取试剂盒购于广州飞扬生物科技有限公司;蛋白酶K购于美国Merck公司;盐酸胍购于上海赛百盛基因技术有限公司;Tris-base购于Biosharp公司;EDTA-Na2,TESO,NaAc,PEG4000购于健阳生物科技有限公司;其他试剂均为国产分析纯。TENSOR27红外光谱仪(美国布鲁克光谱仪器公司);VSM振动样品磁强计(美国Quantum Design公司);NanoDrop2000超微量分光光度计(美国THERMO公司)。纳米粒度分析仪Zetasizer Nano ZS(英国Malvern公司)。
1.2 方法 1.2.1 硅羟基磁珠的制备与表征根据Geng等[5]的研究和预实验最终确定制备过程为将5 mmol FeCl3·6H2O溶于40 mL乙二醇中,随后加入3.6 g NaAc和1.0 g PEG4000,剧烈搅拌至完全溶解。将混合溶剂转移至100 mL反应釜中,在200℃反应8 h,所得沉淀分别用去离子水和乙醇交替洗涤3次。40℃真空干燥12 h,即得到磁性Fe3O4纳米粒子。取部分样品用灭菌超纯水制备成100 mg/mL的溶液,备用。
称取300 mg水热合成法的磁性Fe3O4纳米粒子溶于150 mL 0.1mol/L的HCl中,超声分散10 min,去离子水洗涤两次后,加入240 mL乙酸,超声分散10 min,随后转入三口烧瓶中,缓慢滴加3 mL 28%的浓氨水,称取112 g的TESO置于其中,常温下剧烈搅拌8 h。静置后倒去上层液体所得沉淀分别用去离子水和乙醇交替洗涤3次。40℃真空干燥12 h,即得到磁性Fe3O4@SiO2粒子纳米粒子。取部分样品用灭菌超纯水制备成100 mg/mL的溶液,备用。
磁珠的表征采用红外光谱测试:采用KBr压片法,扫描范围;磁学性能测试:采用振动样品磁强计在298K条件下测定样品的磁学性能。
1.2.2 DNA提取实验步骤取250 μL血液样品到2 mL EP管中,加入25 μL蛋白酶K溶液和适量自制裂解液震荡混匀,65℃孵育15 min;放冷至室温后,沿管壁加入350 μL无水异丙醇立即混匀,然后加入适量体积100 mg/mL磁珠溶液充分振荡,置于磁力架上磁分离,吸除废液;加入适量自制缓冲液混匀,磁分离吸除液;加入800 μL 80%乙醇漂洗,磁分离吸除液体重复漂洗1次,室温晾干后加入50 μL ddH2O洗脱,一定温度条件下水浴10 min,置于磁力架吸附吸出液体得到DNA提纯液体。
1.2.3 核酸提取实验的条件优化通过单因素实验,先后优化磁珠量、裂解液中胍盐浓度、缓冲液体系及洗脱温度,这4个工作条件。采用核酸提取浓度、OD260/280及OD260/230三参数进行评价,当OD260/230在1.8-1.9,OD260/230大于2.0,同时核酸提取浓度越大,说明所提取的核酸纯度高,浓度大,提取效果好。
1.2.3.1 磁珠量的优化自制磁珠粒径大约200 nm,浓度为100 mg/mL,为了考察磁珠用量对全基因组DNA提取效果的影响,实验分别考察5、10、15、20、25和30 μL自制磁珠对核酸提取的效率进行优化。使磁珠用量分别为0.5、1.0、1.5、2.0、2.5和3.0 mg六组,每组设置3个平行对照。
1.2.3.2 缓冲液的优化缓冲液在提取磁珠中是分离和分散磁珠和絮状沉淀的重要一步,本实验采用60 mmol/L盐酸胍缓冲液(pH7.5) 与一定体积无水乙醇配比作为缓冲液,并对缓冲液进行制备和优化配制,实验分别考察含无水乙醇体积比为30%、40%、50%、60%、70%和80%的缓冲液液对核酸提取的效率进行优化。每组实验设置3个平行对照。
1.2.3.3 裂解液的优化本实验采用高浓度低pH盐酸胍溶液(pH4.9) 来充分裂解血液细胞,为了考察裂解液对全基因组DNA提取效果的影响,在实验其他步骤保持不变的情况下,实验分别考察含3、3.5、4、4.5和5 mol/L的盐酸胍裂解液对核酸提取的效率进行优化。每组实验设置3个平行对照。
1.2.3.4 洗脱温度的优化对于磁珠法提取DNA在加入洗脱液洗脱核酸并将EP管放入恒温水浴锅分别以应用温度分别为35℃、50℃、65℃和80℃,对其进行洗脱前的温度处理,应用nanodrop2000测得核酸提取效率判断温度对核酸提取的影响。每组实验设置3个平行对照。
2 结果 2.1 硅羟基基功能化磁性复合颗粒的表征对自制磁珠和硅羟基磁珠同时采用KBr压片法红外光谱测试,结果(图 1-A)显示,上方波形为裸磁珠红外光谱,下方的波形为硅羟基修饰的磁珠,相比两个波形在586 cm-1和1 224 cm-1均有很明显的吸收峰。对自制裸磁珠和硅羟基磁珠分别通过振动样品磁强计(VSM)测得数据(图 1-B)显示,Fe3O4和Fe3O4@SiO2空心微球的饱和磁化强度分别为88.90 emμ/g和75.95 emμ/g。应用马尔文粒度仪检测,结果(图 1-C)显示,硅羟基磁珠光强峰值在527 nm,粒径主要分布在450-550 nm,平均粒径为574 nm。

|
| 图 1 羧基功能化纳米磁珠的表征 A:红外吸收光谱;B:磁滞回线图;C:硅羟基磁珠粒径分布图 |
自制羧基功能化磁珠的浓度为100 mg/mL,在充分混匀的情况分别加入5、10、15、20、25和30 μL得到结果如图 2-A,其中在5-15 μL区间,核酸浓度随加入磁珠的量增加而增大,但在15-30 μL范围内,浓度差异并不明显,几乎趋于平稳,对于OD260/280和OD260/230在磁珠量低于15 μL时趋于稳定,分别保持在1.90-1.91和2.10-2.20之间,而当磁珠量大于15 μL后,OD260/280和OD260/230急剧下降。同时,对提取的样品进行琼脂糖凝胶电泳检测,结果如图 2-B所示。
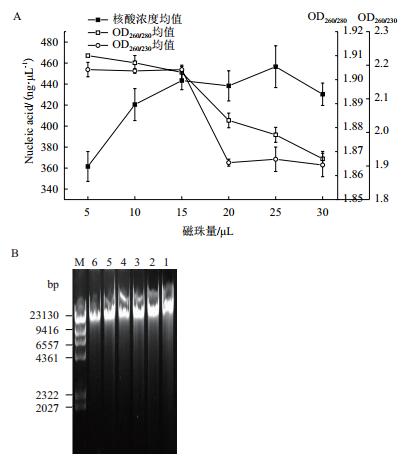
|
| 图 2 试剂盒磁珠量优化 A:磁珠量对提取效率影响差异图;B:磁珠量优化电泳图(1-6:磁珠量为5-30 μL) |
对自制核酸提取试剂盒的缓冲液进行优化,对盐酸胍缓冲液与乙醇混合比例30%-80%等差递增条件进行了实验,采用平均核酸浓度、OD260/280及OD260/230三参数进行评价。所得各项数据如图 3-A所示,当乙醇在缓冲液中占60%时,样本的平均核酸提取浓度最高,且OD260/280为1.90,OD260/230为1.93。同时,对提取的样品进行琼脂糖凝胶电泳检测,结果如图 3-B所示。

|
| 图 3 试剂盒缓冲液乙醇含量优化 A:缓冲液乙醇含量对提取效率影响差异图;B:缓冲液乙醇含量优化电泳图(1-6:缓冲液含乙醇比例分别为30%-80%) |
实验分别考察含3、3.5、4、4.5和5 mol/L的盐酸胍裂解液对血液进行裂解并提取核酸,结果如图 4所示,其中核酸浓度随裂解液中盐酸胍浓度的增加而增大,其浓度最大值表现在盐酸胍浓度为5 mol/L时,对于核酸纯度值OD260/280和OD260/230在盐酸胍浓度高于4 mol/L后趋于稳定,分别保持在1.89-1.90和2.00-2.05之间(图 4-A)。同时,对提取的样品进行琼脂糖凝胶电泳检测,结果如图 4-B所示。

|
| 图 4 试剂盒裂解液浓度优化 A:裂解液浓度对提取效率影响差异图;B:裂解液浓度优化电泳图(1-5:裂解液的浓度分别为3-5 mol/L) |
在核酸提取的最后一步,加入洗脱液后我们对其进行水浴处理,应用温度分别为35℃、50℃、65℃和80℃,结果(图 5) 显示,核酸浓度在洗脱温度为65℃是的值最大,而在80℃的值最小,而对于核酸纯度OD260/280数值稳定在1.90左右,OD260/230在温度升高时有明显的降低趋势,但其数值均保持在2.15-2.25之间(图 5-A)。同时,对提取的样品进行琼脂糖凝胶电泳检测,结果如图 5-B所示。
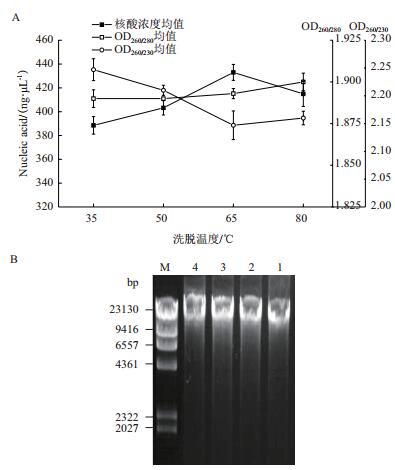
|
| 图 5 试剂盒核酸洗脱温度优化 1-4:洗脱温度分别为35℃、50℃、65℃、80℃;A:核酸洗脱温度对提取效率影响差异图;B:核酸洗脱温度优化电泳图 |
整合上述单因素实验结果的最优工作条件,即磁珠量为1.5 mg,裂解液为含5 mol/L的盐酸胍溶液(pH4.9),缓冲液体系为含60%无水乙醇的60 mmol/L盐酸胍缓冲液(pH7.5),洗脱温度为65℃,作为自制试剂盒的最优提取方案,当提取血量为250 μL时,DNA提取得率为20 μg左右。并与商业化试剂盒比较,结果如表 1所示,其数据差异均满足统计学差异(P < 0.05),并对样品进行琼脂糖凝胶电泳(图 6)。

|
| 图 6 omega试剂盒与自制试剂盒对比 1-3:OMEGA试剂盒;4-6:自制硅羟基磁珠试剂盒 |
综合上述自制试剂盒的最优提取方案,取20 μg的全基因组DNA,采用本试剂盒提取方案,进行3组平行实验,最后所得核酸产量为19.04±0.13,OD260/280为1.82,OD260/230为2.03,最终所得样品富集效率为88.7%-95.9%。
3 讨论在碱性条件下(pH值为9-10),在水介质中使用氯化铁和尿素,可合成Fe2O3纳米粒子[6]。与之相近应用溶剂热法也可以很高效率的制备Fe3O4纳米微粒[7]。本研究中应用水热法制备Fe3O4纳米微粒,并对其表面进行硅羟基修饰,得到可应用于核酸提取的硅羟基纳米磁珠。
细胞核酸的提取是分子生物学重要的一步。传统的几种提取DNA的方法包括:煮沸法、盐析法、酚-氯仿法、异丙醇沉淀方法、滤膜离心柱法[8, 9]。Qie等[10]应用聚酰胺-胺型树枝状高分子PAMAM对DNA的结合效应将其修饰在铁磁性纳米粒子表面,应用修饰后的粒子提取DNA,此方法较为新颖。对各种传统方法进行比较证明磁珠修饰法的提取效率高及过程较为便捷[11]。本实验应用自制的纳米磁珠表面修饰硅羟基进行核酸提取,并对裂解液浓度、缓冲液体系、磁珠量、洗脱温度进行优化。实验结果表示4个因素均对核酸的提取效率有影响。对于实验结果用紫外分光光度计进行定量检测,纯DNA的OD260/280比值约为1.8,正常值为1.8-2.0。该比值小于1.6时表示样品有蛋白质污染,同时当比值大于2.0时,则有RNA污染。而OD260/230表示DNA中的杂质盐离子,正常的比值应大于1.8[12]。
3.1 硅羟基磁珠的表征由图 1-A可知,1 224 cm-1是O-Si-O的伸缩振动峰,其中3 426.0 cm-1是Fe3O4及SiO2表面的-OH峰和Fe-O的特征吸收峰发生重叠,同时586 cm-1和1 224 cm-1处的峰也说明Si和Fe3O4表面的-OH发生反应,从而证实包覆过程已发生[13]。对于振动样品磁强计的测量结果(图 1-B)可看出在硅羟基包裹下磁珠磁力为75.95 emμ/g相比裸磁珠88.90 emμ/g有明显的减弱,且均具有超顺磁性[14]。Fe3O4@SiO2空心微球在磁场条件下由良好的磁响应性,这一性质对磁性离子在生物分离等领域中的应用非常重要。
3.2 磁珠量优化在对于全血提取DNA的优化试验中,Ling等[15]对提取磁珠量进行优化实验,但实验中仅仅取了TOYOBO公司的试剂盒磁珠40 μL和80 μL两个样本,对于实验优化没有一个较长的梯度现象。对于我们自制的硅羟基磁珠的浓度为100 mg/mL,在加入磁珠从5 μL到15 μL的过程中可以明显看到核酸浓度呈上升的趋势,且纯度比值趋于平衡,15 μL以后磁珠浓度有少量的增加,而OD260/280和OD260/230有明显的下降,表示不一定磁珠越多提取效率越高,过量的磁珠会影响后面的缓冲和漂洗过程,阻碍了对盐离子的漂洗,造成样品有盐离子污染,同时也对蛋白酶K溶解蛋白质有一定的影响,故OD260/280出现走低趋势。由此提取中出于核酸纯度和经济效益的关系,磁珠量选优结果为15 μL的磁珠。
3.3 缓冲液优化在核酸提取的过程中,在加入磁珠除废后加入缓冲液,主要是起到分散磁珠和纯化DNA的作用,并有效的分散絮状沉淀[16]。并且这个步骤在实验中需重复两次,对磁珠法提取核酸的效率有很大的影响,从图 3-A中乙醇比例在60%时达到了峰值,且OD260/280均值为1.907±0.006和OD260/230均值为1.933±0.015也几乎位于最高点,图 3-A中泳道1,2,3,6均有拖带现象,证明核酸纯度不够。对于OD260/230仅在乙醇比例为60%比值大于1.8,其他位置均存在盐离子污染,所以缓冲液乙醇比例为60%时提取效率最优。
3.4 裂解液优化Shan等[17]在优化实验中得出裂解液中盐酸胍浓度对核酸提取效率的影响较大,图 4-B中裂解液浓度较低时核酸浓度也较低,且样品OD260/230低于1.8有盐离子污染,并且OD260/280的比值也较低,随裂解液浓度的增加明显可见核酸浓度和纯度比值均随其增加而增大,在盐酸胍浓度为4 mol/L时,纯度比值OD260/280为1.883±0.023、OD260/230为2.023±0.078,且随盐酸胍浓度增加趋于稳定,均在正常纯度范围之内。而核酸浓度在4.5 mol/L时为417.10±8.32,在5 mol/L时为444.00±11.62其差异并不大,在图 4-B的电泳图中345泳道的亮度明显高于12,出于高效率而言5 mol/L是实验的最优条件,而出于经济考虑,采用4.5 mol/L的盐酸胍溶液性价比更高。
3.5 洗脱温度优化从图 5-B结果可看出洗脱温度较其他因素对核酸的提取效率影响较小,随着温度增加,核酸提取效率有一定的提高,但核酸纯度值OD260/280和OD260/230均趋于平衡,且在较好的纯度值范围内,洗脱温度在80℃时核酸浓度有些许的降低,表明温度过高会破坏样品中的核酸。故如需要短时间完成提取则不需要在洗脱过程中加热,以便于节省时间且对核酸浓度影响不大,如需要更高的提取浓度,则在洗脱过程中需要在65℃水浴10 min,以得到最好的核酸提取效率。
3.6 试剂盒对比根据实验数据表明自制的试剂盒核酸浓度为438.63±18.28提取效率优于商业化的试剂盒提取的核酸浓度324.90±13.11。并且OD260/230为1.87,OD260/230达到2.203,即提取纯度很高,所有数据均满足统计学差异(P < 0.05)。
4 结论对磁珠法提取DNA的过程,对裂解液、缓冲液、磁珠量、洗脱温度进行大量实验,得到当裂解液浓度为5 mol/L,缓冲液乙醇含量为60%,磁珠量为15 μL,洗脱温度为65℃时,自制硅羟基纳米磁珠的提取效率最高,且优于商业化试剂盒,当提取血量为250 μL时,DNA提取得率为20 μg左右,样品富集效率为88.7%-95.9%。
| [1] | Ormond KE, Wheeler MT, Hudgins L, et al. Challenges in the clinical application of whole-genome sequencing[J]. Lancet, 2010, 375 (9727): 1749–1751. DOI:10.1016/S0140-6736(10)60599-5 |
| [2] | LI XX, Zhao HY, Yang YT, et al. Comparing the three methods to extract human genomic DNA from whole blood[J]. Progress in Modern Biomedicine, 2013, 13 (27): 5221–5225. |
| [3] | Sadeghi S. Surface modified magnetic Fe3O4 nanoparticles as a selective sorbent for solid phase extraction of uranyl ions from water samples[J]. Journal of Hazardous Materials, 2012, 215-216 (2): 208–216. |
| [4] | Xiao Y, Wang CH, Li X, et al. Research on magnetic separation methods for the extraction of nucleic acids[J]. Advanced Materials Research, 2013, 662 (2): 343–347. |
| [5] | Geng MX, Liu FT. Preparation of Fe3O4 nanoparticles by hydro-thermal method[J]. Journal of University of Jinan, 2009, 8 (4): 401–404. |
| [6] | Rahman M, Jamal A, Khan S, et al. Characterization and applications of as-grown -Fe2O3 nanoparticles prepared by hydrothermal method[J]. Journal of Nanoparticle Research, 2011, 13 (9): 3789–3799. DOI:10.1007/s11051-011-0301-7 |
| [7] | Li G, Shen B, He N, et al. Synthesis and characterization of Fe3O4@SiO2 core-shell magnetic microspheres for extraction of genomic DNA from human whole blood[J]. Journal of Nanoscience & Nanotechnology, 2011, 11 (12): 10295–10301. |
| [8] | Tang JN, Zeng ZG, Wang HN, et al. An effective method for isolation of DNA from pig faeces and comparison of five different methods[J]. Journal of Microbiological Methods, 2008, 75 (3): 432–436. DOI:10.1016/j.mimet.2008.07.014 |
| [9] | Muñoz-Cadavid C, Rudd S, Zaki SR, et al. Improving molecular detection of fungal DNA in formalin-fixed paraffin-embedded tissues:comparison of five tissue DNA extraction methods using panfungal PCR[J]. Journal of Clinical Microbiology, 2010, 48 (6): 2147–2153. DOI:10.1128/JCM.00459-10 |
| [10] | Qie F, Zhang G, Hou J, et al. Extracting genomic DNA of foodstuff by polyamidoamine(PAMAM)-magnetite nanoparticles[J]. Talanta, 2012, 93 (2): 166–171. |
| [11] | Witt S, Neumann J, Zierdt H, et al. Establishing a novel automated magnetic bead-based method for the extraction of DNA from a variety of forensic samples[J]. Forensic Science International Genetics, 2012, 6 (5): 539–547. DOI:10.1016/j.fsigen.2012.01.002 |
| [12] | Wilfinger WW, Mackey K, Chomczynski P. Interpretation of nucleic acid OD260/280 ratios[J]. Protocols and Product Manuals, 2012, 123 (1): 1–13. |
| [13] | Sun J, Yu G, Liu L, et al. Core-shell structured Fe3O4@SiO2 supported cobalt(Ⅱ)or copper(Ⅱ)acetylacetonate complexes:magnetically recoverable nanocatalysts for aerobic epoxidation of styrene[J]. Catalysis Science & Technology, 2014, 4 (5): 2496–2499. |
| [14] | Mikhaylova M, Kim DK, Bobrysheva N, et al. Superparamagnetism of magnetite nanoparticles:dependence on surface modification[J]. Langmuir, 2004, 20 (6): 2472–2477. DOI:10.1021/la035648e |
| [15] | Ling J, Wang H, Zhang S, et al. Optimization of genomic DNA extraction with magnetic bead-based semi-automatic system[J]. Journal of Zhejiang University Medical Sciences, 2012, 41 (3): 320–326. |
| [16] | Li G, Shen B, He N, et al. Synthesis and characterization of Fe3O4@SiO2 core-shell magnetic microspheres for extraction of genomic DNA from human whole blood[J]. Journal of Nanoscience & Nanotechnology, 2011, 11 (12): 10295–10301. |
| [17] | Shan Z, Wu Q, Wang X, et al. Bacteria capture, lysate clearance, and plasmid DNA extraction using pH-sensitive multifunctional magnetic nanoparticles[J]. Analytical Biochemistry, 2010, 398 (1): 120–122. DOI:10.1016/j.ab.2009.11.006 |





