蛋白激发子是一类能激发植物获得性抗性,增强植物自身免疫的蛋白质,在植物保护上发挥着重要作用[1-4]。激发子诱导植物产生抗性的过程主要包括植物受体对激发子的识别、信号传导,以及下游抗性相关基因表达调控等[5-7]。植物对激发子的识别是触发植物免疫信号传导的开关,候选互作蛋白的鉴定与功能研究将为深入了解植物与病原微生物的互作、揭示蛋白激发子诱导植物产生抗性的作用机制奠定坚实的基础。目前,研究比较清楚的激发子受体仅有少数,如细菌鞭毛蛋白(Flagellin)flg22的受体FLS2(Flagellin-sensing 2),细菌转录延伸因子Tu(EF-Tu)elf18的受体EFR,以及水稻上几丁质受体等[8-11]。许多蛋白激发子与植物的互作蛋白尚未鉴定。
真菌蛋白激发子Hrip1(Hypersensitive response inducing protein 1),是本实验室从极细链格孢菌中分离纯化出了一种分子量为17 562.5 Da新型超敏反应诱导蛋白。利用快速扩增cDNA末端反应(RACE)克隆了Hrip1的编码基因(GenBank accession number HQ713431)。Hrip1在烟草上引发了类似于典型超敏反应的坏死斑,DNA laddering在内的细胞凋亡反应,提高烟草对TMV的系统抗性;同时能诱导烟草中NO和ROS的产生,激活水杨酸途径的蛋白激酶以及多种防御相关基因的表达[12]。Hrip1转基因拟南芥对盐和干旱胁迫的抗性显著增强,说明蛋白激发子Hrip1基因在拟南芥中的表达能够改善和提高植株的耐盐抗旱能力[13]。此外,Hrip1能显著提高水稻对稻瘟菌的抗性,诱导抗性基因和蛋白的上调表达(另文发表),为了阐述Hrip1诱导水稻的抗性机制,本研究利用酵母双杂交筛选Hrip1在水稻中的互作蛋白,同时构建互作蛋白的原核表达体系,以期为研究水稻对蛋白激发子Hrip1的识别机制以及Hrip1互作蛋白的进一步鉴定奠定基础。
1 材料与方法 1.1 材料日本晴(Oryza sativa ssp. japonica)种子和原核表达载体pGEX-6P-2由本实验室保存。水稻cDNA文库、酵母菌株Mav203、酵母双杂交载体pDBleu(BD)和pPC86(AD)由中国农业科学院植物保护研究所王国梁研究员惠赠。酵母Minimal SD Base、SD/-Trp、SD/-Leu、SD/-Trp/-Leu、SD/-His/-Trp/-Leu、x-a-gal等购自Clontech。大肠杆菌表达感受态细胞BL21、ProteinIso® GST Resin、Western blot Kit等购自北京全式金生物技术有限公司。
1.2 方法 1.2.1 诱饵载体的构建以本实验室保存的pet32-Hrip1质粒为模板扩增Hrip1片段,用限制性内切酶Nco Ⅰ和Not Ⅰ对Hrip1片段和pDBleu载体(包含BD结构域)进行双酶切,酶切产物回收,T4DNA连接酶25℃链接30 min,连接产物转化大肠杆菌感受态细胞Trans1-T1,涂布卡那霉素50 μg/mL的LB平板,37℃过夜,挑取若干单克隆菌落,酶切验证成功后送上海生工生物工程技术公司进行测序。
1.2.2 诱饵载体的转化和自激活检测 1.2.2.1 酵母感受态细胞的制备从-80℃冰箱中取出酵母菌株MaV203,在YPAD平板上划线培养,待酵母菌落直径约为2-3 mm时,挑单克隆于1.5 mL离心管,30℃过夜,取100 μL菌液于50 mL的YPDA液体培养基中,30℃培养16-20 h至OD600=0.15,3 500 r/min离心20 min,收集菌体,30 mL新鲜YPAD重悬细胞,继续培养至OD600=0.6,3 500 r/min离心20 min,收集菌体,加入15 mL无菌水重悬细胞,继续离心20 min,弃上清,加入1.5 mL 1.1×TE/ LiAc重悬细胞后12 000 r/min离心30 s,弃上清,加入600 μL 1.1×TE/ LiAc,制成酵母感受态细胞。
1.2.2.2 质粒转化酵母在1.5 mL离心管中分别加入100 ng的质粒DNA、10 μL的carrierDNA、500 μL的PEG/ LiAc和50 μL的酵母感受态细胞,轻轻混匀;30℃培养30 min,加入50 μL的DMSO,42℃热激25 min,12 000 ×g离心10 s,弃上清,加入1 mL的YPD Plus Liquid Medium重悬细胞,30℃继续培养1 h,12 000 ×g离心10 s,弃上清,用0.5 mL NacL(0.9%)溶液重悬细胞,取100 μL菌液涂在SD-Leu平板,30℃倒置培养,观察酵母转化子生长情况。
1.2.2.3 诱饵质粒自激活鉴定将质粒pDBleu-Hrip、pPC86;pDBleu、pPC86分别共转化酵母感受态细胞,取100 μL涂于SD-Trp-Leu平板,30℃培养2 d,分别从SD-Trp-Leu平板上挑取包含pDBleu-Hrip、pPC86;pDBleu、pPC86的单菌落,1 mL SD-Trp-Leu过夜培养,取100 μL菌液,12 000 ×g离心10 s,弃上清,500 μL无菌水重悬细胞,重复该步骤2次,使菌液最终体积为200 μL,分别吸取1 μL菌液点在不同浓度3AT(0 mmol/L、10 mmol/L、20 mmol/L、30 mmol/L、40 mmol/L、50 mmol/L、60 mmol/L、70 mmol/L、80 mmol/L)的SD-Trp-Leu-His平板上,30℃倒置培养3-5 d,观察酵母菌落生长情况。
1.2.3 Hrip1水稻互作蛋白的筛选制备包含有诱饵载体pDBleu-Hrip的酵母感受态细胞,在一个转化组中加入将500 μL的酵母感受态细胞、1 μg的水稻cDNA文库质粒、20 μL Yeast carrier DNA和1 mL PEG/ LiAc,转化步骤同上,最终取若干400 μL菌液涂50 mmol/L 3AT的SD-Trp-Leu-His平板,30℃倒置培养3-7 d。从筛库平板上挑选生长速度快且菌落直径较大的单菌落,3 mL SD-Trp-Leu液体培养基过夜培养,取200 μL菌液,用无菌水洗过后,重新点在50 mmol/L 3AT的SD-Trp-Leu-His平板上,能生长的克隆为阳性候选克隆,用天根公司酵母质粒小提试剂盒提取质粒并送测序。测序结果在国际水稻库(http://rice.plantbiology.msu.edu/index.shtml)中进行比对与分析。将感兴趣的候选互作蛋白质粒与pDBleu-Hrip1重新共转化酵母细胞,挑单菌落于SD-Trp-Leu液体培养基,无菌水清洗细胞后,再次点50 mmol/L 3AT的SD-Trp-Leu-His平板和包含有x-a-gal(40 μg/mL)的50 mmol/L 3AT SD-Trp-Leu-His平板,能良好生长的为Hrip1互作蛋白。
1.2.4 Hrip1互作蛋白基因的克隆和原核表达载体的构建取1 mg长势良好的日本晴叶片,用天根植物总RNA试剂盒提取其总RNA,以RNA为模板,使用北京全式金公司生产的反转录试剂盒(TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix)进行第一链cDNA的合成。根据数据库中的互作蛋白基因序列设计特异序列,PCR扩增,反应条件为:95℃预变性2 min,95℃变性20 s,59℃复性20 s,72℃延伸1 min,35个循环,72℃延伸5 min。反应产物经琼脂糖凝胶电泳检测回收,连接pEASY-Blunt Zero载体,转化全式金大肠杆菌感受态细胞Trans1-T1,筛选单克隆送测序。以测序成功的pEASY-Blunt Zero-CSN5质粒为模板,设计特异引物,扩增互作蛋白CSN5基因序列,用限制性内切酶BamH Ⅰ、Sal Ⅰ对片段和原核表达载体pGEX-6P-2(GST标签)双酶切,产物胶回收,连接、转化大肠杆菌感受态细胞Trans1-T1,送测序。
1.2.5 互作蛋白的原核表达与纯化取测序正确的重组载体pGEX-6P-2-CSN5转化大肠杆菌BL21感受态细胞。取菌落PCR检测正确的菌株,接种50 mL氨苄青霉素的(终浓度为100 μg/mL)LB培养液中,37℃震荡培养至OD600约0.6,调整温度为37℃、28℃、16℃,加入终浓度为0.2 mmol/L的IPTG诱导过夜;收集菌体,超声破碎后,分别取上清进行SDS-PAGE分析,确定合适的诱导温度。取菌液接种到50 mL氨苄青霉素的LB培养液中,37℃过夜。按1:100的稀释比例转接到1 L的LB培养液扩大培养,离心收集菌体,用上样缓冲液(50 mmol/L Tris,200 mmol/L NaCl,pH 8.0) 重悬菌体,超声破碎后,12 000 ×g离心1 h,收集上清后过0.45 μm滤膜,加入GST亲和层析柱,孵育后,用洗脱缓冲液洗脱(50 mmol/L Tris,200 mmol/L NaCl,还原性谷胱甘肽GSH 20 mmol/L,pH 8.0),收集蛋白用3 KD超滤管除盐,SDS-PAGE检测分析。
1.2.6 互作蛋白的Western blot检测取纯化后的蛋白样品20 μL,同时取等量空载体菌株的纯化产物作阴性对照,用12%的SDS-PAGE电泳,转膜至PVDF膜(24 V×1 h),含5%脱脂牛奶封闭液封闭2 h。取10 mL经稀释的GST一抗(1:5 000稀释),室温孵育2 h,1×TBST buffer洗涤PVDF膜3次,每次5 min。加入10 mL稀释的HRP二抗(1:5 000稀释),室温孵育1 h,1×TBST buffer洗涤PVDF膜3次,每次5 min。PVDF膜沥干后,置于专用塑料底层,加入显色液检测。
2 结果 2.1 诱饵载体的构建与鉴定以pET32-Hrip1为模板扩增Hrip1基因,得到一条大小约450 bp的条带(图 1-A),与预期相符。用Nco Ⅰ和Not Ⅰ双酶切对Hrip1片段、pDBleu双酶切,T4连接酶连接转化感受态细胞Trans1-T1。37℃过夜,挑取若干单克隆菌落培养,提取重组质粒酶切检测,结果(图 1-B)得到一条500 bp和一条大于8 000 bp的条带,说明Hrip1已经连接到载体pDBleu中。进一步的测序结果表明,目的片段按正确读码框插入pDBleu载体,没有碱基的缺失和突变,成功构建诱饵载体pDBleu-Hrip。
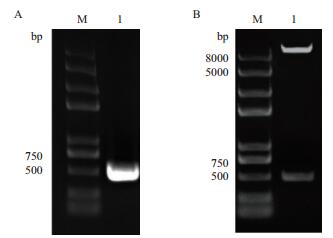
|
| 图 1 Hrip1基因的扩增和重组诱饵载体的双酶切鉴定 A:Hrip1的PCR扩增;M:DNA Marker;1:Hrip1条带B:pDBleu-Hrip的双酶切鉴定;M:DNA Marker;1:酶切结果 |
将pDBleu-Hrip、pPC86;pDBleu、pPC86分别共转化酵母菌株MaV203,两种转化菌株都可以在SD-Trp-Leu平板上长出白色菌落,直径大于2 mm,且含有诱饵蛋白的(pDBleu-Hrip、pPC86) 菌株与对照组(pDBleu、pPC86) 相比,长势没有差别。说明各载体成功转化且诱饵载体对酵母菌株MaV203没有毒性,可以进行后续实验。用SD-Trp-Leu液体培养基分别培养包含诱饵蛋白的菌株(pDBleu-Hrip、pPC86) 和对照菌株(pDBleu、pPC86),分别吸取1 μL菌液点在不同浓度3AT的SD-Trp-Leu-His平板上,30℃倒置培养3-5 d。结果(图 2) 发现,在3AT浓度为50 mmol/L时,包含诱饵蛋白的菌株和对照菌株都不能正常生长,说明50 mmol/L的3AT可以有效抑制MaV203细胞对报告基因的本底表达,因此可以利用3AT浓度为50 mmol/L的SD-Trp-Leu-His平板进行酵母双杂交筛选。
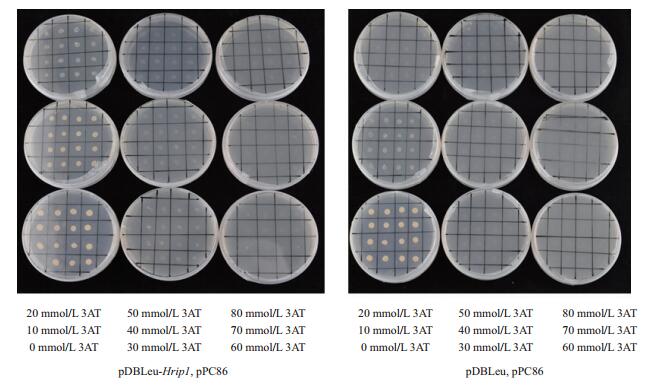
|
| 图 2 诱饵载体的自激活检测 |
将水稻cDNA文库转化到包含诱饵载体pDBleu-Hrip的MaV203后,在50 mmol/L 3AT的SD-Trp-Leu-His平板筛选,得到182个初始阳性克隆,通过提取质粒、序列信息在Rice Genome Annotation Project(http://rice.plantbiology.msu.edu/index.shtml)中搜索比对,去掉移码突变的菌株,得到16个阳性克隆。经重新共转化,只有一个克隆可以在50 mmol/L 3AT SD-Trp-Leu-His上生长,同时在含x-a-gal(40 μg/mL)的筛选板上显蓝色(图 3)。说明筛选到一个水稻文库蛋白与Hrip1存在相互作用。生物信息学分析表明,该互作蛋白在水稻库中的名称为COP9 signalosome complex subunit 5b(CSN5),基因cDNA全长1 083 bp,编码360个氨基酸,分子量40 kD。
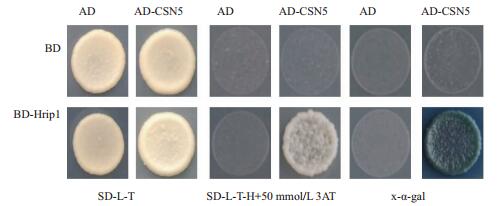
|
| 图 3 Hrip1与CSN5的酵母双杂交互作 |
以反转录得到的水稻cDNA为模板进行PCR扩增,获得大小约为1 000 bp的目的条带(图 4-A)。测序结果表明,目的条带的序列与水稻库中提供的序列完全一致,可以满足构建表达载体的要求。用限制性内切酶BamH Ⅰ、Sal Ⅰ对CSN5片段和原核表达载体pGEX-6P-2(GST标签)双酶切,连接转化后随机选取若干单克隆,培养菌株后提取质粒,重组质粒双酶切鉴定结果(图 4-B)显示,在产物中得到一条大小1 000 bp左右的条带,表明CSN5已经连接到pGEX-6P-2质粒中,进一步的测序结果显示,重组载体的读码框正确,插入序列无碱基缺失或突变,表明成功构建了pGEX-6P-2-CSN5原核表达载体。
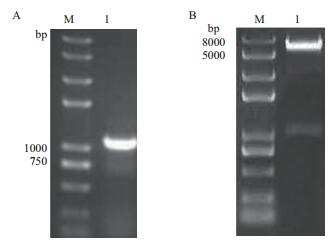
|
| 图 4 CSN5基因的扩增和重组表达载体的双酶切鉴定 A:CSN5基因序列的PCR扩增(M:DNA Marker;1:CSN5条带);B:pGEX-6P-2-CSN5的质粒双酶切鉴定(M:DNA Marker;1:酶切结果) |
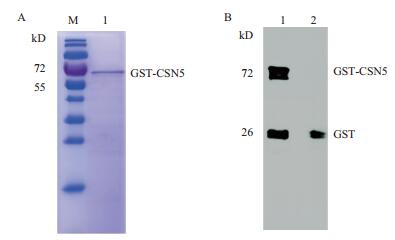
将重组表达载体pGEX-6P-2-CSN5转化感受态细胞BL21,表达菌株分别在16℃、28℃和37℃下诱导过夜,收集菌体,超声破碎,取适量上清液进行SDS-PAGE电泳,结果(图 5-A)显示,在37℃和28℃下均不能检测到融合蛋白的可溶性表达,只有当诱导温度为16℃时,SDS-PAGE显示出表达分子量67.8 kD的融合蛋白(CSN5蛋白的理论分子量为40 kD,pGEX-6P-2上的GST标签分子量为27.8 kD),融合表达蛋白大小与预期一致。为了进一步明确CSN5融合蛋白的可溶性,重新收集16℃诱导的菌体,破碎离心后,分别取上清和沉淀进行电泳分析,以空载体菌株为阴性对照。结果(图 5-B)显示,在阴性对照组中,上清和沉淀均没有检测到融合蛋白的表达。16℃诱导下,融合蛋白在沉淀和上清中均出现一条蛋白条带,而且在沉淀中的表达量更多,说明融合蛋白以包涵体和可溶蛋白的形式表达。为了获取纯化的融合蛋白,将菌株接种到1 L的LB液体培养基集中大量培养,收集上清后用GST亲和层析柱纯化,SDS-PAGE显示得到单一的蛋白条带(图 6-A)。

|
| 图 5 CSN5融合蛋白的诱导表达 A:不同温度诱导条件下CSN5蛋白的融合表达(M:蛋白Marker;1:空载体pGEX-6P-2上清总蛋白;2,3,4:分别是37℃、28℃、16℃诱导下pGEX-6P-2-CSN5上清总蛋白);B:CSN5在大肠杆菌BL21中表达情况(M:蛋白Marker;1:pGEX-6P-2-CSN5上清总蛋白;2:pGEX-6P-2-CSN5沉淀总蛋白;3:空载体pGEX-6P-2上清总蛋白;4:空载体pGEX-6P-2沉淀总蛋白) |
为了进一步验证纯化得到的蛋白是GST-CSN5。使用抗GST标签鼠单克隆抗体为一抗,辣根过氧化物酶HRP标记的羊抗鼠IgG(H+L)为二抗,Western blot检测结果(图 6-B)表明,融合蛋白可以和GST标签单克隆抗体结合,且目的条带大小与融合蛋白分子量一致,说明GST-CSN5在大肠杆菌BL21中得到正确表达。
|
| 图 6 CSN5融合蛋白的纯化和Western blot检测 A:CSN5融合蛋白的纯化(M:蛋白Marker;1:GST-CSN5蛋白);B:CSN5融合蛋白Western blot检测(1:GST-CSN5蛋白;2:GST标签蛋白) |
植物在面对众多潜在病原微生物时,依然可以顽强生存下去,这得益于能高效地识别这些微生物产生的激发子而激活其复杂而精密的先天免疫系统[14]。在植物与病原物相互作用中,植物细胞中的受体蛋白或靶蛋白起到了关键性作用。分布于细胞表面和细胞体内的靶蛋白或受体蛋白识别微生物入侵产生的PAMPs(Pathogen pathogen associated molecular patterns)或DAMPs(danger-associated molecular patterns)而启动信号传递和下游多种防御反应[15]。
Hrip1是本实验室分离到的一种蛋白质激发子,可以显著提高烟草和水稻的抗性,但Hrip1引起植物抗性反应的免疫机理仍不清楚[12, 13]。在本研究中,将Hrip1构建到包含BD结构域的诱饵载体pDBleu上,通过酵母双杂交技术筛选水稻cDNA库,得到了一个蛋白激发子Hrip1的互作蛋白CSN5。CSN5是COP9信号复合体中(COP9 signalosome complex,CSN)的一个亚基。COP9信号复合体由8个蛋白亚基组成CSN1-CSN8,最早发现于拟南芥的光形态构成的研究中[16, 17]。拟南芥的COP9突变体显示出“组成型光形态建成”的表型,这类突变体在黑暗中生长时会像正常光照生长的幼苗一样:下胚轴短小、子叶张开、质体分化以及光调控基因的表达。COP9复合体在高等真核生物中比较保守,主要参与蛋白的泛素化途径[18]。它具有异肽酶的活性,负责将NEDD8(Neural precursor cell expressed,developmentally down-regulated 8) 蛋白从cullin-RING泛素连接酶(CRLs)中去除;以保证CRLs功能的准确性[19, 20]。后续研究证明,CSN广泛参与了真核生物在生长发育中的各种代谢途径,包括光形态构成、花的发育、激素信号转导、细胞周期调控、DNA修复等[21]。CSN5本身是一种金属蛋白酶[22],是CSN的催化活性中心,通过包含的JAMM基序(JAB1/MPN/Mov34 metalloenzyme)将共价连接的RUB1(Related Ubiquitin 1)/NEDD8切割开而行使其催化活性,。实际上,CSN同样在植物的防御反应中发挥重要作用。在烟草中,CSN与SCF泛素连接酶在RAR1和SGT1的参与下,调控R基因介导的抗性反应,抵御多种病原物的入侵[23]。CSN5在拟南芥中可以和源自于阿拉伯半乳杆菌(Hyaloperonospora arabidopsidis,Hpa)以及丁香假单胞菌(Pseudomonas syringae,Psy)的多种效应子直接互作,参与拟南芥对Hpa和Psy的抗性[24]。许多植物对病原物的响应和信号通路依赖于对特定蛋白质的降解,一些病原微生物将植物CSN作为靶标蛋白以阻止寄主的抵抗[21]。本研究通过酵母双杂交筛选以及共转试验证明了蛋白激发子Hrip1能与CSN5相互作用,初步推测CSN5在Hrip1诱导植物免疫反应的过程中发挥着重要作用,植物有可能是在CSN5的参与下完成了对Hrip1的识别,同时启动相应信号传递网络,进而引起下游多种抗性反应,最终使植物具备了系统性获得抗性,这些推测需要今后进一步研究证实。为了进一步验证Hrip1和CSN5的体外互作,本研究建立了CSN5原核表达技术,选取带GST标签的pGEX-6P-2作为表达载体,从水稻cDNA文库中准确克隆到CSN5基因,通过酶切、连接得到正确的pGEX-6P-2-CSN5重组表达质粒。将CSN5融合表达载体转化大肠杆菌表达菌株BL21,分别在16℃、28℃和37℃下过夜诱导,发现只有16℃条件下,诱导表达可以得到可溶性性的目的蛋白。表达蛋白通过GST亲和层析纯化后,得到了单一条带的目的蛋白。Western blot鉴定了原核表达的CSN5蛋白是正确的。本研究鉴定了蛋白激发子Hrip1在水稻中的互作蛋白,并建立了互作蛋白的原核表达技术,研究结果为后续体外验证Hrip1和CSN5的相互作用以及CSN5基因的功能奠定了基础。
4 结论本研究通过酵母双杂交技术,在水稻cDNA文库中筛选到了蛋白激发子Hrip1的互作蛋白CSN5。构建互作蛋白CSN5的原核表达载体pGEX-6P-2-CSN5,并在大肠杆菌表达菌株BL21中成功表达,16℃过夜诱导,通过GST亲和层析得到纯化的可溶性融合蛋白。Western blot鉴定进一步证明了CSN5蛋白在大肠杆菌中得到了正确表达。
| [1] | Zhang Y, Yang X, Liu Q, et al. Purification of novel protein elicitor from Botrytis cinerea that induces disease resistance and drought tolerance in plants[J]. Microbiological Research, 2010, 165 (2): 142–151. DOI:10.1016/j.micres.2009.03.004 |
| [2] | Gómez-Gómez L, Felix G, Boller T. A single locus determines sensitivity to bacterial flagellin in Arabidopsis thaliana[J]. The Plant Journal, 1999, 18 (3): 277–284. DOI:10.1046/j.1365-313X.1999.00451.x |
| [3] | Mao J, Liu Q, Yang X, et al. Purification and expression of a protein elicitor from Alternaria tenuissima and elicitor-mediated defence responses in tobacco[J]. Annals of Applied Biology, 2010, 156 (3): 411–420. DOI:10.1111/aab.2010.156.issue-3 |
| [4] | Bu B, Qiu D, Zeng H, et al. A fungal protein elicitor PevD1 induces Verticillium wilt resistance in cotton[J]. Plant Cell Reports, 2014, 33 (3): 461–470. DOI:10.1007/s00299-013-1546-7 |
| [5] | Okinaka Y, Yang CH, Perna NT, et al. Microarray profiling of Erwinia chrysanthei 3937 genes that are regulated during plant infection[J]. Molecular Plant-Microbe Interactions, 2002, 15 (7): 619–629. DOI:10.1094/MPMI.2002.15.7.619 |
| [6] | Garcia-Brugger A, Lamotte O, Vandelle E, et al. Early signaling events induced by elicitors of plant defenses[J]. Molecular Plant-Microbe Interactions, 2006, 19 (7): 711–724. DOI:10.1094/MPMI-19-0711 |
| [7] | Schwessinger B, Ronald PC. Plant innate immunity:perception of conserved microbial signatures[J]. Plant Biology, 2012, 63 . |
| [8] | Boller T, Felix G. A renaissance of elicitors:perception of microbe-associated molecular patterns and danger signals by pattern-recognition receptors[J]. Annual Review of Plant Biology, 2009, 60 : 379–406. DOI:10.1146/annurev.arplant.57.032905.105346 |
| [9] | Zipfel C, Robatzek S, Navarro L, et al. Bacterial disease resistance in Arabidopsis through flagellin perception[J]. Nature, 2004, 428 (6984): 764–767. DOI:10.1038/nature02485 |
| [10] | Kunze G, Zipfel C, Robatzek S, et al. The N terminus of bacterial elongation factor Tu elicits innate immunity in Arabidopsis plants[J]. The Plant Cell, 2004, 16 (12): 3496–3507. DOI:10.1105/tpc.104.026765 |
| [11] | Zhang XC, Wu X, Findley S, et al. Molecular evolution of lysin motif-type receptor-like kinases in plants[J]. Plant Physiology, 2007, 144 (2): 623–636. DOI:10.1104/pp.107.097097 |
| [12] | Kulye M, Liu H, Zhang Y, et al. Hrip1, a novel protein elicitor from necrotrophic fungus, Alternaria tenuissima, elicits cell death, expression of defence-related genes and systemic acquired resistance in tobacco[J]. Plant, Cell & Environment, 2012, 35 (12): 2104–2120. |
| [13] | Peng XC, Qiu DW, Zeng HM, et al. Inducible and constitutive expression of an elicitor gene Hrip1 from Alternaria tenuissima enhances stress tolerance in Arabidopsis[J]. Transgenic Research, 2015, 24 (1): 135–145. DOI:10.1007/s11248-014-9824-x |
| [14] | Boller T, He SY. Innate immunity in plants:an arms race between pattern recognition receptors in plants and effectors in microbial pathogens[J]. Science, 2009, 324 (5928): 742–744. DOI:10.1126/science.1171647 |
| [15] | Dodds PN, Rathjen JP. Plant immunity:towards an integrated view of plant-pathogen interactions[J]. Nature Reviews Genetics, 2010, 11 (8): 539–548. DOI:10.1038/nrg2812 |
| [16] | Kwok S F, Solano R, Tsuge T, et al. Arabidopsis homologs of a c-Jun coactivator are present both in monomeric form and in the COP9 complex, and their abundance is differentially affected by the pleiotropic cop/det/fus mutations[J]. The Plant Cell, 1998, 10 (11): 1779–1790. DOI:10.1105/tpc.10.11.1779 |
| [17] | Wei N, Deng XW. COP9:a new genetic locus involved in light-regulated development and gene expression in Arabidopsis[J]. The Plant Cell, 1992, 4 (12): 1507–1518. DOI:10.1105/tpc.4.12.1507 |
| [18] | Wei N, Serino G, Deng XW. The COP9 signalosome:more than a protease[J]. Trends in Biochemical Sciences, 2008, 33 (12): 592–600. DOI:10.1016/j.tibs.2008.09.004 |
| [19] | Schwechheimer C, Serino G, Callis J, et al. Interactions of the COP9 signalosome with the E3 ubiquitin ligase SCFTIR1 in mediating auxin response[J]. Science, 2001, 292 (5520): 1379–1382. DOI:10.1126/science.1059776 |
| [20] | Lyapina S, Cope G, Shevchenko A, et al. Promotion of NEDD8-CUL1 conjugate cleavage by COP9 signalosome[J]. Science, 2001, 292 (5520): 1382–1385. DOI:10.1126/science.1059780 |
| [21] | Franciosini A, Serino G, Deng XW. COP9 Signalosome Network[J]. Molecular Biology, 2014 : 313–332. |
| [22] | Cope GA, Suh GSB, Aravind L, et al. Role of predicted metalloprotease motif of Jab1/Csn5 in cleavage of Nedd8 from Cul1[J]. Science, 2002, 298 (5593): 608–611. DOI:10.1126/science.1075901 |
| [23] | Craig A, Ewan R, Mesmar J, et al. E3 ubiquitin ligases and plant innate immunity[J]. Journal of Experimental Botany, 2009, 60 (4): 1123–1132. DOI:10.1093/jxb/erp059 |
| [24] | Mukhtar MS, Carvunis AR, Dreze M, et al. Independently evolved virulence effectors converge onto hubs in a plant immune system network[J]. Science, 2011, 333 (6042): 596–601. DOI:10.1126/science.1203659 |




