2. 中国科学院遗传与发育生物学研究所 遗传资源中心,北京 100101;
3. 中国科学院大学,北京 100049
2. Genetic Resource Center, Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101;
3. University of Chinese Academy of Sciences, Beijing 100049
嗜乳脂蛋白家族包含嗜乳脂蛋白 (Butyrophilin,BTN)、类嗜乳脂蛋白 (Butyrophilin-like,BTNL) 及Skint (Selection and upkeep of intraepithelial T cells) 等成员,属于免疫球蛋白超家族[1]。首先被发现的嗜乳脂蛋白是牛嗜乳脂蛋白1A1(Butyrophilin 1A1,BTN1A1),因其可与乳脂结合,控制乳脂分泌而被定义为嗜乳脂蛋白[2, 3]。之后的研究发现,多数嗜乳脂蛋白基因定位于主要组织相容性复合体 (Major histocompatibility complex,MHC) 区段内[4],且胞外结构域与B7共刺激分子同源,暗示该类基因可能参与免疫调节过程[5]。目前针对嗜乳脂蛋白家族的研究主要集中于人和小鼠中[6]。根据Ensembl数据库 (http://asia.ensembl.org/index.html),人的基因组共包含12个嗜乳脂蛋白基因:BTN1A1、BTN2A1、BTN2A2、BTN3A1、BTN3A2、BTN3A3、BTNL2、BTNL3、BTNL8、BTNL9、ERMAP (红细胞膜相关蛋白) 和MOG (髓鞘少突胶质细胞糖蛋白),其中BTN3A1具有激活γ9δ2 T细胞的功能[7, 8],BTNL2的剪切突变与结节病相关[9],BTNL8可增强T细胞活化[10],MOG是风疹病毒感染宿主的受体[11]。鼠的基因组中包含18个嗜乳脂蛋白基因:Btn1a1、Btn2a2、Btnl1、Btnl2、Btnl4、Btnl6、Btnl9、Btnl10、ERMAP、MOG和Skint1-Skint8,其中Btn1a1、Btn2-a2和Btnl2具有抑制T细胞活化的功能[12-14],Skin-t1可调节表皮γδT细胞的发育[15, 16],Btnl1具有抑制肠上皮细胞炎症因子表达的功能[17],Btnl1与Btnl6组成异源复合体促进T细胞的增殖[18]。
在猪的基因组中,经Ensembl数据库预测得到14个嗜乳脂蛋白家族基因,但皆缺乏相关的功能研究。其中猪Btnl5位于7号染色体正链:28093234-28108144,定位于MHC区段内。根据EST数据信息,Btnl5基因主要在肠道中表达[19]。研究显示,同在肠道表达的鼠源Btnl1可抑制炎症因子KC/CXCL1、MIP1β/CCL4、IL-15和IL-6的表达[17],推测Btnl1可能通过抑制核因子κB (NF-κB) 从而抑制上述炎症因子的表达[17]。NF-κB是转录调控因子,参与多种基因的转录调控。在肠道中,模式识别受体识别病原微生物后激活NF-κB,从而激活IL-1、IL-6和TNFα等细胞因子的分泌,而上述细胞因子可促进NF-κB进一步活化,导致炎症的放大和持续[20]。在炎症性肠病 (Inflammatory bowel disease,IBD) 和肠炎模型中皆伴随有NF-κB的过度活化[21, 22],因此,NF-κB是肠道炎症的关键调控因子。为探索Btnl5基因的序列特征,以及是否具有与鼠源Btnl1相似的生物学功能,本研究对Btnl5基因cDNA全长进行克隆,并对其序列进行生物信息学分析,进一步检测该基因在不同组织中的表达水平及其对NF-κB信号通路的影响,旨为研究猪Btnl5基因的功能以及该基因在猪肠道疾病中的作用提供理论依据。
1 材料与方法 1.1 材料选取体重均匀,生长状况良好的1月龄巴马香猪3头,屠宰后立即摘取心脏、肝脏、脾脏、肺、肾脏、空肠、结肠、大脑、肌肉、胸腺和肠系膜淋巴结保存于液氮中。肠上皮细胞系IPEC-1由中国农业大学伍国耀老师惠赠。NF-κB启动子报告基因、MyD88质粒、β-gal质粒、pcDNA3.1-myc-his质粒由本实验室保存。RNAiso plus、M-MLV反转录酶、Prime STAR高保真PCR酶、T-载体PCR产物克隆试剂盒、去除基因组DNA的反转录试剂盒、SYBR定量PCR试剂盒、限制性内切酶Xho Ⅰ、EcoR Ⅰ购自Takara公司 (中国大连)。RLM-RACE试剂盒、胎牛血清、DMEM/F12培养基、Lipofectamine 2000购自life公司 (美国)。Bright-Glo荧光素酶检测试剂盒、RNA提取试剂盒购自promega公司 (美国)。质粒小提试剂盒、琼脂糖回收试剂盒购自OMEGA公司 (美国)。
1.2 方法 1.2.1 引物设计参照Ensembl中提供的猪Btnl5预测序列,使用Primer Premier 5.0软件设计引物,扩增Btnl5片段,根据扩增得到的序列设计5'和3'端基因特异性的RACE引物。根据定量PCR要求设计Btnl5和内参基因GAPDH的引物。序列分析确定Btnl5的开放读码框后,设计包含限制性内切酶位点的上下游引物,将Btnl5的开放读码框连入pcDNA3.1-myc-his真核表达载体中。引物由Invitrogen公司合成,引物序列见表 1。
空肠组织总RNA按照promega RNA提取试剂盒提取,分别使用1%琼脂糖电泳及紫外分光光度计检测RNA完整性、纯度及浓度。空肠cDNA第一链按照M-MLV反转录酶说明书反转录获得,用于Btnl5片段扩增。5'RACE cDNA第一链及3'RACE cDNA第一链按照RLM-RACE试剂盒说明书反转录获得。PCR扩增使用Prime STAR高保真酶,反应体系皆为50 μL:2 × Prime STAR Max Premix 25 μL,上下游引物各1.25 μL,cDNA 2 μL,dd H2O 20.5 μL。Btnl5片段扩增条件:98℃预变性1 min;98℃变性10 s,59℃退火5 s,72℃延伸15 s,32个循环;72℃延伸1 min。5'RACE扩增退火温度为55℃,其余条件如Btnl5片段扩增。3'RACE扩增以Gene Racer 3'Primer和3'RACE Primer 1为引物扩增16个循环,PCR产物回收后作为模板以3' RACE Primer 2和3'Nested Primer为引物扩增18个循环,其他扩增条件如Btnl5片段扩增。所有PCR产物经1%琼脂糖凝胶电泳、琼脂糖凝胶回收后进行加A反应,与PMD19-T载体连接,转化大肠杆菌DH5α感受态细胞,送往生工生物工程 (上海) 股份有限公司进行测序鉴定。
1.2.3 生物信息学分析将测序得到的3段序列使用SeqMan[23]拼接,得到Btnl5基因的cDNA全长。比对Btnl5的cDNA全长序列与基因组序列,得到外显子、内含子相对位置及长度。使用NCBI的ORF Finder软件 (https://www.ncbi.nlm.nih.gov/orffinder/) 预测基因的开放读码框,并将基因编码序列翻译为相应的氨基酸序列。使用pI/Mw网站 (http://web.expasy.org/compute_pi/) 计算Btnl5蛋白的分子量和理论等电点。使用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/) 网站预测Btnl5是否为跨膜蛋白。使用SMART网站 (http://smart.embl-heidelberg.de/) 预测Btnl5的结构域及大小。
1.2.4 相对定量PCR按照RNAiso plus说明书,分别提取心脏、肝脏、脾脏、肺、肾脏、空肠、结肠、大脑、肌肉、胸腺及肠系膜淋巴结总RNA,检测RNA完整性及纯度。使用去除基因组DNA的反转录试剂盒合成cDNA第一链。以持家基因GAPDH为内参,对Btnl5 mRNA在各个组织中的表达量进行相对定量分析,引物见表 1。定量PCR体系10 μL:SYBR Premix Ex Taq 5 μL,上下游引物各0.2 μL,cDNA模板1 μL,ddH2O 3.6 μL。定量PCR反应在Bio-Rad CFX96进行,反应程序为:95℃预变性3 min;95℃变性15 s,60℃退火/延伸30 s,60℃检测30 s,40个循环;65℃-95℃,0.5℃/5 s绘制溶解曲线。每个组织设二次技术重复,相对定量结果采用2-ΔCT法进行计算。
1.2.5 Btnl5-myc真核表达载体的构建以Btnl5-T载体为模板,以Btnl5-myc-F和Btnl5-myc-R为引物,使用高保真酶扩增Btnl5编码区。扩增体系50 μL:2 × Prime STAR Max Premix 25 μL,上下游引物各1.25 μL,模板2 μL,dd H2O 20.5 μL。扩增条件:98℃预变性1 min;98℃变性10 s,59℃退火5 s,72℃延伸15 s,32个循环;72℃延伸1 min。产物经电泳鉴定后进行胶回收,回收产物和pcDNA3.1-myc-his载体分别经Xho Ⅰ、EcoR Ⅰ双酶切后使用T4 DNA连接酶连接,转化大肠杆菌DH5α感受态细胞,酶切鉴定并测序鉴定。
1.2.6 细胞培养及转染猪肠上皮细胞系IPEC-1培养于完全培养基 (DMEMF/12,10% FBS,青链霉素双抗) 中,培养条件为37℃,5% CO2。按照每孔5×104个细胞的密度接种于24孔板,待细胞覆盖率达到70%-80%时,按照Lipofectamine 2000说明书进行转染,实验分3组,每组3个复孔。每孔转染质粒NF-κB 50 ng、β-gal 25 ng,空白对照组同时转染pcDNA3.1-myc-his 300 ng,MyD88对照组同时转染MyD88 100 ng、pcDNA3.1-myc-his 200 ng,实验组同时转染MyD88 100 ng、Btnl5-myc 200 ng。
1.2.7 荧光素酶报告基因检测IPEC-1细胞系转染24 h后,使用生理盐水清洗,加入100 μL细胞裂解液 (20 mmol/L HEPES,150 mmol/L NaCl,2 mmol/L EDTA,1% Nonidet P-40,10%甘油,蛋白酶抑制剂,pH 7.5),4℃震荡裂解细胞30 min,12 000 r/min,4℃离心10 min,取上清检测荧光素酶活性及β-gal活性。荧光素酶活性检测:20 μL蛋白上清与20 μL荧光素酶底物混匀后立即用荧光检测仪检测活性。β-gal活性检测:20 μL蛋白上清、100 μL ONPG加450 μL Z buffer (60 mmol/L Na2HPO4,40 mmol/L NaH2PO4,10 mmol/L KCl,1 mmol/L MgSO4,pH 7) 颠倒混匀后37℃静置反应至溶液变黄,加1 mol/L的碳酸钠200 μL终止反应,酶标仪检测410 nm处吸收值。荧光素酶活性与β-gal活性比值即为报告基因的表达活性。
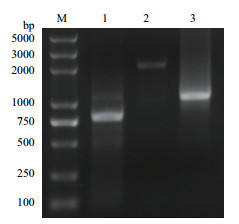
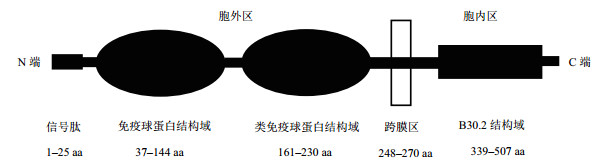
2 结果 2.1 Btnl5基因cDNA全长克隆及序列分析实验分3段克隆Btnl5全长:5'RACE、Btnl5片段和3' RACE (图 1),5'RACE在750 bp左右有明显条带。Btnl5片段在2 000 bp左右有特异性条带。3' RACE经巢式PCR扩增后在1 000 bp处得到明显条带。3段均送多个阳性克隆进行测序鉴定。将测序得到的3段序列拼接得到Btnl5基因cDNA全长共3 334 bp,与基因组序列比对得到11个外显子,外显子长度分别为302、144、95、348、282、201、21、27、27、27和1 860 bp (图 2)。经NCBI网站的ORF Finder预测,5'非编码区456 bp,3'非编码区1 198 bp,开放读码框为1 680 bp,编码559个氨基酸残基的多肽链。pI/Mw网站推算Btnl5蛋白分子量为61.7 kD,理论等电点为5.1。TMHMM Server v. 2.0预测1-247氨基酸为胞外区,248-270氨基酸为跨膜区,271-559氨基酸为胞内区。经SMART预测胞外区包含信号肽、两段免疫球蛋白结构域,胞内区为B30.2结构域 (图 3)。
|
| 图 1 Btnl5基因cDNA全长的克隆 M:BM5000 DNA相对分子质量标准;1:5'RACE扩增产物;2:Btnl5片段扩增产物;3:3'RACE扩增产物 |

|
| 图 2 Btnl5基因外显子、内含子长度及编码区相对位置 白色框和灰色框分别对应编码区和非编码区;框内或框上方数字代表外显子的碱基数;框下方数字代表内含子的碱基数 |
|
| 图 3 预测Btnl5蛋白结构域及各结构域长度 |
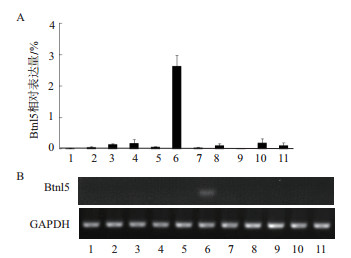
定量PCR结果显示,所检测的心脏、肝脏、脾脏、肺、肾脏、空肠、结肠、大脑、肌肉、胸腺及肠系膜淋巴结11种组织中,Btnl5主要表达于空肠,在其余10种组织中表达水平极低 (图 4-A)。Btnl5与内参GAPDH PCR产物经2%琼脂糖电泳显示,Btnl5与GAPDH扩增条带在100 bp左右,GAPDH在11个组织中表达相似,Btnl5只在空肠有明显条带 (图 4-B)。
|
| 图 4 定量PCR分析猪Btnl5的表达 A:不同组织中Btnl5定量PCR结果;B:不同组织中Btnl5和GAPDH的PCR产物电泳图 (1:心脏;2:肝脏;3:脾脏;4:肺;5:肾脏;6:空肠;7:结肠;8:大脑;9:肌肉;10:胸腺;11:肠系膜淋巴结) |
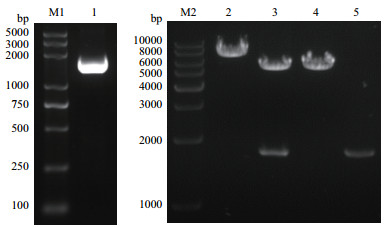
以Btnl5-T质粒为模板进行PCR扩增,在2 000 bp处得到单一条带,与Btnl5编码区长度一致 (图 5)。回收的PCR产物与pcDNA3.1-myc-his连接的重组质粒经Xho Ⅰ、EcoR Ⅰ双酶切,1%琼脂糖凝胶电泳鉴定,重组质粒双酶切产物在5 000 bp和2 000 bp左右有清晰条带,分别与空载体双酶切所产生条带和Btnl5扩增产物大小相同 (图 5),且经测序验证Btnl5插入位置、序列均正确。
|
| 图 5 Btnl5 ORF扩增和Btnl5-myc表达载体酶切鉴定 M1:BM5000 DNA相对分子质量标准;1:Btnl5扩增产物;M2:1kb DNA相对分子质量标准;2:重组质粒单酶切产物;3:重组质粒双酶切产物;4:空载体双酶切产物;5:Btnl5扩增产物 |
实验表明,肠上皮细胞系IPEC-1转染MyD88后,NF-κB信号通路活化,报告基因表达增高,而同时转染Btnl5时,报告基因表达量降低约50%,差异显著 (P=0.009,图 6)。
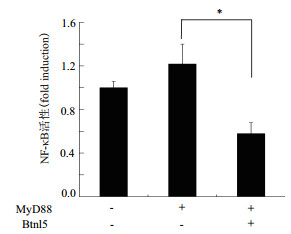
|
| 图 6 Btnl5抑制NF-κB信号通路 |
目前对嗜乳脂蛋白家族的研究数据较少,但普遍认为嗜乳脂蛋白是新的免疫调节蛋白家族[6, 24]。本研究克隆得到猪Btnl5基因cDNA全长共3 334 bp,其中包含456 bp的5'非编码区和1 198 bp的3'非编码区,以及1 680 bp的开放读码框,编码559个氨基酸。经软件预测Btnl5蛋白是一个跨膜蛋白,胞外是两段免疫球蛋白结构域,胞内是B30.2结构域。含有免疫球蛋白结构域的膜蛋白属于免疫球蛋白超家族,该类蛋白通过结合目的蛋白参与识别、粘附、信号传递等功能[25]。而胞内的B30.2结构域是真核生物中最常见的结构域之一[26],但其功能目前尚不清楚,Perfetto等分析认为该结构域作为接头蛋白,与其他蛋白形成蛋白复合体发挥作用[26]。因此推测Btnl5可能通过与其他蛋白相互结合发挥功能。
在检测的11个组织 (心脏、肝脏、脾脏、肺、肾脏、空肠、结肠、大脑、肌肉、胸腺和肠系膜淋巴结) 中,Btnl5仅在空肠检测到较高表达水平,具有明显的组织特异性,与EST信息相符[19]。肠道是机体对抗外来入侵的第一道屏障,哺乳动物肠道内含有大量细菌,在肠道内执行重要的新陈代谢功能。这些细菌中的多数在其他组织中会引起强烈的宿主反应,却能与肠道细胞并存,甚至促进肠道的稳定。其机制远未研究清楚,但越来越清楚的是NF-κB在维持肠道稳定方面起到了关键性的作用[27]。
本研究发现Btnl5对MyD88激活的NF-κB信号通路有抑制作用。NF-κB信号通路对细胞存活、肠道紧密连接具有重要作用[28],同时可通过调控炎症因子的表达对抗微生物感染,防止致病菌侵入。但过量的炎症因子会破坏细胞间的紧密连接,致使肠上皮细胞渗透性增加[29],增加癌症及自身免疫疾病发生的风险[30]。为防止NF-κB信号通路过度活化,机体会同时启动NF-κB的负向调节。如NF-κB的活化启动IκBα(Inhibitor of NF-κB) 的表达,终止下游基因转录[31];Lawrence等[32]研究发现,活化的IKKα加速p65的降解;此外,A20亦可负调控NF-κB的表达[33]。通过报告基因检测发现,Btnl5同样具有抑制NF-κB信号通路的作用,推测Btnl5可能参与NF-κB的负调控过程。但Btnl5对NF-κB下游基因是否具有广泛的抑制作用及其抑制NF-κB信号通路的机制,还有待进一步研究。
4 结论克隆了猪Btnl5基因cDNA全长3 334 bp,包含1 680 bp的开放读码框,编码559个氨基酸残基的多肽链。在检测的11个组织 (心脏、肝脏、脾脏、肺、肾脏、空肠、结肠、大脑、肌肉、胸腺、肠系膜淋巴结) 中,Btnl5只在空肠中具有较高水平的表达,并且具有抑制NF-κB信号通路的作用。
致谢 本研究在实验和文稿修改过程中得到了中国科学院生物物理研究所李翀研究员及本实验室许崇凤、张瑞莹助理研究员的无私帮助和大力支持,本文工作得到国家重点基础研究发展计划 (973)(2013CB835200) 资助,在此深表感谢。| [1] | Afrache H, Gouret P, Ainouche S, et al. The butyrophilin (BTN) gene family: from milk fat to the regulation of the immune response[J]. Immunogenetics, 2012, 64 (11): 781–794. DOI:10.1007/s00251-012-0619-z |
| [2] | Heid HW, Winter S, Bruder G, et al. Butyrophilin, an apical plasma membrane-associated glycoprotein characteristic of lactating mammary glands of diverse species[J]. Biochim Biophys Acta, 1983, 728 (2): 228–238. DOI:10.1016/0005-2736(83)90476-5 |
| [3] | Jeong J, Rao AU, Xu J, et al. The PRY/SPRY/B30. 2 domain of butyrophilin 1A1(BTN1A1) binds to xanthine oxidoreductase: implications for the function of BTN1A1 in the mammary gland and other tissues[J]. J Biol Chem, 2009, 284 (33): 22444–22456. DOI:10.1074/jbc.M109.020446 |
| [4] | Rhodes DA, Stammers M, Malcherek G, et al. The cluster of BTN genes in the extended major histocompatibility complex[J]. Genomics, 2001, 71 (3): 351–362. DOI:10.1006/geno.2000.6406 |
| [5] | Arnett HA, Viney JL. Immune modulation by butyrophilins[J]. Nature reviews Immunology, 2014, 14 (8): 559–569. DOI:10.1038/nri3715 |
| [6] | Rhodes DA, Reith W, Trowsdale J. Regulation of Immunity by Buty-rophilins[J]. Annu Revi Immunol, 2016, 28 (34): 151–172. |
| [7] | Wang H, Morita CT. Sensor function for butyrophilin 3A1 in prenyl pyrophosphate stimulation of human Vgamma2Vdelta2 T cells[J]. Journal of Immunology, 2015, 195 (10): 4583–4594. DOI:10.4049/jimmunol.1500314 |
| [8] | Sandstrom A, Peigne CM, Leger A, et al. The intracellular B30. 2 domain of butyrophilin 3A1 binds phosphoantigens to mediate activation of human V gamma 9V delta 2T Cells[J]. Immunity, 2014, 40 (4): 490–500. DOI:10.1016/j.immuni.2014.03.003 |
| [9] | Valentonyte R, Hampe J, Huse K, et al. Sarcoidosis is associated with a truncating splice site mutation in BTNL2[J]. Nature Genetics, 2005, 37 (4): 357–364. DOI:10.1038/ng1519 |
| [10] | Chapoval AI, Smithson G, Brunick L, et al. BTNL8, a butyrophilin-like molecule that costimulates the primary immune response[J]. Molecular Immunology, 2013, 56 (4): 819–828. DOI:10.1016/j.molimm.2013.08.003 |
| [11] | Cong HL, Jiang Y, Tien P. Identification of the myelin oligodendrocyte glycoprotein as a cellular receptor for rubella virus[J]. Journal of Virology, 2011, 85 (21): 11038–11047. DOI:10.1128/JVI.05398-11 |
| [12] | Smith IA, Knezevic BR, Ammann JU, et al. BTN1A1, the mammary gland butyrophilin, and BTN2A2 are both inhibitors of T cell activation[J]. J Immunol, 2010, 184 (7): 3514–3525. DOI:10.4049/jimmunol.0900416 |
| [13] | Sarter K, Leimgruber E, Gobet F, et al. Btn2a2, a T cell immunomodulatory molecule coregulated with MHC class Ⅱ genes[J]. J Exp Med, 2016, 213 (2): 177–187. DOI:10.1084/jem.20150435 |
| [14] | Swanson RM, Gavin MA, Escobar SS, et al. Butyrophilin-like 2 modulates B7 costimulation to induce Foxp3 expression and regulatory T cell development in mature T cells[J]. Journal of Immunology, 2013, 190 (5): 2027–2035. DOI:10.4049/jimmunol.1201760 |
| [15] | Barbee SD, Woodward MJ, Turchinovich G, et al. Skint-1 is a highly specific, unique selecting component for epidermal T cells[J]. Proc Natl Acad Sci USA, 2011, 108 (8): 3330–3335. DOI:10.1073/pnas.1010890108 |
| [16] | Turchinovich G, Hayday AC. Skint-1 identifies a common molecular mechanism for the development of interferon-gamma-secreting versus interleukin-17-secreting gammadelta T cells[J]. Immunity, 2011, 35 (1): 59–68. DOI:10.1016/j.immuni.2011.04.018 |
| [17] | Bas A, Swamy M, Abeler-Dorner L, et al. Butyrophilin-like 1 encodes an enterocyte protein that selectively regulates functional interactions with T lymphocytes[J]. Proc Natil Acad Sci USA, 2011, 108 (11): 4376–4381. DOI:10.1073/pnas.1010647108 |
| [18] | Lebrero-Fernandez C, Bergstrom JH, Pelaseyed T, et al. Murine butyrophilin-like 1 and btnl6 form heteromeric complexes in small intestinal epithelial cells and promote proliferation of local T lymphocytes[J]. Frontiers in Immunology, 2016, 7 (12): 1–13. |
| [19] | Gorodkin J, Cirera S, Hedegaard J, et al. Porcine transcriptome analysis based on 97 non-normalized cDNA libraries and assembly of 1, 021, 891 expressed sequence tags[J]. Genome Biology, 2007, 8 (4): 90–105. |
| [20] | Jobin C, Sartor R. The I kappa B/NF-kappa B system: a key determinant of mucosal inflammation and protection[J]. Am J Physiol-Cell Physiol, 2000, 278 (3): C451–C462. |
| [21] | Rogler G, Brand K, Vogl D, et al. Nuclear factor kappa B is activated in macrophages and epithelial cells of inflamed intestinal mucosa[J]. Gastroenterology, 1998, 115 (2): 357–369. DOI:10.1016/S0016-5085(98)70202-1 |
| [22] | Schreiber S, Nikolaus S, Hampe J. Activation of nuclear factor kappa B in inflammatory bowel disease[J]. Gut, 1998, 42 (4): 477–484. DOI:10.1136/gut.42.4.477 |
| [23] | Swindell SR, Plasterer TN. SEQMAN. Contig assembly[J]. Methods in Molecular Biology, 1997, 70 (70): 75–89. |
| [24] | Di Marco Barros R, Roberts NA, Dart RJ, et al. Epithelia use butyrophilin-like molecules to shape organ-specific γδ T cell compartments[J]. Cell, 2016, 167 (1): 203–218. DOI:10.1016/j.cell.2016.08.030 |
| [25] | Barclay AN. Membrane proteins with immunoglobulin-like domains-a master superfamily of interaction molecules[J]. Seminars in Immunology, 2003, 15 (4): 215–223. DOI:10.1016/S1044-5323(03)00047-2 |
| [26] | Perfetto L, Gherardini PF, Davey NE, et al. Exploring the diversity of SPRY/B30. 2-mediated interactions[J]. Trends in Biochemical Sciences, 2013, 38 (1): 38–46. DOI:10.1016/j.tibs.2012.10.001 |
| [27] | Eckmann L, Neish AS. NF-kappa B and Mucosal Homeostasis[J]. Current Topics in Microbiology & Immunology, 2011, 349 (349): 145–158. |
| [28] | Peterson LW, Artis D. Intestinal epithelial cells: regulators of barrier function and immune homeostasis[J]. Nature Reviews Immunology, 2014, 14 (3): 141–153. DOI:10.1038/nri3608 |
| [29] | Gitter AH, Bendfeldt K, Schulzke JD, et al. Leaks in the epithelial barrier caused by spontaneous and TNF-alpha-induced single-cell apoptosis[J]. Faseb Journal, 2000, 14 (12): 1749–1753. DOI:10.1096/fj.99-0898com |
| [30] | Karin M, Greten FR. NF-kappaB: linking inflammation and immunity to cancer development and progression[J]. Nature Reviews Immunology, 2005, 5 (10): 749–759. DOI:10.1038/nri1703 |
| [31] | Sun SC, Ganchi PA, Ballard DW, et al. NF-κB controls expression of inhibitor I κB α: evidence for an inducible autoregulatory pathway[J]. Science, 1993, 259 (5103): 1912–1915. DOI:10.1126/science.8096091 |
| [32] | Lawrence T, Bebien M, Liu GY, et al. IKKα limits macrophage NF-κB activation and contributes to the resolution of inflammation[J]. Nature, 2005, 434 (7037): 1138–1143. DOI:10.1038/nature03491 |
| [33] | Majumdar I, Paul J. The deubiquitinase A20 in immunopathology of autoimmune diseases[J]. Autoimmunity, 2014, 47 (5): 307–319. DOI:10.3109/08916934.2014.900756 |





