稻瘟病是世界范围内最严重的水稻病害之一,每年造成严重的水稻产量损失。该病是由子囊菌稻瘟病菌 (Magnaporthe oryzae) 侵染水稻引起的。稻瘟病菌具有许多植物病原真菌生命循环的重要特点,其侵染过程和致病分子机制在丝状真菌中具有典型性。近年来稻瘟病菌和水稻已经成为研究植物病原真菌与寄主互作的理想模式生物[1, 2]。目前,科学家们对蛋白质翻译后修饰的研究日趋深入,而由精氨酸甲基转移酶 (Protein arginine methyltransferases,PRMTs) 催化的蛋白质精氨酸甲基化近年来也有很多报道。蛋白质精氨酸甲基化属于一种典型蛋白质翻译后修饰,广泛存在于真核细胞中并调控蛋白质功能。精氨酸甲基化主要分为单甲基化、对称和不对称双甲基化3种修饰类型。蛋白质精氨酸甲基转移酶催化过程为催化S-腺苷甲硫胺酸 (S-adenosylmethionine,SAM) 提供的甲基基团转移到核质蛋白包括组蛋白的精氨酸侧链胍基基团上。
目前为止,PRMTs基因已在多种生物体基因组序列中得到鉴定。人类含有9种确定的PRMTs,但是推测可能更多的PRMTs还未被鉴定到[3, 4]。哺乳动物细胞中PRMTs可以分为3种类型:Ⅰ型PRMT催化形成单甲基精氨酸和不对称双甲基精氨酸,包括PRMT1(主要的Ⅰ型PRMT),PRMT2,PRMT3,CARM1/PRMT4,PRMT6和PRMT8;Ⅱ型PRMT催化形成单甲基和对称双甲基精氨酸,主要包括PRMT5[5]和PRMT9[6];Ⅲ型只能催化胍基基团的单甲基化,包括PRMT7[7]。PRMTs参与了众多不同细胞水平的调控,如染色质信号转导、PRMT1、PRMT2、CARM1、PRMT5、PRMT6和PRMT7可以甲基化组蛋白末端[8, 9];核糖体机制,PRMT3催化40S核糖体蛋白S2[10];DNA修复和mRNA剪切[11]。
近年来,研究表明人的PRMTs与众多疾病的发生密切相关。常见肿瘤包括前列腺癌和乳腺癌受激素调控,而PRMTs则是核受体的共激活子,并且可能在肿瘤中表达增强。据报道,过量表达PRMT4与不依赖雄性激素的前列腺癌密切相关[12],而抑制PRMT1和PRMT4的小分子能够抑制雌激素和雄激素受体介导的转录活性[13]。另外,过量表达PRMT5能够促进不依赖于停泊位点的细胞的生长,表明PRMT5可能是通过抑制肿瘤抑制子的表达来负调控细胞形态的变化[14]。同时,PRMTs也间接地影响着心血管疾病的发生。Zhang等[15]研究发现,靶向PRMT1可能会恢复巨核细胞的顶端分化,进而找到治疗急性巨核细胞白血病的有效方法。此外,甲基化水平对神经系统的影响也是比较敏感。PRMT5可以对RNA聚合酶Ⅱ亚基POLR2A羧基端的精氨酸残基R1810进行对称地双甲基化修饰,此修饰可能影响转录终止进而引发神经退行性疾病[16]。
在酿酒酵母数据库中只发现3种精氨酸甲基转移酶,Hmt1、Hsl7和Rmt2[17]。Hmt1与哺乳动物的PRMT1同源,属于Ⅰ型PRMT[18]。Hmt1对于酵母细胞生长并非必需,但在NPL3或CBP80突变背景下HMT1缺失会导致酵母致死[19];Hsl7与哺乳动物PRMT5同源,属于Ⅱ型PRMT;Rmt2属于Ⅳ型精氨酸甲基转移酶[20],该酶催化精氨酸δ氮原子发生单甲基化。Rmt2仅在酿酒酵母中被鉴定到,且以颗粒和点状结构聚集在酵母细胞中[21],其同源物尚未在哺乳动物中报道。HMT1是酵母中主要的精氨酸甲基转移酶,其功能涉及核质转运、转录激活和延伸、mRNA剪切和信号转导等过程[21-23],底物主要是核不均一核糖核蛋白 (hnRNPs),其C端常富含RGG重复序列并作为精氨酸甲基化位点[24]。
在植物中精氨酸甲基转移酶的研究较少,模式植物拟南芥中AtPRMT1a/b与AtPRMT4a/b分别是人PRMT1和PRMT4的同源蛋白,而研究比较清楚的是AtPRMT3、5、10。AtPRMT3突变体致使拟南芥生长迟缓,并且对部分氨基糖苷类的抗生素耐受性增强,可以通过影响前体rRNA加工进而控制核糖体生物合成[25];AtPRMT5的缺失可导致生长受阻和晚花等,且与前体mRNA的剪切有关[26];AtPRMT10属于植物特有的Ⅰ型PRMT,可催化H4R3,与拟南芥开花有关[27]。
在植物病原真菌中PRMT基因也是高度保守的,但是其生物功能在稻瘟菌中尚未报道。王光辉等[28]2012年初步研究了禾谷镰刀菌 (Fusarium graminearum) PRMT的生物学功能,4种PRMT基因中只有amt1突变体表型明显,侵染力和毒力都大大降低,而其余3种突变体都与野生型无明显差异。构巢曲霉 (Aspergillus nidulans) 中已报道的有3种精氨酸甲基转移酶,即RmtA、RmtB和RmtC。rmtA,rmtB和rmtC敲除突变体在营养生长和生殖上都没有明显缺陷,但rmtA,rmtC突变体对过氧化氢的敏感性增强,且高温处理下,rmtC突变体菌丝生长明显减慢[29]。黄曲霉 (Aspergillus flavus) 中rmtA的缺失可导致该菌分生孢子数增加,菌核数减少,并且影响黄曲霉毒素B1的产生,进一步影响其次级代谢[30]。白色念珠菌 (Candida albicans) 中所报道的与酵母HMT1、RMT2同源的CaHMT1和CaRMT2对白色念珠菌的生长都没有影响,但是CaHMT1是该菌主要的功能性精氨酸甲基转移酶[31]。
然而,精氨酸甲基转移酶是否影响稻瘟病菌的致病性尚未可知。本研究通过生物信息学方法确定稻瘟病菌可能的编码精氨酸甲基转移酶基因,并对其进行生物信息学分析,最后,通过荧光定量PCR方法分析稻瘟病菌PRMTs在各个生育阶段的表达水平,旨为进一步研究稻瘟病菌精氨酸甲基转移酶的功能奠定基础。
1 材料与方法 1.1 材料本研究所用稻瘟病菌Guy11菌株由中国农业科学院植物保护研究所植物抗病功能基因组保存。
1.2 方法 1.2.1 菌丝的收集在超净工作台中将干燥保存在滤纸片上的Guy11菌株置于燕麦培养基上,在25℃培养室中倒置暗培养2 d后,再光照培养5 d。将培养好的Guy11用灭菌水涂断,接种到CM液体完全培养基中,28℃,180 r/min黑暗条件下培养2 d,过滤即可得到新鲜菌丝。用滤纸吸干水分后液氮速冻,保存于-80℃待用。
1.2.2 孢子的收集将Guy11接种到番茄燕麦培养基上,25℃光照培养7-10 d后,用灭菌水将培养基表面的菌丝洗去,晾干后用双层纱布封口。待光照培养2-3 d后,可用一定量灭菌水充分洗下孢子,miracloth滤布过滤后,离心滤液即可得到新鲜的孢子。用滤纸吸干水分后液氮速冻,保存于-80℃待用。
1.2.3 附着胞的收集用血球计数板观察,将孢子悬浮液浓度调至1×105个/mL,均匀喷在人工疏水表面上,25℃黑暗条件下保湿培养3 h或12 h后卡片刮取,收集水珠,用3层滤纸过滤后即可收集到不同阶段的附着胞。液氮速冻,保存于-80℃待用。
1.2.4 侵染菌丝的收集用0.01%的Tween20把孢子悬浮液调至浓度为1×105个/mL,喷雾接种到大麦一叶平展期叶片背面,25℃黑暗条件下保湿培养,24 h之后给予光照处理,分别在接种后18 h、24 h和42 h取样。取样时,小心撕下大麦叶片表皮,液氮速冻后,保存于-80℃待用。
1.2.5 RNA提取与qRT-PCR本实验采用TRIzol法提取稻瘟病菌Guy11的7个不同生育阶段 (营养菌丝、分生孢子、3 h/12 h附着胞和18 h/24 h/42 h侵染菌丝) 的总RNA,通过RNA反转录试剂盒 (Promega公司) 获得cDNA,稀释后作为qRT-PCR模板。荧光定量PCR引物 (表 1) 使用Primer5.0软件进行设计,并由北京六合华大基因科技有限公司合成。
本实验使用TaKaRa公司的SYBR Premix Ex TaqTM试剂盒进行qRT-PCR反应。反应体系为20 μL,以稻瘟菌Actin基因MGG_03982为内参基因,并由MyiQ2双色实时荧光定量PCR仪 (Bio-Rad,美国) 完成,表达量的计算按照2-ΔΔCt进行[32]。
1.2.6 生物信息学分析在NCBI网站上,利用已发表的酿酒酵母 (Saccharomyces cerevisiae) 中3种精氨酸甲基转移酶的氨基酸序列[17]在稻瘟菌数据库中进行BLASTP搜索,得到4种与酵母PRMTs同源的稻瘟病菌PRMTs;利用ClustalW进行序列比对;利用同样方法获取其他物种中的精氨酸甲基转移酶序列;利用MEGA5.0软件构建系统进化树。
2 结果 2.1 MoPRMTs基因的克隆根据酿酒酵母的3种精氨酸甲基转移酶 (HM-T1,RMT2和HSL7) 的氨基酸序列,搜索比对 (Bl-astP) 稻瘟病菌全基因组数据库 (https://data.broadinstitute.org/annotation/genome/magnaporthe_comparative/MultiHome.html),得到4种稻瘟病菌精氨酸甲基转移酶,基因号为MGG_04584,MGG_17318,MGG_09290和MGG_03894,分别命名为MoPRMT1,MoPRMT2,MoPRMT3和MoPRMT4。MoPRMT1的cDNA全长为1 038 bp,编码一个含345个氨基酸的蛋白,与酵母HMT1相似性高达58%;MoPRMT2亦是同源HMT1而来,相似性为40%;经序列比对可知,MoPRMT3和MoPRMT4均含有精氨酸甲基转移酶家族特有结构域,且分别与RMT2和HSL7同源,序列相似性分别为37%和27%。
2.2 MoPRMTs基因的生物信息学分析每个精氨酸甲基转移酶都包含一段非常保守的甲基转移酶结构域,该结构域能结合甲基供体S-腺苷甲硫氨酸 (SAM) 和蛋白底物的子域[33]。利用ClustalW进行MoPRMTs氨基酸序列比对分析发现,其保守结构域包括Motif Ⅰ、Motif Post Ⅰ、Motif Ⅱ和Motif Ⅲ,而THW loop则是PRMTs特有结构域 (图 1)。

|
| 图 1 稻瘟病菌MoPRMTs氨基酸序列比对分析 |
本实验通过构建系统发育树 (图 2) 来明确稻瘟病菌4种精氨酸甲基转移酶与其他物种中PRMTs的亲缘关系,将稻瘟病菌MoPRMTs编码的蛋白与人类 (Homo sapiens)、酿酒酵母 (Saccharomyces cerevisiae)、禾谷镰刀菌 (Fusarium graminearum)、粗糙脉孢菌 (Neurospora crassa) 和构巢曲霉 (Aspergillus nidulans) 中的同源蛋白进行序列比对,进而构建系统发育树。从进化树分析可得,酿酒酵母仅含3种PRMTs;稻瘟菌PRMTs同粗糙脉孢菌、禾谷镰刀菌等丝状真菌一样,都含有4种精氨酸甲基转移酶,且亲缘关系最近;而在人类基因组中发现9种PRMTs[6]。
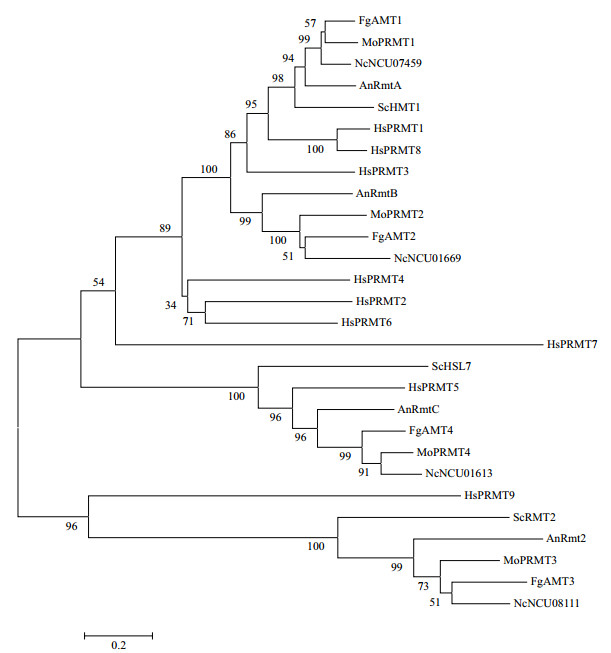
|
| 图 2 不同物种PRMTs氨基酸序列系统进化树分析 Hs:Homo sapiens(人类);Mo:Magnaporthe oryzae(稻瘟病菌);Sc:Saccharomyces cerevisiae(酿酒酵母);Fg:Fusarium graminearum(禾谷镰刀菌);Nc:Neurospora crassa(粗糙脉孢菌);An:Aspergillus nidulans(构巢曲霉) |
本研究利用实时荧光定量PCR技术,研究稻瘟病菌MoPRMTs基因在不同生长和侵染时期的表达水平。分析结果 (图 3) 表明,稻瘟病菌中4个MoPRMTs基因的表达谱差异较大,且表达峰值也处于不同时间点。MoPRMT1在侵染后24 h表达量达到最高峰,而其他阶段的表达水平基本一致;有趣的是MoPRMT2,MoPRMT3和MoPRMT4在侵染后24 h几乎没有表达,而在禾谷镰刀菌中,与MoPRMT1同源的AMT1,其敲除突变体的致病力明显下降[28],可推测MoPRMT1对于稻瘟病菌侵染后侵染菌丝的生长可能起重要作用。

|
| 图 3 稻瘟病菌MoPRMTs基因在不同时期的表达分析 Myc:mycelium (菌丝);Spo:spore (孢子);AP:appressoria (附着胞);IH:invasive hyphae (侵染菌丝) |
MoPRMT2与其他3个基因相比,各阶段的表达量都较低,且各时期的表达水平也没有很明显的变化;MoPRMT3和MoPRMT4在成熟附着胞时期表达量都达到最高 (图 3),由此可推测,这两个基因对于附着胞的成熟有很重要的作用,可能与侵染栓的形成密切相关。此外,MoPRMT3在芽管时期也有相对高的表达,可见MoPRMT3对于稻瘟病菌附着胞形成也有一定作用;MoPRMT4在侵染后42 h也出现表达峰值,推测该基因对于稻瘟病菌侵染菌丝在寄主体内扩展起重要作用。
3 讨论精氨酸甲基转移酶催化蛋白质甲基化修饰在真核生物的多种细胞过程中起重要作用。本研究通过生物信息学分析发现,稻瘟病菌中含有4个PRMTs,将稻瘟病菌MoPRMTs基因编码的蛋白与人类、酿酒酵母、禾谷镰刀菌、粗糙脉孢菌和构巢曲霉中的同源蛋白进行序列比对和进化树分析发现,稻瘟病菌与禾谷镰刀菌亲缘关系最近,与人类的亲缘关系最远。MoPRMT1和MoPRMT2都与HMT1同源,而MoPRMT3和MoPRMT4分别与RMT2和HSL7同源。
最早研究的是酿酒酵母中的HMT1,HMT1对酵母的生长并非必需,但其功能涉及广泛。HMT1催化的底物主要是hnRNPs,包括Hrp1,Nab2和Npl3等,通过甲基化hnRNPs组分,进而影响前体mRNA修饰,转录的延伸和终止[34-39];RMT2主要催化核糖体蛋白Rpl12[40];Hsl7则对于出芽酵母的芽颈生长、磷酸化和Swe1的降解至关重要[41]。
而禾谷镰刀菌AMT1则是与HMT1和MoPRMT1同源的关键性精氨酸甲基转移酶,AMT1敲除突变体的营养生长减慢,无性生殖和有性生殖都没有变化,对氧化物和高渗溶液敏感性增加,毒素和侵染力都明显降低;且定位于细胞核和细胞质,调控hnRNPs组分的核质转运;而其他3种PRMT均无表型;amt1/amt2双敲除突变体表型与AMT1相同,说明二者不存在功能冗余[28]。在构巢曲霉中,rmtA,rmtB和rmtC敲除突变体在营养生长和生殖上都没有缺陷[29]。以上真菌中PRMTs的研究结果可为稻瘟病菌中MoPRMTs基因功能研究提供借鉴。
稻瘟病菌侵染地上部分一般是通过分生孢子传播途径,扩散到水稻叶片等组织,在适宜湿度环境下,分生孢子黏附在水稻叶片表面,依靠其自身营养而萌发形成芽管,继而芽管再进行特异性分化产生具有黑色素的附着胞[42],附着胞形成侵染栓并穿透叶片等组织的角质层和表皮细胞壁,侵入表皮细胞。稻瘟病菌侵入后菌丝迅速扩展蔓延,在水稻细胞中生长并侵染邻近细胞进而深入叶肉细胞[43, 44]。
在稻瘟病菌的不同发育阶段,4个MoPRMTs基因的表达模式和表达量各不相同。MoPRMT1在24 h侵染菌丝中的表达量远高于其他阶段,在其他阶段表达量几乎一致,对照上述稻瘟病菌侵染过程,可推测MoPRMT1在稻瘟病菌侵入寄主细胞后的生长过程中起重要作用;MoPRMT2与其他3个基因相比,各阶段的表达量都较低,仅在成熟附着胞时期和侵染后18、24 h表达量略高;MoPRMT3在芽管和附着胞发育阶段表达量明显升高,说明该基因可能参与附着胞的形成和成熟;MoPRMT3和MoPRMT4在成熟附着胞时期表达量都达到最高,由此可推测,二者参与了稻瘟病菌侵入寄主细胞的关键阶段;MoPRMT4在侵染后42 h也出现表达峰值,推测该基因对于稻瘟病菌侵染菌丝在寄主体内的扩展也可能起重要作用。
4 结论本研究通过与酵母的3个精氨酸甲基转移酶同源比对发现,稻瘟病菌中含有4个PRMTs。序列比对分析表明,这4个精氨酸甲基转移酶都含有PRMTs家族保守结构域。系统发育树分析结果表明,稻瘟病菌PRMTs在丝状真菌中非常保守,亲缘关系最近。实时荧光定量PCR分析这4个PRMTs在稻瘟病菌不同生长和发育时期的表达谱,表明MoPRMT1在侵染后24 h表达量达到最高峰,而其他阶段的表达水平基本一致;MoPRMT2与其他3个基因相比,各阶段的表达量都较低,且各时期的表达水平也没有很明显的变化;MoPRMT3在芽管和附着胞发育阶段表达量明显较高,MoPRMT3和MoPRMT4在成熟附着胞时期表达量都达到最高,MoPRMT4在侵染后42 h也出现表达峰值。
| [1] | Valent B, Chumley FG. Molecular genetic analysis of the rice blast fungus, Magnaporthe grisea[J]. Annual Review of Phytopathology, 1991, 29 (29): 443–467. |
| [2] | Dean RA, Talbot NJ, Ebbole DJ, et al. The genome sequence of the rice blast fungus Magnaporthe grisea[J]. Nature, 2005, 434 (7036): 980–986. DOI:10.1038/nature03449 |
| [3] | Petrossian TC, Clarke SG. Uncovering the human methyltransfera-some[J]. Molecular & Cellular Proteomics Mcp, 2010, 10 (1): M110. 000976. |
| [4] | Richon VM, Johnston D, Sneeringer CJ, et al. Chemogenetic analysis of human protein methyltransferases[J]. Chemical Biology & Drug Design, 2011, 78 (2): 199–210. |
| [5] | Branscombe TL, Frankel A, Lee JH, et al. PRMT5(Janus kinase-binding protein 1) catalyzes the formation of symmetric dimethylarginine residues in proteins[J]. Journal of Biological Chemistry, 2001, 276 (35): 32971–32976. DOI:10.1074/jbc.M105412200 |
| [6] | Yang Y, Hadjikyriacou A, Xia Z, et al. PRMT9 is a type Ⅱ methyltransferase that methylates the splicing factor SAP145[J]. Nature Communications, 2015, 6 : 6428. DOI:10.1038/ncomms7428 |
| [7] | Zurita-Lopez CI, Sandberg T, Kelly R, et al. Human protein arginine methyltransferase 7(PRMT7) is a type Ⅲ enzyme forming ω-NG-monomethylated arginine residues[J]. Journal of Biological Chemistry, 2012, 287 (11): 7859–7870. DOI:10.1074/jbc.M111.336271 |
| [8] | Lorenzo AD, Bedford MT. Histone arginine methylation[J]. Febs Letters, 2011, 585 (13): 2024–2031. DOI:10.1016/j.febslet.2010.11.010 |
| [9] | Yang Y, Bedford MT. Protein arginine methyltransferases and cancer[J]. Nature Reviews Cancer, 2012, 13 (1): 37–50. DOI:10.1038/nrc3409 |
| [10] | Swiercz R, Cheng D, Kim D, et al. Ribosomal protein rpS2 is hypomethylated in PRMT3-deficient mice[J]. Journal of Biological Chemistry, 2007, 282 (23): 16917–16923. DOI:10.1074/jbc.M609778200 |
| [11] | Bedford MT, Clarke SG. Protein arginine methylation in mammals: who, what, and why[J]. Molecular Cell, 2009, 33 (1): 1–13. DOI:10.1016/j.molcel.2008.12.013 |
| [12] | Hong H, Kao C, Jeng MH, et al. Aberrant expression of CARM1, a transcriptional coactivator of androgen receptor, in the development of prostate carcinoma and androgen-independent status[J]. Cancer, 2004, 101 (1): 83–89. DOI:10.1002/(ISSN)1097-0142 |
| [13] | Cheng D, Yadav N, King RW, et al. Small molecule regulators of protein arginine methyltransferases[J]. Journal of Biological Chemistry, 2004, 279 (23): 23892–23899. DOI:10.1074/jbc.M401853200 |
| [14] | Pal S, Vishwanath SN, Erdjument-Bromage H, et al. Human SWI/SNF-associated PRMT5 methylates histone H3 arginine 8 and negatively regulates expression of ST7 and NM23 tumor suppressor genes[J]. Mol Cell Biol, 2004, 24 (21): 9630–9645. DOI:10.1128/MCB.24.21.9630-9645.2004 |
| [15] | Zhang L, Tran NT, Su H, et al. Cross-talk between PRMT1-mediated methylation and ubiquitylation on RBM15 controls RNA splicing[J]. Elife Sciences, 2015, 4 : pii: e07938. |
| [16] | Zhao DY, Gish G, Braunschweig U, et al. SMN and symmetric arginine dimethylation of RNA polymerase Ⅱ C-terminal domain control termination[J]. Nature, 2015, 529 (7584): 48–53. DOI:10.1038/nature16469 |
| [17] | Sayegh J, Clarke SG. Hsl7 is a substrate-specific type Ⅱ protein arginine methyltransferase in yeast[J]. Biochemical & Biophysical Research Communications, 2008, 372 (4): 811–815. |
| [18] | Cimato TR, Tang J, Xu Y, et al. Nerve growth factor-mediated increases in protein methylation occur predominantly at type Ⅰ argi-nine methylation sites and involve protein arginine methyltransfe-rase 1[J]. J Neurosci Res, 2002, 67 (4): 435–442. DOI:10.1002/(ISSN)1097-4547 |
| [19] | Mcbride AE, Weiss VH, Kim HK, et al. Analysis of the yeast arginine methyltransferase Hmt1p/Rmt1p and its in vivo function[J]. Journal of Biological Chemistry, 2000, 275 (5): 3125–3136. |
| [20] | Zobel-Thropp P, Gary JD, Clarke S. Delta-N-Methylarginine is a novel posttranslational modification of arginine residues in yeast proteins[J]. J Biol Chem, 1998, 273 (45): 29283–29286. DOI:10.1074/jbc.273.45.29283 |
| [21] | Bedford MT, Richard S. Arginine methylation an emerging regulator of protein function[J]. Molecular Cell, 2005, 18 (3): 263–272. DOI:10.1016/j.molcel.2005.04.003 |
| [22] | Boisvert F, Chénard CA, Richard S. Protein interfaces in signaling regulated by arginine methylation[J]. Sci STKE, 2005, 2005 (271): re2. |
| [23] | Yu MC, Bachand F, Mcbride AE, et al. Arginine methyltransferase affects interactions and recruitment of mRNA processing and export factors[J]. Genes & Development, 2004, 18 (16): 2024–2035. |
| [24] | Shen EC, Henry MF, Weiss VH, et al. Arginine methylation facilitates the nuclear export of hnRNP proteins[J]. Genes & Development, 1998, 12 (5): 679–691. |
| [25] | Hang R, Liu C, Ahmad A, et al. Arabidopsis protein arginine methyltransferase 3 is required for ribosome biogenesis by affecting precursor ribosomal RNA processing[J]. Proceedings of the National Academy of Sciences, 2014, 111 (45): 16190–16195. DOI:10.1073/pnas.1412697111 |
| [26] | Deng X, Dean C. Arginine methylation mediated by the Arabidopsis homolog of PRMT5 is essential for proper pre-mRNA splicing[J]. Proc Natl Acad Sci, 2010, 107 (44): 19114–19119. DOI:10.1073/pnas.1009669107 |
| [27] | Niu L, Lu F, Pei Y, et al. Regulation of flowering time by the protein arginine methyltransferase AtPRMT10[J]. Embo Reports, 2007, 8 (12): 1190–1195. DOI:10.1038/sj.embor.7401111 |
| [28] | Wang G, Wang C, Hou R, et al. The AMT1 arginine methyltransfe-rase gene is important for plant infection and normal hyphal growth in Fusarium graminearum[J]. PLoS One, 2012, 7 (5): e38324. DOI:10.1371/journal.pone.0038324 |
| [29] | Bauer I, Graessle S, Loioll P, et al. Novel insights into the functio-nal role of three protein arginine methyltransferases in Aspergillus nidulans[J]. Fungal Genet Biol, 2010, 47 : 551–561. DOI:10.1016/j.fgb.2010.03.006 |
| [30] | Satterlee T, Cary JW, Calvo AM. RmtA, a putative arginine methyl-transferase, regulates secondary metabolism and development in Aspergillus flavus[J]. PLoS One, 2016, 11 : e0155575. DOI:10.1371/journal.pone.0155575 |
| [31] | Mcbride AE, Zuritalopez C, Regis A, et al. Protein arginine methylation in Candida albicans: role in nuclear transport[J]. Eukaryotic Cell, 2007, 6 : 1119–1129. DOI:10.1128/EC.00074-07 |
| [32] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C (T)) method[J]. Methods, 2001, 25 (4): 402–408. DOI:10.1006/meth.2001.1262 |
| [33] | Bachand F. Protein arginine methyltransferases: from unicellular eukaryotes to humans[J]. Eukaryotic Cell, 2007, 6 : 889–898. DOI:10.1128/EC.00099-07 |
| [34] | Green DM, Marfatia KA, Crafton EB, et al. Nab2p is required for poly (A) RNA export in Saccharomyces cerevisiae and is regulated by arginine methylation via Hmt1p[J]. Journal of Biological Chemistry, 2002, 277 (10): 7752–7760. DOI:10.1074/jbc.M110053200 |
| [35] | Kessler MM, Henry MF, Shen E, et al. Hrp1, a sequence-specific RNA-binding protein that shuttles between the nucleus and the cytoplasm, is required for mRNA 3'-end formation in yeast[J]. Genes & Development, 1997, 11 (19): 2545–2556. |
| [36] | González CI, Ruiz-Echevarría MJ, Vasudevan S, et al. The yeast hnRNP-like protein Hrp1/Nab4 marks a transcript for nonsense-mediated mRNA decay[J]. Mol Cell, 2000, 5 (3): 489–499. DOI:10.1016/S1097-2765(00)80443-8 |
| [37] | Gross S, Moore CL. Rna15 interaction with the a-rich yeast polyadenylation signal is an essential step in mRNA 3'-end formation[J]. Mol Cell Biol, 2002, 21 (23): 8045–8055. |
| [38] | Hector RE, Nykamp KR, Dheur S, et al. Dual requirement for yeast hnRNP Nab2p in mRNA poly (A) tail length control and nuclear export[J]. Embo Journal, 2002, 21 (7): 1800–1810. DOI:10.1093/emboj/21.7.1800 |
| [39] | Wong CM, Tang HMV, Kong KYE, et al. Yeast arginine methyltransferase Hmt1p regulates transcription elongation and termination by methylating Npl3p[J]. Nucleic Acids Research, 2010, 38 (7): 2217–2228. DOI:10.1093/nar/gkp1133 |
| [40] | Chern MK, Chang KN, Liu LF, et al. Yeast ribosomal protein L12 is a substrate of protein-arginine methyltransferase 2[J]. Journal of Biological Chemistry, 2002, 277 (18): 15345–15353. DOI:10.1074/jbc.M111379200 |
| [41] | Kucharczyk R, Gromadka R, Migdalski A, et al. Disruption of six novel yeast genes located on chromosome Ⅱ reveals one gene essential for vegetative growth and two required for sporulation and conferring hypersensitivity to various chemicals[J]. Yeast, 1999, 15 (10B): 987–1000. DOI:10.1002/(ISSN)1097-0061 |
| [42] | Howard RJ, Bourett TM, Ferrari MA. Infection by magnaporthe: an in vitro analysis[M]. Electron Microscopy of Plant Pathogens, 1991: 251-264. |
| [43] | Heath MC, Valent B, Howard RJ, et al. Correlations between cytologically detected plant-fungal interactions and pathogenicity of Magnaporthe grisea toward weeping lovegrass[J]. Phytopathology, 1990, 80 (12): 1382–1386. DOI:10.1094/Phyto-80-1382 |
| [44] | Heath MC, Valent B, Howard RJ, et al. Interactions of two strains of Magnaporthe grisea with rice, goosegrass, and weeping lovegrass[J]. Can J Bot, 2011, 68 (8): 1627–1637. |





