OsRhoGAP (Rho GTPase) 是水稻Rho/Rop蛋白内在酶的激活蛋白。OsRhoGAP最有代表性的功能就是作为信号网络中Rho/Rop的活性调控组分,参与到依赖于细胞骨架重组和极性囊泡运输等基本细胞过程中,如细胞极性、细胞形态发生和细胞运动[1]。最初对RhoGAP的认识来自对动物细胞的研究,研究发现RhoGAPs的功能是作为Rho/ROP的相互作用蛋白,通过与GTP结合形式的Rho/ROP结合,激活后者内在的GTP酶活性,促进其转换成为GDP结合的失活形式,从而涉及多种细胞过程,包括运动、收缩、生长、分化和发育[2]。迄今,已从真核生物中分离鉴定的RhoGAPs有70余种,但是,对植物RopGAP的研究非常有限。
目前普遍认为植物RhoGAP/RopGAP涉及多种植物细胞生理过程,如参与了Rho/ROP对极性生长的调节。有研究显示,拟南芥REN1(ROP1 enhancer 1) 编码的RopGAP蛋白能够抑制花粉管顶端的AtROP1活性,该基因的缺失会影响花粉管的极性生长[3]。Ulrich通过转基因实验证实,烟草NtRhoGAP1的保守氨基酸突变会导致花粉管膨大[4],这与烟草Rho基因过表达的结果一致,说明NtRhoGAP1与NtRAC5的功能相关。大麦RopGAP蛋白MAGAP1与Rop家族成员RACB相互作用,调控根毛延伸过程[5]。此外,有研究表明,RopGAP还参与植物生物及非生物胁迫应答。Baxter-Burrell等[6]发现,拟南芥AtRopGAP4参与氧胁迫的耐受性调控;Hoefle等[5]则证实大麦中与微管结合的MAGAP1能通过调节微管骨架的动态变化,抵御真菌侵袭。植物中对烟草、拟南芥RopGAPs的研究已取得重要进展,但是有关水稻RhoGAP/RopGAP的研究尚未见报道。
水稻不仅是主要的粮食作物,而且是重要的模式植物。水稻育性控制的研究对于探讨水稻产量形成具有重要意义。研究表明,水稻ROP基因OsRacD与水稻育性相关[7-9]。为了研究该基因在育性控制过程中的功能和作用机制,以该基因组成型激活形式G15V-OsRacD为诱饵进行酵母双杂交筛选,从雌雄蕊形成期水稻幼穗中分离到一些潜在的相互作用蛋白编码基因[10, 11],其中包括OsRhoGAP1(GenBank登录号AY364310)。本研究基于实验室的前期工作基础,对水稻Rho蛋白OsRacD潜在互作蛋白编码基因OsRhoGAP1进行了生物信息学和表达模式分析,旨在为深入研究该基因的功能及作用机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 植物材料水稻材料为粳稻栽培品种日本晴 (Oryza sativa L. japonica.cv. Nipponbare),种植于河南师范大学生命科学学院水稻实验基地 (北纬35.18,东经113.52)。
1.1.2 试剂TRIzol总RNA提取试剂、实时定量反转录试剂盒SYBR PrimeScript RT-PCR Kit、SYBR® Premix Ex TaqTM均购自大连宝生物公司。实验所用的植物激素脱落酸 (Abscisic acid,ABA)、吲哚乙酸 (Indoleacetic acid,IAA)、水杨酸 (Salicylic acid,SA)、赤霉素 (Gibberellin,GA)、6-苄氨基嘌呤 (6-benzyl aminopurine,6-BA) 和茉莉酸甲酯 (Methyl Jasmonate,MeJA) 均购于北京鼎国生物公司。引物由北京金唯智生物公司合成。
1.2 方法 1.2.1 水稻材料的培养及处理水稻种子以75%酒精消毒后,于2015年5月在水稻实验基地播种,并进行常规管理。在水稻生长的幼苗期 (30 d)、幼穗期 (60 d)、成熟期 (90 d) 分别采集根、叶以及雌雄蕊形成期的幼穗,液氮速冻后于-80℃保存备用。
植物激素处理采用的是水培法培育的水稻幼苗,水稻种子以75%酒精消毒后,于28℃黑暗培养2-3 d,萌发后转移到人工气候箱中培养 (光照16 h,28℃ /黑暗8 h,25℃,光照强度16 000 lx)。整个过程以Yoshida[12]培养液培养,每天更换新鲜的培养液。待生长至三叶一心期 (约14 d) 进行激素 (ABA、IAA、SA、GA、6-BA、MeJA) 处理,处理方法为100 μmol/L植物激素反复喷洒水稻幼苗叶片,确保植株通过气孔接受到激素刺激,分别取处理后0 h、3 h、6 h、12 h的叶组织,用液氮速冻后于-80℃保存备用。
1.2.2 生物信息学分析将OsRhoGAP1基因的cDNA序列提交生物信息学网站NCBI (http://www.ncbi.nlm.nih.gov/),以Blastn检索同源序列,并通过ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html) 确定其开放阅读框;以Blastp和ScanProsite软件分析OsRhoGAP1基因编码蛋白的保守结构域;并在水稻基因组序列数据库 (http://www.ncbi.nlm.nih.gov/genome/seq/BlastGen/BlastGen.cgi?taxid=4530) 中检索,使用DNAMAN软件分析OsRhoGAP1的基因结构,将其基因结构与检索到的同源序列进行多序列比对,采用clustal1.81软件和MEGA5.2.2软件生成系统进化树。
1.2.3 总RNA的提取及cDNA的合成按照TRIzol总RNA提取试剂说明书,分别提取水稻根、叶以及雌雄蕊形成期的幼穗的总RNA,经DNA酶消化后,按照SYBR PrimeScript RT-PCR Kit试剂盒说明书要求,反转录为cDNA。
1.2.4 OsRhoGAP1表达的检测以水稻OsAct1[13]为内参基因,采用qRT-PCR检测OsRhoGAP1基因的表达特征性。荧光定量PCR反应体系为: cDNA 2 μL;2 x SYBR® Premix Ex TaqTM 10 μL;PCR Forward/ Reverse primer (10 μmol/L) 各0.4 μL (表 1);ROX Reference DyeⅡ(50x)0.4 μL;DEPC水6.8 μL。使用ABI 7500实时PCR仪 (Applied Biosystems) 进行PCR扩增。反应采用两步法PCR扩增标准程序: 95℃ 30 s;95℃: 5 s;60℃: 34 s,进行40个循环。
记录并保存各组扩增的CT值。数据分析采用ABI PRISM 7500型PCR仪自带的SDS Software v 1.4软件,以2-△△Ct法[14]分析基因表达水平,数据统计采用Microsoft Excel软件,显著性分析采用Spass v19.0软件。
2 结果 2.1 OsRhoGAP1的序列特征和同源性分析将OsRhoGAP1基因cDNA序列进行Blastp检索和ScanProsite工具分析,发现其中包含植物RhoGAPs两个典型的保守结构域,即CRIB基序和RhoGAP结构域 (图 1)。根据已报道的植物RopGAP序列特征,推测OsRhoGAP1的N端还缺少部分氨基酸。将OsRhoGAP1与通过Blastn检索到的相似性100%的序列进行比对与拼接,再通过ORF finder软件分析,预测OsRhoGAP1的cDNA编码区全长1 353 bp,编码450个氨基酸和1个终止密码。其中,89-102位和134-315位氨基酸分别对应CRIB基序和RhoGAP结构域。
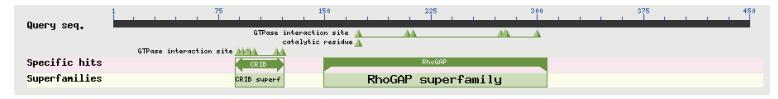
|
| 图 1 OsRhoGAP1基因编码蛋白保守结构域分析 |
将OsRhoGAP1的氨基酸序列在NCBI数据库中进行其同源性的比对分析,发现来自单子叶植物,如玉米、小麦、谷子的同源序列较多,而来自双子叶植物的序列相对较少。选择其中15个蛋白的序列与OsRhoGAP1进行比对,DNAMAN软件分析的结果显示,与单子叶植物中RhoGAP相比,OsRhoGAP1与水稻XP_015620392和EEC96414相似性分别达100%和90%,与高粱蛋白XP_002442-244、谷子蛋白XP_004962865和山羊草蛋白EMT-12220的相似性为99%,与玉米XP_008672984的相似性为96%;但在双子叶植物中,OsRhoGAP1与拟南芥、烟草、木薯中Rop蛋白的相似性在86%以下。采用DNAMAN软件进行多序列比对,结果 (图 2) 显示它们的高同源性区段集中在RhoGAP结构域,该结构域中包含3个保守的特征序列EGIF/YR,RELP和LNKMN。

|
| 图 2 OsRhoGAP1与其他植物RhoGAP氨基酸序列同源比对结果 参与同源比对的分别为:水稻 (OsRhoGAP1,XP_006664074,XP_015620392,EEC69414)、玉米 (NP_001151189,XP_008672984)、高粱 (XP_002442244)、谷子 (XP_004962865)、山羊草 (EMT12220)、二穗短柄草 (XP_003575834)、拟南芥 (AAC79102)、烟草 (XP_009618734)、芝麻 (XP_011079758)、落花生 (XP_016184376)、木薯 (OAY49614)、可可 (EOX94911)。黑色框代表 100%的氨基酸同源性;红色框和蓝色框分别代表有较少和较多的不同氨基酸的替换 |
利用clustal1.81软件和MEGA5.2.2软件以NJ法生成系统进化树进行Bootstrap检测。结果 (图 3) 显示,在分析的所有序列中,OsRhoGAP1与水稻蛋白XP_015620392和EEC96414的进化地位最接近。
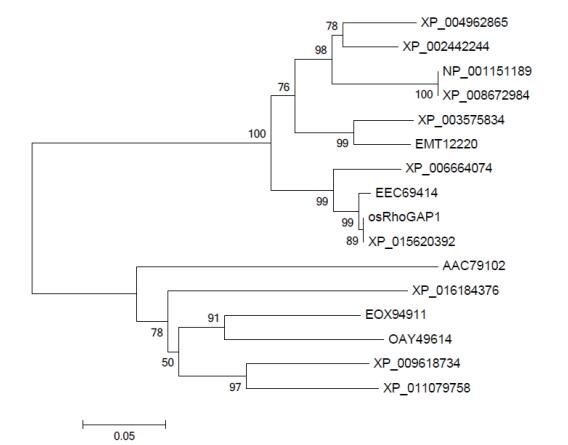
|
| 图 3 OsRhoGAP1基于其他物种中的RhoGAP氨基酸序列聚合进化树分析结果 |
以组成型表达的水稻肌动蛋白OsAct1基因[13]为内参,通过实时定量PCR检测OsRhoGAP1基因在水稻生长至幼苗期、幼穗期和成熟期时的根、叶、幼穗组织中的表达水平。结果 (图 4) 显示,OsRhoGAP1在水稻的多种组织器官及不同的发育阶段中均有表达,但表达水平差异明显。在幼苗期,该基因主要在根中的表达,在根中的表达量为叶中的1.6倍;在幼穗期,该基因主要在幼穗和叶中表达,表达量分别达到根中的7.1倍和3.6倍;在成熟期,该基因主要在根中的表达,达到了叶中表达量的25倍。说明OsRhoGAP1的表达具有时空特异性,主要在细胞快速分裂的幼根、幼叶、幼穗组织以及成熟根中高表达,提示该基因可能与水稻生长发育分化有密切联系。
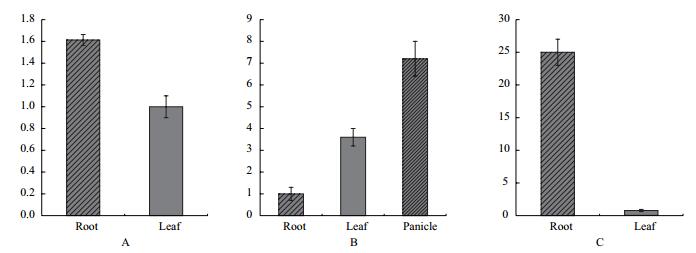
|
| 图 4 OsRhoGAP1在水稻不同发育阶段不同组织中的表达 A:幼苗期;B:幼穗期;C:成熟期 |
为了检测植物激素对OsRhoGAP1基因表达的影响,采用6种激素处理水稻幼苗,以实时定量PCR检测幼苗地上部分在处理后0 h、3 h、6 h、12 h内OsRhoGAP1基因的表达水平。结果 (图 5) 显示,经ABA、SA、GA和MeJA处理后,OsRhoGAP1基因表达水平均有明显上调,但幅度不同。其中经ABA、SA以及MeJA处理后,OsRhoGAP1基因的表达水平均在处理后6 h达到峰值,表达水平达到3倍以上,而后下降;而经GA处理后,OsRhoGAP1基因的表达量持续上升直到12 h后达到峰值,表达水平达到6.4倍。经IAA和6-BA处理后,OsRhoGAP1基因表达水平持续下调,到12 h后达到原来的0.05%以下。以上结果说明,在幼苗期,OsRhoGAP1基因对6种激素 (ABA、IAA、SA、GA、6-BA、MeJA) 响应比较灵敏,其中,ABA、SA、GA和MeJA均使该基因上调,而IAA和6-BA使该基因下调。
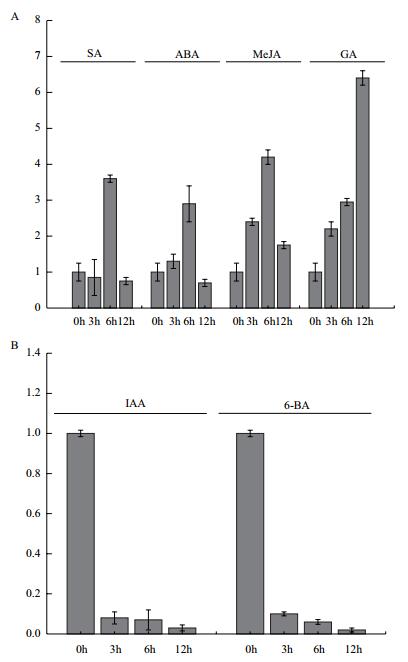
|
| 图 5 OsRhoGAP1在6种植物激素 (SA、ABA、MeJA、GA、IAA、6-BA) 处理下的表达分析 |
Rho GTPase作为植物ROP家族的一员,与哺乳动物Cdc42和Rac GTPases密切相关,RhoGAP在植物的许多生理过程中发挥了重要的作用,如Ca2+依赖的花粉管的生长、根毛发育、H2O2介导的细胞死亡、活性氧 (ROS) 的生成及植物激素应答等[15-18]。在动物和真菌中的RhoGAP蛋白中很少发现CRIB结构域,但是在大部分高等植物中,如烟草、拟南芥中两种结构域是同时存在的,RhoGAP高亲和性特异性的调节不仅需要GAP结构域中的精氨酸催化剂碱基,而且还需要CRIB结构域的催化机制[19, 20]。
本研究对实验室前期文库筛选获得的OsRhoGAP1进行序列分析和拼接,并进行同源检索,确定该基因含有RhoGAP和CRIB两个保守结构域,以及RhoGAP结构域中3个保守的典型催化序列。现有研究认为,作为植物RhoGAP特有的结构,CRIB结构域能够调控RopGAP与Rop的互作。例如,拟南芥AtRopGAP1的CRIB结构域增强了它对AtRop1的活性[15];AtRopGAP1和AtRopGAP2的CRIB结构域保守位点突变会影响其与Rop蛋白的结合[15, 19];烟草NtRhoGAP1的CRIB结构域提高了它对Rop蛋白Rac5的活性[4]。推测OsRhoGAP1通过CRIB结构域介导自身与OsRacD结合,并通过RhoGAP结构域催化位点激活OsRacD内在GTP酶活性,促进其结合的GTP水解为GDP,最终导致OsRacD失活。同时,前期实验结果显示,OsRhoGAP1只与激活形式的OsRacD结合,推测OsRhoGAP1只是单纯介导GTP向GDP的转换,而不参与其他过程。所以,本实验所分离的OsRhoGAP1属于RhoGAP亚家族成员。
同源性分析显示,OsRhoGAP1与其他植物RhoGAPs在RhoGAP结构域区段同源程度较高,这与Ulrich研究[4]相符,烟草RhoGAP1的RhoGAP结构域与拟南芥AtRopGAP1、水稻AAX95236、百脉根AAC62624的RhoGAP结构域的相似性分别达到72%、63%和62%。OsRhoGAP1与双子叶植物RhoGAP相似性达到96%以上;但与单子叶植物RhoGAP相似性在86%以下。高度保守的RhoGAP催化结构域提示了RopGAP在功能或调控效应上的保守性。
水稻的生长发育是有规律的,在不同的发育阶段具有不同的特性,如水稻幼苗期的主要发育特点是根系的生长、分蘖数增加、叶片增多;在幼穗期,水稻植株最显著特征是出现幼穗的分化;在成熟期,水稻植株主要进行生殖生长,即籽粒的形成和充实,此时地上部分基本停止生长。已有研究表明RopGAP对植物顶端生长具有调控作用[3-5],RhoGAP可通过调节F-肌动蛋白动力学来调控花粉管的生长[21]。因此,我们通过对OsRhoGAP1在水稻不同发育时期根、叶以及幼穗组织中的表达分析显示,在幼苗期,OsRhoGAP1主要在根和叶中表达;在幼穗期,OsRhoGAP1的表达明显集中于幼穗组织;在成熟期,OsRhoGAP1主要在根中表达。上述结果表明,水稻OsRhoGAP1基因的表达特性与水稻生长发育紧密联系,尤其在快速分裂生长的组织中保持高表达水平。这可能与OsRacD的表达有一定的协同关系,暗示二者可能共同参与了水稻组织生长分化相关的调控过程。
植物激素是调控植物生长发育的重要信号分子,参与调控植物特定的生理过程。对该基因启动子作用元件进行预测分析显示,该基因启动子区域有很多激素应答元件,故本实验检测了6种植物激素对OsRhoGAP1表达的影响。结果显示,ABA、SA、GA和MeJA能上调OsRhoGAP1的表达,尤其是GA上调幅度最为明显,达6.4倍。已有资料显示,GA参与种子发芽、茎的延长、花与果实的成熟过程[22, 23],ABA和SA有增强植物抗逆性的功能[24, 25],提示该基因的功能可能与激素信号介导的相关生理过程和抗逆性有关。本实验室前期研究显示,OsRacD的表达也受多种激素调控,ABA、GAs、6-BA能显著上调OsRacD地上部分的表达量,IAA和SA则下调该基因在地上部分的表达量[26]。根据现有结果,ABA、GA均使OsRhoGAP1与OsRacD的表达上调,IAA使OsRhoGAP1与OsRacD的表达下调,鉴于OsRhoGAP1与OsRacD的结合特性,我们推测在植物激素的应答反应中,OsRhoGAP1可能通过调控OsRacD的活性来实现生物学功能,其作用机制有待于进一步研究。
综上所述,OsRhoGAP1在水稻幼穗组织中高表达,提示OsRhoGAP1可能与水稻幼穗的发育分化过程相关。该基因的表达与ABA、SA、GA、MeJA、IAA和6-BA等植物激素密切相关,提示其功能可能涉及到植物生长发育中的多种过程。同时,在表达模式和激素响应方面,OsRhoGAP1与OsRacD的表达有一定的协同关系,OsRhoGAP1与OsRacD的相互作用机制,有待进一步的研究。目前,本实验室正在通过利用各种现代分子生物学技术对该基因进行功能鉴定,为系统解析其作用机制提供实验依据。
4 结论对OsRhoGAP1序列进行比对与拼接,预测OsRhoGAP1的cDNA编码区全长1 353 bp,编码450个氨基酸,具有植物RhoGAP蛋白典型的结构特征,即CRIB基序和RhoGAP结构域。水稻中OsRhoGAP1基因的表达具有时空特异性,主要在细胞快速分裂的幼根、幼叶、幼穗组织以及成熟根中高表达,尤其是幼穗组织。该基因的表达受ABA、SA、GA和MeJA的诱导,受IAA和6-BA的抑制。
| [1] | Nagawa S, Xu T, Yang ZB. RHO GTPase in plants: conservation and invention of regulators and effectors. Small GTPases, 2010, 1 : 78–88. DOI:10.4161/sgtp.1.2.14544 |
| [2] | Amin E, Jaiswal M, Derewenda U, et al. Deciphering the molecular and functional basis of RhoGAP family proteins: a systematic approach towards selective inactivation of Rho family proteins. The Journal of Biological Chemistry, 2016, 291 : 20353–20371. DOI:10.1074/jbc.M116.736967 |
| [3] | Hwang JU, Vernoud V, Szumlanski A, et al. A tip-localized RhoGAP controls cell polarity by globally inhibiting Rho GTPase at the cell apex. Current Biology Cb, 2008, 18 : 1907–1916. DOI:10.1016/j.cub.2008.11.057 |
| [4] | Klahre U, Kost B. Tobacco RhoGTPase activating protein1 spatially restricts signaling of RAC/Rop to the apex of pollen tubes. Plant Cell, 2006, 18 : 3033–3046. DOI:10.1105/tpc.106.045336 |
| [5] | Hoefle C, Huesmann R. A barley ROP GTPase activating protein associates with micro-tubules and regulates entry of the barley powdery mildew fungus into leaf epidermal cells. Plant Cell, 2011, 23 : 2422–2439. DOI:10.1105/tpc.110.082131 |
| [6] | Baxter-Burrell A, Yang Z, Springer PS, et al. RopGAP4-dependent Rop GTPase rheostat control of Arabidopsis oxygen deprivation tolerance. Science, 2002, 296 : 2026–2028. DOI:10.1126/science.1071505 |
| [7] | 米志勇, 王树生, 吴乃虎. 水稻低分子量G T P结合蛋白基因OsRacD的分离. 科学通报, 2000, 45(19): 2047–2055. DOI:10.3321/j.issn:0023-074X.2000.19.005 |
| [8] | Ye JR, Huang MJ, Wu NH. Fertility analysis of the Arabidopsis transformed with antisense rice osRACD gene. Progress in Natural Science, 2003, 13 : 424–428. |
| [9] | 叶建荣, 黄美娟, 赵淑慧, 等. osRACD基因表达与光敏核不育水稻光周期育性转换的相关性. 自然科学进展, 2004, 14(2): 166–172. |
| [10] | Liang WH, Tang CR, et al. Cloning and characterization of a new actin gene from Oryza sativa L. Progress In Natural Science, 2004, 14 : 867–874. DOI:10.1080/10020070412331344471 |
| [11] | 梁卫红, 唐朝荣, 吴乃虎. 两种水稻GDP解离抑制蛋白基因的分离及特征分析. 中国生物化学与分子生物学报, 2004, 20(6): 785–791. |
| [12] | Yoshida S, Forno DA, Cock JH. Laboratory manual for physiological studies of rice[M]. International Rice Research Institute, 1976. |
| [13] | Mcelroy D, Blowers AD, Jenes B, et al. Construction of expression vectors based on the rice Actin1(Act1)5' region for use in monocot transformation. Molecular and General Genetics, 1991, 231 : 150–160. DOI:10.1007/BF00293832 |
| [14] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta DeltaC (T)) method. Methods, 2001, 25 : 402–408. DOI:10.1006/meth.2001.1262 |
| [15] | Wu G, Li H, Yang Z. Arabidopsis RopGAPs are a novel family of rho GTPase-activating proteins that require the Cdc42/Rac-interadtive binding motif for rop-specific GTPase stimulation. Plant Physiology, 2000, 124 : 1625–1636. DOI:10.1104/pp.124.4.1625 |
| [16] | 张之为, 赵君, 樊明寿, 等. 植物小G蛋白的研究进展. 西北植物学报, 2009, 29(3): 622–628. |
| [17] | Nibau C, Wu HM, Cheung AY. RAC/ROP GTPases: 'hubs' for signal integration and diversification in plants. Trends Plant Sci, 2006, 11 : 309–315. DOI:10.1016/j.tplants.2006.04.003 |
| [18] | Yang Z, Fu Y. ROP/RAC GTPase signaling. Curr Opin Plant Biol, 2007, 10 : 490–494. DOI:10.1016/j.pbi.2007.07.005 |
| [19] | Schaefer A, Miertzschke M, Berken A, et al. Dimeric plant RhoGAPs are regulated by its CRIB effector motif to stimulate a sequential GTP hydrolysis. Journal of Molecular Biology, 2011, 411 : 808–822. DOI:10.1016/j.jmb.2011.06.033 |
| [20] | Hoefle C, Huesmann C, Schultheiss H, et al. A barley rOP GTPase activating protein associates with microtubules and regulates entry of the barley powdery mildew fungus into leaf epidermal cells. Plant Cell, 2011, 23 : 2422–2439. DOI:10.1105/tpc.110.082131 |
| [21] | Ying G, Yang Z. ROP GTPase regulation of pollen tube growth through the dynamics of tip-localized F-actin. Journal of Experimental Botany, 2003, 54 : 822–824. |
| [22] | Yamauchi Y, Ogawa M, Kuwahara A, et al. Activation of gibberellin biosynthesis and response pathways by low temperature during imbibition of Arabidopsis thaliana seeds. Plant Cell, 2004, 16 : 367–378. DOI:10.1105/tpc.018143 |
| [23] | Itoh H, Ueguchi-Tanka M, Sato Y, et al. The gibberellins signaling pathway is regulated by the appearance and disappearance of SLENDER RICE1 in nuclei. Plant Cell, 2002, 14 : 57–70. DOI:10.1105/tpc.010319 |
| [24] | Bright J, Desikan R, Hancock JT, et al. ABA-induced NO generation and stomatal closure in Arabidopsis are dependent on H2O2 synthesis. Plant Journal, 2006, 45 : 113–122. DOI:10.1111/j.1365-313X.2005.02615.x |
| [25] | Du H, Liu H, Xiong L. Endogenous auxin and jasmonic acid levels are differentially modulated by abiotic stresses in rice. Frontiers in Plant Science, 2013, 4 : 397–397. |
| [26] | 林群婷, 梁卫红, 李辉, 等. 水稻Rop基因OsRac5的表达特性. 植物生理学报, 2013, 49(12): 1400–1406. |





