SF2蛋白(Serine/Arginine Splicing Factor 1)是富含丝氨酸、精氨酸SR(Serine/Arginine)蛋白家族的成员之一,作为剪切因子能够促进剪接体的召集和前体mRNA的组成性剪接[1, 2]。在人类基因组中,约有94%的前体mRNA可发生组成性剪接,可使同一前体mRNA分子产生不同剪接体,从而增加了基因表达的复杂性和蛋白的多样性[3]。SF2蛋白作为多功能RNA结合蛋白广泛参与mRNA转录后的调控[4],包括mRNA初级体的剪接、出核、翻译和降解等生物合成过程[5-9]。因此深入研究SF2蛋白相关RNA,对进一步揭示其生物学功能具有重要的意义。
RNA免疫共沉淀技术被广泛应用于蛋白-RNA相互作用的研究[10, 11],将RNA免疫共沉淀技术与高通量测序技术相结合,能够获取与目的蛋白所结合RNA的信息[12]。然而,在已发表的279个CLIP数据集中,经过PCR重复之后有高达83.8%的CLIP-seq数据因捕获的RNA特异性较差而被舍弃[13]。因此为提高CLIP数据的有效性,获得低背景,高富集效率的RNA样本,建立高效灵敏的质检方法十分重要。捕获的RNA样本中,无法预测并筛选出稳定表达的基因作为内参基因,因此不能直接利用常规的qPCR进行目标RNA的定量检测。目前常采用PCR结合琼脂糖电泳技术的半定量方案检测RNA是否捕获[14],但无法定量检测RNA的富集效率以及非特异性RNA的富集情况。该质控无法精确预测文库构建的质量,增加了实验风险性。针对此问题我们提出可以在Srsf1抗体捕获的产物和IgG抗体的产物中等比例引入外源酿酒酵母RNA,并以酿酒酵母RNA的β-actin作为定量内参进行qPCR检测目标RNA的富集效率。针对引入外源RNA作为内参的方法,如Holstege等[15]采用生物素标记外源RNA的方法,检测全基因组的表达水平;Kanno等[16]利用外源mRNA进行绝对定量,但未见在RIP富集效率检测中利用酵母菌RNA作为内参的报道。
本研究针对RIP捕获SF2蛋白相互作用的RNA为样本,以酿酒酵母RNA的β-actin作为内参,采用qPCR技术精准定量检测目标RNA的富集效率。
1 材料与方法 1.1 材料 1.1.1 实验材料HeLa细胞;酿酒酵母(BY4741)。
1.1.2 试剂及仪器RNAiso Plus、Yeast RNAiso Kit试剂购自TaKaRa公司、逆转录试剂盒购自ThermoScientific公司、SuperReal PreMix SYBR Green购自TIANGEN公司、高糖DMEM培养基购自美国Hyclone公司、胎牛血清购自美国Gibco公司、SF2抗体、normal IgG抗体均购自Santa cruz公司、Protein A+G Agarose、RIPA lysis buffer等蛋白相关试剂购自碧云天公司、蛋白酶抑制剂、RNA抑制剂均购自Thermo公司、引物则都由英潍捷基(上海)贸易有限公司合成纯化;非接触式全自动破碎仪购自Diagenode公司、Nanodrop 2000c超微量分光光度计购自Thermo Fisher Scientific、实时荧光定量PCR仪购自Applied Biosystems公司;其余常用化学试剂均购自国药集团。
1.2 方法 1.2.1 酵母RNA的提取采用RNAiso Plus、Yeast RNAiso Kit进行RNA抽提。以Nanodrop 2000c超微量分光光度计和0.1%甲醛变性琼脂糖凝胶电泳鉴定RNA的质量和完整性,-80℃保存备用。
1.2.2 qPCR检测外源酿酒酵母RNA以Srsf1和PABP[17, 18]做阳性参照(参考文献已证明与SF2蛋白相互作用的RNA),PQLC1[17]作为阴性参照;qPCR采用SuperReal PreMix SYBR Green试剂盒,ABI 7500 real-time PCR system和7500 System Software-SDS 2.2(Applied Biosystems)分析RNA样本,反应体系和反应条件依据说明书进行。
1.2.3 RIP得到SF2蛋白及其靶RNA采用0.1%甲醛交联免疫共沉淀(RNA immunoprecipitation,RIP)技术,获取得到以下产物:(1)细胞裂解上清:总蛋白(Input-pr),总RNA(input-RNA);(2)RIP上清蛋白(Unbounding-pr);(3)RIP沉淀:SF2抗体特异性捕获的SF2蛋白(SF2-ip-pr),RNA(SF2-ip-RNA),IgG抗体吸附的非特异性RNA(IgG-ip-RNA)。以Nanodrop 2000c超微量分光光度计和1%甲醛变性琼脂糖凝胶电泳鉴定RNA的质量和完整性,-80℃保存备用。
1.2.4 SDS-PAGE、Western blot、考马斯亮蓝检测目标蛋白SF2Input-pr、Unbounding-pr、SF2-ip-pr蛋白样品在12%的SDS-PAGE上分离得到两个相同的SDS-PAGE胶,其一转移至PVDF膜,一抗(鼠源)按1: 1 000稀释比孵育过夜,用辣根过氧化物酶标记山羊抗小鼠二抗室温孵育1 h,利用ECL发光试剂盒显色;其二浸没在考马斯亮蓝试剂中染色1 h后用脱色液过夜脱色,均用Image Lab 4.1进行分析。
1.2.5 qPCR验证ip-RNA的富集SF2-ip-RNA、Ig-G-ip-RNA等比例加入外源酿酒酵母RNA。采用逆转录试剂盒逆转录cDNA,操作步骤依据说明书进行。以Srsf1和PABP做阳性参照,PQLC1作为阴性参照;qPCR采用SuperReal PreMix SYBR Green试剂盒,ABI 7900 real-time PCR system和7900 System Software-SDS 2.2(Applied Biosystems)分析SF2-ip-RNA和IgG-ip-RNA样本,反应体系和反应条件依据说明书进行。
2 结果 2.1 外源酿酒酵母RNA中目标RNA表达量的检测qPCR结果(图 1)显示,HeLa细胞中的目标RNA Srsf1、β-actin(Hela-actin)、PABP、PQLC1在HeLa细胞input样本中高表达,在酿酒酵母中表达量和阴性结果保持一致;而外源酵母RNA中的β-actin(Teast-actin)在酿酒酵母RNA中高表达,在HeLa细胞中不表达。上述结果表明外源酵母RNA不干扰目的RNA的检测。
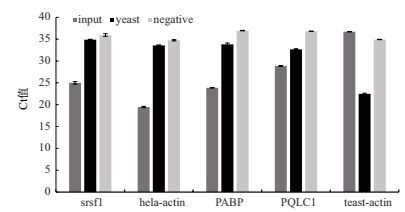
|
| 图 1 HeLa内源RNA表达水平 Y轴表示qPCR所检测的基因达到阈值时的循环数(Ct值);X轴表示检测的基因 |
Western blot检测结果(图 2-A)显示,ip-beads(ip蛋白和beads偶联复合物)最亮,unbounding-pr样品中没有检测到SF2蛋白;考马斯染色检测结果(图 2-B)显示,SF2-ip-pr只检测到一条蛋白,在input-pr总蛋白对应有相同条带,unbounding-pr未检测到该蛋白,成功捕获单一目的蛋白。
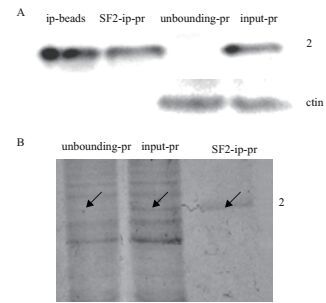
|
| 图 2 Western印记检测SF2蛋白(A)和考马斯检测总蛋白(B) A:ip-beads:RIP沉淀和beads的混合物;SF2-ip-pr:RIP沉淀;unboundingpr:RIP上清;input:总细胞裂解物。B:箭头所指表示SF2蛋白 |
qPCR的检测结果如表 1,图 3所示,检测得到阳性基因Srsf1和PABP在SF2阳性抗体和IgG阴性抗体产物中相差6个Ct值以上,浓度差异可达60倍以上;而阴性基因PQLC1只相差1.7个Ct值;且检测基因的溶解曲线如图 3-B所示,检测基因均为单一产物。
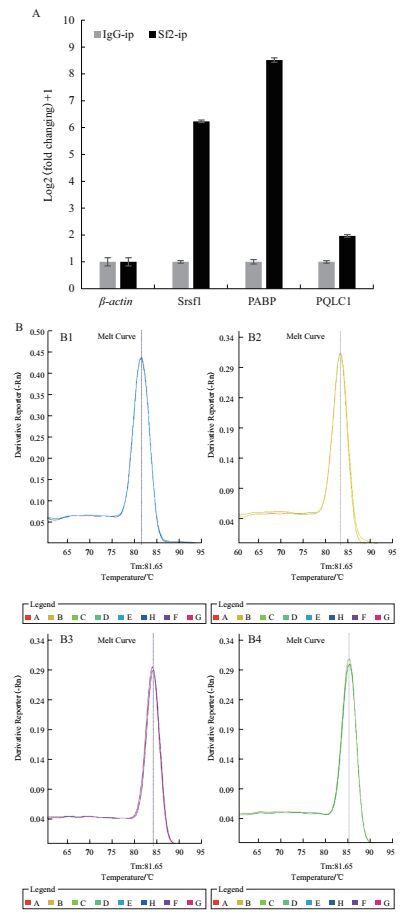
|
| 图 3 富集效率的检测 A:qRT-PCR检测RNA富集(Y轴表示Srsf1、PABP、PQLC1在SF2阳性抗体和IgG阴性抗体产物中的浓度差异[Log2(fold changing)+1];X轴表示检测的目的基因);B:目的基因熔解曲线(横坐标表示Tm值(DNA双链解链50%的温度),纵坐标表示相对荧光信号;B1-4分别表示β-actin、Srsf1、PABP、PQLC1的熔解曲线) |
目前研究蛋白-RNA相互作用的常用实验方法是蛋白交联免疫共沉淀结合高通量二代测序技术[19, 20]。获得低背景,高特异性,高富集效率的RNA样本,可提高文库构建的质量,降低实验的风险性,显著降低测序成本,极大地提高生物和技术的可重复性,并且可以使低丰度RNA结合蛋白(RNA-binding proteins,RBPs)和很少靶标RNA的RBPs的RBP位点更容易识别,精准的获得与目标蛋白相互作用的RNA信息[21, 22]。qPCR能够高效灵敏检测目的RNA的富集,而目的RNA是利用不同抗体(SF2特异性抗体和IgG阴性抗体)捕获而得,无法筛选出像GAPDH等能够稳定存在于捕获产物中的基因作为内参,则不能利用qPCR进行相对定量检测。因此引入外源RNA作为参考进行相对定量,可精准检测捕获的目的RNA的富集。
本研究结果显示,外源酿酒酵母RNA中未检测出目标RNA,说明引入种属差异大的外源RNA对于目的RNA的检测没有影响。同时对阳性基因富集效率的检测,得到阳性基因Srsf1、PABP[17, 18]在SF2阳性抗体和IgG阴性抗体产物中相对浓度差异可达60倍以上,而阴性基因PQLC1[17]只相差1.7个Ct值,相对浓度差异远远低于60倍,表明阳性参照Srsf1、PABP高效富集,说明基于0.1%甲醛交联免疫共沉淀成功捕获与SF2特异性相互作用的低背景RNA。因此,本研究方案引入外源RNA结合qPCR技术定量评估RIP富集RNA的质量,得到低背景,高特异性,高富集效率的RNA样本,克服了半定量PCR结合琼脂糖电泳技术无法精确定量检测RNA富集效率及非特异性RNA富集情况的问题。
4 结论本研究利用qPCR技术,以外源酿酒酵母(BY4741)RNA作为内参,能快速、准确定量检测RIP-RNA的富集效率。对于改善提升RIP技术的应用具有明显的价值。
| [1] | Das S, Krainer AR. Emerging functions of SRSF1, splicing factor and oncoprotein, in RNA metabolism and cancer. Molecular Cancer Research:MCR, 2014, 12 (9): 1195–1204. DOI:10.1158/1541-7786.MCR-14-0131 |
| [2] | Long JC, Caceres JF. The SR protein family of splicing factors:master regulators of gene expression. The Biochemical Journal, 2009, 417 (1): 15–27. DOI:10.1042/BJ20081501 |
| [3] | Oltean S, Gammons M, Hulse R, et al. SRPK1 inhibition in vivo:modulation of VEGF splicing and potential treatment for multiple diseases. Biochem Soc T, 2012, 40 : 831–835. DOI:10.1042/BST20120051 |
| [4] | Sonenberg N, Hinnebusch AG. New modes of translational control in development, behavior, and disease. Mol Cell, 2007, 28 (5): 721–729. DOI:10.1016/j.molcel.2007.11.018 |
| [5] | Hastings ML, Krainer AR. Pre-mRNA splicing in the new millennium. Curr Opin Cell Biol, 2001, 13 (3): 302–309. DOI:10.1016/S0955-0674(00)00212-X |
| [6] | Ram O, Ast G. SR proteins:a foot on the exon before the transition from intron to exon definition. Trends Genet, 2007, 23 (1): 5–7. DOI:10.1016/j.tig.2006.10.002 |
| [7] | Huang YQ, Steitz JA. SRprises along a messenger's journey. Mol Cell, 2005, 17 (5): 613–615. DOI:10.1016/j.molcel.2005.02.020 |
| [8] | Sanford JR, Ellis JD, Cazalla D, et al. Reversible phosphorylation differentially affects nuclear and cytoplasmic functions of splicing factor 2/alternative splicing factor. Proc Natl Acad Sci USA, 2005, 102 (42): 15042–15047. DOI:10.1073/pnas.0507827102 |
| [9] | Sanford JR, Gray NK, Beckmann K, et al. A novel role for shuttling SR proteins in mRNA translation. Gene Dev, 2004, 18 (7): 755–768. DOI:10.1101/gad.286404 |
| [10] | Tenenbaum SA, Carson CC, Lager PJ, et al. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. P Natl Acad Sci USA, 2000, 97 (26): 14085–1490. DOI:10.1073/pnas.97.26.14085 |
| [11] | Gerstberger S, Hafner M, Tuschl T. A census of human RNA-binding proteins. Nature Reviews Genetics, 2014, 15 (12): 829–845. DOI:10.1038/nrg3813 |
| [12] | Kishore S, Jaskiewicz L, Burger L, et al. A quantitative analysis of CLIP methods for identifying binding sites of RNA-binding proteins. Nat Methods, 2011, 8 (7): 559–564. DOI:10.1038/nmeth.1608 |
| [13] | Van Nostrand EL, Pratt GA, Shishkin AA, et al. Robust transcripto-me-wide discovery of RNA-binding protein binding sites with enha-nced CLIP (eCLIP). Nature Methods, 2016, 13 (6): 508–514. DOI:10.1038/nmeth.3810 |
| [14] | Niranjanakumari S, Lasda E, Brazas R, et al. Reversible cross-linking combined with immunoprecipitation to study RNA-protein interactions in vivo. Methods, 2002, 26 (2002): 182–190. |
| [15] | Holstege FCP, Jennings EG, Wyrick JJ, et al. Dissecting the regulatory circuitry of a eukaryotic genome. Cell, 1998, 95 (5): 717–728. DOI:10.1016/S0092-8674(00)81641-4 |
| [16] | Kanno J, Aisaki K, Igarashi K, et al. "Per cell"normalization method for mRNA measurement by quantitative PCR and microarrays. BMC Genomics, 2006, 7 : 64–77. DOI:10.1186/1471-2164-7-64 |
| [17] | Anczukow O, Akerman M, Clery A, et al. SRSF1-regulated alternative splicing in breast cancer. Mol Cell, 2015, 60 (1): 105–117. DOI:10.1016/j.molcel.2015.09.005 |
| [18] | Sanford JR, Coutinho P, Hackett JA, et al. Identification of nuclear and cytoplasmic mRNA targets for the shuttling protein SF2/ASF. PLoS One, 2008, 3 (10): e3369. DOI:10.1371/journal.pone.0003369 |
| [19] | Michalak P. RNA world-the dark matter of evolutionary genomics. J Evol Biol, 2006, 19 (6): 1768–1774. DOI:10.1111/jeb.2006.19.issue-6 |
| [20] | Ule J, Jensen KB, Ruggiu M, et al. CLIP identifies Nova-regulated RNA networks in the brain. Science, 2003, 302 (5648): 1212–1215. DOI:10.1126/science.1090095 |
| [21] | Lebedeva S, Jens M, Theil K, et al. Transcriptome-wide analysis of regulatory interactions of the RNA-binding protein HuR. Mol Cell, 2011, 43 (3): 340–352. DOI:10.1016/j.molcel.2011.06.008 |
| [22] | Wu X, Chesoni S, Rondeau G, et al. Combinatorial mRNA binding by AUF1 and Argonaute 2 controls decay of selected target mRNAs. Nucleic Acids Research, 2013, 41 (4): 2644–2658. DOI:10.1093/nar/gks1453 |





