2. 云南省林业科学院 云南省森林植物培育与开发利用重点实验室,昆明 650201
2. Yunnan Province Key Laboratory of Forest Plant Cultivation and Utilization, Yunnan Academy of Forestry, Kunming 650201
思茅松(Pinus kesiya var. langbianensis)是松科松属常绿乔木,为卡西亚松(P. kesiya)的地理变种,主要分布在云南中南部海拔1 800 m以下地区,具有生长快、材质好、纹理正直等特点,木材纤维长且强度高,是云南省重要的造林、采脂、纸浆材及用材针叶树种,在云南省林业生产和生态环境建设中起着极其重要的作用[1, 2]。思茅松的遗传育种研究一直受到重视,已从育种策略[3]、核型分析[4]和等位酶分析[5]等方面开展了研究。随着科技的进步,分子标记技术已在近年来开始用于思茅松遗传多样性研究。尽管建立了思茅松简单重复序列区间扩增(Inter-simple sequence repeat,ISSR)[6]和扩增片段长度多态性(Amplified fragment length polymorphism,AFLP)[7]的PCR扩增体系,但已有的思茅松遗传多样性研究仅限于随机扩增多态性(Randomly amplified polymorphic DNA,RAPD)一种方法[8],还没有用简单序列重复(Simple sequence repeat,SSR)标记分析思茅松遗传多样性的报道,因而缺乏思茅松SSR标记的特异性引物。
SSR标记又称微卫星标记,具有稳定性高、多态性丰富、位点特异性、共显性遗传和检测方便等特点,是广泛应用于动植物遗传多样性分析、遗传图谱构建、亲权鉴定、基因定位及克隆等的理想分子标记[9, 10]。引物开发是进行SSR分子标记研究的前提。早期开发SSR引物的方法主要有2种,一种是cDNA文库构建和克隆测序的方法,另一种是通过搜索公共数据库中的表达序列标签(EST)进行开发[11]。前者步骤复杂、工作量大、开发成本高,后者需要大量EST数据的支持。由于思茅松分子研究基础较为薄弱,目前尚无其SSR标记开发及应用的报道。鉴于当前高通量测序技术发展迅速,测序成本显著降低,利用高通量测序技术对植物进行转录组测序,其所产生的巨量数据中包含了大量的EST序列,基于此进行SSR标记开发无疑是便捷有效的方法[12, 13]。因此,对思茅松EST-SSR标记进行系统性识别并开发专属、稳定和有效的标记引物,对于推进思茅松遗传多样性及遗传结构探索、开展种质资源的研究以及选育优良新品种(系)等工作有重要的理论和现实意义。
本研究以思茅松转录组测序数据为基础,对59 636条Unigenes中的SSRs位点进行系统搜索和引物批量设计,对它们在转录组中的分布特征进行统计及分析,并且随机挑选224对引物进行PCR验证,旨在为思茅松的SSR分子标记开发和利用奠定基础。
1 材料与方法 1.1 材料转录组测序材料为云南省普洱市普文镇普文林场的高产脂单株“KHR01”,采集其树干韧皮部和形成层组织,液氮保存。2014年5月,经华大基因公司(北京)通过Illumina Hiseq2000进行转录组测序,共获得59 636条Unigene,总长42 526 184 nt,平均长度713 nt。
SSR扩增所用思茅松样本于2015年8月采自景谷林场和普文林场的2个人工林居群,每个居群各取21株单株样本。样品取健康的当年生针叶,硅胶干燥保存,用于DNA提取。
1.2 方法 1.2.1 DNA提取采用DNAsecure Plant Kit(北京天根)试剂盒,提取思茅松各单株的基因组DNA,经1%琼脂糖凝胶电泳检测合格后,稀释成20 ng/μL,置于-20℃保存备用。
1.2.2 SSR序列来源使用软件Micro Satellite(MISA)对59 636条Unigene进行SSR位点查找。查找标准:单、二、三、四、五、六核苷酸的最小重复次数分别为12、6、5、5、4、4次,并且SSR位点侧翼序列长度≥50 bp。
用Primer5和Oligo7软件进行引物设计和评价。设计引物时设置的主要参数为:GC含量40%-70%,退火温度58-60℃,引物长18-25 bp,预期扩增产物长度100-300 bp,且无二级结构和二聚体。引物命名为YAFP加序号,如YAFP-001。
1.2.3 SSR引物筛选随机选取224对SSR引物,由Invitrogen(上海)公司合成。PCR扩增所用的2×Taq PCR Master Mix购自北京康为世纪生物科技有限公司。PCR反应体系(20 μL)为:Master Mix10 μL,DNA模版1 μL,Primer F与Primer R各0.4 μL,ddH2O 8.2 μL。利用BioRad基因扩增仪(T100)进行扩增,PCR扩增程序为:94℃预变性5 min;94℃ 30 s,60℃ 30 s,72℃ 1 min,35个循环;72℃延伸5 min。
用6个思茅松单株DNA样品的混合模板对224对引物进行初筛,PCR产物用2%琼脂糖凝胶于5 V/cm下电泳60 min,电泳结束后EB染色,用GENE GENIUS凝胶成像系统进行观察拍照,检测有无明显目的条带。有目的条带的引物再用思茅松12个DNA模版进行SSR引物复筛,检测是否具有多样性。
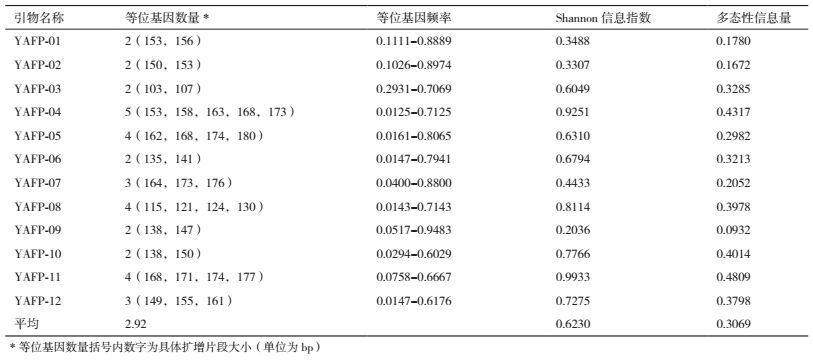
1.2.4 SSR遗传多样性分析选择12对条带清晰、扩增结果稳定且具有多态性位点的引物,合成荧光标记引物,以2个居群的42个思茅松单株DNA样品为模板,按照上述PCR反应体系及扩增程序进行SSR位点扩增。取1 μL PCR扩增产物,于ABI3730 DNA分析仪上进行毛细管电泳,并收集数据。用GeneMapper 4. 0软件对收集的原始数据进行分析,通过内标读取SSR标记片段的大小。按照大写英文字母顺序记录等位基因,即同一位点最大的等位基因记为A,其余等位基因则依次记为B,C,D等;只有1个峰值的按照纯合基因型处理。用POPGENE 1.31软件统计分析等位基因数(Na)、Shannon信息指数(I),利用PowerMarker 3.25软件计算多态性信息量(PIC)。
2 结果 2.1 思茅松转录组中SSR位点的分布特点经过MISA软件筛选,在思茅松转录组的59 636个Unigene序列中发现共3 745 SSR位点,SSR的分布频率(SSR的个数与总Unigene的数量比)为6.28%。思茅松转录组中SSR重复单元的分布特征如表 1所示。可以看出,思茅松转录组中SSR的主要重复类型是三核苷酸重复,占SSR位点总数的49%,其次为二核苷酸重复,占SSR总数的24%,其余核苷酸重复类型的数量较少,总计27%(表 1)。
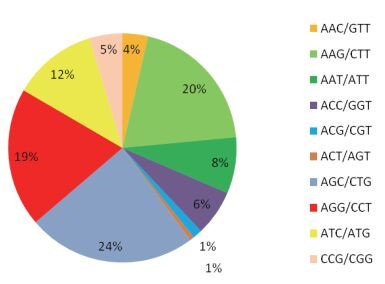
在出现频率最多的三核苷酸重复中,出现频率最高的核苷酸类型是AGC/CTG,占三核苷酸重复类型的24%;其次为AAG/CTT,占三核苷酸重复类型的20%;出现频率第三和第四的分别是AGG/CCT和ATC/ATG,分别占三核苷酸重复类型的19%和12%;其余核苷酸类型的数量较少,总计25%(图 1)。
|
| 图 1 三核苷酸重复中不同重复单元比例 |
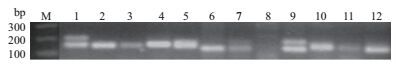
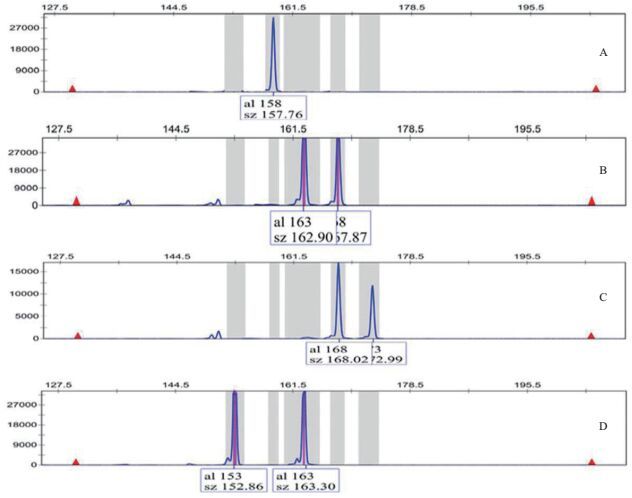
用6个思茅松单株样本的DNA混合模板对224对引物进行初筛,其中123对引物扩增效果明显,能够获得带型清晰、与预期大小相符的目的片段,扩增率为54.91%。之后选用思茅松12个单株样本的DNA模板对这123对引物进行复筛,得到带型清晰且扩增稳定的引物共29对,占所有设计引物的12.95%。图 2展示了引物YAFP-04扩增12个模板的琼脂糖凝胶电泳结果。图 3展示了引物YAFP-04分别扩增4个样品的毛细管电泳结果。这29对引物扩增出的目的片段长度基本在100到280 bp,但经ABI3730毛细管电泳分析后,其中17对引物的扩增片段大小不符合SSR重复类型的倍数大小或存在较多的无效扩增,仅12对引物(表 2)的扩增效率不低于85%,扩增结果的峰型清晰且具有多态性,用于后续的思茅松群体遗传多样性分析。
|
| 图 2 引物YAFP-04扩增12个样本的琼脂糖凝胶电泳结果 M:marker;1-6:景谷种群;7-12:普文种群 |
|
| 图 3 引物YAFP-04对4个样本的基因型分型结果 A:JG07,B:JG12、C:JG43,D:PW80 |
通过GeneMapper4.0软件进行数据读取,采用POPGENE 1.31和PowerMarker软件进行统计分析,42个样本在12个SSR位点检测到的遗传变异见表 3。12对引物共检测到35个等位基因,平均每对引物检测到2.92个等位基因,其中引物YAFP-04在42个样本中检测的等位基因数量最多,为5个(图 3)。12个位点的多态性信息量(PIC)为0.093 2-0.480 9,平均为0.306 9。根据Botstein等[14]的理论,有4个标记为低度多态位点(0 < PIC < 0.25),8个标记为中度多态位点(0.25 < PIC < 0.5),无高度多态位点(PIC > 0.5),YAFP-11位点的PIC最高(0.480 9)。
思茅松转录组中微卫星种类丰富且数量众多,从单核苷酸重复到6核苷酸重复都有出现,主要重复类型为二、三核苷酸重复类型,占到所有重复类型的73%,这与张振等[15]对红松(Pinus koraiensis)转录组SSR标记的分析结果一致。同为松属植物,红松EST中的SSR重复类型也是以二核苷酸与三核苷酸重复类型居多,且以三核苷酸重复类型为主。大别山五针松(Pinus dabeshanensis)[16]也呈现相同结果,甘蔗(Saccharum officinarum)[17]和普通小麦(Triticum aestivum)[18]等农作物同样是以三核苷酸重复类型为主,其三核苷酸重复类型分别为90%和67%。二核苷酸重复单元中AT/TA和AG/TC(占全部二核苷酸重复单元的88.28%)占主导地位,但GC/CG单元只出现一次,这一结果与黄海燕等人[19]在杜仲(Eucommia ulmoides)的转录组SSR中GC/CG单元只出现一次的结果一致,表现出明显的偏倚性,这一结果可能与思茅松本身基因组特性有关。
从转录组数据中搜索SSR位点,搜索结果与搜索标准有关[19]。本项研究中,从思茅松59 636个Unigene序列中发现3 745个SSR位点,分布频率为6.28%,略高于马尾松(Pinus massoniana)(3.62%)[20],日本落叶松(Larix kaempferi)(3.85%)[21]及火炬松(Pinus taeda)(4.32%)[22]。思茅松转录组数据的Unigene序列总长为42 526 184 bp,平均11.36 kb发现1个SSR位点,其SSR分布距离与杨树(Populus,14.0 kb)[23]、银杏(Ginkgo biloba,12.02 kb)[24]和胡椒(Piper nigrum,12.86 kb)[25]相近,低于橡胶(Hevea brasiliensis,3.93 kb)[26]、茶树(Camellia sinensis,3.68 kb)[27]和柑桔(Citrus,5.7 kb)[28],高于油菜(Brassica napus)[29]、杜仲(26.13 kb)[19]等。这一方面说明本项研究设定的SSR位点搜寻标准较为适宜,另一方面也提示,当搜寻结果获得的SSR位点数量较少而难以开发满意数量的SSR标记时,可适当调整搜索标准。
通过转录组测序数据开发SSR标记是一种较为快捷有效的方法[30-32]。但由于针叶树种的基因组序列(10 000-40 000 Mb)较为巨大,是拟南芥(Arabidopsis thaliana,114.5 Mb)的100倍以上,且重复DNA序列较多,导致针叶树种的SSR标记开发成功率较低[33, 34]。海岸松(Pinus pinaster)的成功率为7%[35],中山杉(Taxodium‘zhongshansa’)为16%[36],冷杉(Abies alba)为24%[37]。本研究也表明思茅松SSR标记开发成功率较低,仅为5.36%(12/224)。但所获得的12对引物经毛细管电泳检测具有多态性,且成功的在2个居群42个思茅松样本中进行了遗传多样性分析,后续可作为遗传图谱构建、基因定位及克隆等研究的理想分子标记。
4 结论通过对思茅松转录组测序分析数据的Unigenes序列进行SSR位点搜索,得到3 745个SSR位点,SSR重复类型以三核苷酸为主(49%);其次是二核苷酸(24%),主要重复单元分别为AGC/CTG和AT/TA。对其中的224个位点进行引物设计,最终筛选出12个扩增效率高、扩增条带清晰的多态性位点。这12个SSR标记成功地运用到思茅松2个居群的42个样本中,印证了利用转录组数据开发SSR标记的可行性,为思茅松种质资源的遗传多样性水平分析及变异水平的研究奠定了技术基础。
| [1] | 吴兆录. 思茅松研究现状的探讨. 林业科学, 1994, 30(2): 151–157. |
| [2] | 孟梦, 陈宏伟, 王达明, 等. 优良的纸浆材树种——思茅. 西南造纸, 2004, 33(3): 13–14. |
| [3] | 许玉兰, 段安安. 思茅松遗传改良研究现状及育种策略的探讨. 西南林学院学报, 2004, 24(4): 68–72. |
| [4] | 吴丽圆, 赵文书, 顾志建. 思茅松不同居群的染色体核型研究. 云南林业科技, 1999, 28(2): 32–35. |
| [5] | 陈少瑜, 赵文书, 张树红. 思茅松种子园遗传结构及遗传多样性. 云南大学学报:自然科学版, 2001, 23(6): 472–477. |
| [6] | 王曙光, 杨文武, 普晓兰. 思茅松大配子体DNA的提取及ISSR反应体系的建立. 四川林业科技, 2012, 33(2): 90–95. |
| [7] | 姚鹏强, 李培, 王曙光, 等. 思茅松AFLP分子标记体系的建立. 西南林业大学学报, 2012, 32(6): 30–33. |
| [8] | 朱云凤, 陈少瑜, 郝佳波, 等. 思茅松优良无性系的聚类分析及精英无性系特异标记. 西部林业科学, 2016, 45(4): 141–146. |
| [9] | Morgante M, Olivieri AM. PCR-amplified microsatellites as markers in plant genetics. The Plant Journal, 1993, 3 (1): 175–182. DOI:10.1046/j.1365-313X.1993.t01-9-00999.x |
| [10] | Powell W, Machray GC, Provan J, et al. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1996, 1 (7): 215–222. DOI:10.1016/S1360-1385(96)86898-0 |
| [11] | 陈怀琼, 隋春, 魏建和. 植物SSR引物开发策略简述. 分子植物育种, 2009, 7(4): 845–851. |
| [12] | 骆蒙, 贾继增. 植物基因组表达序列标签 (EST) 计划研究进展. 生物化学与生物物理进展, 2001, 28(4): 494–497. |
| [13] | 董斌, 李荣喜, 黄永芳, 等. 分子标记在油茶研究中的应用. 生物技术通报, 2015, 31(6): 74–80. |
| [14] | Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32 (3): 314–331. |
| [15] | 张振, 张含国, 莫迟红, 等. 红松转录组SSR分析及EST-SSR标记开发. 林业科学, 2015, 51(8): 114–120. |
| [16] | Xiang X, Zhang Z, Wang Z, et al. Transcriptome sequencing and development of EST-SSR markers in Pinus dabeshanensis, an endangered conifer endemic to China. Molecular Breeding, 2015, 35 (8): 1–10. |
| [17] | Cordeiro GM, Casu R, Mclntyre CL, et al. Microsatellite markers from sugarcane (Saccharum spp.) ESTs cross transferable to erianthus and sorghum. Plant Science, 2001, 160 (6): 1115–1123. DOI:10.1016/S0168-9452(01)00365-X |
| [18] | Gupta PK, Rustgi S, Sharma S, et al. Transferable EST-SSRs markers for the study of polymorphism and genetic diversity in bread wheat. Molecular Genetics and Genomics, 2003, 270 (4): 315–323. DOI:10.1007/s00438-003-0921-4 |
| [19] | 黄海燕, 杜红岩, 乌云塔娜, 等. 基于杜仲转录组序列的SSR分子标记的开发. 林业科学, 2013, 49(5): 176–181. DOI:10.11707/j.1001-7488.20130523 |
| [20] | 刘公秉, 季孔庶. 基于松树EST序列的马尾松SSR引物开发. 分子植物育种, 2009, 7(4): 833–838. |
| [21] | 杨秀艳, 孙晓梅, 张守攻, 等. 日本落叶松EST-SSR标记开发及二代优树遗传多样性分析. 林业科学, 2011, 47(11): 52–58. DOI:10.11707/j.1001-7488.20111109 |
| [22] | 林元震, 郭海, 刘纯鑫, 等. 火炬松热胁迫cDNA文库的EST-SSR预测. 华南农业大学学报, 2009, 30(3): 41–44. |
| [23] | Cardle L, Ramsay L, Milbourne D, et a1. Computational and experimental characterization of physically clustered simple sequence repeats in plants. Genetics, 2000, 156 (2): 847–854. |
| [24] | 樊洪泓, 李廷春, 李正鹏, 等. 银杏EST序列中微卫星的分布特征. 基因组学与应用生物学, 2009, 28(5): 869–873. |
| [25] | 伍宝朵, 郝朝运, 范睿, 等. 胡椒EST-SSR标记的开发和应用. 热带作物学报, 2015, 36(2): 237–243. |
| [26] | 安泽伟, 赵彦宏, 程汉, 等. 橡胶树EST-SSR标记的开发与应用. 遗传, 2009, 31(3): 311–319. |
| [27] | 杨华, 陈琪, 韦朝领, 等. 茶树转录组中SSR位点的信息分析. 安徽农业大学学报[J], 2011, 38(6): 882-886. |
| [28] | Chen CX, Zhou P, Cho YA, et a1. Mining and characterizing microsatellites from citrus ESTs. Theoretical and Applied Genetics, 2006, 112 (7): 1248–1257. DOI:10.1007/s00122-006-0226-1 |
| [29] | 李小白, 张明龙, 崔海瑞. 油菜EST资源的SSR信息分析. 中国油料作物学报, 2007, 29(1): 20–25. |
| [30] | Parchman TL, Geist KS, Grahnen JA, et al. Transcriptome sequencing in an ecologically important tree species:assembly, annotation, and marker discovery. BMC Genomics, 2010, 11 (3): 180. |
| [31] | Postolache D, Leonarduzzi C, Piotti A, et al. Transcriptome versus genomic microsatellite markers:highly informative multiplexes for genotyping Abies alba Mill. and congeneric species. Plant Molecular Biology Reporter, 2014, 32 (3): 750–760. DOI:10.1007/s11105-013-0688-7 |
| [32] | Pinosio S, González-Martínez SC, Bagnoli F, et al. First insights into the transcriptome and development of new genomic tools of a widespread circum-Mediterranean tree species, Pinus halepensis Mill. Molecular Ecology Resources, 2014, 14 (4): 846–856. DOI:10.1111/men.2014.14.issue-4 |
| [33] | Guevara MA, Chagné D, Almeida MH, et al. Isolation and characterization of nuclear microsatellite loci in Pinus pinaster Ait. Molecular Ecology Notes, 2005, 5 (1): 57–65. DOI:10.1111/men.2005.5.issue-1 |
| [34] | Nystedt B, Street NR, Wetterbom A, et al. The Norway spruce genome sequence and conifer genome evolution. Nature, 2013, 497 (7451): 579–584. DOI:10.1038/nature12211 |
| [35] | Mariette S, Chagné D, Decroocq S, et al. Microsatellite markers for Pinus pinaster Ait. Annals of Forest Science, 2001, 58 (2): 203–206. DOI:10.1051/forest:2001119 |
| [36] | Cheng Y, Yang Y, Wang Z, et al. Development and characterization of EST-SSR Markers in Taxodium "Bzhongshansa". Plant Molecular Biology Reporter, 2015, 33 (6): 1–11. |
| [37] | Postolache D, Leonarduzzi C, Piotti A, et al. Transcriptome versus genomic microsatellite markers:highly informative multiplexes for genotyping Abies alba Mill. and congeneric species. Plant Molecular Biology Reporter, 2014, 32 (3): 750–760. DOI:10.1007/s11105-013-0688-7 |