砷(As)广泛存在于岩石圈、水圈和生物圈。长期接触砷可以导致各种癌症的发生,如膀胱癌、肺癌、皮肤癌等。目前,世界上包括孟加拉、印度、阿根廷、美国和中国等许多国家面临着严重的砷污染威胁。有报道称中国土壤中砷浓度的平均值为11.2 mg/kg,约为世界平均值(6 mg/kg)的2倍[1]。因此砷污染的治理迫在眉睫。
自然环境中的砷主要以As5+和As3+两种无机价态形式存在,其中后者毒性大约为前者的100倍[2]。砷酸盐的毒性源于其作为磷酸盐的结构类似物,可以取代磷酸盐,抑制氧化磷酸化过程;而亚砷酸盐能够与半胱氨酸的巯基结合,破坏或干扰蛋白和酶的功能[3],从而发挥毒性作用。目前砷污染的主要治理原理是通过物理、化学、生物等技术降低砷的毒性或增加其稳定性。研究发现在砷污染较严重的地区,一些特殊的“耐砷微生物”[4]在砷化合态的相互转化过程中发挥着重要作用[5]。目前报道较多的耐砷微生物有芽孢杆菌属、假单胞菌属、红球菌属、苍白杆菌属、节细菌属、放线菌属、微杆菌属等[6-9]。微生物的耐砷特性与砷吸收、氧化还原、甲基化和外排等过程密切相关[10]。微生物体内主要通过ars操纵子介导砷代谢过程,其中砷酸盐还原酶(ArsC)[11]催化As5+还原成As3+,而As3+又被亚砷酸盐外排泵(ArsB、ACR3)[12]排出细胞外,还有其他的一些相关基因arsR和arsD(调节基因),arsA(ATP酶基因),arsH[13]、arsO等也参与了辅助代谢过程。另外一种机制是由亚砷酸盐氧化酶AioBA[11]催化介导的,它可以将高毒的As3+氧化成低毒、较稳定的As5+。而Qin等[14]的研究发现,在细菌Rhodopseudomonas palustris CGA009中As3+可以被转甲基化酶ArsM经过不同程度的甲基化,最终转变成气态的三甲基砷排出细胞,从而达到耐砷效果。近几年,由于微生物的耐砷机制而演化出利用微生物进行砷污染的修复技术也受到了越来越多的关注。除此之外,有些耐砷微生物还有植物促生作用,可以促进蜈蚣草[15]、粉叶蕨[16]、大叶井口边草[17]等一些砷富集植物对砷的吸收,从而达到微生物—植物协同作用修复砷污染的目的。
在土壤砷污染的来源中,矿业开采是主要原因之一。湖南的康家湾铅锌矿是中国的第四大矿区,至今已有100多年的开采历史,在开采过程中,周围环境被各种重金属污染,如铅、锌、砷等,对人们的生活构成很大的威胁。目前国内外关于砷污染的环境中高耐砷性菌株分离的研究较少。本研究以该矿区作为研究对象,目的是分离筛选耐高浓度砷的细菌,并对其进行鉴定,通过研究其对砷的氧化还原能力、砷耐性基因及其他的与砷耐性相关的特性,从而确定目标菌株,进行下一步砷代谢机制的研究,旨为土壤砷污染的生物修复奠定理论基础。
1 材料与方法 1.1 材料 1.1.1 供试土壤采自湖南康家湾水口山铅锌矿(26°33'57.9" N 112°36'23.7" E)周围矿渣区10-20 cm的表层土壤。采样点周围覆盖冶炼矿渣,重金属含量较高,表层植物较少。去除土壤表面矿渣,现场测定pH值,用无菌铲取约5 kg左右的新鲜表层土壤,置于无菌袋中,做好标记,将无菌袋放入冰盒中空运回实验室。取一部分新鲜样品,立刻进行细菌分离,剩余的样品存于4℃冰箱中,用于化学分析,土壤的理化性质见表 1。
LB液体培养基:酵母提取物5 g,氯化钠10 g,胰蛋白胨10 g,蒸馏水1 000 mL,pH 7.2。CDM培养基[18]:溶液A(0.081 2 mol/L七水硫酸镁,0.187 mol/L氯化铵,0.07 mol/L无水硫酸钠,0.574 mmol/L磷酸氢二钾,4.57 mmol/L二水合氯化钙和0.446 mol/L乳酸钠),121℃灭菌20 min;溶液B(4.8 mmol/L七水硫酸亚铁)和溶液C(0.95 mol/L碳酸氢钠),用0.45 μm过滤器过滤除菌。分别配制上述3种溶液,1 L CDM液体培养基中含有100 mL溶液A、2.5 mL溶液B、10 mL溶液C和887.5 mL蒸馏水,调pH至7.2。上述培养基若制备固体培养基,加入14 g/L的琼脂粉即可。
1.1.3 主要试剂和仪器细菌基因组DNA提取试剂盒和购自天根生化科技有限公司,PCR扩增和克隆相关试剂购自大连宝生物有限公司(TaKaRa),引物由上海英潍捷基公司合成,测序服务由生工生物工程(上海)股份有限公司提供。PCR仪和凝胶成像系统分别为Bio-Rad PCR仪和上海Tocan360凝胶成像系统。
1.2 方法 1.2.1 供试土壤理化性质的测定将新鲜土样进行烘干、过筛、消化(1:1的HNO3-H2SO4)处理,用火焰原子吸收分光光度法[19]测定供试土壤中的重金属含量。用总有机碳分析仪(TOC-VE,Shimadzu,日本)检测有机碳含量;而总氮和磷的含量用傅里叶变换近红外光谱仪(NIRLabN-200,BUCHI,瑞士)即可测得。
1.2.2 土壤耐砷菌株的分离和纯化取1 g新鲜土样溶于99 mL无菌水中,锥形瓶中加入10粒左右的玻璃珠,150 r/min,28℃振荡培养30 min。将土壤悬液连续稀释至浓度10-3,10-4和10-5,然后取0.1 mL稀释液涂布于含有40 mmol/L Na2HAsO4·7H2O的CDM平板上,28℃倒置培养3-7 d后划线分离。为了进一步筛选出对砷具有较高耐受性的菌株,分别用含不同梯度Na2HAsO4·7H2O(65、130和260 mmol/L)的CDM固体培养基对初筛分离的菌株进一步分离培养,纯化。所有实验重复3次。
1.2.3 对砷和其他重金属耐受性检测对筛选出来的耐砷能力较强的菌株进行高浓度砷和其他重金属的耐受性检测。将分离纯化的菌株接种于分别含有400、600、800 mmol/L Na2HAsO4·7H2O和5、10、20 mmol/L NaAsO2的CDM琼脂平板上,28℃培养3-7 d。对其他重金属(镉、汞、铜、镍和钡)的耐受性测定分别选用化合物Cd(NO3)2·4H2O、HgCl2、Cu(NO3)2·3H2O、NiCl2和BaCl2的浓度为1-20 mmol/L,共20个梯度,每两个梯度间隔1 mmol/L,用平板划线法将菌株接种于含有不同重金属浓度的CDM固体培养基上,重复3次,28℃培养3-7 d。
1.2.4 耐砷菌株的鉴定和耐砷基因的检测采用16S rRNA基因的PCR扩增和测序分析法对筛选出来的耐砷细菌进行菌种鉴定。利用细菌基因组DNA提取试剂盒提取耐砷菌株的基因组DNA,并以其作为模板,以27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1 492R(5'-CGGCTACCTTGTTACGACTT-3')[20]为引物进行50 μL体系的PCR扩增。扩增程序:94℃变性4 min;94℃ 1 min,55℃ 1 min,72℃ 1.5 min,循环30次;72℃ 10 min。为了获得几乎全长的16S rRNA基因,将PCR扩增产物用DNA纯化试剂盒(TaKaRa)进行纯化,然后用切胶回收试剂盒(TaKaRa)回收纯化DNA片段,并用pMD18-T克隆试剂盒(TaKaRa)将纯化片段链接到pMD18-T载体中,10 μL的连接体系包含1 μL T4 DNA Ligase Buffer(10×),7.5 μL DNA,0.5 μL pMD-18T,1 μL T4 DNA连接酶,16℃连接16 h,然后用氯化钙法转化到大肠杆菌感受态细胞(购自天根生化科技有限公司)中,挑取单克隆进行菌落PCR检测,筛选出阳性克隆,然后送到生工生物工程股份有限公司利用引物M13-47(5'-CGCCAGGGTTTTCCCAGTCACGAC-3')/RV-M(5'-AGCGGATAACAATTTCACACAGGA-3')进行测序。用DNAMAN软件对测序结果进行拼接,将得到的序列提交到http://www.eztaxon-e.ezbiocloud. net/[21]进行序列分析,然后用MEGA 5.0[22]软件中的邻接法(Neighbor-Joining)构建系统发育树[23]。
对于砷耐受性相关基因(As3+氧化酶基因aioA、As5+还原酶基因arsC、外排基因arsB/ACR3(1)/ACR3(2)、甲基化酶基因arsM)的检测,我们分别选取了9对引物(表 2)进行PCR扩增。25 μL PCR反应体系包含14.75 μL 2×Taq PCR Mix,上下游引物各0.2 mmol/L,25 ng DNA模板,克隆及测序方法同16S rRNA鉴定。
采用砷钼蓝法[7]进行测定。其原理为五价砷与钼酸铵作用生成砷钼酸络合物,然后被抗坏血酸还原成钼蓝,在沸水浴中显色并在865 nm下吸收峰值达到最大。挑取单菌落接种于LB液体培养基中,28℃,150 r/min培养24 h。将菌液4 750 × g离心10 min,用PIPES(20 mmol/L,pH 7.0)缓冲液洗涤菌体两次,悬浮于分别含有1 mmol/L NaHAsO2和1 mmol/L Na2HAsO4·7H2O的PIPES中,并调整菌液浓度至OD600 = 1.0,28℃,150 r/min培养。分别在培养24、48和72 h时取1 mL菌液于1.5 mL离心管中,4 750 ×g离心10 min去沉淀,将上清用PIPES缓冲液稀释10倍,用于测定As5+和As3+含量。具体测定方法:处理一:取300 μL稀释后的上清液加入100 μL KIO3溶液(5 mmol/L KIO3溶于50 mmol/L HCl),目的是将上清液中的As3+氧化成As5+;处理二:取300 μL稀释后的上清液加入100 μL 25 mmol/L HCl,对样品进行酸化。两份样品均25℃处理10 min,再分别加入600 μL钼蓝试剂(每升含有6 g钼酸铵、10.8 g抗坏血酸、0.136 g酒石酸锑钾、67.3 mL浓硫酸)立刻放入水浴锅中,78℃处理10 min,然后冰浴5 min,用分光光度计在865 nm下测定其吸光度值。所有处理均进行3次重复。以砷(Na2HAsO4·7H2O)浓度(0、20、40、60、80和100 mmol/L)为横坐标,吸光度OD865为纵坐标,绘制标准曲线。处理一和处理二分别测定的是样品中总As和As5+浓度,而As3+的浓度则根据两个处理的吸光度差值计算可得。
1.2.6 菌株产IAA能力测定根据康贻军等[29]的方法对砷氧还能力较强的菌株进行产吲哚乙酸(IAA)能力定量测定。耐砷菌株产IAA浓度测定:用固体LB培养基活化耐砷菌株,然后接种于含有0.5 g/L色氨酸的LB液体培养基中,150 r/min 28℃培养48 h。5 000×g离心15 min取2 mL上清液于10 mL离心管中,再加入等体积的Salkowshi比色液(每升10. 8 mol /L H2SO4含4.5 g FeCl3),室温避光放置30 min。根据混合液的颜色可初步判断产IAA量(若菌株产IAA,混合液则显示红色);然后用分光光度计测定OD530值,定量检测IAA。以空白培养基作对照,重复3次。以纯IAA浓度(0、20、40、60和80 μg/mL)为横坐标,OD530为纵坐标作标准曲线。根据IAA标准曲线的OD530值计算菌液中IAA浓度。
2 结果 2.1 抗砷菌株的分离和筛选利用含有40 mmol/L Na2HAsO4·7H2O的CDM平板,从采集的土壤样品中共初筛获得152株耐砷细菌。然后用更高浓度的As5+对152株菌进行了进一步的筛选,在65、130和260 mmol/L Na2HAsO4·7H2O的CDM琼脂培养基中分别筛选出了42、26和6株砷耐受性菌株(图 1)。
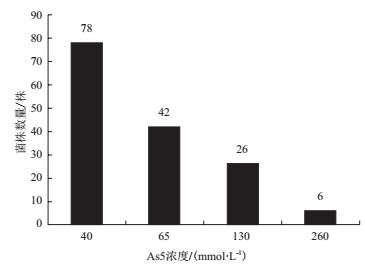
|
| 图 1 根据不同浓度的As5+筛选的菌株数量 |
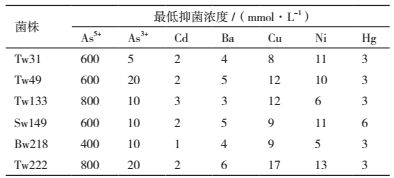
对筛选出的6株砷高耐受性菌株进行砷的最低抑菌浓度和耐其他重金属性能的测定。从表 3可以看出大多数菌株对As5+的耐受性在600 mmol/L以上,甚至高达800 mmol/L(Tw133和Tw222);对As3+的耐受性除了Tw31(5 mmol/L)以外,也均在10 mmol/L以上。这些耐砷菌株还对多种重金属具有耐受性,其中对镍和铜耐受性较高,对汞和钡产生中等程度的耐受性,而对低浓度的镉较为敏感。
根据16S rRNA基因序列构建的系统发育树结果表明这6株耐砷菌株分属于5个不同的属:假单胞菌属、苍白杆菌属、芽孢杆菌属、威廉氏菌属和节细菌属(图 2)。
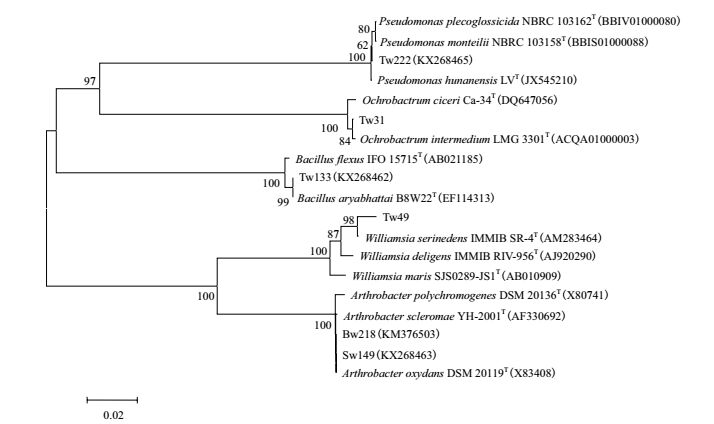
|
| 图 2 耐砷菌株与相关菌的系统发育树 |
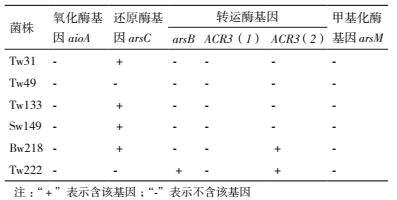
利用9对引物检测以上6株细菌的砷耐受性相关基因,结果(表 4)显示,4株耐砷菌株含有还原酶基因arsC,其中Tw133中arsC由引物对arsC4r/arsC4f扩增得到,而其他三株菌Tw31、Sw149和Bw218均用amlt42f/amlt376r引物得到。Tw222菌株同时含有两个外排基因arsB和ACR3(2),菌株Bw218仅扩增得到外排基因ACR3(2)。在这6株菌中均没有扩增得到氧化酶基因和甲基化酶基因,并且在菌株Tw49中没有检测到任何耐砷基因。
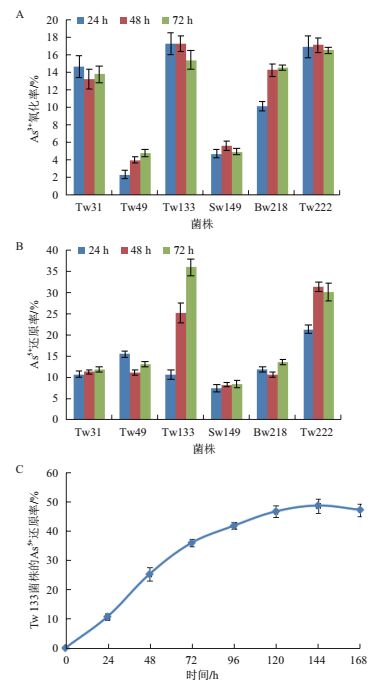
用砷钼蓝法对6株耐高浓度砷的细菌进行砷氧化还原能力测定。结果(图 3)显示,这6株菌均表现出了不同程度的氧化还原能力,其中菌株Bacillus sp. Tw133和Pseudomonas sp. Tw222的氧化还原率明显高于其他菌株。在72 h内,Bacillus sp. Tw133和Pseudomonas sp. Tw222均可氧化17 %左右的As3+(图 3-A),并可还原大约35 %的As5+(图 3-B)。由于Bacillus sp. Tw133菌株在72 h时的还原率表现出明显的增长趋势,因此又对其进行了更细致地测定,结果表明Bacillus sp. Tw133在144 h还原率达到最大值48.66 %(图 3-C)。由此可见,Bacillus sp. Tw133和Pseudomonas sp. Tw222菌株在砷氧化还原方面表现出了较明显的优势。
|
| 图 3 耐砷菌株分别对As的氧化还原能力 A:6株耐砷菌株分别在24、48、72 h对1 mmol/L As3+的氧化率;B:6株耐砷菌株分别在24、48、72 h对1 mmol/L As5+的还原率;C:Tw133菌株每隔24 h测得的对1 mmol/L As5+的还原能力 |
IAA是一种植物内源激素,与植物的生长发育相关,一定浓度的IAA可以促进植物的生长。通过对优势菌株Bacillus sp. Tw133和Pseudomonas sp. Tw222进行产IAA能力的测定。如图 4所示,B管的红色明显比C深,根据显色结果初步判断,Bacillus sp. Tw133的产IAA能力比Pseudomonas sp. Tw222强。根据IAA标准曲线计算可得,Bacillus sp. Tw133可以产生高达42.86 μg/mL的IAA,明显高于Pseudomonas sp. Tw222(24.36 μg/mL)。而由此可推断该两株菌对植物有一定的促生作用。

|
| 图 4 菌株产IAA能力显色结果 A:空白对照;B:Tw133菌株产IAA;C:Tw222菌株产IAA |
随着社会经济的发展,土壤砷污染问题越来越严重,其毒性对环境和人类健康产生极大的威胁。面对这种日益严峻的土壤砷污染问题,迫切需要一种高效、经济、环保的方法进行修复。近几年,越来越多的研究表明,自然界中的微生物在砷的迁移转化过程中发挥了重要作用[30],因此通过细菌的代谢活动减少或去除砷污染受到了越来越多的关注。多种耐砷菌株的分离和鉴定有利于深入研究其代谢机制,从而促进砷污染的生物修复。
本研究从湖南省康家湾铅锌矿土壤中共筛选出152株菌,其中耐高浓度砷的菌株细菌共6株。16S rRNA序列分析显示,这6株耐砷菌株属于5个不同的种属,分别为假单胞菌属、苍白杆菌属、芽孢杆菌属、威廉氏菌属和节细菌属。其中苍白杆菌属[31]、芽孢杆菌属[9]、节细菌属[6]和假单胞菌属[32]均有报道。目前关于威廉氏菌属的砷耐受性尚未有文献报道,因此该研究中获得的Williamsia sp. Tw49丰富了耐砷细菌的资源。
从重金属污染环境中分离得到的细菌普遍存在耐砷及其他重金属的特性。有报道称一些细菌对砷的耐受性分别可达10 mmol/L(As3+)和100 mmol/L(As5+)[33],甚至在Paul等[8]的研究中发现Acinetobacter,Microbacterium,Pseudomonas和Rhizobium对砷酸盐耐受性可高达600 mmol/L。而本研究筛选得到的6株菌均表现出了更大的砷耐受性,其中对As5+和As3+的最大耐受性分别为800 mmol/L(Bacillus sp. Tw133和Pseudomonas sp. Tw222)和20 mmol/L(Williamsia sp. Tw49和Pseudomonas sp. Tw222),如此高的砷耐受性特征为今后研究该菌的砷代谢机制提供了理论支持,也为发现细菌中新的砷代谢机理提供了可能性。
为了适应砷污染的环境,细菌形成了各种砷代谢机制,目前最常见的耐砷机理包括砷的氧化、还原、甲基化等[2]。通过耐砷基因检测,其中5株耐砷细菌中含有至少一个与砷耐受性相关的基因。6株耐砷菌株均表现出了不同程度的氧化还原能力,尤其是Bacillus sp. Tw133和Pseudomonas sp. Tw222的氧化率达17 %左右,但是未检测到砷氧化酶基因和甲基化酶基因,可能是在这些菌株中存在其他类型的砷氧化酶基因[34]和甲基化酶基因或者选用的引物不合适[35]。根据As5+还原能力检测结果可知,耐砷菌株Bacillus sp. Tw133的还原率在144 h时达到最大值48.66 %,明显高于耐砷菌株Enterobacter coacae sp. MC204(38.1 %)[7],而且这与该菌株中检测到砷还原酶基因(arsC)结果相符。尽管Williamsia sp. Tw49对砷表现出了较高的耐受性(As5+ = 600 mmol/L,As3+ = 10 mmol/L),但是用所选引物进行PCR扩增,没有检测到任何砷耐受性基因,推测该菌株可能存在未知的耐砷机制,有待于进一步深入研究。
植物修复技术是指利用植物清除土壤中的污染物质或使污染物质无毒化的技术[36],但是较高浓度的重金属限制了一些耐重金属植物的正常生长,而从污染地区分离到的一些细菌具有植物促生特性(PGP),如产IAA、ACC(1-氨基环丙烷羧酸)、铁载体等[37],可以促进植物对重金属的吸收,因此利用微生物—植物协同作用对重金属污染土壤进行修复也是一种较为理想的选择。文一等[38]的研究表明,链霉菌Streptomyces sp.能够促进砷超积累植物蜈蚣草对砷的进一步吸收。本研究中筛选出的耐砷菌株Bacillus sp. Tw133和Pseudomonas sp. Tw222产IAA的量分别可达42.86 μg/mL和24.36 μg/mL,均高于植物根际促生菌Pseudomonas sp. WP6[29]产IAA的量20.92 μg/mL。初步表明这两株优势菌株均具有明显的促生能力,为植物—微生物联合修复砷污染提供一定的理论依据。实验后期将通过测序确定这两株优势菌的耐砷基因簇,以便研究它们的耐砷机制,并用基因工程手段对基因簇进行改造,提高其代谢砷的能力,为砷污染的生物修复奠定基础。
4 结论本实验从湖南康家湾铅锌矿区总共筛选出152株细菌菌株,其中6株耐砷菌株对As5+和As3+的耐受性分别可高达800 mmol/L和20 mmol/L,并且对其他重金属也有不同程度的抗性。这6株砷高耐受性细菌属于5个不同的属,所含有的砷耐受性基因主要为还原酶基因(arsC)和外排基因(arsB、ACR3(2))。Bacillus sp. Tw133和Pseudomonas sp. Tw222对砷的氧化还原能力相对较高,尤其Bacillus sp. Tw133在144 h的As5+还原率最高可达48.66 %,并且两株菌产IAA的量分别为42.86 μg/mL和24.36 μg/mL,表现出了较明显的优势。
| [1] | 魏复盛, 陈静生, 吴燕玉, 等. 中国土壤环境背景值研究. 环境科学, 1991, 12(4): 12–19. |
| [2] | 蔡林, 王革娇. 抗砷性微生物及其抗砷分子机制研究进展. 微生物学通报, 2009, 36(8): 1253–1259. |
| [3] | Krumova K, Nikolovska M, Groudeva V. Isolation and identification of arsenic-transforming bacteria from arsenic-contaminated sites in Bulgaria. Biotechnol Biotechnol Equip, 2008, 22 (2): 721–728. DOI:10.1080/13102818.2008.10817541 |
| [4] | Zargar K, Hoeft S, Oremland R, et al. Identification of a novel arsenite oxidase gene, arxA, in the Haloalkaliphilic, arsenite-oxidizing bacterium Alkalilimnicola ehrlichii strain MLHE-1. Journal of Bacteriology, 2010, 192 (14): 3755–3762. DOI:10.1128/JB.00244-10 |
| [5] | 张潆月, 班允赫, 史荣久, 等. 具有砷(Ⅴ)还原能力的硫酸盐还原菌筛选及生长特性研究. 河南科学, 2015, 33(4): 553–558. |
| [6] | Cai L, Liu GH, Rensing C, et al. Genes involved in arsenic transformation and resistance associated with different levels of arsenic-contaminated soils. BMC Microbiol, 2009, 9 (4): 1–11. |
| [7] | Jareonmit P, Mehta M, Sadowsky MJ, et al. Phylogenetic and phenotypic analyses of arsenic-reducing bacteria isolated from an old tin-mine area in Thailand. World J Microbiol Biotechnol, 2012, 28 (5): 2287–2292. DOI:10.1007/s11274-012-1034-1 |
| [8] | Paul D, Poddar S, Sar P. Characterization of arsenite-oxidizing bacteria isolated from arsenic-contaminated groundwater of West Bengal. J Environ Sci Health Part A-Toxic/Hazardous Substances Environ Eng, 2014, 49 (13): 1481–1492. DOI:10.1080/10934529.2014.937162 |
| [9] | Sanyal SK, Mou TJ, Chakrabarty RP, et al. Diversity of arsenite oxidase gene and arsenotrophic bacteria in arsenic-affected Bangladesh soils. AMB Express, 2016, 6 (1): 1–11. DOI:10.1186/s13568-015-0169-5 |
| [10] | 韩永和, 王珊珊. 微生物耐砷机理及其在砷地球化学循环中的作用. 微生物学报, 2016, 56(6): 901–910. |
| [11] | Cavalca L, Corsini A, Zaccheo P, et al. Microbial transformations of arsenic:perspectives for biological removal of arsenic from water. Future Microbiol, 2013, 8 (6): 753–768. DOI:10.2217/fmb.13.38 |
| [12] | Desoeuvre A, Casiot C, Héry M. Diversity and distribution of arsenic-related genes along a pollution gradient in a river affected by acid mine drainage. Microb Ecol, 2016, 71 : 672–685. DOI:10.1007/s00248-015-0710-8 |
| [13] | Yang HC, Rosen BP. New mechanisms of bacterial arsenic resistance. Biomed J, 2016, 39 (1): 5–13. DOI:10.1016/j.bj.2015.08.003 |
| [14] | Qin J, Rosen BP, Zhang Y, et al. Arsenic detoxification and evolution of trimethylarsine gas by a microbial arsenite S-adenosylmethionine methyltransferase. Proc Natl Acad Sci USA, 2006, 103 (7): 2075–2080. DOI:10.1073/pnas.0506836103 |
| [15] | 廖晓勇, 陈同斌, 谢华, 等. 磷肥对砷污染土壤的植物修复效率的影响:田间实例研究. 环境科学学报, 2004, 24(3): 455–462. |
| [16] | 宋书巧, 周永章, 周兴, 等. 土壤砷污染特点与植物修复探讨. 热带地理, 2004, 24(1): 6–9. |
| [17] | 纪冬丽, 孟凡生, 薛浩, 等. 国内外土壤砷污染及其修复技术现状与展望. 环境工程技术学报, 2016, 6(1): 90–99. |
| [18] | Weeger W, Lievremont D, Perret M, et al. Oxidation of arsenite to arsenate by a bacterium isolated from an aquatic environment. Biometals, 1999, 12 (2): 141–149. DOI:10.1023/A:1009255012328 |
| [19] | 景丽杰, 马甲. 火焰原子吸收分光光度法测定污染土壤中5种重金属. 中国土壤与肥料, 2009, 1: 74–77. DOI:10.3969/j.issn.1673-6257.2009.01.019 |
| [20] | Yanagi M, Yamasato K. Phylogenetic analysis of the family Rhizobiaceae and related bacteria by sequencing of 16S rRNA gene using PCR and DNA sequencer. FEMS Microbiol Lett, 1993, 107 (1): 115–20. DOI:10.1111/fml.1993.107.issue-1 |
| [21] | Kim OS, Cho YJ, Lee K, et al. Introducing EzTaxon-e:a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species. Int J Sys Evol Microbiol, 2012, 62 (3): 716–721. |
| [22] | Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 2011, 28 (10): 2731–2739. DOI:10.1093/molbev/msr121 |
| [23] | Saitou N, Nei M. The neighbor-joining method:A new method for reconstructing phylogenetic trees. Mol Biol Evol, 1987, 4 (4): 406–425. |
| [24] | Inskeep WP, Macur RE, Hamamura N, et al. Detection, diversity and expression of aerobic bacterial arsenite oxidase genes. Environ Microbiol, 2007, 9 (4): 934–943. DOI:10.1111/emi.2007.9.issue-4 |
| [25] | Sun YM, Polishchuk EA, Radoja U, et al. Identification and quantification of arsC genes in environmental samples by using real-time PCR. J Microbial Methods, 2004, 58 (3): 335–349. DOI:10.1016/j.mimet.2004.04.015 |
| [26] | Escudero LV, Casamayor EO, Chong G, et al. Distribution of microbial arsenic reduction, oxidation and extrusion genes along a wide range of environmental arsenic concentrations. PLoS ONE, 2013, 8 (10): e78890. DOI:10.1371/journal.pone.0078890 |
| [27] | Achour AR, Bauda P, Billard P. Diversity of arsenite transporter genes from arsenic-resistant soil bacteria. Res Microbiol, 2007, 158 (2): 128–137. DOI:10.1016/j.resmic.2006.11.006 |
| [28] | Jia Y, Huang H, Zhong M, et al. Microbial arsenic methylation in soil and rice rhizosphere. Environ Sci Technol, 2013, 47 (7): 3141–3148. |
| [29] | 康贻军, 程杰, 梅丽娟, 等. 植物根际促生菌的筛选及鉴定. 微生物学报, 2010, 50(7): 853–861. |
| [30] | 杨婧, 朱永官. 微生物砷代谢机制的研究进展. 生态毒理学报, 2009, 4(6): 761–769. |
| [31] | Branco R, Chung AP, Morais PV. Sequencing and expression of two arsenic resistance operons with different functions in the highly arsenic-resistant strain Ochrobactrum tritici SCII24T. BMC Microbiol, 2008, 8 (1): 1–12. DOI:10.1186/1471-2180-8-1 |
| [32] | 管思琪, 罗蕾, 邢辉, 等. 湖南石门磺厂矿区尾矿库抗砷菌株的分离、鉴定及性质研究. 江苏农业科学, 2014, 42(4): 300–303. |
| [33] | Jackson CR, Dugas SL. Phylogenetic analysis of bacterial and archaeal arsC gene sequences suggests an ancient, common origin for arsenate reductase. BMC Evol Biol, 2003, 3 (1): 1–10. DOI:10.1186/1471-2148-3-1 |
| [34] | Sultana M, Vogler S, Zargar K, et al. New clusters of arsenite oxidase and unusual bacterial groups in enrichments from arsenic-contaminated soil. Arch Microbiol, 2012, 194 (7): 623–635. DOI:10.1007/s00203-011-0777-7 |
| [35] | Cordi A, Pagnout C, Devin S, et al. Determination of physiological, taxonomic, and molecular characteristics of a cultivable arsenic-resistant bacterial community. Environ Sci Poll Res, 2015, 22 (18): 13753–13763. DOI:10.1007/s11356-014-3840-5 |
| [36] | 洪雪花, 张婉静, 张爱, 等. 环境砷的存在状态、生物转化和修复研究进展. 云南农业大学学报, 2011, 26(4): 567–571. |
| [37] | Shagol CC, Krishnamoorthy R, Kim K, et al. Arsenic-tolerant plant-growth-promoting bacteria isolated from arsenic-polluted soils in South Korea. Environ Sci Pollut Res, 2014, 21 (15): 9356–9365. DOI:10.1007/s11356-014-2852-5 |
| [38] | 文一, 廖晓勇, 阎秀兰. 链霉菌的抗砷特性及其对蜈蚣草富集砷的作用. 生态毒理学报, 2013, 8(2): 186–193. DOI:10.7524/AJE.1673-5897.20130322002 |