增强子在真核生物的基因调控中,是一个重要的顺式元件,起到调节相关基因表达的开关作用[1],能对靶向基因进行时空表达的调控[2]。增强子的活性则受到多种因素的影响,只有在特定条件下才能增加靶向基因的表达,因此增强子的活性是处在不断变化之中[2]。增强子的获得对研究基因表达调控特性具有重要意义,但用传统的方法去获得和注解增强子是相当困难的。增强子捕获(Enhancer trapping,ET)是用来确定一段DNA序列中是否包含增强子功能的一项技术,是一种研究增强子控制细胞中基因时空表达模式特征的有效方法[3]。增强子捕获载体通常由迷你启动子驱动报告基因随机整合到基因组中用以提供一种直接简单的手段来测定控制基因表达的调节模块[4]。最早在1992年,Korn等[5]已尝试在小鼠上建立增强子检测方法,随后Grabher等[6]在青鳉(Medaka)中成功应用了以转座子为介导的增强子捕获技术,在斑马鱼上也有SB转座子[7],TOL2转座子[8]介导的增强子捕获的报道。
斑马鱼属脊椎动物亚门,辐鳍鱼纲(Actinopt-erygii),鲤科,短担尼鱼属(Danio)的一种硬骨鱼,因其全身分布有深蓝色与银白色相间的纵纹,酷似非洲草原斑马而得名斑马鱼。至20世纪80年代斑马鱼被引入实验室,现如今已成为研究脊椎动物发育学和胚胎学的模式生物[9, 10]。斑马鱼基因序列与人类基因的高度同源性,达到了87%的相似性,近年来被用于作为研究人类各种疾病的模型,广泛的应用于生命科学领域[11]。斑马鱼的受精卵容易大量获得、发育快、胚胎透明,在受精24 h后已经有器官的发生,发育至5 d时主要的器官已经形成并发挥相应的作用,这些特点便于对斑马鱼进行遗传学操作及实时监测发育过程。本实验以斑马鱼为模式生物,初步探讨SB转座子介导的增强子捕获技术的有效性,从而为高通量捕获增强子和基因功能研究奠定基础。
1 材料与方法 1.1 材料Tuebingen斑马鱼(Danio rerio)购自国家斑马鱼资源中心;SB转座子介导的增强子捕获转基因斑马鱼F1代SK-3系为本实验室制备。
基因组提取试剂盒购自宝生物工程有限公司,限制性内切酶Sau3A1,T4 DNA Ligase,Annealing Buffer均购自NEB有限公司,无RNA酶DNAse1,RNAsein,NaOH,链酶蛋白酶,多聚甲醛,PTU,HEPES,Tween20,tRNA,肝素钠,苯酚,氯仿,HCl,BCIP,NBT等购自Sigma公司;地高辛标记RNA混合液,蛋白酶K,羊抗地高辛标记物抗体等购自Roche公司;转录缓冲液,T7 RNA聚合酶等购自Promega公司;去离子甲酰胺购自CarloErba;羊血清,无水甲醇,NaCl,KCl,MgCl2,无水乙醇等购自国药。
1.2 方法 1.2.1 F2代鱼胚收集和荧光检测将SK-3系与TU系野生型斑马鱼进行交配,获得F2代胚胎,培养于E3溶液中至特定的发育时期以备荧光检测。在体式荧光显微镜(M165FC,Lecia,德国)下观察不同时期的胚胎GFP的表达并做好记录。本实验中观察的胚胎GFP的表达时期分别是24 hpf、2 d、3 d、4 d、5 d、7 d。在检测完荧光之后,收集5-10尾F2代斑马鱼继续饲养至20 d后提取基因组备用。
1.2.2 Splinkerette PCR 1.2.2.1 PCR引物序列根据转座元件序列设计两轮PCR引物,接头序列参考文献[12],引物序列见表 1。
本实验采用TaKaRa的基因组提取试剂盒提取饲养20 d的F2代斑马鱼的基因组,将获得的DNA经Nanodrop核酸测定仪(Nanodrop1000,美国)测得浓度为380 ng/µL,OD260/OD280=1.85,-20℃保存备用。
1.2.2.3 基因组打断50 µL反应体系为: Genomic DNA 5 µL,10×NEB BUFFER 5 µL,Sau3A1 3 µL,ddH2O补齐至50 µL。将反应体系置于37℃过夜处理,之后用TaKaRa的DNA片段纯化试剂盒进行纯化并用45 µL ddH2O洗脱。
1.2.2.4 接头合成合成体系为50 µL: SPLNK-BOT 2 µL,SPLNKGATC-TOP 2 µL,Annealing Buffer 46 µL。反应程序: 95℃,3 mins;自然降至室温,约30 mins,-20℃保存。
1.2.2.5 接头连接(Ligated genomic DNA)50 µL体系包括: Digested genomic DNA 37 µL,10X T4Ligase Buffer 5 µL,annealed splinkerette oligonucleotide 6 µL,T4 DNA Ligase(400 U/µl)1.5 µL,连接反应条件: 16℃,16 h。
1.2.2.6 PCR扩增第一轮PCR(Round 1 PCR)体系50 µL为Ligated genomic DNA 10 µL,ddH2O 11 µL,2× Taqmix 25 µL,F12 2 µL,SB1N 2 µL,PCR扩增程序: 95℃预变性4 min;95℃ 30 s,58℃ 30 s,72℃ 2 min,35个循环;最后72℃延伸10 min。第2轮PCR(Round 2 PCR)体系50 µL为Round 1 PCR product 1 µL,ddH2O 20 µL,2x Taqmix 25 µL,F14 2 µL,SB2N 2 µL,PCR扩增程序: 95℃预变性4 min;95℃ 30 s,58℃ 30 s,72℃ 90 s,35个循环;最后72℃延伸10 min。反应完成后,将PCR产物在1.5%琼脂糖凝胶电泳中进行电泳并切胶回收,测序。
1.2.3 增强子注解将测序获得的染色体侧翼DNA序列通过BLASTN与ENSEMBL的斑马鱼基因组数据库(GRCz10) 进行比对。从Ensembel浏览器上下载斑马鱼(Zebrafish)、青鳉(Medaka)、东方红鳍鲀(Fugu)的侧翼序列上下游各50KB区域的基因组序列(www.ensembl.org),并将所得到的序列进行比对分析(http://genome.lbl.gov/vista/mvista/submit.shtml)。
1.2.4 原位杂交根据插入位点处的内源基因Ednraa编码序列设计RNA原位杂交探针。采用地高辛和T7RNA聚合酶标记合成反义RNA探针。参考文献[13]进行斑马鱼胚胎原位杂交,通过荧光体式显微镜(M165FC,Lecia,德国)采集图片。
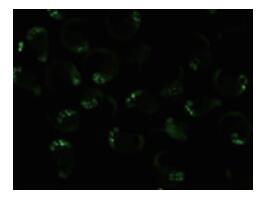
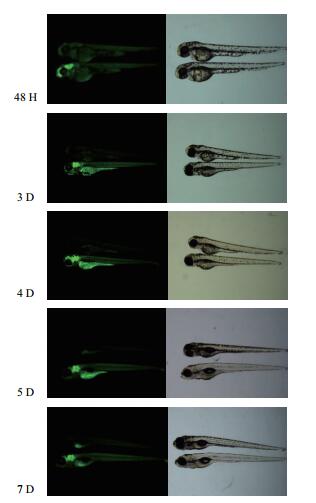
2 结果 2.1 F2代胚胎不同发育时期GFP表达将SK-3系F1与TU系斑马鱼进行杂交,收集胚胎于E3培养液中培养至特定的发育时期进行观察,结果(图 1)表明从24 hpf开始,在荧光显微镜下可观察到斑马鱼胚胎脑部有明显的荧光表达,荧光位置分别是前脑、中脑和后脑。在2 dpf、3 dpf、4 dpf、5 dpf、7 dpf等发育时期荧光仍集中于脑部,表达强度基本无变化,而表达部位有所改变(图 2),体现出基因表达调控的时空特性。
|
| 图 1 SK-3斑马鱼胚胎24 hpf的脑部荧光表达情况 |
|
| 图 2 SK-3增强子捕获转基因斑马鱼不同发育时期的荧光表达情况 |
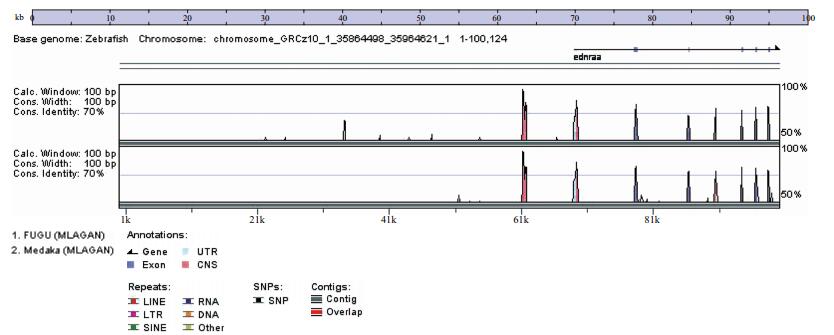
将测序结果中得到的染色体侧翼DNA序列在ENSEMBL的斑马鱼基因组数据库(GRCz10) 中进行比对,表明捕获载体插入1号染色体,捕获的增强子位于内源基因ednraa上游20 kb处。再从Ensembel浏览器上下载注解所需的序列文件,经分析获得增强子注解图(图 3)。
|
| 图 3 SK-3增强子捕获转基因斑马鱼的增强子注解 |
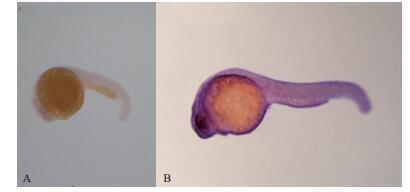
针对注解得到的结果,根据Ednraa基因编码序列设计其反义RNA探针,进行原位杂交验证,结果见图 4。阴性对照组胚胎的荧光集中于头部,而Ednraa反义RNA探针的原位杂交结果显示胚胎整体明显着色,且头部着色较深。
|
| 图 4 sk-3系F2代胚胎24 hpf原位杂交图 A:阴性对照;B:探针杂交 |
近年来,利用转座子介导法进行斑马鱼基因捕获、增强子捕获,获得突变体,进而研究基因功能及表达调控已获得较大进展。研究表明来自Tc1/Mariner超家族的SB转座子[14],可在不同物种中具有转座活性,已广泛应用于鱼类、两栖类、哺乳动物等的转基因研究[15-19]。由于转座子在宿主基因组的高效插入,在斑马鱼上利用转座子介导增强子捕获技术鉴定了大量增强子[20]。Parinov等[8]利用TOL2转座子介导斑马鱼增强子捕获,建立突变品系,研究斑马鱼发育的相关功能基因。Balciunas等[7]利用SB转座子,在斑马鱼中进行增强子捕获研究,建立了9个突变品系。本课题组采用SB介导的增强子捕获技术获得了大量斑马鱼插入突变体,通过报告基因GFP可确定捕获的增强子和内源基因时空表达特性,大大提高了筛选效率。我们已通过表型筛选建立了若干个突变品系,本研究所用品系表型为头部区域发绿色荧光,对该品系进行注解,获得了脑部发育调控增强子,并在其下游定位了一个内源基因。利用转座子介导在斑马鱼上进行增强子捕获研究是目前最有效的增强子捕获技术,筛选突变表型斑马鱼系,为研究相关基因的调控模式提供了很好的工具。Naouel等[21]通过增强子捕获技术捕获转基因斑马鱼品系(ub49),发现载体插入位点附近内源性基因kctd15a与转基因斑马鱼荧光蛋白表达模式密切相近,在随后的发育阶段在神经组织中也有相似的时空表达特征。Asakawa等[22]和Takeuchi等[23]先后分别利用Tol2转座子介导Gal4-UAS系统在斑马鱼上进行脑部神经组织电路分析和增强子捕获研究。本研究建立的斑马鱼品系SK-3,其插入位点附件存在内源基因Ednraa,对该基因进行RNA原位杂交,结果表明该基因具有明显的转录活性,且在头部表达水平较高,提示该基因在脑部的表达可能被位于其上游的增强子调控,而该增强子具有脑部表达特异性。因此,本研究捕获得增强子可能与脑部早期发育有关,该增强子的表达特性和内源基因的确切功能尚需进一步验证。
相比用F1代转基因斑马鱼进行注解,本实验直接利用增强子捕获转基因斑马鱼F2代对其进行注解,F2代的GFP表达的表型更稳定,在采集荧光图片以及后续的基因组注解上可以耗费更少的时间和精力,大大提高了研究效率。本研究获得了脑部特异表达GFP的增强子捕获系,通过其不同发育阶段的表达模式,可以揭示增强子的调控模式及对早期胚胎组织器官发育相关的功能基因。本研究与报道的增强子捕获系不同[7],从而丰富了增强子研究素材。本研究通过生物信息学方法对SK-3系转座子插入位点进行了较为详细的注解,获得了斑马鱼早期胚胎脑部发育相关的增强子可能的位置及其调控的内源性基因Ednraa,该基因与人类基因组中EDNRA蛋白有高达73%的同源性,而Gordon等[24]研究发现EDNRA的突变会导致伴有秃头症的颌面脂肪症,而本研究注释得到的斑马鱼的内源基因Ednraa,因其高度的同源性使得斑马鱼更好地作为研究人类疾病的模式动物。由于GFP表达模式可直接表明该基因时空表达特性,RNA原位杂交初步结果也说明GFP与Ednraa的表达模式基本一致,进一步说明该基因可能受到所捕获的增强子元件的调控,提示基因捕获技术对研究基因功能高度有效。
4 结论本研究通过SB介导的增强子捕获技术获得了脑部稳定表达GFP的斑马鱼增强子捕获品系SK-3;通过生物信息学注解获得了所捕获的增强子的染色体位置及其下游调控的内源基因Ednraa;GFP与Ednraa的表达模式具有一致性。SK-3系可为斑马鱼脑部神经发育情况的研究提供良好的动物模型。
| [1] | 贾春平, 曾溢滔. 增强子作用机制的研究进展. 生命科学, 2002, 14(2): 73–76. |
| [2] | 孙长斌, 张曦. 超级增强子研究进展. 遗传, 2016, 38(12): 1056–1068. |
| [3] | CY Liu, Song GL, Mao L, et al. Generation of an enhancer-trapping vector for insertional mutagenesis in zebrafish. PLoS One, 2015, 10 (10): 1–21. |
| [4] | Bellen HJ. Ten years of enhancer detection: lessons from the fly. Plant Cell, 1999, 11 (12): 2271–2281. DOI:10.1105/tpc.11.12.2271 |
| [5] | Korn R, Schoor M, Neuhaus H, et al. Enhancer trap integrations in mouse embryonic stem cells give rise to staining patterns in chimaeric embryos with a high frequency and detect endogenous genes. Mechanisms of Development, 1992, 39 (1-2): 95–109. DOI:10.1016/0925-4773(92)90029-J |
| [6] | Grabher C, Henrich T, Sasado T, et al. Transposon-mediated enhancer trapping in medaka. Gene, 2003, 322 : 57–66. DOI:10.1016/j.gene.2003.09.009 |
| [7] | Balciunas D, Davidson AE, Sivasubbu S, et al. Enhancer trapping in zebrafish using the Sleeping Beauty transposon. BMC Genomics, 2004, 5 (1): 62. DOI:10.1186/1471-2164-5-62 |
| [8] | Parinov S, Kondrichin I, Korzh V., et al. Tol2 transposon-mediated enhancer trap to identify developmentally regulated zebrafish genes in vivo. Developmental Dynamics, 2004, 231 (2): 449–59. DOI:10.1002/dvdy.v231:2 |
| [9] | Kimmel CB, Ballard WW, Kimmel SR, et al. Stages of embryonic development of the zebrafish. Dev Dyn, 1995, 203 (3): 253–310. DOI:10.1002/aja.1002030302 |
| [10] | Grunwald DJ, Eisen JS. Headwaters of the zebrafish -emergence of a new model vertebrate. Nat Rev Genet, 2002, 3 (9): 717–724. |
| [11] | 李礼, 罗凌飞. 以斑马鱼为模式动物研究器官的发育与再生. 遗传, 2013, 35(4): 421–432. |
| [12] | Potter CJ, Luo L. Splinkerette PCR for mapping transposable elements in Drosophila. PLoS One, 2010, 5 (4): e10168. DOI:10.1371/journal.pone.0010168 |
| [13] | Thisse C, Thisse B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nat Protoc, 2008, 3 (1): 59–69. DOI:10.1038/nprot.2007.514 |
| [14] | Ivics Z, Hackett PB, Plasterk RH, et al. Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells. Cell, 1997, 91 (4): 501–510. DOI:10.1016/S0092-8674(00)80436-5 |
| [15] | Ivics Z, Garrels W, Mátés L, et al. Germline transgenesis in pigs by cytoplasmic microinjection of Sleeping Beauty transposons. Nat Protoc, 2014, 9 (4): 810–827. DOI:10.1038/nprot.2014.010 |
| [16] | Tschida BR, Largaespada DA, Keng VW. Mouse models of cancer: Sleeping Beauty transposons for insertional mutagenesis screens and reverse genetic studies. Semin Cell Dev Biol, 2014, 27 : 86–95. DOI:10.1016/j.semcdb.2014.01.006 |
| [17] | Mátés L. Rodent transgenesis mediated by a novel hyperactive Sleeping Beauty transposon system. Methods Mol Biol, 2011, 738 (2): 87–99. |
| [18] | Carlson DF, Garbe JR, Tan WF, et al. Strategies for selection marker-free swine transgenesis using the Sleeping Beauty transposon system. Transgenic Res, 2011, 20 (5): 1125–1137. DOI:10.1007/s11248-010-9481-7 |
| [19] | Izsvák Z, Chuah MK, VandenDriessche T, et al. Efficient stable gene transfer into human cells by the Sleeping Beauty transposon vectors. Methods, 2009, 49 (3): 287–297. DOI:10.1016/j.ymeth.2009.07.001 |
| [20] | 沈丹, 陈才, 王赛赛, 等. Tc1/Mariner转座子超家族的研究进展. 遗传, 2017, 39(1): 1–13. |
| [21] | Naouel G, Zhao XF, Staale E, et al. Zebrafish enhancer trap line showing maternal and neural expression of kctd15a. Dev Growth Differ, 2012, 54 (2): 241–252. DOI:10.1111/dgd.2012.54.issue-2 |
| [22] | Asakawa K, Suster ML, Mizusawa K, et al. Genetic dissection of neural circuits by Tol2 transposon-mediated Gal4 gene and enhancer trapping in zebrafish. Proc Natl Acad Sci USA, 2008, 105 (4): 1255–1260. DOI:10.1073/pnas.0704963105 |
| [23] | Takeuchi M, Matsuda K, Yamaguchi S, et al. Establishment of Gal4 transgenic zebrafish lines for analysis of development of cerebellar neural circuitry. Dev Biol, 2015, 397 (1): 1–17. DOI:10.1016/j.ydbio.2014.09.030 |
| [24] | Gordon CT, Weaver KN, Zechi-Ceide RM, et al. Mutations in the endothelin receptor type A cause mandibulofacial dysostosis with alopecia. The American Journal of Human Genetics, 2015, 96 (4): 519–531. DOI:10.1016/j.ajhg.2015.01.015 |





