免疫球蛋白基因超家族(IgSF)是一类分子结构中含有类似免疫球蛋白结构域(Ig结构域)、不依赖Ca2+的细胞黏附分子。IgSF的所有成员都有3个共同保守区域:免疫球蛋白样结构域(Ig)、跨膜结构域(Transmembrane)和胞质尾区(Cytoplasmicregion)。IgSF的蛋白功能主要体现在免疫系统中,负责参与相关免疫应答及免疫调节反应。在先天性免疫和获得性免疫受体中的IgSF有很多类型,如免疫球蛋白(Ig)和T细胞抗原受体(TCR)、自然杀伤细胞受体和细胞黏附分子等[1-3]。NITRs作为IgSF家族中的一员,在相关结构上高度保守。主要包括1-2个胞外免疫球蛋白结构域(1个IgV和1个Ig(V / C2) 域)、1个跨膜区和1个含有1-2个免疫受体酪氨酸抑基序(ITIMs)的胞质尾区。在功能研究上,斑马鱼ITIM在哺乳动物细胞系中的表达验证了ITIM在体外具有调节免疫抑制信号功能[4, 5];斑点叉尾鮰NITR[6]的发现确定了它的免疫生物学功能,并有11种不同类型的NITR基因在斑点叉尾鮰上被鉴定出来,这些不同类型的NITR基因在胞外Ig结构域、连接区(Joining regions)、跨膜结构域和ITIM的数量上展现出多样性的特征。目前NITR已在7种硬骨鱼类中被鉴定发现:河豚(Spheroides nephelus)[7, 8],斑马鱼(Danio rerio)[9],斑点叉尾鮰(Ictalurus punctatus)[6],虹鳟(Oncorhynchus mykiss)[10]、鲤(Cyprinus carpio)[11]、日本比目鱼(Paralichthys olivac-eus)[12]和青鳉(Oryzias latipes)[13]。但是迄今为止NITR及其同系物并没有在哺乳动物中被鉴定发现[14]。
近年来,针对NITRs的功能及其在进化过程中作用的研究正成为热点。Shen等[15]在鲶鱼NK-like细胞中检测到NITR,但是NITRs的特定细胞谱系并不确定。另外,NITR的特异性结合配体现在也不清楚,但是可以确定它们能与某些异体细胞表面上的靶位结合[16]。Rojo等[17]发现斑马鱼对鳗利斯顿氏菌发生免疫应答后的NITR在转录水平上发生上调,并推测NITR在免疫方面有着重要作用。Litman等[14]发现NITRs的整体结构和胞质信号与哺乳动物的自然杀伤性细胞免疫球蛋白样受体(KIRs)类似,推测NITR可能是先天性免疫和适应性免疫系统进化过程中重要的组成阶段。
罗非鱼作为世界上广泛养殖的经济鱼类,同时也是我国南方重要的养殖鱼类之一,具有很高的经济价值[20]。鉴于NITR在硬骨鱼类中的免疫功能中的特殊地位,本研究对罗非鱼NITR分子进行了克隆鉴定,并对该基因及其氨基酸序列特征进行了生物信息学分析;同时研究了该基因在罗非鱼健康组织中的表达情况,旨在为进一步研究罗非鱼NITR分子生物学功能奠定基础。
1 材料与方法 1.1 材料本实验所用健康尼罗罗非鱼购自湛江市东风市场,体质量规格约100±10 g/尾;克隆载体pMD18-T Vector购自TaKaRa公司(大连);Easy Taq酶、Ex Taq酶来自北京全式金公司;PCR仪购自Bio-Rad公司;DNA胶回收试剂盒购自Thermo公司。
1.2 方法 1.2.1 罗非鱼Total RNA的提取和cDNA的合成实验用罗非鱼在实验室条件下(28±2)℃暂养4周后,取健康尼罗罗非鱼脾脏组织10-20 mg,再按照北京全式金TransZol Up Plus RNA Kit试剂盒说明书提取罗非鱼脾脏的Total RNA。将提取的罗非鱼脾脏Total RNA按照北京全式金EasyScript First-Strand cDNA Synthesis SuperMix试剂盒说明方法反转成cDNA第一链。
1.2.2 罗非鱼NITR基因全长的克隆依据本实验室罗非鱼转录组数据预测的NITR分子,利用Primer Premier 5.0设计罗非鱼NITR基因克隆引物On-NITR-1F、On-NITR-1026R(表 1),然后进行PCR扩增,PCR反应条件参照北京全式金2×EasyTaq PCR SuperMix说明书进行,之后电泳检测PCR产物,将目的条带切胶回收后与pMD18-T载体连接,连接产物转化到DH5α中,挑取单克隆进行菌落PCR鉴定后,将阳性克隆送广州生工测序。
阳性克隆的测序结果用DNAMAN Version 6软件进行拼接,从而获得On-NITR cDNA全长序列。通过NCBI(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行On-NITR序列比对分析;通过ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)进行开放阅读框确定;通过ExPASy ProtParam tool(http://web.expasy.org/protparam/)进行On-NITR氨基酸序列推导和相关理化性质分析;通过Softberry(http://linux1.softberry.com/berry.phtml?topic=psite & group=programs & subgroup=proloc)进行氨基酸基本功能位点分布预测;通过SignalP 4.0 Server(http://www.cbs.Dtu.dk/services/signalp)进行信号肽序列预测;通过TMHMM Server 2.0(http://www.cbs.dtu.dk/services/TMHMM)进行跨膜结构域预测;通过Inter ProScan Sequence search(http://www.ebi.ac.uk/Tools)和SMART(http://smart.embl-heidelberg.de/)进行蛋白功能结构域分析;通过SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)、SWISSM-MODEL(http://swissmodel.expasy.org/Interactive)进行二级结构、三维结构预测分析;通过Clutalx和Genedoc软件进行氨基酸同源序列比对分析。
1.2.4 罗非鱼NITR的组织表达分析重新利用Primer 5.0软件设计一对特异性引物On-RT-NITR-F、On-RT-NITR-R。以β-actin为内参基因,通过ABI 7500 Real-Time定量PCR仪分析进行qRT-PCR。分析On-NITR在试验中的各个组织中的表达分布。每个样本设置3个复孔作为平行。RT-PCR条件参照TransScript Green Two-Step qRT-PCR SuperMix试剂盒进行。具体的反应条件为:预变性94℃ 30 s;94℃ 5 s,60℃ 34 s,40个循环;95℃ 15 s(Dissociation Stage)。通过2-△△Ct方法计算罗非鱼NITR基因在不同组织中的相对表达量。
2 结果 2.1 On-NITR基因的克隆用On-NITR-1F、On-NITR-1026R作为引物,罗非鱼脾脏组织cDNA作为模板进行PCR扩增,获得的产物测序后经NCBI Blast分析判定该扩增产物为罗非鱼NITR基因。用本试验中罗非鱼NITR基因序列命名为On-NITR(GenBank登录号: KX989509),该基因全长1 119 bp,5' 非编码区为39 bp,3' 非编码区为55 bp,ORF为1 026 bp,可编码341个氨基酸(图 1)。

|
| 图 1 罗非鱼NITR及其推导的氨基酸序列 注:阴影部分为信号肽 |
ProtParam在线分析结果显示,罗非鱼NITR蛋白的理论分子量为37.38 kD,理论等电点(PI)为8.28。推测的氨基酸序列中丝氨酸(Ser)的含量最高,比例为10%;亮氨酸(Leu)含量次之,为9.4%;色氨酸(Trp)含量最低,为0.6%。其中带正电荷的氨基酸残基(Arg+Lys)有34个,带负电荷的氨基酸残基(Asp+Glu)有30个。不稳定指数为36.85,归类为1个稳定的蛋白。该蛋白脂溶指数(Aliphatic index)为96.04,总的平均亲水值为0.098,很有可能是疏水性蛋白且疏水性不强。ProtScale分析表明罗非鱼NITR蛋白疏水性氨基酸残基数目在整体上稍微略高于亲水性氨基酸残基数目,疏水性不是很明显(图 2),为疏水性蛋白,与理化分析的结果一致,因此推测罗非鱼NITR基因编码的蛋白质为疏水性蛋白质。

|
| 图 2 罗非鱼NITR蛋白亲疏水性预测 |
使用Signal P 4.1 Server预测该序列的信号肽(Signal peptide),发现前19个氨基酸为信号肽序列,为分泌型蛋白;利用TMHMM Server 2.0预测跨膜结构域,发现在第251-273位氨基酸之间存在跨膜结构域(图 3)。SoftBerry-Psite预测该序列功能位点,发现该序列酪蛋白激酶Ⅱ磷酸化位点6个,N-糖基化位点和蛋白激酶C磷酸化位点各有5个,N-豆蔻酰化位点4个,cAMP和cGMP相关磷酸化位点、酪氨酸激酶磷酸化位点以及原生质膜脂筏附着位点各有1个,CAAX box 6个,C末端定位信号序列微体6个。蛋白质亚细胞定位预测On-NITR蛋白在细胞中各位置的分布概率:细胞膜(64%),高尔基体(46%)、内质网(膜)(68.5%),内质网(腔)(10%),由Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对On-NITR进行亚细胞定位预测的结果为细胞膜,这与该蛋白作为分泌型免疫受体的预测结果相互佐证。
|
| 图 3 罗非鱼NITR蛋白扩膜结构域预测 |
通过SOPMA分析罗非鱼NITR蛋白的二级结构,发现该蛋白二级结构中由109个氨基酸组成延伸链(Extended strand),占31.96%;由97个氨基酸组成α螺旋(Alpha helix),占28.45%;由95个氨基酸组成无规则卷曲结构(Random coil),占27.86%;由40个氨基酸组成β折叠结构((Beta turn)占11.73%。利用SWISS-MODLE在线预测蛋白质三级结构工具发现,On-NITR蛋白属于免疫折叠蛋白,且与斑马鱼NITR蛋白具有一定的相似性,分析发现On-NITR蛋白结构模型与预测的蛋白质二级结构结果一致(图 4)。

|
| 图 4 罗非鱼NITR蛋白(A)与斑马鱼NTIR蛋白(B)三维结构 |
将本研究On-NITR基因编码的氨基酸序列与NCBI数据库中已经公布的其他物种的NITR氨基酸序列进行BLAST比对发现它们的相似度为27%-46%(表 2),这与目前报道的其他物种间NITR的比对结果一致。
运用ClustalW软件将罗非鱼NITR的氨基酸序列与虹鳟(Oncorhynchus mykiss)、斑马鱼(Danio rerio)、牙鲆(Paralichthys olivaceus)及桔点圆鲀(Spherodoies nephelus)等物种的NITR氨基酸序列进行多重序列比对(图 5)。综合NCBI对各物种的BLAST和InterPro在线软件对各物种氨基酸的功能分析发现该序列含有1个信号肽区域和2个Ig-domain区为IgV(25-139)、Ig(147-247),且分别包含1个甘氨酸连接桥区(GXG和FGXG),1个跨膜结构区(251-273),以及1个胞质尾区(274-341),该胞质尾区含有NITR典型的ITIM基序(305-312)(免疫受体酪氨酸抑制基序,Immunoreceptor tyrosine-based inhibitor motifs)和1个ITIM类似基序itim(321-326),且具有较高的保守性(图 5)。

|
| 图 5 罗非鱼NITR与其他物种的同源性比较 |
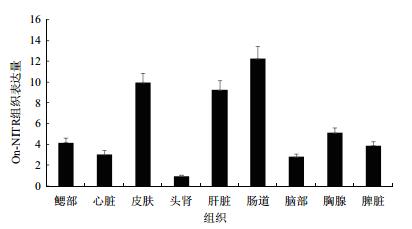
通过ABI 7500Real-Time定量PCR仪进行qRT-PCR分析,检测健康罗非鱼各组织NITR基因的表达量,结果(图 6)显示,On-NITR基因在所取的9个组织中均能表达,在肠道中表达量最高,其次是皮肤、肝脏,在胸腺、鳃、脾脏、心脏、脑中的表达量较少,在头肾组织中的表达量最少。
|
| 图 6 罗非鱼NITR在不同组织中的表达情况 |
本研究对依托实验室转录组数据预测的罗非鱼NITR分子进行了克隆鉴定,并推导出编码的NITR氨基酸序列。BLAST分析显示On-NITR序列与其他已报道的硬骨鱼类之间的同源性为27%-46%。
尽管On-NITR与硬骨鱼类的比对呈现较低的同源性,但是结构上都具有胞外免疫球蛋白结构域(Ig domain)—IgV和Ig(V/C2),并包含相应的甘氨酸连接桥基序(The glycine bulges of the joining mitif)[21],1个跨膜结构域以及1个含有免疫抑制酪氨酸基序(ITIM)的胞质尾区,并且高度保守。On-NITR的两个胞外Ig结构域内各含有一个甘氨酸连接桥区域GXG(GQG128)和FGXG(FGNG241),并含有8个保守的半胱氨酸残基,这与斑马鱼、虹鳟等[6, 8]其他硬骨鱼类相一致。所有的半胱氨酸残基都位于胞外区域,表明On-NITR蛋白可能与其他物种的Ig区半胱氨酸具有相同的功能—形成二硫键结合区;此外,On-NITR(N-X-S/T-X)的5个N-糖基化位点也全部都位于胞外Ig区,其他已报道的硬骨鱼类含有2-7个,这些Ig区域的糖基化位点与该受体的黏附作用和识别其他抗原的功能息息相关[22],这意味着On-NITR的Ig区可能在细胞间的黏附作用中起着关键作用。
在On-NITR的胞质尾区,含有1个免疫抑制酪氨酸抑制基序(ITIM,LHYAAL 312)和1个类似ITIM基序—itim(CVYSRV 326)。On-NITR的ITIM区结构上与Ravetch等[23]提出的ITMI基序结构(I/V/L/S)XYXX(V/L)相一致,具有较高保守性,而itim基序不尽相同。Blery等[24]发现(I/V/S)TYSX(L/V)型基序只是属于7种自然杀伤性细胞受体基序的一种,itim基序的出现表明ITIM基序在硬骨鱼类中也存在一定的多样性[25]。已有报道将含有正常的ITIM基序受体和ITIM基序中酪氨酸残基发生突变的受体进行对比研究发现,ITIM基序在调节自然杀伤细胞受体抑制信号和结合其他免疫球蛋白受体方面具有重要作用[26, 27],On-NITR的ITIM基序与上游的跨膜结构域相隔33个氨基酸,itim和ITIM中所含有的酪氨酸之间相距23个氨基酸,这与Bléry等[24]报道的哺乳动物自然杀伤细胞受体上的距离长度非常类似,通过蛋白空间构象与功能的密切联系,可以推测On-NITR上的ITIM基序在功能上可能也相对保守。
有意思的是,On-NITR蛋白的结构特征与先天性免疫中的自然杀伤细胞免疫抑制受体(KIRs)类似[28],与白细胞受体集群所编码的免疫抑制受体所具有的调控位点和其他结构特征一样,但是KIRs不含有IgV结构域和甘氨酸连接桥基序(J motif)。此外,On-NITR蛋白的结构特征与适应性免疫中的T细胞受体(TCRs)和免疫球蛋白(Igs)也非常相近[9],都含有IgV结构域和甘氨酸连接桥基序。说明NITR蛋白很可能兼有先天性免疫和获得性免疫蛋白结构特征。
组织分布结果显示,On-NITR在罗非鱼的肠道、皮肤、肝脏中表达量较高;其次是胸腺、鳃、脾脏、心脏及脑,在头肾中的表达量最低。目前已报道的不同鱼类的NITR基因在各组织中的表达情况差异较大,日本比目鱼[12]和鲤[11]NITR在头肾、脾脏和肠道中发生高水平表达,而斑点叉尾鮰[6]NITR1在头肾、脾脏中表达量较高,但是NITR 2-4在肠道和头肾中表达量很低,虹鳟[10]脾脏和头肾中NITR1、NITR4都很高,但是NITR2、NITR3表达量很低。推测On-NITR的表达情况可能与NITR的类型有关。此外,NITR主要在硬骨鱼类的巨噬细胞、B细胞、T细胞及NK-like细胞中被检测发现,并且在各种不同细胞中的表达情况也具有一定差异[6],暗示着On-NITR表达的差异情况很可能是由于不同组织的细胞种类不同而造成的。
4 结论本研究成功克隆并获得罗非鱼NITR基因,该基因cDNA全长1 119 bp,ORF为1 026 bp,可编码341个氨基酸。利用生物信息学相关方法对On-NITR蛋白结构和功能特点进行分析发现On-NITR具有1个信号肽区域、2个胞外Ig-domain区、1个跨膜结构域,以及一个有NITR典型的免疫受体酪氨酸抑制基序(ITIM)的胞质尾区,且具有较高的保守性。组织表达分析发现On-NITR在健康尼罗罗非鱼组织中均有表达,在肠道、皮肤、肝脏表达水平较高,在胸腺、鳃、脾脏、心脏和脑组织中的表达量较低,在头肾组织中的表达量最低。
| [1] | Colonna M. Specificity and function of immunoglobulin superfamily NK cell inhibitory and stimulatory receptors. Immunological Reviews, 1997, 155 (1): 127–133. DOI:10.1111/imr.1997.155.issue-1 |
| [2] | Daeron M. Fc receptor biology. Ann Rev Immunol, 1997, 15 . |
| [3] | Lee SJ, Benveniste EN. Adhesion molecule expression and regulation on cells of the central nervous system. Journal of Neuroimmunology, 1999, 98 (2): 77–88. DOI:10.1016/S0165-5728(99)00084-3 |
| [4] | Wei S, et al. The zebrafish activating immune receptor Nitr9 signals via Dap12. Immunogenetics, 2007, 59 (10): 813–821. DOI:10.1007/s00251-007-0250-6 |
| [5] | Djeu JY, Jiang K, Wei S. A view to a kill: signals triggering cytotoxicity. Clin Cancer Res, 2002, 8 (3): 636–640. |
| [6] | Hawke NA, Yoder JA, Haire RN, et al. Extraordinary variation in a diversified family of immune-type receptor genes. Proc Nati Acad Sci USA, 2001, 98 (24): 13832–13837. DOI:10.1073/pnas.231418598 |
| [7] | Hawke NA, Yoder JA, Litman GW. Expanding our understanding of immunoglobulin, T-cell antigen receptor, and novel immune-type receptor genes: A subset of the immunoglobulin gene superfamily. Immunogenetics, 1999, 50 (4): 124–133. |
| [8] | Strong SJ, Mueller MG, Litman RT, et al. A novel multigene family encodes diversified variable regions. Proceedings of the Nati Acad Sci USA, 1999, 96 (26): 15080–15085. DOI:10.1073/pnas.96.26.15080 |
| [9] | Yoder JA, Litman GW, Yoder JA., et al. Immune-type receptor genes in zebrafish share genetic and functional properties with genes encoded by the mammalian leukocyte receptor cluster. Proc Natil Acad Sci USA, 2001, 98 (12): 6771–6776. DOI:10.1073/pnas.121101598 |
| [10] | Yoder JA, Mueller GM, Nichols KM, et al. Cloning novel immune-type inhibitory receptors from the rainbow trout, Oncorhynchus mykiss. Immunogenetics, 2002, 54 (9): 662–670. DOI:10.1007/s00251-002-0511-3 |
| [11] | Stet RJ, Hermsen T, Westphal AH, et al. Novel immunoglobulin-like transcripts in teleost fish encode polymorphic receptors with cytoplasmic ITAM or ITIM and a new structural Ig domain similar to the natural cytotoxicity receptor NKp44. Immunogenetics, 2005, 57 (1-2): 77–89. DOI:10.1007/s00251-005-0771-9 |
| [12] | Piyaviriyakul P, et al. A novel immune-type receptor of Japanese flounder(Paralichthys olivaceus)is expressed in both T and B lymphocytes. Fish Shellfish Immunol, 2007, 22 (5): 467–476. DOI:10.1016/j.fsi.2006.05.007 |
| [13] | Desai S, Heffelfinger AK, Orcutt TM, et al. The medaka novel immune-type receptor(NITR)gene clusters reveal an extraordinary degree of divergence in variable domains. Bmc Evolutionary Biology, 2008, 8 (1): 177–185. DOI:10.1186/1471-2148-8-177 |
| [14] | Litman GW, Hawke NA, Yoder JA. Novel immune-type receptor genes. Immunological Reviews, 2001, 181 (1): 250–259. DOI:10.1034/j.1600-065X.2001.1810121.x |
| [15] | Shen L, Stuge TB, Bengtén E, et al. Identification and characteriza-tion of clonal NK-like cells from channel catfish(Ictalurus punctatus). Dev Comp Immunol, 2004, 28 (2): 139–152. DOI:10.1016/S0145-305X(03)00119-8 |
| [16] | Litman GW, Cannon JP, Dishaw LJ. Reconstructing immune phylogeny: new perspectives. Nature Reviews Immunology, 2005, 5 (11): 866–879. DOI:10.1038/nri1712 |
| [17] | Rojo I, et al. Innate immune gene expression in individual zebrafish after Listonella anguillarum, inoculation. Fish Shellfish Immunol, 2007, 23 (6): 1285–1293. DOI:10.1016/j.fsi.2007.07.002 |
| [18] | Barten R, Torkar M, et al. Divergent and convergent evolution of NK-cell receptors. Trends Immunol, 2001, 22 (1): 52–57. DOI:10.1016/S1471-4906(00)01802-0 |
| [19] | Hsu KC, Chida S, Geraghty DE, et al. The killer cell immunoglobulin-like receptor(KIR)genomic region: gene-order, haplotypes and allelic polymorphism. Immunological Reviews, 2002, 190 (1): 40–52. DOI:10.1034/j.1600-065X.2002.19004.x |
| [20] | 黄锦炉. 罗非鱼无乳链球菌病病原学、病理学及cpsE基因的原核表达研究[D]. 雅安: 四川农业大学, 2012. |
| [21] | Chothia C, Novotný J, Bruccoleri R, et al. Domain association in immunoglobulin molecules: The packing of variable domains. J Mol Biol, 1985, 186 (3): 651–663. DOI:10.1016/0022-2836(85)90137-8 |
| [22] | Barclay AN. Ig-like domains: evolution from simple interaction molecules to sophisticated antigen recognition. Proc Natil Acad Sci USA, 1999, 96 (26): 14672–14674. DOI:10.1073/pnas.96.26.14672 |
| [23] | Ravetch JV, Lanier LL. Immune Inhibitory Receptors. Science, 2000, 290 (5489): 84–89. DOI:10.1126/science.290.5489.84 |
| [24] | Bléry M, Olcese L, et al. Early signaling via inhibitory and activat-ing NK receptors. Hum Immunol, 2000, 61 (1): 51–64. DOI:10.1016/S0198-8859(99)00157-3 |
| [25] | Rudd PM, Elliott T, Cresswell P, et al. Glycosylation and the imm-une system. J Protein Chem, 1998, 17 (6): 2370–2376. |
| [26] | Yusa S, Catina TL, Campbell KS. KIR2DL5 can inhibit human NK cell activation via recruitment of Src homology region 2-containing protein tyrosine phosphatase-2(SHP-2). Journal of Immunology, 2004, 172 (12): 7385–7392. DOI:10.4049/jimmunol.172.12.7385 |
| [27] | Takai, Toshiyuki. Paired immunoglobulin-like receptors and their MHC classI recognition. Immunology, 2005, 4 : 433–440. |
| [28] | Tomasello E, Bléry M, Viély F, et al. Erratum: Signaling pathways engaged by NK cell receptors: double concerto for activating receptors, inhibitory receptors and NK cells. Seminars in Immunology, 2000, 12 (2): 139–147. DOI:10.1006/smim.2000.0216 |