MYB类转录因子是迄今为止在植物中发现的成员数量最多的转录因子。自1987年Paz-Ares等[1]从玉米中克隆得到与色素合成相关的MYB类转录因子基因 (C1) 以来,已经从多种植物中分离和鉴定了大量的MYB类转录因子,如拟南芥中198个[2]、水稻中183个[3]、苹果中229个[4]、甜橙中177个[5]、谷子 (Setaria italica) 中209个[6],获得MYB类转录因子的成员数量在100以上是普遍的。结构上,MYB转录因子各家族成员的N-端具有高度保守的结构域,即DNA binding结构域,也称为MYB结构域,由1-4个不等的MYB重复单元组成。根据MYB结构域所含重复单元个数,将MYB转录因子划分为4种类型: 4R-MYB (4个MYB重复单元)、3R-MYB (3个MYB重复单元)、R2R3-MYB (2个MYB重复单元) 以及MYB-related (1个或部分MYB重复单元),其中R2R3-MYB类转录因子数量最多[7-9]。
随着对植物中更多的MYB转录因子的功能解析,人们发现不同类型的MYB转录因子具有不同的功能,参与生长发育、细胞形态建成、激素和环境因子应答等许多生命活动过程的调节,特别是参与植物初级和次级代谢的调节[10]。研究表明,拟南芥的类黄酮合成途径相关基因 (如EBGs和LBGs) 受AtMYB11及AtMYB75等调控[8, 9],棉花的GmMYB042基因的过表达使转基因烟草的类黄酮代谢途径部分关键酶基因 (如PAL、CHS和FLS) 的表达量明显上升[10],玉米的ZmMYB111和ZmMYB148可同时调节PAL基因,涉及类黄酮和木质素的合成[11]。
蒙古沙冬青 (A. mongolicus) 是我国西北荒漠地区唯一的强旱生常绿阔叶灌木,同时也是阿拉善荒漠区特有的建群物种,对维持当地生态平衡发挥至关重要的作用。沙冬青长期生活在干旱、低温等逆境环境中,具有极强的抗旱、抗寒能力,其形态结构和生理方面都有与之相适应的特征,这为提高水分的利用率、应对水分亏缺、极端严寒、高温、风蚀沙埋等逆境环境提供了条件[12, 13],因此沙冬青是研究植物抗逆机理和筛选抗逆基因的资源植物,具有重要的科学研究价值。
近年来,基于现代分子生物学技术的发展,通过转录组测序构建了蒙古沙冬青正常与逆境胁迫下的转录本数据库,为筛选抗逆响应基因、研究抗逆应答机理提供了大量的数据[14-17],同时,人们研究了沙冬青中重要的功能基因 (包括编码转录因子基因) 的结构和响应逆境的表达特征,如AmCBL1[18]和AmDREB2[19]等,但关于蒙古沙冬青中的MYB转录因子基因的研究还未见报道。近来,本实验室的研究表明,干旱胁迫下MiR858通过上调MYB转录因子的表达而调节类黄酮类物质的合成[20]。由于植物中MYB类转录因子数量最多,且参与多种生物学过程,因此,本研究基于本实验室前期构建的蒙古沙冬青转录本数据库,对获得的AmMYB4-like基因进行了相关研究,旨在为阐释MYB类转录因子在蒙古沙冬青逆境应答中的特点提供数据支持。
1 材料与方法 1.1 材料蒙古沙冬青 (A. mongolicus) 种子采自宁夏回族自治区中卫县。播种于盛有蛭石:珍珠岩:营养土 (3:1:1) 浸湿的混合土壤的塑料盆中,光照16 h (23℃)/黑暗8 h (18℃),光照强度93 μmol·m-2·s-1,空气湿度30%-40%。种子发芽后,每周浇一次水。生长8周后,分别以20% PEG6000模拟干旱胁迫、以4℃低温培养进行低温胁迫,胁迫时间分别为0 h、1 h、6 h、24 h、7 d,以0 h为对照。分别收获叶片和根组织,液氮速冻,-80℃冷冻保存。每个处理设置3个生物学重复。
Trizol试剂购自Invitrogen公司,FastQuant cDNA第一链合成试剂盒、荧光定量试剂盒SYBR Green和无缝重组试剂盒 (EasyGeno) 购自天根生化 (北京) 有限公司,KOD FX Neo PCR克隆酶购自东洋纺 (上海) 生物科技有限公司,胶回收试剂盒购自爱思进生物技术 (杭州) 有限公司,大肠杆菌 (Escherichia coli) DH5α感受态细胞购自北京博迈德科技发展有限公司,质粒小量抽提试剂盒购自百泰克生物技术有限公司,AT克隆载体购自北京全式金生物。植物表达载体pPZP212为本实验室保存。
1.2 方法 1.2.1 基因分离根据本实验室构建的蒙古沙冬青干旱转录本数据库,筛选表达量较高的MYB基因序列作为候选基因,设计引物序列 (表 1)。用Trizol法提取蒙古沙冬青叶片和根总RNA,cDNA第一链合成按天根FastQuant RT kit说明书进行。反应体系 (50 μL):第一链cDNA 1 μL、10 mmol/L正向引物1.5 μL、10 mmol/L反向引物1.5 μL、2 mmol/L dNTPs 10 μL、2×PCR Buffer for KOD Fx Neo 25 μL、1 U/μL KOD FX Neo 1 μL及ddH2O 10 μL。PCR扩增条件: 94℃预变性2 min,98℃变性10 s,61℃退火30 s,68℃延伸60 s,循环34次。扩增产物经1%琼脂糖凝胶电泳检测后,回收目的片段,送至生工 (上海) 测序。
将获得的基因序列在NCBI网站进行BLAST查询,获得相关同源基因信息。使用在线的ORF (Open reading frame,ORF) 查询网站确定基因的ORF (http://www.ncbi.nlm.nih.gov/orffinder/)。用于进化树分析的序列来源于NCBI数据库。用MEGA5.1软件进行蛋白序列比对及进化树分析,用邻接法 (Neighbor-Joining) 构建系统进化树。使用在线软件 (http://web.expasy.org/compute_pi/) 预测等电点及相对分子质量大小,使用在线软件 (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc/) 对亚细胞定位进行预测,使用NetPhos3.1Serve在线软件 (http://www.cbs.dtu.dk/services/NetPhos/) 预测磷酸化位点。使用在线软件 (http://weblogo.berkeley.edu/examples.html) 进行保守结构域分析。
1.2.3 胁迫条件下的基因表达分析用Trizol法提取20% PEG6000及4℃低温处理的蒙古沙冬青叶片和根的总RNA,反转录合成单链cDNA。将单链cDNA产物浓度稀释5倍后作为荧光定量PCR反应的模板。在荧光定量PCR仪MYIQ2(Bio-Rad) 上进行荧光定量PCR扩增,引物序列见表 1。反应体系 (20 μL): 2×FastFire qPCR PreMix (with SYBR Green I)10 μL、ddH2O 7 μL、10 mmol/L上下游特异引物 (见表 1) 各1 μL和cDNA 1 μL。扩增条件: 95℃预变性1 min,95℃变性5 s,55℃退火10 s,72℃延伸15 s,循环40次。以蒙古沙冬青AmeIF1基因为内参基因[21]。采用2-△△Ct方法计算目的基因的相对表达量。
1.2.4 植物表达载体的构建利用无缝重组试剂盒 (EasyGeno) 使用无缝重组方法将目的基因连接到植物表达载体pPZP212上,选择Xba Ⅰ和Sac Ⅰ作为插入位点,引物序列见表 1。重组反应体系 (10 μL): 0.5 μL线性化载体 (110 ng/μL)、2 μL插入片段 (180 ng/μL)、5 μL 2×EasyGeno Assembly Mix、2.5 μL ddH2O。具体步骤参考说明书进行。将重组载体转化E. coli DH5α,在含有100 μg/mL壮观霉素的LB固体培养基上筛选转化菌落,进行菌液PCR验证及测序验证。
2 结果 2.1 AmMYB4-like基因的扩增以蒙古沙冬青叶片总RNA反转录后获得的cDNA为模板,AmMYB4-like-F和AmMYB4-like-R为引物进行PCR扩增。图 1显示,扩增的目的条带大小约1 000 bp。经过测序,该序列长度为1 145 bp。将得到的序列在NCBI网站进行Blast检索,该序列与大豆GmMYB4-like的核苷酸序列相似性最高 (89%),因此将获得的基因命名为AmMYB4-like。
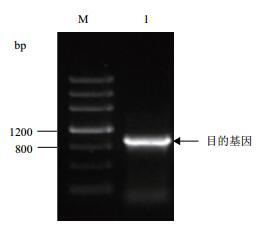
|
| 图 1 AmMYB4-like PCR扩增产物凝胶电泳图 M: DNA Marker Ⅲ;1: PCR产物 |
所获得的蒙古沙冬青编码MYB蛋白的目的基因全长为1 145 bp,其中5'UTR长度为157 bp,3'UTR长度为28 bp,开放阅读框长度为960 bp,编码319个氨基酸 (图 2)。在线软件分析表明,该蛋白的等电点 (pI) 为5.09,相对分子质量 (MW) 为78.530 21 kD。对其进行MYB结构域分析,AmMYB4-like含有R2和R3保守结构域,属于典型的R2R3-MYB类转录因子 (图 2,图 3)。

|
| 图 2 AmMYB4-like的核苷酸序列及推测的蛋白序列 阴影区分别表示R2和R3保守结构域;具有下划线的氨基酸残基为功能残基;“波浪线”表示用于ORF区扩增的引物序列;“□”表示用于qRT-PCR的引物序列 |
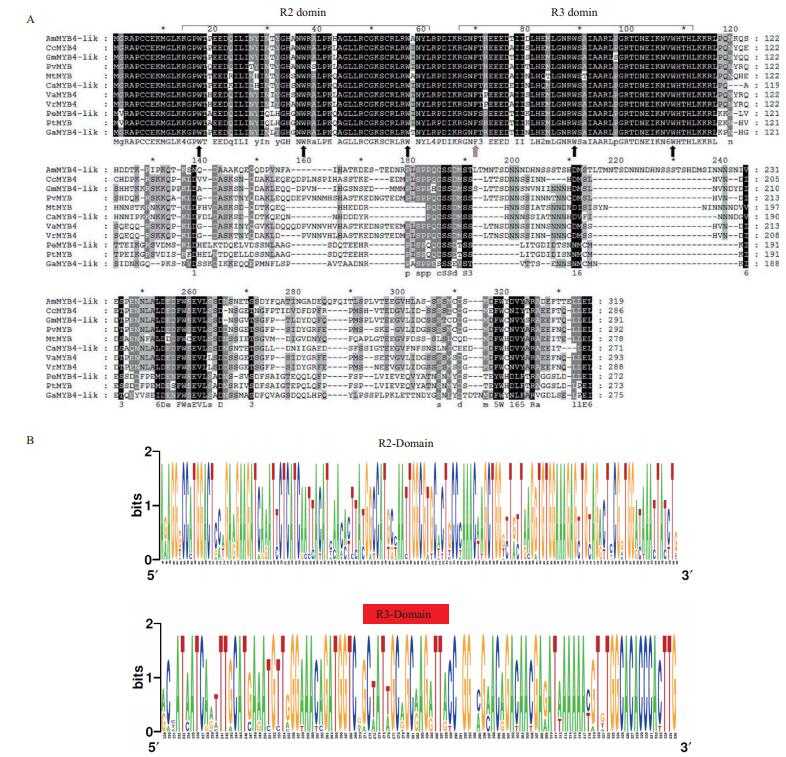
|
| 图 3 AmMYB4-like与其他物种已知MYB蛋白的多序列比对 (A) 及R2和R3保守结构域分析 (B) s 共有序列:黑色阴影;R2和R3保守结构域:框线;箭头:与bHLH蛋白相互作用的氨基酸残基;黑色箭头:色氨酸残基 (W),灰色箭头:苯丙氨酸残基 (F) 大豆 (Glycine max) GmMYB4-like (NP_001304532.1),木豆 (Cajanus cajan) CcMYB4(KYP70735.1),菜豆 (Phaseolus vulgaris) PvMYB (XP_007162544.1),蒺藜状苜蓿 (Medicago truncatula) MtMYB (XP_013449896.1),鹰嘴豆 (Cicer arietinum) CaMYB4-like (XP_012569365.1),红豆 (Vigna angularis) VaMYB4(XP_017411137.1),绿豆 (Vigna radiata var. radiate) VrMYB4(XP_014495663.1),胡杨 (Populus euphratica) PeMYB4-like (XP_011025983.1),三叶杨 (Populus trichocarpa) PtMYB (XP_002306849.2),木本棉 (Gossypium arboretum) GaMYB4-like (XP_017640194.1) |
对AmMYB4-like进行亚细胞定位预测,结果表明其定位于细胞核中。利用在线软件预测AmMYB4-like具有41个磷酸化修饰位点,其中Ser 24个,Thr 15个,Tyr 2个。
2.2.2 AmMYB4-like的同源序列比对与系统进化分析在NCBI中搜索与AmMYB4-like相近的10个编码MYB的氨基酸序列与本研究获得的AmMYB4-like进行多序列比对。结果显示 (图 3-B),所有序列的N-端都具有R2和R3保守结构域,皆属于R2R3-MYB类转录因子。在R2保守结构域中含有3个色氨酸残基 (W),相互间的间隔为19个氨基酸。在R3保守结构域中的第一个色氨酸残基 (W) 被苯丙氨酸 (F) 所取代,另外2个色氨酸残基 (W) 不变。此外多序列比对显示各序列在C-端变异较大 (图 3-A)。对N-端的R2和R3保守结构域进一步采用在线软件分析氨基酸序列特点,结果显示保守性很强 (图 3-B)。
为研究AmMYB4-like与其他植物MYB序列的进化关系,除了上述10个序列外,又选取了4个编码MYB的氨基酸序列:拟南芥 (Arabidopsis thaliana) AtMYB14(NP_180676.1),烟草 (Nicotiana tabacum) NtMYB4-like (NP_001312823.1),玉米 (Zea mays) ZmMYB4(NP_001296459.1) 和水稻 (Oryza sativa) OsMYB4(XP_015633465.1),采用邻接法 (N-J) 构建进化树。结果 (图 4) 显示,双子叶植物的MYB4序列和单子叶植物的MYB4序列明显的聚为2类,而属于豆科植物的8个序列聚为1类,亲缘关系较近。尽管本研究中的AmMYB4-like与所研究的豆科植物的7个MYB4序列亲缘关系最近,但表现独为一支。

|
|
图 4 AmMYB4-like的系统进化树分析
大豆 (Glycine max) GmMYB4-like (NP_001304532.1),木豆 (Cajanus cajan) CcMYB4(KYP70735.1),菜豆 (Phaseolus vulgaris) PvMYB (XP_007162544.1),蒺藜状苜蓿 (Medicago truncatula) MtMYB (XP_013449896.1),鹰嘴豆 (Cicer arietinum) CaMYB4-like (XP_012569365.1),红豆 (Vigna angularis) VaMYB4(XP_017411137.1),绿豆 (Vigna radiata var. radiate) VrMYB4(XP_014495663.1),胡杨 (Populus euphratica) PeMYB4-like (XP_011025983.1),三叶杨 (Populus trichocarpa) PtMYB (XP_002306849.2),木本棉 (Gossypium arboretum) GaMYB4-like (XP_017640194.1),拟南芥 (Arabidopsis thaliana) AtMYB14(NP_180676.1),烟草 (Nicotiana tabacum) NtMYB4-like (NP_001312823.1),玉米 (Zea mays) ZmMYB4(NP_001296459.1) 和水稻 (Oryza.sativa) OsMYB4(XP_015633465.1) 三角表示为本研究的AmMYB4-like |
采用qRT-PCR技术分析正常条件下AmMYB4-like在叶片和根中的表达特点。结果 (图 5) 表明,AmMYB4-like在叶片和根中都表达,且叶片中的表达量较高,约为根中的1.46倍。以20% PEG6000模拟干旱胁迫,处理1 h时与对照相比,AmMYB4-like在叶片和根中都表现为上调表达,分别为对照水平的3.08倍和4.04倍。而胁迫处理6 h时,AmMYB4-like在叶片中的表达量降低、与对照水平相同,而根中的表达量继续增加,为对照水平的4.23倍。当胁迫处理24 h时,AmMYB4-like在叶片中的表达量与对照基本相同,而根中的表达量降低为对照水平的59.8%。当胁迫处理时间维持7 d时,AmMYB4-like在叶片中的表达量降低,为对照水平的61.2%,根中的表达量增加,为对照水平的2.52倍 (图 6)。
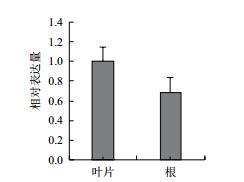
|
| 图 5 AmMYB4-like在不同组织中的表达特征 |
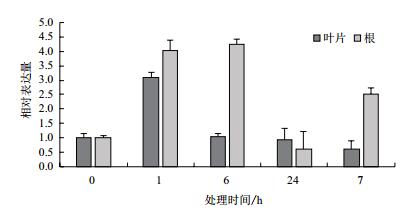
|
| 图 6 20%PEG6000胁迫下AmMYB4-like在不同组织中的表达特征 |
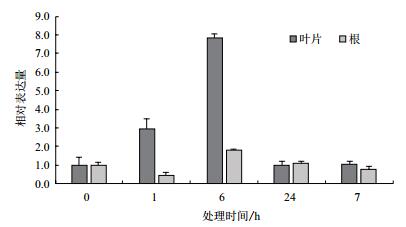
采用qRT-PCR技术分析4℃低温处理下AmMYB4-like在叶片和根中的表达特点。结果 (图 7) 显示,AmMYB4-like在叶片和根中表达不同。4℃低温处理1 h和6 h时,与对照相比,AmMYB4-like在叶片表现为上调表达,分别为对照水平的2.93倍和7.86倍,当胁迫时间延长,AmMYB4-like表达量降低,与对照水平基本相同。而根中的AmMYB4-like表达量在胁迫6 h增加,为对照水平的1.77倍,在其他处理条件下都低于或近于对照水平。
|
| 图 7 4℃低温胁迫下AmMYB4-like在不同组织中的表达特征 |
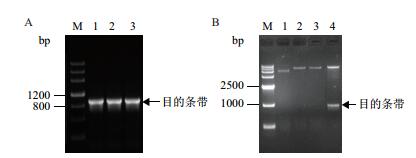
采用无缝重组法构建表达载体pPZP212-AmMYB4-like,转化E. coli DH5α,在含有Spec的LB固体培养基上筛选转化菌落,菌液PCR验证结果 (图 8-A) 显示,在1 000 bp位置附近具有与目的基因大小一致的条带,进一步测序显示结果正确,同时对构建好的重组载体进行了酶切验证,结果 (图 8-B) 显示在1 000 bp位置附近具有与目的基因大小一致的片段,以上结果表明pPZP212-AmMYB4-like植物表达载体构建成功。
|
| 图 8 pPZP212-AmMYB4-like重组载体菌液PCR验证 (A) 及酶切验证 (B) A. M: DNA Marker Ⅲ;1、2、3为PCR产物. B. M: D15 000 DNA Marker;1:质粒;2:质粒Sac Ⅰ单酶切;3:质粒Xba Ⅰ单酶切;4:质粒 |
植物的MYB转录因子是具有成员数量最多的转录因子超家族,在4种类型的MYB转录因子中,属于R2R3-MYB转录因子数量最多,但不同物种的数量不同,如大豆 (G. max)244个[22],棉花 (Gossypium raimondii)205个[23],毛果杨 (Populus trichocarpa)192个[24],玉米 (Z. mays)157个[25],拟南芥126个[26],葡萄 (Vitis vinifera)108个[27],水稻 (O.sa-tiva)88个[28]。有研究者认为R2R3-MYB转录因子数量多少与基因数量多少有关[23],也就是说物种基因组中所含有的基因数量越多,R2R3-MYB转录因子数量增加。
研究表明,R2R3-MYB转录因子具有重要的生物学功能,包括参与初级和次级代谢、生物和非生物胁迫、细胞命运和发育过程等[7]。R2R3-MYB转录因子除了在N端具有保守的DNA-binding结构域外,在C端则有转录激活/抑制区[23]。许多功能基因的启动子中含有特殊的MYB结合元件 (核心序列TAACTG),在逆境胁迫情况下,MYB转录因子可以与该元件结合从而激活胁迫应答基因的表达[29]。
本研究从蒙古沙冬青中克隆得到1个编码MYB转录因子基因AmMYB4-like,生物信息学分析表明,其编码蛋白序列含有高度保守的R2和R3的MYB结构域,属于R2R3-MYB类转录因子。蒙古沙冬青属于豆科植物,系统进化树分析显示AmMYB4-like与同属于豆科MYB4亲缘关系最近,而与其他双子叶植物物种的亲缘关系相对较远,与单子叶植物 (水稻和玉米) MYB4的亲缘关系最远,提示植物MYB4的进化可能存在种属特异性。
杜海等[10]在研究大豆GmMYB042时发现其受到PEG8000、NaCl及低温诱导。He等[23]选取了棉花 (G. raimondi) 中25个属的R2R3-MYB转录因子,分别研究了在叶片和根中应答干旱和高盐胁迫下的表达特征,结果表明,这些成员在叶片和根中具有不同的响应干旱和高盐的表达模式,说明可以通过不同的信号途径参与胁迫应答。本研究中,正常条件下AmMYB4-like在根和叶中都表达,但在不同胁迫条件下,具有不同的表达模式。以20%PEG6000模拟干旱处理,叶片和根中的AmMYB4-like上调表达,叶片中的AmMYB4-like在1 h时达到最高水平,为对照水平的3.08倍,而根中的AmMYB4-like在6 h达到最高水平,为对照水平的4.23倍,表明叶片和根中的AmMYB4-like参与干旱胁迫应答。在4℃低温处理下,叶片中的AmMYB4-like表达量增加,处理6 h时达到最高水平,为对照水平的7.86倍,但根中AmMYB4-like尽管在4℃低温处理6 h表达提高,但相对较少。上述结果说明蒙古沙冬青叶片中的AmMYB4-like既响应低温胁迫,也响应干旱胁迫,而根中的AmMYB4-like主要参与干旱胁迫应答。本研究以7 d作为较长的胁迫时间研究了AmMYB4-like的表达特征,结果显示,只有根中的AmMYB4-like在干旱胁迫7 d时检测出上调表达,为对照水平的2.52倍,说明根中的AmMYB4-like参与较长时间的干旱胁迫应答。根作为植物吸水的重要组织部位,对于干旱耐受能力十分重要,蒙古沙冬青是强旱生植物,其根的干旱耐受性十分重要,干旱胁迫下根中的相关基因的表达对其耐受性可能具有一定的意义。
4 结论从蒙古沙冬青中分离到1个编码MYB转录因子基因,命名为AmMYB4-like。该序列开放阅读框编码319个氨基酸,具有典型的MYB转录因子保守的R2R3结构域,属于R2R3-MYB转录因子。qRT-PCR分析表明,叶片中的AmMYB4-like参与低温和干旱胁迫应答,而根中的AmMYB4-like主要参与干旱胁迫应答。构建了pPZP212-AmMYB4-like植物表达载体。
| [1] | Paz-Ares J, Ghosal D, Wienand U, et al. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators. EMBO J, 1987, 6 (12): 3553–3558. |
| [2] | Chen YH, Yang XY, He K, et al. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family. Plant Mol Biol, 2006, 60 (1): 107–124. DOI:10.1007/s11103-005-2910-y |
| [3] | Cao ZH, Zhang SZ, Wang RK, et al. Genome wide analysis of the apple MYB transcription factor family allows the identification of MdoMYB121 gene conferring abiotic stress tolerance in plants. PLoS One, 2013, 8 (7): e69955. DOI:10.1371/journal.pone.0069955 |
| [4] | Hou XJ, Li SB, Liu SR, et al. Genome-wide classification and evolutionary and expression analyses of citrus MYB transcription factor families in sweet orange. PLoS One, 2014, 9 : e112375. DOI:10.1371/journal.pone.0112375 |
| [5] | Muthamilarasan M, Khandelwal R, Yadav CB, et al. Identification and molecular characterization of MYB transcription factor superfamily in C4 model plant foxtail millet (Setaria italica L.). PLoS One, 2014, 9 : e109920. DOI:10.1371/journal.pone.0109920 |
| [6] | Du H, Zhang L, Liu L, et al. Biochemical and molecular characterization of plant MYB transcription factor family. Biochemistry (Mosc), 2009, 74 (1): 5–16. |
| [7] | Ambawat S, Sharma P, Yadav NR, et al. MYB transcription factor genes as regulators for plants: an overview. Physiol Mol Biol Plants, 2013, 19 (3): 307–321. DOI:10.1007/s12298-013-0179-1 |
| [8] | Lepiniec L, Debeaujon I, Routaboul JM, et al. Genetics and biochemistry of seed flavonoids. Annu Rev Plant Biol, 2006, 57 (1): 405–430. DOI:10.1146/annurev.arplant.57.032905.105252 |
| [9] | Gonzalez A, Zhao M, Leavitt JM, et al. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings. Plant J, 2008, 53 (5): 814–827. DOI:10.1111/tpj.2008.53.issue-5 |
| [10] | 杜海, 冉凤, 马珊珊, 等. GmMYB042基因对类黄酮生物合成的调控作用. 作物学报, 2016, 42(1): 1–10. DOI:10.7606/j.issn.1009-1041.2016.01.01 |
| [11] | Zhang JJ, Zhang SS, Li H, et al. Identification of transcription factors ZmMYB111 and ZmMYB148 involved in phenylpropanoid metabolism. Front Plant Sci, 2016, 7 : 131–134. |
| [12] | 周宜君, 刘春兰, 冯金朝, 等. 沙冬青抗旱、抗寒机理的研究进展. 中国沙漠, 2001, 21(3): 312–315. |
| [13] | 郭生祥, 刘志银, 郝昕. 沙冬青的研究进展. 甘肃林业科技, 2005, 30(4): 5–8. |
| [14] | Zhou YJ, Gao F, Liu R, et al. De novo sequencing and analysis of root transcriptome using 454 pyrosequencing to discover putative genes associated with drought tolerance in Ammopiptanthus mongolicus. BMC Genomics, 2012, 13 : 266. DOI:10.1186/1471-2164-13-266 |
| [15] | Gao F, Wang J, Wei SJ, et al. Transcriptomic analysis of drought stress responses in Ammopiptanthus mongolicus leaves using the RNA-Seq technique. PLoS One, 2015, 10 : e0124382. DOI:10.1371/journal.pone.0124382 |
| [16] | Wu YQ, Wei W, Pang X, et al. Comparative transcriptome profiling of a desert evergreen shrub, Ammopiptanthus mongolicus, in response to drought and cold stresses. BMC Genomics, 2014, 15 : 671. DOI:10.1186/1471-2164-15-671 |
| [17] | Pang T, Ye CY, Xia X, et al. De novo sequencing and transcriptome analysis of the desert shrub, Ammopiptanthus mongolicus, during cold acclimation using Illumina/Solexa. BMC Genomics, 2013, 14 : 488. DOI:10.1186/1471-2164-14-488 |
| [18] | Chen JH, Sun Y, Sun F, et al. Tobacco plants ectopically expressing the Ammopiptanthus mongolicus AmCBL1 gene display enhanced tolerance to multiple abiotic stresses. Plant Growth Regul, 2011, 6 (3): 259–269. |
| [19] | 李章磊, 高飞, 曹玉震, 等. 蒙古沙冬青AmDREB2. 1基因的克隆及表达分析. 生物技术通报, 2015, 31(3): 108–114. |
| [20] | Gao F, Wang N, Li HY, et al. Identification of drought-responsive microRNAs and their targets in Ammopiptanthus mongolicus by using high-throughput sequencing. Sci Rep, 2016, 6 : 34601. DOI:10.1038/srep34601 |
| [21] | Shi J, Liu M, Shi J, et al. Reference gene selection for qPCR in Ammopiptanthus mongolicus under abiotic stresses and expression analysis of seven ROS-scavenging enzyme genes. Plant Cell Rep, 2012, 31 (7): 1245–54. DOI:10.1007/s00299-012-1245-9 |
| [22] | Du H, Yang SS, Liang Z, et al. Genome-wide analysis of the MYB transcription factor superfamily in soybean. BMC Plant Biol, 2012, 12 : 106. DOI:10.1186/1471-2229-12-106 |
| [23] | He Q, Jones DC, Li W, et al. Genome-Wide Identification of R2R3-MYB genes and expression analyses during abiotic stress in Gossypium raimondii. Sci Rep, 2016, 6 : 22980. DOI:10.1038/srep22980 |
| [24] | Wilkins O, Nahal H, Foong J, et al. Expansion and diversification of the Populus R2R3-MYB family of transcription factors. Plant Physiol, 2009, 149 (2): 981–993. |
| [25] | Du H, Feng BR, Yang SS, et al. The R2R3-MYB transcription factor gene family in maize. PLoS One, 2012, 7 : e37463. DOI:10.1371/journal.pone.0037463 |
| [26] | Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana. Curr Opin Plant Biol, 2001, 4 (5): 447–456. DOI:10.1016/S1369-5266(00)00199-0 |
| [27] | Matus JT, Aquea F, Arce-Johnson P. Analysis of the grape MYB R2R3 subfamily reveals expanded wine quality-related clades and conserved gene structure organization across Vitis and Arabidopsis genomes. BMC Plant Biol, 2008, 8 (1): 1–15. DOI:10.1186/1471-2229-8-1 |
| [28] | Katiyar A, Smita S, Lenka SK, et al. Genome-wide classification and expression analysis of MYB transcription factor families in rice and Arabidopsis. BMC Genomics, 2012, 13 : 544. DOI:10.1186/1471-2164-13-544 |
| [29] | 乔孟, 于延冲, 向凤宁. 拟南芥R2R3-MYB类转录因子在环境胁迫中的作用. 生命科学, 2009, 21(1): 145–150. |





