狭义的RNA干扰(RNA interference,RNAi),是指由长双链RNA(double-stranded RNA,dsRNA)产生的小RNA介导序列特异性mRNA降解,进而引发基因沉默的现象。广义的RNAi,是指由小非编码RNA(Small non-coding RNA,sncRNA)诱导mRNA降解、抑制翻译或促使异染色质形成等,进而引发基因沉默的现象[1-3]。本文所描述的RNAi指的是广义RNAi。
1998年,Fire和Mello等[4]在线虫(Caenorhabditis elegans)的研究中首次描述了RNAi现象。他们在研究反义RNA作用的过程中发现,正义RNA和反义RNA的混合物,即dsRNA对基因的沉默作用远远强于任一单链RNA,并将dsRNA(实际是由dsRNA产生的小RNA)引起的基因沉默现象称为RNAi。此后,人们在果蝇(Drosophila melanogaster)、锥虫(Trypanosoma)、涡虫(Planaria)、水螅(Hydra)、斑马鱼(Barchydanio rerio)、拟南芥(Arabidopsis thaliana)、青蛙(Rana nigromaculata)、小鼠及人类等多种真核生物中均发现了RNAi现象[5, 6]。RNAi是真核生物独有的基因沉默现象,普遍存在于真核生物中,是真核生物调控内源基因表达及抵御外源核酸入侵的高效自我保护系统[7, 8]。本文从广义的角度对RNAi的作用机制、作用过程的关键因子及其应用的研究进展进行综述,以期为RNAi的进一步深入研究提供参考。
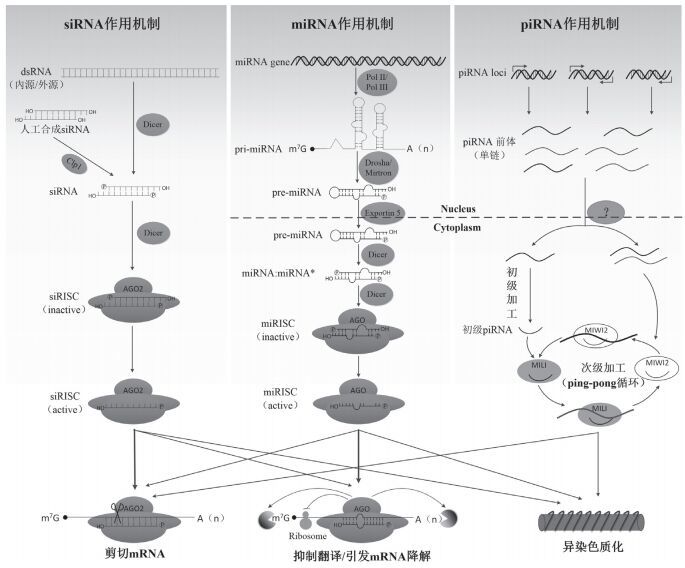
1 RNAi的作用机制RNAi由长度为20-30 nt的sncRNA触发,根据sncRNA生物合成和作用机制的不同,可将其分为siRNA(small interfering RNA)、miRNA(microRNA)和piRNA(PIWI-interacting RNA)3种[9]。
1.1 siRNA的作用机制siRNA的作用机制见图 1。由于RNA病毒入侵、基因组中反向重复序列转录、转座子转录、自退火转录或实验转染等原因,使得细胞中出现内源性或外源性的dsRNA,核酸内切酶Dicer识别dsRNA,并将其剪切成长度为21-25 nt、5′端含有一个磷酸基团、3′端含有一个羟基并且突出2 nt的siRNA[9-12]。siRNA的这一结构对于RNAi是必要的,缺乏5′端磷酸化的siRNA或平端siRNA无论在细胞内外都无法启动RNAi[13]。人工导入的siRNA需要经过细胞质中的激酶Clp1加工成5′端磷酸化的siRNA才能参与诱导靶基因沉默[14]。具有一定结构的siRNA随后在Dicer的帮助下与AGO2等结合,构成siRNA诱导沉默复合体(siRNA-induced silencing complex,siRISC),siRISC中的siRNA经AGO2作用分解成两条单链,正义链被释放出去,反义链则留在siRISC中。仅含反义链的siRISC被激活,在反义链的引导下通过碱基互补配对原则与靶基因结合,进而诱导靶基因的沉默。沉默发生后,靶基因被释放,使得siRISC能与另一个靶基因结合,开始诱导新一轮的基因沉默[10, 15, 16]。
siRISC诱导基因沉默的具体方式包括以下3种:(1)反义siRNA与靶mRNA完全互补配对(一般发生在mRNA的编码区或开放阅读框中),siRISC中具有核酸内切酶活性的AGO2剪切靶mRNA,启动靶mRNA的降解,一旦最初降解形成,细胞中的核酸外切酶就会攻击剩余片段以完成整个降解过程,从而导致靶基因沉默,这是siRISC的主要作用方式[17-20]。(2)反义siRNA与靶mRNA不完全配对(一般发生在mRNA的3′非编码区),此时的siRISC将抑制靶mRNA的翻译或引发靶mRNA的降解。siRISC通过与翻译起始复合物竞争mRNA的5′帽子结构、影响核糖体大小亚基的聚合等作用在翻译的起始阶段抑制翻译,或者通过减缓核糖体在翻译中的移动速度、促进核糖体的提早解离等作用在翻译的其他阶段抑制翻译。siRISC还可以促进脱帽酶切除mRNA的5′帽子结构,脱腺苷酶切除mRNA的3′端poly(A)尾巴,使mRNA失去5′和3′端的保护而被核酸外切酶攻击,最终导致mRNA的降解[21-23]。(3)除了作用于mRNA在转录后水平调控基因表达外,siRISC也可作用于DNA在转录水平调控基因的表达。siRISC通过激活甲基化酶或抑制去甲基化酶的作用,引发DNA的甲基化,DNA的甲基化进一步导致染色质蛋白的甲基化、乙酰化、磷酸化和泛素化等,从而促进异染色质的形成,引发基因沉默[21, 24-26](图 1)。
双链siRNA进入siRISC后,经AGO2作用解开成两条单链,反义链留在siRISC中引导siRISC沉默靶基因,正义链被释放出去。被释放的正义链可能很快被降解,也可能作为引物,在RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase,RdRP)的催化下以靶mRNA为模板扩增得到dsRNA,dsRNA又被Dicer降解成siRNA进入RNAi循环,从而使得RNAi的效果级联放大[10, 27-29]。
1.2 miRNA的作用机制与siRNA主要来自外源性dsRNA不同,miRNA主要源于生物自身的基因组[31]。细胞核内编码miRNA的基因在RNA聚合酶Ⅱ或聚合酶Ⅲ(polymerase Ⅱ/ polymerase Ⅲ,Pol Ⅱ/Pol Ⅲ)的作用下转录得到pri-miRNA,pri-miRNA是一个具有帽子结构、poly(A)尾巴、一个或多个茎环结构的大分子RNA,长度从数百到数万个碱基不等。pri-miRNA经Drosha或Mirtron作用,去除帽子结构和poly(A)尾巴,得到长度约为70 nt的pre-miRNA,pre-miRNA通常具有茎环结构,且其3′端突出2 nt。pre-miRNA被转运蛋白Exportin 5转运到细胞质中,在细胞质中被Dicer剪切成长度约为22 nt的miRNA:miRNA*双链[21, 32]。这种双链miRNA的分子结构与siRNA相似,同样是5′端具有磷酸基团,3′端具有羟基且突出2 nt,不同的是siRNA的两条链完全互补配对,而miRNA的两条链只是部分互补配对[33]。miRNA:miRNA*双链很快与AGO(AGO1-4,主要是AGO1)等结合形成miRNA诱导沉默复合体(miRNA-induced silencing complex,miRISC),随后miRNA:miRNA*双链中5′端碱基配对稳定性较强的正义链miRNA*迅速被降解,5′端碱基配对稳定性较弱的反义链miRNA则保留在miRISC中。仅含反义链的miRISC被激活,在反义链miRNA的引导下通过碱基互补配对原则识别靶基因,诱导靶基因沉默[9, 21](图 1)。
siRNA主要通过完全互补配对的方式与靶基因结合,并通过直接剪切作用诱发靶基因的降解,从而保护体内基因组不受转座子活动或病毒感染等外源性因素的影响。与之不同,miRNA主要通过不完全配对的方式与靶mRNA结合,抑制靶mRNA的翻译或引发靶mRNA的降解,从而内源性的调控体内基因的表达[34, 35]。miRNA与靶mRNA不完全配对时的具体作用机制与siRNA与靶mRNA不完全配对时的具体作用机制类似,亦是通过与翻译起始复合物竞争5′帽子结构或影响核糖体的正常功能等作用抑制靶mRNA的翻译,以及通过促进相应酶切除mRNA的5′和3′端保护结构等作用诱发靶基因的降解[21, 36-38]。与siRNA一致,miRNA也可以与靶基因完全互补配对,通过直接剪切作用诱发靶基因的降解[39]。此外,miRNA也能作用于DNA,通过诱导DNA甲基化,促进异染色质的形成,从而在转录水平调控基因的表达[40](图 1)。
1.3 piRNA的作用机制RNAi过程中,3种sncRNA均与Argonaute蛋白家族结合,Argonaute蛋白家族包括AGO和PIWI两类亚家族,siRNA和miRNA与AGO亚家族结合,而piRNA则与PIWI亚家族结合[41, 42]。piRNA途径是动物特有的,主要存在于动物的生殖细胞中[43]。piRNA的具体作用机制尚未阐明,目前对piRNA的认识尚处于初级阶段。
基因间的重复序列、活跃的转座子基因或piRNA基因簇转录得到的单链piRNA前体,经具有核酸酶活性的PIWI蛋白加工,得到长度约为24-32 nt的piRNA。与siRNA和miRNA不同,piRNA的生成不需要Dicer的参与。piRNA的生物合成涉及初级加工和次级加工两条途径。初级加工途径产生初级piRNA,初级piRNA经次级加工途径(亦称ping-pong循环)扩增。初级加工及初级piRNA与PIWI蛋白的结合发生在细胞质中,但初级加工过程所需的因子尚未清楚。在ping-pong循环中,初级piRNA依次被PIWI蛋白MILI和MIWI2(即PIWIL2和PIWIL4)剪切其5′端,由此决定piRNA的5′端结构,决定piRNA 3′端结构的核酸酶目前未知[30, 44]。piRNA可以通过直接剪切靶mRNA或者促进异染色质的形成等方式诱导基因沉默,从而在转录后水平或转录水平调控基因的表达[45, 46]。
2 RNAi过程的关键因子RNAi过程中有多个关键因子参与,其中最为重要的是Dicer、Argonaute蛋白家族和RdRP。
2.1 DicerDicer是一个高度保守的ATP依赖的核酸内切酶,隶属于RNaseⅢ家族。在RNAi途径中,siRNA和miRNA的生成依赖Dicer的剪切活性。Dicer的分子量在200 kD左右,具有4个特征结构域,从N端到C端依次为DEXD/H解旋酶结构域、PAZ结构域、两个串联的RNaseⅢ(RNaseⅢa和RNaseⅢb)结构域及dsRBD结构域,其中PAZ结构域有助于识别底物,dsRBD结构域结合底物,DEXD/H解旋酶结构域使底物解螺旋,RNaseⅢ结构域则参与了底物的剪切。Dicer的作用底物为dsRNA,为了剪切dsRNA的两条链,需要两个Dicer共同起作用,形成二聚体。Dicer二聚体中含有4个复合的剪切活性中心(RNaseⅢ活性中心),但在剪切过程中,仅两个活性中心发挥作用,另外两个活性中心失活,因为两个活性中心间隔22 nt,因此Dicer剪切产生的siRNA和miRNA的长度为22 nt。然而,Dicer结构的微小改变会导致剪切活性中心间隔的变化,从而使得剪切产物的长度略有差异[1, 29, 47]。
2.2 Argonaute蛋白家族Argonaute是RNAi过程中的核心组分,siRNA、miRNA或piRNA锚定在Argonaute上引导Argonaute作用于靶基因,从而引发靶基因沉默。Argonaute蛋白家族包括AGO和PIWI两类亚家族。AGO亚家族在各组织中均表达,它与siRNA或miRNA结合,主要通过影响mRNA的稳定或抑制翻译在转录后水平调控基因表达。PIWI亚家族主要在动物的生殖细胞中表达,它与piRNA一起作用诱导转座子沉默[48-50]。Argonaute的分子量约为100 kD,具有PAZ和PIWI两个特征结构域。其中,PAZ结构域与Dicer共享,能直接契合Dicer剪切形成的RNA 3′端突出2 nt;PIWI结构域具有类似RNase H的催化核心结构,能对靶mRNA进行剪切,但在进化过程中,部分Argonaute家族成员由于PIWI结构域中的氨基酸序列发生变化而使其丧失了原有的核酸内切酶活性[21, 51, 52]。在人类的4个AGO蛋白(AGO1-4)中,仅AGO2具有剪切活性,但在果蝇中,所有的AGO蛋白和PIWI蛋白均具有剪切活性。除了PIWI结构域介导的剪切,Argonaute还可与其他蛋白因子相互作用,通过诱导翻译抑制、mRNA降解、异染色质形成等方式沉默基因[30, 53]。
2.3 RdRPRdRP是RNAi信号扩增的核心分子。它可以RISC释放出的正义siRNA为引物,靶mRNA为模板,通过“下降PCR”的方式合成dsRNA;还可通过非引物依赖的方式,以一些异常的单链mRNA为模板合成dsRNA。这些dsRNA被加工成新的siRNA进入RNAi循环,如此反复从而使RNAi效应放大。参与RNAi的RdRP同系物存在于真菌、线虫和植物中,目前还未在果蝇和哺乳动物体内发现RdRP同系物,其体内缺失的参与RNAi的RdRP同系物可由其他RdRP替代,如一些病毒RdRP的水平转移。实际上,有证据表明果蝇和脊椎动物中存在RdRP类似物,但是这些物质是否参与及如何参与RNAi过程仍有待研究[1, 29, 54]。
3 RNAi的应用 3.1 在基因功能研究中的应用随着分子生物学的发展,人们完成了众多生物的全基因组测序工作,对这些全基因组序列进行基因功能分析相当重要。通过RNAi特异沉默基因表达,比较基因沉默前后生物表型的变化,可以阐明基因的功能。与传统的基因敲除手段相比,RNAi技术具有快速、高效、特异性强、操作简易等优点,已成为研究基因功能的强有力工具,在后基因组时代中发挥重要作用[55]。
Kimber等[56]利用RNAi技术沉默马铃薯白线虫(Globodera pallida)的FMRF酰胺类多肽(FMRFamide-like peptides,FLP)基因发现,马铃薯白线虫的运动功能受到了阻碍,表明flp对马铃薯白线虫的正常运动功能是必须的。Zhang等[57]鉴定了东亚飞蝗(Locusta migratoria manilensis)几丁质合成酶1基因(Chitin synthase 1 gene,LmCHS1)的2个可变剪接体LmCHS1A和LmCHS1B,并利用RNAi技术对它们的功能进行了分析,结果显示注射LmCHS1、LmCHS1A和LmCHS1B dsRNA的幼虫死亡率分别为95%、88%和51%,其中,注射LmCHS1A和LmCHS1 dsRNA的幼虫表型相似,均表现为发育迟缓,蜕皮过程中旧角质层无法脱离,蜕皮之后新的角质层薄,后腿弯曲,无法行走,身体扭曲,而注射LmCHS1B dsRNA的幼虫表皮褶皱,无法完成蜕皮,以上结果表明,LmCHS1的可变剪接体在东亚飞蝗的生长和发育过程中均发挥重要作用。Dong等[58]克隆了番茄(Solanum lycopersicum)的MADS-box基因SlMADS1,为了进一步探讨SlMADS1的功能,他们利用RNAi技术沉默该基因发现,SlMADS1沉默的番茄果实成熟时间缩短,参与果实成熟的乙烯合成基因和乙烯应答基因表达水平均上调,乙烯产量比野生型增加近2-4倍,这些结果提示SlMADS1是番茄果实成熟的重要负调控因子。
3.2 在信号传导通路研究中的应用信号传导是一个十分复杂的过程,传统的缺失突变技术和RNAi技术相结合可以方便确定信号传导通路中各个基因的上下游关系[59]。Clemens等[60]用dsRNA干扰果蝇细胞系来研究胰岛素信号传导通路,得到的结果与已知胰岛素信号传导通路完全一致,表明RNAi技术可用于分析复杂的信号传导通路。转染是研究信号传导通路的传统方法,但该方法具有重组蛋白非生理聚集及转染效率不高的缺点,RNAi技术克服上述缺点,并且具有简单、快速、重复性好等优势,是研究信号传导通路的有力手段[59]。
3.3 在疾病治疗中的应用RNAi技术能高效、特异地下调靶基因的表达,是一种十分有前景的疾病治疗方法。与其他治疗方法相比,RNAi治疗效果持续时间长,使得治疗费用降低,且治疗所需的sncRNA剂量小,降低或消除了不良反应的发生。目前,基于RNAi的治疗研究主要针对癌症、病毒感染和眼部疾病等[10]。
polo样激酶1(polo-like kinase 1,PLK1)和纺锤体驱动蛋白(Kinesin spindle protein,KSP)是必要的细胞周期蛋白,与癌症的发生密切相关。抑制PLK1和KSP的活性将诱导有丝分裂阻滞和癌细胞凋亡,因此,PLK1和KSP可以作为癌症治疗的靶点。研究者对靶向PLK1和KSP的siRNA进行化学修饰,并将修饰的siRNA通过静脉注射到小鼠肿瘤模型中,结果显示,siRNA有效沉默了靶基因,抑制了PLK1和KSP的生物学活性,引起广泛的有丝分裂受阻和肿瘤细胞凋亡,从而有力抵抗了癌症[61]。人类细小病毒B19(Human parvovirus B19,B19V)可引发急性和慢性心肌炎,并伴随内皮功能紊乱,目前还没有治疗B19V感染的有效方法。Brandt等[62]将靶向B19V结构蛋白VP2基因的短发卡RNA(Short hairpin RNA,shRNA)经腺病毒载体介导进入感染了B19V的UT7/Epo-S1细胞中,结果显示,VP2的mRNA水平显著降低,B19V的复制受到明显抑制,提示靶向结构B19-VP2基因的RNAi技术可用于治疗B19V感染。艾滋病是一种严重威胁人类生命安全的传染病,由人类免疫缺陷病毒(Human immunodeficiency virus,HIV)引起。HIV主要攻击人体T淋巴细胞,破坏免疫系统,从而使人体感染各种疾病,最终导致艾滋病。高效抗逆转录病毒疗法(Highly active antiretroviral therapy,HAART)是治疗艾滋病的常用方法,但该方法具有耐药性等缺陷,因此仍然需要寻找更好的治疗手段[63]。将靶向HIV-1 Nef的siRNA导入人类T细胞中,有效抑制了细胞中HIV-1的复制,表明RNAi介导的基因疗法是治疗艾滋病的潜在手段,值得深入研究[64, 65]。眼部因其具有局限的密闭空间,便于局部用药,因此成为RNAi治疗的理想靶器官[66]。整合素连接激酶(Integrin linked kinase,ILK)具有调节细胞增生、分化、迁移和凋亡的作用。Zheng等[67]将靶向ILK的siRNA通过慢病毒载体介导进入视网膜Müller细胞后,细胞中ILK的mRNA和蛋白水平均显著下降,α-平滑肌肌动蛋白应力纤维减少,细胞迁移和细胞介导的凝胶收缩明显受到抑制,这提示RNAi可以作为与Müller细胞分化相关的眼部纤维增生性疾病的治疗新方法。
4 结语RNAi技术具有高效、简捷、特异性强等优点,是研究基因功能、信号传导通路和治疗疾病的有力工具,但目前仍存在一些无法完满解决的问题,如RNAi的详尽作用机制,包括RISC的具体组分及各组分的结构和功能、piRNA初级加工过程所需因子、决定piRNA 3′端结构的核酸酶等都尚未清楚,以及RNAi应用过程中存在的脱靶效应[68]。相信随着研究的不断深入,这些问题将逐渐得到解决,RNAi技术将更好地为人们所用。
| [1] | Nejepinska J, Flemr M, Svoboda P. The Canonical RNA Interference Pathway in Animals[M]// Mallick B, Ghosh Z. Regulatory RNAs-Basics, Methods and Applications. Berlin:Springer, 2012:111-149. |
| [2] | 崔喜艳, 孙小杰, 刘忠野, 等. RNAi机制及在植物中应用的研究概述. 吉林农业大学学报, 2013, 35(2): 160–166. |
| [3] | Czech B, Hannon GJ. Small RNA sorting:matchmaking for Argonautes. Nature Reviews Genetics, 2010, 12 (1): 19–31. |
| [4] | Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391 (6669): 806–811. DOI:10.1038/35888 |
| [5] | Brantl S. Antisense-RNA regulation and RNA interference. Biochimica et Biophysica Acta, 2002, 1575 (1-3): 15–25. DOI:10.1016/S0167-4781(02)00280-4 |
| [6] | Cerutti H, Casas-Mollano JA. On the origin and functions of RNA-mediated silencing:from protists to man. Current Genetics, 2006, 50 (2): 81–99. DOI:10.1007/s00294-006-0078-x |
| [7] | Torres-Martínez S, Ruiz-Vázquez RM. RNAi pathways in Mucor:A tale of proteins, small RNAs and functional diversity. Fungal Genetics and Biology, 2015, 90 : 44–52. |
| [8] | 李绍祥, 郭汉斌, 曹建彪. RNA干扰的途径和机制. 昆明医科大学学报, 2012, 33(11): 148–151. DOI:10.3969/j.issn.1003-4706.2012.11.042 |
| [9] | Jinek M, Doudna JA. A three-dimensional view of the molecular machinery of RNA interference. Nature, 2009, 457 (7228): 405–412. DOI:10.1038/nature07755 |
| [10] | Pereira P, Queiroz JA, Figueiras A, et al. Affinity approaches in RNAi-based therapeutics purification. Journal of Chromatography B, 2016, 1021 (10): 45–56. |
| [11] | 哀建国, 杨勇. RNA介导的植物基因沉默作用及其应用. 浙江林学院学报, 2005, 22(1): 123–128. |
| [12] | Ramachandran PV, Ignacimuthu S. RNA Interference—A Silent but an Efficient Therapeutic Tool. Applied Biochemistry and Biotechnology, 2013, 169 (6): 1774–1789. DOI:10.1007/s12010-013-0098-1 |
| [13] | Nykanen A, Haley B, Zamore PD. ATP requirements and small interfering RNA structure in the RNA interference pathway. Cell, 2001, 107 (3): 309–321. DOI:10.1016/S0092-8674(01)00547-5 |
| [14] | Schaffer AE, Eggens VRC, Caglayan AO, et al. CLP1 founder mutation links tRNA splicing and maturation to cerebellar development and neurodegeneration. Cell, 2014, 157 (3): 651–663. DOI:10.1016/j.cell.2014.03.049 |
| [15] | Ku SH, Jo SD, Lee YK, et al. Chemical and structural modifications of RNAi therapeutics. Advanced Drug Delivery Reviews, 2015, 78 (3): 250–258. |
| [16] | Hannon GJ. RNA interference. Nature, 2002, 418 (6894): 244–251. DOI:10.1038/418244a |
| [17] | Gish RG, Yuen M, Chan HLY, et al. Synthetic RNAi triggers and their use in chronic hepatitis B therapies with curative intent. Antiviral Research, 2015, 121 : 97–108. DOI:10.1016/j.antiviral.2015.06.019 |
| [18] | Liu J, Carmell MA, Rivas FV, et al. Argonaute2 is the catalytic engine of mammalian RNAi. Science, 2004, 305 (5689): 1437–1441. DOI:10.1126/science.1102513 |
| [19] | Haley B, Zamore PD. Kinetic analysis of the RNAi enzyme comp-lex. Nature Structural & Molecular Biology, 2004, 11 (7): 599–606. |
| [20] | Liu Y, Tan H, Tian H, et al. Autoantigen La promotes efficient RNAi, antiviral response, and transposon silencing by facilitating multiple-turnover RISC catalysis. Molecular Cell, 2011, 44 (3): 502–508. DOI:10.1016/j.molcel.2011.09.011 |
| [21] | 赵雅, 吴立刚. 基因表达调控多面手——microRNA和siRNA的作用机制. 生命科学, 2010, 22(7): 628–633. |
| [22] | Petersen CP, Bordeleau M, Pelletier J, et al. Short RNAs repress translation after initiation in mammalian cells. Molecular Cell, 2006, 21 (4): 533–542. DOI:10.1016/j.molcel.2006.01.031 |
| [23] | Wu L, Fan J, Belasco JG. Importance of translation and nonnucleolytic ago proteins for on-target RNA interference. Current Biology, 2008, 18 (17): 1327–1332. DOI:10.1016/j.cub.2008.07.072 |
| [24] | Kim DH, Villeneuve LM, Morris KV, et al. Argonaute-1 directs siRNA-mediated transcriptional gene silencing in human cells. Nature Structural & Molecular Biology, 2006, 13 (9): 793–797. |
| [25] | 史毅, 金由辛. RNA干扰与siRNA (小干扰RNA) 研究进展. 生命科学, 2008, 20(2): 196–201. |
| [26] | Morris KV, Chan SW, Jacobsen SE, et al. Small interfering RNA-induced transcriptional gene silencing in human cells. Science, 2004, 305 (5688): 1289–1292. DOI:10.1126/science.1101372 |
| [27] | Sherman JH, Munyikwa T, Chan SY, et al. RNAi technologies in agricultural biotechnology:The Toxicology Forum 40th Annual Summer Meeting. Regulatory Toxicology and Pharmacology, 2015, 73 (2): 671–680. DOI:10.1016/j.yrtph.2015.09.001 |
| [28] | Zhang C, Ruvkun G. New insights into siRNA amplification and RNAi. RNA Biology, 2012, 9 (8): 1045–1049. DOI:10.4161/rna.21246 |
| [29] | 和琼姬, 燕飞, 陈剑平. RNA干扰机制及其主要蛋白因子研究进展. 浙江农业学报, 2011, 23(2): 415–420. |
| [30] | Kim VN, Han J, Siomi MC. Biogenesis of small RNAs in animals. Nature Reviews Molecular Cell Biology, 2009, 10 (2): 126–139. DOI:10.1038/nrm2632 |
| [31] | 何苗, 王子卫. miRNA与siRNA胃癌相关研究的现状及进展. 遗传, 2011, 33(9): 925–930. |
| [32] | 唐彬, 肖斌, 邹全明, 等. Mirtron——miRNA合成的新途径. 生命的化学, 2009, 29(3): 315–317. |
| [33] | Yang J, Lai EC. Alternative miRNA biogenesis pathways and the interpretation of core miRNA pathway mutants. Molecular Cell, 2011, 43 (6): 892–903. DOI:10.1016/j.molcel.2011.07.024 |
| [34] | Sarett SM, Nelson CE, Duvall CL. Technologies for controlled, local delivery of siRNA. Journal of Controlled Release, 2015, 218 : 94–113. DOI:10.1016/j.jconrel.2015.09.066 |
| [35] | 张超, 庞全海. siRNA与miRNA在生物体基因调控中沉默机制的比较. 中国生物化学与分子生物学报, 2012, 28(5): 393–398. |
| [36] | Huntzinger E, Izaurralde E. Gene silencing by microRNAs:contributions of translational repression and mRNA decay. Nature Reviews Genetics, 2011, 12 (2): 99–110. DOI:10.1038/nrg2936 |
| [37] | Humphreys DT, Westman BJ, Martin DI, et al. MicroRNAs control translation initiation by inhibiting eukaryotic initiation factor 4E/cap and poly (A) tail function. Proceedings of the National Academy of Sciences, 2005, 102 (47): 16961–16966. DOI:10.1073/pnas.0506482102 |
| [38] | Piao X, Zhang X, Wu L, et al. CCR4-NOT deadenylates mRNA associated with RNA-induced silencing complexes in human cells. Molecular and Cellular Biology, 2010, 30 (6): 1486–1494. DOI:10.1128/MCB.01481-09 |
| [39] | 吴丹, 胡兰. miRNA及其研究进展. 中国畜牧兽医, 2006, 33(1): 40–42. |
| [40] | Jover-Gil S, Candela H, Ponce MR. Plant microRNAs and development. International Journal of Developmental Biology, 2005, 49 (5-6): 733–744. DOI:10.1387/ijdb.052015sj |
| [41] | 廖珂, 柴志欣, 钟金城. 小分子非编码RNA与雄性不育. 生物技术通报, 2016, 32(1): 33–40. |
| [42] | Aravin AA, Hannon GJ, Julius B. The Piwi-piRNA pathway provides an adaptive defense in the transposon arms race. Science, 2007, 318 (5851): 761–764. DOI:10.1126/science.1146484 |
| [43] | Ketting RF. The Many Faces of RNAi. Developmental Cell, 2011, 20 (2): 148–161. DOI:10.1016/j.devcel.2011.01.012 |
| [44] | Siomi MC, Sato K, Pezic D, et al. PIWI-interacting small RNAs:the vanguard of genome defence. Nature Reviews Molecular Cell Biology, 2011, 12 (4): 246–258. DOI:10.1038/nrm3089 |
| [45] | Nishida KM, Saito K, Mori T, et al. Gene silencing mechanisms mediated by Aubergine-piRNA complexes in Drosophila male gonad. RNA, 2007, 13 (11): 1911–1922. DOI:10.1261/rna.744307 |
| [46] | Brent BT, Findley SD, Ling J, et al. Drosophila PIWI associates with chromatin and interacts directly with HP1a. Genes & Development, 2007, 21 (18): 2300–2311. |
| [47] | 袁婺洲, 吴秀山. RNAi机制研究的最新进展. 生命科学研究, 2003, 7(1): 8–14. |
| [48] | Höck J, Meister G. The Argonaute protein family. Genome Biology, 2008, 9 (2): 1–8. |
| [49] | Bak CW, Yoon TK, Choi Y. Functions of PIWI proteins in spermatogenesis. Clinical & Experimental Reproductive Medicine, 2011, 38 (2): 61–67. |
| [50] | Seto AG, Kingston RE, Lau NC. The Coming of Age for Piwi Proteins. Molecular Cell, 2007, 26 (5): 603–609. DOI:10.1016/j.molcel.2007.05.021 |
| [51] | Zhou X, Liao Z, Jia Q, et al. Identification and characterization of Piwi subfamily in insects. Biochemical & Biophysical Research Communications, 2007, 362 (1): 126–131. |
| [52] | Pak J, Maniar JM, Mello CC, et al. Protection from Feed-Forward Amplification in an Amplified RNAi Mechanism. Cell, 2012, 151 (4): 885–899. DOI:10.1016/j.cell.2012.10.022 |
| [53] | Witold F, Bhattacharyya SN, Nahum S. Mechanisms of post-transcriptional regulation by microRNAs:are the answers in sight?. Nature Reviews Genetics, 2008, 9 (2): 102–114. |
| [54] | Concetta L, Paterson BM. Identification of an RNA-dependent RNA polymerase in Drosophila involved in RNAi and transposon suppression. Proceedings of the National Academy of Sciences, 2009, 106 (37): 15645–15650. DOI:10.1073/pnas.0904984106 |
| [55] | 马丽, 张春庆. RNA干扰机制及应用研究进展. 北方园艺, 2012(10): 191–193. |
| [56] | Kimber MJ, Mckinney S, Mcmaster S, et al. flp gene disruption in a parasitic nematode reveals motor dysfunction and unusual neuronal sensitivity to RNA interference. Faseb Journal, 2007, 21 (4): 1233–1243. DOI:10.1096/fj.06-7343com |
| [57] | Zhang JZ, Liu XJ, Zhang JQ, et al. Silencing of two alternative splicing-derived mRNA variants of chitin synthase 1 gene by RNAi is lethal to the oriental migratory locust, Locusta migratoria manilensis(Meyen). Insect Biochemistry & Molecular Biology, 2010, 40 (11): 824–833. |
| [58] | Dong TT, Hu ZL, Deng L, et al. A tomato MADS-box transcription factor, SlMADS1, acts as a negative regulator of fruit ripening. Plant Physiology, 2013, 163 (2): 1026–1036. DOI:10.1104/pp.113.224436 |
| [59] | 李方华, 侯玲玲, 苏晓华, 等. RNA干扰的研究进展及应用. 生物技术通讯, 2010, 21(5): 740–745. |
| [60] | Clemens JC, Worby CA, Simonson-Leff N, et al. Use of double-stranded RNA interference in Drosophila cell lines to dissect signal transduction pathways. Proceedings of the National Academy of Sciences, 2000, 97 (12): 6499–6503. DOI:10.1073/pnas.110149597 |
| [61] | Judge AD, Marjorie R, Iran T, et al. Confirming the RNAi-mediated mechanism of action of siRNA-based cancer therapeutics in mice. Journal of Clinical Investigation, 2009, 119 (3): 661–673. DOI:10.1172/JCI37515 |
| [62] | Brandt MRG, Kirste AG, Pozzuto T, et al. Adenovirus vector-mediated RNA interference for the inhibition of human parvovirus B19 replication. Virus Research, 2013, 176 (1-2): 155–160. DOI:10.1016/j.virusres.2013.05.020 |
| [63] | 王健, 邹雯. 中医药治疗艾滋病的研究进展. 中国艾滋病性病, 2014, 20(10): 61–63. |
| [64] | Brake OT, Legrand N, Eije KJV, et al. Evaluation of safety and efficacy of RNAi against HIV-1 in the human immune system (Rag-2-/-γc-/-) mouse model. Gene Therapy, 2008, 16 (1): 148–153. |
| [65] | Das AT, Brummelkamp TR, Westerhout EM, et al. Human immunodeficiency virus type 1 escapes from RNA interference-mediated inhibition. Journal of Virology, 2004, 78 (5): 2601–2605. DOI:10.1128/JVI.78.5.2601-2605.2004 |
| [66] | 孔怡淳, 赵堪兴. RNA干涉技术及眼科应用. 中华实验眼科杂志, 2014, 32(4): 370–374. |
| [67] | Zheng YP, Liu H, Zeng H, et al. Downregulation of lentivirus-mediated ILK RNAi on tractional force generation in human retinal Müller cells. Acta Pharmacologica Sinica, 2009, 30 (12): 1625–1633. DOI:10.1038/aps.2009.154 |
| [68] | 尚仁福, 吴立刚. RNA干扰的机制及其应用. 生命科学, 2016, 28(5): 576–583. |