蛋白质作为基因功能执行者,有必要通过蛋白质的翻译后修饰(post-translational modification,PTM)形成结构更复杂、功能更多样的能够更精准更专一发挥作用的蛋白质。PTM就是通过给一个或多个氨基酸残基加上修饰基团对翻译后的蛋白质进行共价加工的过程,改变蛋白质的理化性质,影响蛋白质的空间构象和活性状态,亚细胞定位、折叠及其稳定性以及蛋白质互作[1, 2]。常见的PTM有磷酸化修饰、甲基化修饰和泛素化修饰等。蛋白质通过磷酸化与去磷酸化,调控信号转导、基因表达和细胞周期等诸多生命过程,是生物体内最重要的共价修饰方式之一。人类超过50%的蛋白质发生过磷酸化修饰,有100 000个潜在的磷酸化位点[3]。组蛋白的甲基化修饰既可抑制也可增强基因表达,是重要的表观遗传学调控方式[4]。泛素化是细胞蛋白质在发挥降解功能中非常重要的翻译后修饰,重要性亦堪比磷酸化。这类修饰在动物和人体的研究较多且进展较快。
活性氧(reactive oxygen species,ROS)是化学性质活泼、氧化能力很强的含氧代谢产物,在植物体内具有双重功能,即可通过氧化生物大分子等方式损害植物正常的生命活动,也可发挥如信号传递等生理功能[5]。随着对ROS作为信号分子研究的深入发现,其可在植物细胞内扩散,作为第二信使介导多种信号通路,与蛋白巯基状态、谷胱甘肽(glutathione,GSH)、多种抗氧化酶类和激素及转录因子等相关,对激素或者环境胁迫做出多种应答[5]。ROS对蛋白质的这种翻译后修饰,可以通过氧化产生羰基化蛋白等不可逆氧化修饰破坏蛋白结构进而危害植物体的正常生命活动,也可以通过自身的调节机制来适应这种氧化胁迫,特别是通过蛋白质的可逆氧化修饰实现适应。可见植物在环境适应性进化中通过ROS促进蛋白质氧化还原修饰,达到既有效降低氧化胁迫,又保护蛋白的目的。但是目前对植物蛋白质氧化修饰的研究还处于初始阶段,有关具体修饰位点的研究也不多见。鉴于蛋白质的PTM,特别是可逆氧化修饰对逆境下植物生物功能的正常发挥乃至适应和存活都至关重要,ROS调节蛋白质的可逆氧化修饰研究越来越受到人们的重视,在机理和研究手段上都取得了一定进展。各种常见的ROS有H2O2、O2-和1O2、·OH等,它们的半衰期分别以毫秒、微秒、纳秒来计量,且O2-、1O2和·OH的氧化活性强于H2O2[6],所以H2O2相对更为稳定;相较于另外3种ROS,H2O2可穿梭过膜,其作用范围更广。这些ROS都可对氨基酸进行氧化修饰,但鉴于H2O2的稳定性和可跨膜性,目前植物蛋白质氧化修饰的研究主要集中在H2O2造成的氧化修饰上[7-10]。有人以野生型拟南芥为对照组,过氧化氢酶(catalase,CAT)突变型为胁迫处理组,利用蛋白质组对植物甲硫氨酸亚砜(met sulfoxide,Met-SO)的动态进行观察进而分析甲硫氨酸(methionine,Met)的氧化程度[11]。本文根据近年来国内外主要相关研究进展,就ROS对植物蛋白质氧化修饰的方式位点以及可逆氧化修饰的调节机理及对植物抗逆的重要意义进行综述,着重探讨ROS对植物蛋白质可逆氧化修饰的机理、在植物信号转导中的作用及研究方法。
1 植物蛋白质氧化修饰的方式和位点 1.1 ROS对植物蛋白质氧化修饰的信号调节机理正常情况下植物细胞与胞外环境相比,细胞内环境主要靠GSH和硫氧还蛋白(thioredoxin,Trx)的半胱氨酸(cysteine,Cys)构成巯基氧化还原反应系统将其维持在强还原状态[12],不会受到氧化胁迫。而且植物也会利用酶促与非酶促的抗氧化系统[13]来清除一定量的ROS,但在干旱等胁迫条件下过量ROS的产生,使植物细胞处于氧化胁迫状态,很多生物分子如糖类、不饱和脂类、蛋白质和核酸等都成为ROS攻击的目标,引发膜脂过氧化、蛋白质氧化、核酸损伤和酶失活,甚至导致许多生物大分子尤其是蛋白质被氧化而失去功能,并激活程序性细胞死亡。相较于脱氧核糖核酸(deoxyribonucleic acid,DNA)和脂类,蛋白质的氧化更普遍,占68%[14],特别是含硫氨基酸如Met与Cys残基最容易遭到破坏而导致蛋白质相关属性变化和功能失常,可能都是因这两种氨基酸残基被氧化所致。
ROS和活性氮(reactive nitrogen species,RNS)促进可逆的氧化还原PTM的发生,包括Cys、酪氨酸(tyrosine,Tyr)的亚硝基化,Cys、Met的氧化[15, 16]。这种ROS对蛋白质进行的可逆氧化修饰,既保护了蛋白质不受损伤,在条件合适时继续发挥生理功能;又起到抗氧化剂的作用,降低了ROS对机体的进一步损伤[17]。
1.2 ROS对蛋白质氧化修饰的方式和位点蛋白质分子中起关键作用的氨基酸成分对ROS损害特别敏感,因而ROS以多种方式直接或间接地对蛋白质进行攻击从而导致蛋白质修饰。直接修饰包括氨基酸残基的氧化、含硫基团的氧化和二硫键形成以及谷胱甘肽化等;间接修饰主要是指蛋白与脂肪酸过氧化裂解产物结合形成羰基化蛋白。不同的ROS对特定氨基酸侧链有特殊影响,以芳香氨基酸和含硫氨基酸最为突出。大多数蛋白质的氧化作用是不可逆的,经常与蛋白质功能的永久丧失相关,可以导致损害蛋白质的消除或积累;只有一些含硫氨基酸的蛋白质氧化是可逆的[5],如Cys、Met的氧化,可以起到保护Cys避免发生不可逆氧化修饰和调节蛋白功能的双重作用[18]。目前ROS对植物蛋白质氨基酸可逆氧化修饰的研究主要集中在Met和Cys上,本文参考相关文献并梳理了ROS对Cys和Met的氧化修饰,如图 1[19]。除此之外,Cys的亚硝基化和谷胱甘肽化也是常见的氧化修饰方式。

|
| 图 1 ROS对甲硫氨酸和半胱氨酸的可逆氧化修饰 |
芳香族氨基酸是最容易被氧化损伤的氨基酸[20],如Tyr很容易发生苯环间交联形成二酪氨基酸或其酚环的3位上加1个硝基(-NO2)形成硝化氨基酸,而被作为蛋白质氧化损伤的指标[21];ROS对色氨酸(tryptophan,Trp)残基的氧化修饰是不可逆的非随机过程[14],近年来也有文献说明Trp的氧化修饰是可逆的[16],所以厘清其中的抗氧化机制非常重要。芳香族氨基酸(Trp和Tyr)被氧化后,疏水性发生改变,进而影响蛋白质的结构;Tyr磷酸化在信号转导上起着重要的作用;在ROS参与下,Tyr很容易发生苯环间交联而形成二酪氨基酸,可被作为蛋白质氧化损伤的指标[21]。芳香族氨基酸的氧化产物在酸水解环境中也很稳定,通常作为蛋白质氧化的生物标志物,如Moller以Trp作为蛋白质标志物研究了蛋白质组的氧化[14]。Tyr氧化修饰可以作为胁迫条件下的生物标志,Aulak等[22]和Kanski等[23]采用基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/Ionization time of flight mass spectrometry,MALDI-TOF-MS)、十二烷基磺酸钠-聚丙烯酰氨凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)和串联液相色谱四级杆质谱联用(liquid chromatography-liquid chromatography quadrupole,LC-LCQ)等蛋白质组学技术对硝化Tyr进行了研究。Tyr的硝基化氧化修饰和Met残基可逆氧化修饰一样具有抗氧化剂的功能。
1.2.2 ROS对某些蛋白质的不可逆羰基化修饰蛋白质的羰基化修饰主要包括特定位点氨基酸的修饰、肽链断裂、交联和聚合、电荷改变以及对蛋白水解作用的敏感度增加,从而使蛋白结构改变、功能失常和降解。羰基化的主要方式有:ROS介导的赖氨酸(lysine,Lys)、脯氨酸(proline,Pro)、精氨酸(arginine,Arg)和苏氨酸(threonine,Thr)残基的氧化[16];脂质过氧化裂解产物(主要为活性醛类如4-羟基烯醛)与蛋白质氨基酸残基(组氨酸(histid-ine,His)、Cys、Lys)的侧链基团发生米歇尔加成(michaeI addition)和/或希夫碱(schiff base)反应[5];Lys等的糖基化[24]。由于蛋白质羰基化不能被细胞中的酶系统修复,这些作用对蛋白质造成的损伤也是一个不可逆过程。环境胁迫下植物组织羰基化蛋白含量增多,已被广泛用作检测蛋白质氧化损伤的标志。
1.2.3 ROS对Met的可逆氧化修饰 1.2.3.1 ROS对Met可逆氧化修饰过程Met是生物体必需氨基酸之一,无论游离态还是暴露在蛋白质表面的Met都极易被ROS氧化形成Met-SO、更强的氧化剂还可将Met-SO进一步氧化成甲硫氨酸砜(methionine sulfone,Met-SO2)导致氨基酸结构改变和蛋白质功能受损[25]。Met-SO可以被生物体内Met亚砜还原酶(methionine sulfoxide reductase,Msr)家族逆转[26],还原亚砜结构并恢复受损蛋白质的活性,以抵御逆境造成的各种氧化胁迫[27],保证机体的正常运转;然而Met-SO2不能被Msr系统还原,属于不可逆的氧化修饰。
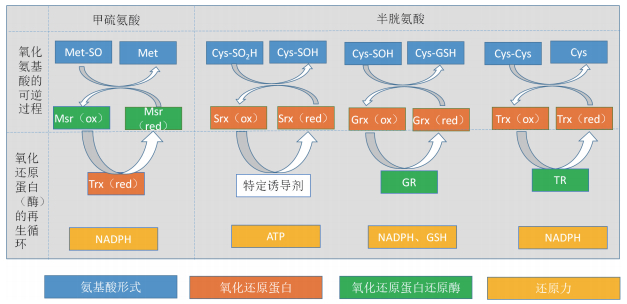
1.2.3.2 Msr在Met可逆氧化修饰中的作用根据多篇文献总结并绘制Msr在蛋白质可逆氧化修饰过程中的作用机制如图 2[17, 25, 28]。Msr参与被氧化破坏的蛋白质或多肽中Met的修复过程[26, 28],可以使氧化的Met-SO还原为Met,进而恢复蛋白质的天然构象与功能,为该还原过程提供还原力的主要为Trx[29],其次是谷氧还蛋白(glutaredoxin,Grx)以及抗坏血酸-谷胱甘肽循环(ascorbate-glutathione cycle,AsA-GSH cycle)系统中的GSH或还原型辅酶Ⅱ(nicotinamide adenine dinucleotide phosphate,NADPH)等[17]。Met-SO具有手性,以对映异构体(Met-S-SO与Met-R-SO)形式混合存在。Msr的催化过程需要Cys残基的参与,当用丝氨酸(serine,Ser)取代其上的Cys时,Msr的活性完全被抑制,但是当Cys上的硫元素被硒元素取代时可以保持Msr的酶活性[30-32]。Msr有MsrA和MsrB两种类型,它们催化不同异构体的Met-SO还原为Met,从而恢复蛋白质的天然构象与功能[27]。Msr催化游离及多肽链中Met-SO的还原类型有3种,分别为:1-Cys-Msr、2-Cys-Msr、3-Cys-Msr[28],其中以2-Cys-Msr的应用最为普遍。2-Cys-Msr含有两个Cys残基,其中一个为催化Cys残基;另一个为循环Cys残基。2-Cys-Msr的催化机制具体如下:首先,催化Cys残基对Met-SO的硫原子进行亲核攻击,产生Msr-MetSO复合体;其次,由Msr上的循环Cys残基对此复合物的硫原子进行亲核攻击,产生含分子内二硫键的Msr、Met和水分子;最后,由还原剂Trx实现Msr的还原再生,整个循环结束。
|
| 图 2 Trx、Srx、Msr等在蛋白质可逆氧化修饰过程中发挥作用的机理及再生机制 Met-SO:Met-sulfoxide,甲硫氨酸亚砜;Met:Methionine,甲硫氨酸;Msr:Methionine sulfoxide reductase,甲硫氨酸亚砜还原酶;Trx:Thioredoxin,硫氧还蛋白;NADPH:Nicotinamide adenine dinucleotide phosphate;还原型辅酶Ⅱ;Cys-SO2H:Cys-sulfinic acid,半胱氨酸亚磺酸;Cys-SOH:Cys-sulfenic acid,半胱氨酸次磺酸;Srx:Sulfiredoxin,硫氧还蛋白;ATP:Adenosine triphosphate;腺苷三磷酸;GSH:glutathione,谷胱甘肽;Grx:Glutaredoxin,谷氧还蛋白;GR:Glutaredoxin reductase,谷氧还蛋白还原酶;GSH:Glutathione,谷胱甘肽;TR:Thioredoxin reductase,硫氧还蛋白还原酶 |
Stam-ler[33]首先提出了蛋白质巯基亚硝基化修饰,认为一氧化氮(nitric oxide,NO)作用于蛋白质疏水区的Cys巯基使之由-SH变成-SNO。根据作用于巯基的程度又可将亚硝基化分为两种:一种是蛋白质巯基被NO直接亚硝基化作用;另一种是由低分子质量巯基亚硝基化化合物向蛋白质的转亚硝基化作用[34]。作用位点多在蛋白质Cys上,又分单亚硝基化修饰和多亚硝基化修饰[35-37]。此种修饰包括亚硝基化和去亚硝基化,是一种可逆修饰。蛋白质巯基亚硝基化产物一旦形成后比较稳定,能活化钙离子通道[38]。
1.2.4.2 ROS对Cys的谷胱甘肽化修饰谷胱甘肽化(glutathionylation)即GSH在谷氧还蛋白酶(glutaredoxin protease,GrxP)的催化下与蛋白质的Cys形成二硫键,该过程是可逆的[1]。GSH修饰对酶活性造成的影响可以由还原剂逆转,而氧化作用则会导致氧化还原酶的不可逆失活[39]。目前可以采用与S-亚硝酰化相似的分析方法,在生物素标记蛋白富集前对蛋白进行酶解,富集谷胱甘肽化肽段,从而鉴定到发生修饰的位点[40]。氧化应激过程和正常生理状态下都可发生谷胱甘肽化与去谷胱甘肽化,谷胱甘肽化是由Grx、Trx和蛋白质二硫键异构酶(protein disulfide isomerase,PDI)等催化产生的,其中Grx催化活性及特异性最强[41]。
1.2.4.3 ROS对Cys的可逆氧化修饰及各种氧化还原蛋白的作用Cys的可逆氧化还原作用机制如图 2所示,Cys在氧化条件下可形成二硫键、亚氧硫基(-SOH)、亚硫酸基(-SO2H)、硫酸基(-SO3H),其中硫酸基是Cys的不可逆氧化形式,可能与生物活性的丧失有关;另外3种均为可逆过程,其可逆过程主要依赖3种氧还蛋白:Trx、硫氧还蛋白(sulfiredoxin,Srx)和Grx。值得注意的是,Trx、Grx、Srx的靶目标是氧化修饰后的Cys残基,过氧化物氧化还原酶(peroxiredoxin,Prx)也是Cys氧化修饰经常出现的氧还蛋白[42],虽然其作用底物主要是过氧化物酶,但也有研究发现Prx也可以参与可逆反应,催化Cys-SO2H的可逆氧化[43],因此本文将Prx列入蛋白质中Cys残基可逆氧化修饰的氧化还原蛋白中。Trx与Srx的中文翻译名字相同,均为硫氧还蛋白[44, 45],但就两者依赖的再生机制不同可以看出,Trx是依赖NADPH还原的硫氧还蛋白,Srx是依赖腺苷三磷酸(adenosine triphosphate,ATP)再生的硫氧还蛋白。硫氧还蛋白系统(thioredoxin system,Trxs)和谷氧还蛋白系统(glutaredoxin system,Grxs)是目前已知的胞内负责还原二硫化物的主要系统。Trx和Grx上都含有Cys残基,其上的2个Cys残基可通过转移2个电子和2个质子可逆地形成二硫化合物,使巯基和二硫化合物相互转换,为一系列生理反应提供氧化还原对能可逆地催化许多氧化还原反应。Trxs是由Trx、NADPH和硫氧还蛋白还原酶(thioredoxin reductase,TR)3部分组成的,还原态的Trx通过巯基供氢使其它含有二硫键的蛋白被还原;氧化态的Trx被NADPH还原,继续发挥作用。该系统能够稳定细胞内环境,调节细胞生长及信号传导过程来保护细胞[46]。通常在Trxs中电子传递路径为:NADPH→TR→Trx→含二硫键的靶蛋白。Grxs包括NADPH、GSH、谷胱甘肽还原酶(glutaredoxin reductase,GR)和Grx。Grx系统中,电子传递路径为:NADPH→GR→GSH→Grx→含二硫键的靶蛋白。
Srx是一类多功能酸性蛋白[12],在实现Cys亚磺酸的还原中起着重要的作用,其作用机理主要是:(1)ATP与亚磺酸反应并使亚磺酸磷酸化;(2)Srx与磷酸化产物作用生成中间复合体;(3)利用Trx/GSH等还原上述中间产物,生成次磺酸,Srx的还原是在Srx的特异性诱导剂的作用下实现的;(4)次磺酸通过形成二硫键被Trx/GSH等进一步还原为Cys。其电子传递路径如下式所述:Cys-SO2H(+ATP)→ Cys-SO2-PO32-(+Srx)→ Cys-SO-S-Srx(+Trx/GSH)→ Cys-SOH。
总而言之,目前对这些氧化还原蛋白Trx、Grx、Srx和Prx的研究各有侧重,四者的关系应是各行其道,互相辅助。ROS胁迫下,Prx发挥清除ROS的功能,Srx在还原亚磺酸时需要Trx辅助,Trx与Grx需要共同的还原力NADPH。由此可见,Cys的可逆氧化修饰中各种氧化还原蛋白有两种类型(图 2),一种是直接参与氧化型Cys残基还原过程的氧还蛋白类,如Trx、Grx和Srx等;另一种是间接参与氧化型Cys还原过程的氧还蛋白还原酶类,如GR和TR。
1.2.5 ROS对Cys的不可逆氧化修饰Cys在氧化条件下可形成二硫键、亚氧硫基(-SOH)、亚硫酸基(-SO2H)、硫酸基(-SO3H),其中亚硫酸基和硫酸基是Cys的不可逆氧化形式,可能与生物活性的丧失有关[47]。为了系统地研究细胞的氧化还原调节,Yano等[48]采用硫醇特异的荧光标记,并结合两种类型的双向电泳非还原双向电泳(non-reducing reducing two-dimensional electrophoresis)和等电聚焦十二烷基磺酸钠聚丙烯酰胺凝胶电泳(isoelectric focusing Sodium dodecyl salt-polyacrylamide gel electrophoresis,IEF SDS-PAGE),成功鉴定到花生中受Trx调控的20多个蛋白。Lee等[47]对一种含有二硫键的植物蛋白质进行了研究,采用硫醇亲和色谱方法富集目的蛋白,共鉴定到65个公认的含二硫键的蛋白质。Moan等[49]则采用双标记法对啤酒酵母中的硫醇氧化蛋白进行富集和定量,质谱鉴定到64个蛋白质。这些都是ROS对Cys进行了不可逆氧化修饰的佐证。
2 植物蛋白质可逆氧化修饰的意义和方法 2.1 植物蛋白质可逆氧化修饰提高植物抗逆性植物蛋白质的可逆氧化修饰主要发生在含硫氨基酸(Met、Cys)上,ROS可促进含硫氨基酸残基的氧化,主要依赖Trx、Grx、Prx、Srx、Msr等酶类[26, 46, 50]产生相应的氧化产物。
2.1.1 Met残基的可逆氧化修饰起着抗氧化剂和信号传递的作用ROS胁迫下,Met残基可以直接消耗氧化剂,调节氧化还原反应进行,起抗氧化剂作用[51]。Valley等[52]提出了Met-芳香族氨基酸模型:Met在蛋白质结构组成中位于易于被氧化的芳香族氨基酸如Trp、Tyr和苯丙氨酸(phenylalanine,Phe)等氨基酸的周围,并营造出疏水环境稳定蛋白质的结构,在ROS的胁迫下,可以通过牺牲Met实现对芳香族氨基酸的保护,从而实现其抗氧化的功能。在α2巨球蛋白(α2macroglobulin,A2M)、谷氨酰胺合成酶(glutamine synthetase,GS)[51]和热休克蛋白(heat shock protein,HSP)[53]等蛋白质中证明存在这种结构并发挥抗氧化功能[51]。Msr在Met可逆氧化修饰中起着重要作用,部分Msr基因敲除实验发现Msr在植物抗性方面的表现,进一步证明了Met的抗氧化作用[54, 55]。
ROS胁迫下,Met也可通过与Met-SO的可逆氧化还原过程调节细胞氧化还原状态。Met残基位于Ser/Thr磷酸化位点附近,起着疏水识别作用,它的氧化与还原参与细胞信号转导[56]。Tarafdar等[57]研究证明MsrA可以促进Met与Met-SO之间的可逆氧化还原反应,进而实现细胞氧化还原的调节。Met残基在被氧化后会致使蛋白质相关属性如结构和疏水性等方面发生变化,抑制激酶的识别和磷酸化从而将氧化信号传递到目标蛋白上,调节激酶的活性,从而将氧化胁迫信号与蛋白质磷酸化偶联起来[28, 31, 58]。Met氧化后可以改变钾离子通道的功能。Met残基被氧化后会使钾离子通道的通道蛋白失活,这一过程可以被Msr逆转恢复[59]。Met残基氧化后会使依赖Ca2+/钙调素(calmodulin,CaM)的蛋白激酶2(calcium/calmodulin-dependent protein kinase Ⅱ,CaMKⅡ)的酶活性上调,该活性也可被Msr逆转恢复;CaM与钙调素结合蛋白(calmodulin-binding protein,CaMBP)的亲和力下降[27, 60]。
2.1.2 Cys残基的氧化修饰起着抗氧化和信号传递的作用Met的抗氧化机制即为位于易于氧化的氨基酸周围保护它们,自身的可逆从而实现了对氧化能力的抵抗;Cys的抗氧化机理即为参与可逆氧化氨基酸的还原过程,保护可逆氨基酸的正常可逆,自己对其他可逆氨基酸的恢复过程从而实现对氧化胁迫的抵抗能力。如Met的可逆还原主要依赖Msr的作用,Msr的作用机制需要依赖Cys残基的辅助;Cys的可逆还原依赖Trx、Srx以及Prx的作用,这3种氧还蛋白均含有Cys[42, 44, 61, 62]残基,作用机制同样也需要Cys残基的辅助作用。
Cys残基的可逆氧化修饰在植物中也起着信号传递控制开关的作用和控制蛋白质转录和稳定性的作用[63],丝裂原活化蛋白激酶(mitogen-activated protein kinases,MAPKs)级联信号在植物信号传递中发挥重要的作用,ROS通过氧化蛋白质Tyr磷酸酶(protein tyrosine phosphatases,PTPs)上的Cys残基使其失活,进而将氧化还原信号传递到MAPKs并使其活化[64]。
2.2 基于蛋白质组学的氧化修饰研究方法 2.2.1 一般蛋白质氧化修饰研究方法蛋白质组学通过研究整体蛋白质活动来揭示生命运动规律,是解析功能基因组表达的必要手段[65]。存在的问题主要有两个:即PTM蛋白的富集分离问题和二级质谱的裂解效率问题。目前广泛使用的肽段裂解技术包括碰撞诱导解离(collision induced dissociation,CID)和源后衰变(post-source decay,PSD)两种[1]。蛋白质组学研究技术的主要手段是使用色谱或者凝胶方式进行富集,使用质谱或者凝胶方式进行分析检测。PTM蛋白质具有不均一性及相对丰度低的特性,对PTM蛋白质进行富集分离十分必要。色谱分离的原理有3类:依据分子量差异进行色谱分离;依据蛋白质的亲疏水性进行反相色谱分离富集过程中;依据电荷差异进行色谱分离。一些常见的蛋白质组学研究方法如图 3。一些非蛋白质组学的研究方法也可应用于PTM研究中,图 3列出了3种常用的方法。随着蛋白质组表达谱研究技术的日益成熟和规模化,PTM蛋白质组学研究技术已经取得了可喜的进展。目前蛋白质组学研究平台侧重在磷酸化、泛素化和乙酰化修饰的研究,同时为蛋白质可逆氧化修饰的研究提供了重要的思路和手段,可逆蛋白质氧化修饰方兴未艾。

|
| 图 3 蛋白质研究一般方法与蛋白质组方法的比较 MALDI TOF/TOF:Matrix-assisted laser desorption Ionization-time of flight/ time of flight(基质辅助激光解吸电离串联飞行时间质谱);LC-MS/MS:Liquid chromatography tandem mass spectrometry(液相色谱-串联质谱联用技术);2D-PAGE:Two-dimensional polyacrylamide gel electrophoresis(双向聚丙烯酰胺凝胶电泳);DIGE:Differential in-gel electrophoresis(胶上差示电泳技术);SILAC:Stable isotope labeling with amino acids in cell cultures(细胞培养氨基酸稳定同位素标记);iTRAQ:Isobaric tags for relative for relative and absolute quantification(同位素标记相对和绝对定量技术);ICAT:Isotope coded affinity tags(同位素编码的亲和标签技术);MRM:Multiple reaction monitoring(多重反应监测);SRM:Selective reaction monitor(选择反应监测);ELISA:enzyme linked immunosorbent assay(酶联免疫吸附测定);WB:Western Blot(蛋白免疫印迹) |
考虑生物体的代谢变化常常是由于修饰程度的改变引起的,因而定量研究PTM尤为重要。定量研究的方法主要是基于凝胶的定量蛋白质组学和基于质谱的定量蛋白质组学。基于凝胶的定量蛋白质组学技术的核心在染色上,鉴于双向电泳染全蛋白,染料与蛋白质疏水作用结合和胶上差示电泳技术(differential in-gel electrophoresis,DIGE)荧光染料,只标记Lys的染色原理,不适用于蛋白质可逆氧化修饰的研究。基于质谱的定量蛋白质组学又分为相对定量和绝对定量。相对定量的方法包括用同位素标记肽段氨基酸的同位素标记相对和绝对定量(isobaric tags for relative for relative and absolute quantification,iTRAQ)技术,因对蛋白质的选择性很低,标记所有的氨基末端,前提是将可逆修饰的蛋白质用合适的方法分离出来,再用iTRAQ技术来分析是可行的;同位素编码的亲和标签(isotope coded affinity tags,ICAT)技术也利用同位素标记氨基酸,ICAT技术本身标记的就是Cys,所以可以用来研究可逆氧化修饰蛋白质,但是ICAT技术不能用来研究不可逆的氧化修饰,原因是Cys的大基团特性使ICAT修饰有困难;因此发展出绝对定量的三重四级杆质多重反应监测(multiple reaction monitoring,MRM)技术,用烷基化试剂标记Cys,烷基化修饰蛋白质,还原后再修饰,做质谱,同时结合亲和层析,此法可用于Cys可逆和不可逆氧化修饰的研究中。细胞培养氨基酸稳定同位素标记(stable isotope labeling with amino acids in cell cultures,SILAC)技术采用体内同位素标记,比较依赖细胞的培养,在植物中应用不多;因此进行体内同位素标记的定量蛋白质组学技术,对细胞培养的依赖性很强,不太适宜在植物上应用。由此可知,将一般蛋白质组学研究技术直接应用于可逆氧化修饰蛋白质组学研究上有一定的局限性,应该结合蛋白质组学技术的原理和科研目的,发展出适于Met、Cys等可逆氧化修饰及硝化修饰的蛋白质组学研究技术。
Met氧化生成Met亚砜后分子量增加16 Da,可以据此特性进行色谱分离[65];Met氧化后从疏水性的Met变为亲水性强的Met-SO[66],利用此特性可使用对角线色谱法(combined fractional diagonal chromatography,COFRADIC)对氧化型氨基酸残基进行富集[67];利用Msr对Met亚砜的亲和性也可达到对Met可逆氧化修饰的研究目的[67]。由于不同研究对象其氨基酸特性不同,有时需要进行一定的标记或者修饰便于目标蛋白的富集。半胱氨酸的蛋白质组学研究技术即是如此。目前应用蛋白质组学技术鉴定半胱氨酸残基的可逆氧化修饰时,MRM方法应用的最多,常使用N-乙酰基马来酰亚胺(N-acetyl-Maleimide,NEM)等烷化剂对自由巯基进行保护,再利用还原剂如二硫苏糖醇(dithiothreitol,DTT)等对氧化型半胱氨酸残基进行还原,以及利用荧光试剂等便于富集分离,再结合质谱检测手段进行鉴定及定量[47-49, 68]。Yano等[48]采用硫醇特异的荧光标记,并结合两种类型的双向电泳(non-reducing/reducing 2-DE和IEF/SDS-PAGE),成功鉴定到花生中受硫氧还蛋白调控的20多个蛋白[1, 48]。Lee等[47]对拟南芥含有二硫键的蛋白质进行了研究,采用硫醇亲和色谱富集目的蛋白,共鉴定到65个公认的含二硫键的蛋白[1, 47]。Moan等[49]采用双标记的方法对啤酒酵母中的硫醇氧化蛋白分别进行富集和定量,总共鉴定出64个蛋白质[1, 49]。
硝基化氧化修饰与谷胱甘肽化修饰也是一种可逆氧化修饰。Jaffrey等[69]和Hao等[70]对硝基化氧化修饰研究技术进行了提出和发展,完善了S-亚硝酰化蛋白质的研究策略,大概包括以下步骤[71]:(1)封闭所有自由巯基,选择性还原S—NO键;(2)引入生物素标签;(3)通过抗生物素蛋白富集目标蛋白质;(4)结合一维或二维凝胶电泳技术、液相色谱技术和质谱技术等手段进行亚硝酰化蛋白质的鉴定。与S-亚硝酰化相似,谷胱甘肽化修饰的蛋白质可以采用相似的研究手段,蛋白酶解后用生物素标记蛋白从而完成谷胱甘肽化肽段的富集[1, 40]。Cheng等[72]用生物素标记谷胱甘肽-S-转移酶后采用一种类似免疫印迹的方法检测谷胱甘肽化蛋白[1, 72];Klatt等[73]借助S-亚硝化谷胱甘肽(S-nitrosoglutathione,GSNO)是GS-供体,可以与蛋白质形成杂和的二硫键的特性,把GSNO与琼脂糖凝胶相结合,通过GSNO柱来富集目标蛋白质[1, 73]。
3 展望植物蛋白质氧化修饰中,蛋白质可逆氧化修饰对植物抵御逆境造成的氧化胁迫意义重大,对它的研究分三个层面,一是通过特定位点的突变实验来确定有无可逆氧化调控作用;二是利用蛋白质组学的方法测定氧化修饰的程度;三是利用综合实验通过控制氧化还原的蛋白酶等的氧化还原特性来解析还原机理及再生机理。
基于蛋白质可逆氧化的研究前景,认为在Trp的可逆氧化机理上值得挖掘;在Srx的再生机理上值得深究;对光合相关蛋白的可逆氧化还原的研究值得关注,特别是蛋白质可逆氧化修饰中还原性蛋白的再生与光合作用相偶联,它们与ROS的复杂关系值得系统研究。
| [1] | 刘金凤, 王京兰, 钱小红, 等. 翻译后修饰蛋白质组学研究的技术策略. 中国生物化学与分子生物学报, 2007, 23(2): 93–100. |
| [2] | Mann M, Jensen ON. Proteomic analysis of post-translational modifications. Nature Biotechnology, 2003, 21 (3): 255–261. DOI:10.1038/nbt0303-255 |
| [3] | Kalume DE, Molina H, Pandey A. Tackling the phosphoproteome: tools and strategies. Current Opinion in Chemical Biology, 2003, 7 (1): 64–69. DOI:10.1016/S1367-5931(02)00009-1 |
| [4] | 宋博研, 朱卫国. 组蛋白甲基化修饰效应分子的研究进展. 遗传, 2011, 33(4): 285–292. |
| [5] | Møller IM, Jensen PE, Hansson A. Oxidative modifications to cellular components in plants. Annual Review of Plant Biology, 2007, 58 : 459–481. DOI:10.1146/annurev.arplant.58.032806.103946 |
| [6] | 丁海东, 刘慧, 朱晓红, 等. 植物细胞蛋白质氧化及其蛋白质组学研究进展. 中国农学通报, 2011, 27(33): 187–193. |
| [7] | Waszczak C, Akter S, Eeckhout D, et al. Sulfenome mining in Arabidopsis thaliana. Proceedings of the National Academy of Sciences, 2014, 111 (31): 11545–11550. DOI:10.1073/pnas.1411607111 |
| [8] | Ghesquière B, Gevaert K. Proteomics methods to study methionine oxidation. Mass Spectrometry Reviews, 2014, 33 (2): 147–156. DOI:10.1002/mas.v33.2 |
| [9] | Gupta V, Carroll KS. Sulfenic acid chemistry, detection and cellular lifetime. Biochimica et Biophysica Acta (BBA)-General Subjects, 2014, 1840 (2): 847–875. DOI:10.1016/j.bbagen.2013.05.040 |
| [10] | Pan J, Carroll KS. Chemical biology approaches to study protein cysteine sulfenylation. Biopolymers, 2014, 101 (2): 165–172. DOI:10.1002/bip.v101.2 |
| [11] | Jacques S, Ghesquière B, De Bock PJ, et al. Protein methionine sulfoxide dynamics in Arabidopsis thaliana under oxidative stress. Molecular & Cellular Proteomics, 2015, 14 (5): 1217–1229. |
| [12] | 张佳娣. 活性氧的信号传导途径. 安徽农业科学, 2010, 38(16): 8283–8285. DOI:10.3969/j.issn.0517-6611.2010.16.002 |
| [13] | Noctor G, Mhamdi A, Foyer CH. Oxidative stress and antioxidative systems: recipes for successful data collection and interpretation. Plant, Cell & Environment, 2016, 66 (10): 1140–1160. |
| [14] | Rinalducci S, Murgiano L, Zolla L. Redox proteomics: basic principles and future perspectives for the detection of protein oxidation in plants. Journal of Experimental Botany, 2008, 59 (14): 3781–3801. DOI:10.1093/jxb/ern252 |
| [15] | Navrot N, Finnie C, Svensson B, et al. Plant redox proteomics. Journal of Proteomics, 2011, 74 (8): 1450–1462. DOI:10.1016/j.jprot.2011.03.008 |
| [16] | 付强, 邹颉, 赵杰宏, 等. 植物氧化还原蛋白质组学的研究进展. 贵州农业科学, 2013, 41(12): 17–20. DOI:10.3969/j.issn.1001-3601.2013.12.005 |
| [17] | Weissbach H, Resnick L, Brot N. Methionine sulfoxide reductases: history and cellular role in protecting against oxidative damage. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2005, 1703 (2): 203–212. DOI:10.1016/j.bbapap.2004.10.004 |
| [18] | Ghezzi P, Bonetto V. Redox proteomics: identification of oxidatively modified proteins. Proteomics, 2003, 3 (7): 1145–1153. DOI:10.1002/pmic.200300435 |
| [19] | Couturier J, Chibani K, Jacquot JP, et al. Cysteine-based redox regulation and signaling in plants. Frontiers in Plant Science, 2013, 4 : 105. |
| [20] | Liebster J, Kopoldova J. The radication chemistry of amino acids. Advances in Radiation Biology, 2013, 1 : 157. |
| [21] | 李冰冰, 赵倩, 张龙富. 活性氧与蛋白质氧化损伤. 平顶山工学院学报, 2005, 14(5): 16–17. |
| [22] | Aulak KS, Miyagi M, Yan L, et al. Proteomic method identifies proteins nitrated in vivo during inflammatory challenge. Proceedings of the National Academy of Sciences, 2001, 98 (21): 12056–12061. DOI:10.1073/pnas.221269198 |
| [23] | Kanski J, Hong SJ, Schöneich C. Proteomic analysis of protein nitration in aging skeletal muscle and identification of nitrotyrosine-containing sequences in vivo by nanoelectrospray ionization tandem mass spectrometry. Journal of Biological Chemistry, 2005, 280 (25): 24261–24266. DOI:10.1074/jbc.M501773200 |
| [24] | Friso G, van Wijk KJ. Posttranslational protein modifications in plant metabolism. Plant Physiology, 2015, 169 (3): 1469–1487. |
| [25] | Rao RSP, Møller IM, Thelen JJ, et al. Convergent signaling pathways——interaction between methionine oxidation and serine/threonine/tyrosine O-phosphorylation. Cell Stress & Chaperones, 2015, 20 (1): 15–21. |
| [26] | 黄舒, 李刚, 朱建堂, 等. 植物Msr (methionine sulfoxide redu-ctase) 基因家族的研究进展. 生命的化学, 2015, 35(3): 313–319. |
| [27] | 王海波, 邹竹荣, 龚明. 小桐子甲硫氨酸亚砜还原酶A的基因克隆及生物信息学分析. 基因组学与应用生物学, 2015, 34(4): 821–829. |
| [28] | Drazic A, Winter J. The physiological role of reversible methionine oxidation. Biochimica et Biophysica Acta, 2014, 1844 (8): 1367–1382. DOI:10.1016/j.bbapap.2014.01.001 |
| [29] | Muthuramalingam M, Matros A, Scheibe R, et al. The hydrogen peroxide-sensitive proteome of the chloroplast in vitro and in vivo. Frontiers in Plant Science, 2013, 4 : 54. |
| [30] | Kim HY, Fomenko DE, Yoon YE, et al. Catalytic advantages provided by selenocysteine in methionine-S-sulfoxide reductases. Biochemistry, 2006, 45 (46): 13697–13704. DOI:10.1021/bi0611614 |
| [31] | Tarrago L, Laugier E, Rey P. Protein-repairing methionine sulfoxide reductases in photosynthetic organisms: gene organization, reduction mechanisms, and physiological roles. Molecular Plant, 2009, 2 (2): 202–217. DOI:10.1093/mp/ssn067 |
| [32] | Lee BC, Dikiy A, Kim HY, et al. Functions and evolution of selenoprotein methionine sulfoxide reductases. Biochimica et Biophysica Acta (BBA)-General Subjects, 2009, 1790 (11): 1471–1477. DOI:10.1016/j.bbagen.2009.04.014 |
| [33] | Stamler JS. Redox signaling: nitrosylation and related target interactions of nitric oxide. Cell, 1994, 78 (6): 931–936. DOI:10.1016/0092-8674(94)90269-0 |
| [34] | Perissinotti LL, Turjanski AG, Estrin DA, et al. Transnitrosation of nitrosothiols: characterization of an elusive intermediate. Journal of the American Chemical Society, 2005, 127 (2): 486–487. DOI:10.1021/ja044056v |
| [35] | 陈畅, 黄波, 韩佩韦, 等. 蛋白质巯基亚硝基化——一种典型氧化还原依赖的蛋白质翻译后修饰. 生物化学与生物物理进展, 2006, 33(7): 609–615. |
| [36] | 黄楚森, 朱维平, 徐玉芳, 等. 蛋白质巯基及其氧化性修饰的化学检测方法. 药学学报, 2012, 47(3): 280–290. |
| [37] | 黄波, 陈畅. 一氧化氮的功能及其作用机制 (Ⅱ)——蛋白质巯基亚硝基化修饰. 生物物理学报, 2012, 28(4): 268–277. |
| [38] | Xu L, Eu JP, Meissner G, et al. Activation of the cardiac calcium release channel (ryanodine receptor) by poly-S-nitrosylation. Science, 1998, 279 (5348): 234–237. DOI:10.1126/science.279.5348.234 |
| [39] | 梁颖, 李玉花. 植物中磷酸甘油醛-3-磷酸脱氢酶 (GAPDH) 在氧化胁迫下的生理功能. 植物生理学通讯, 2009, 45(10): 1027–1032. |
| [40] | Hamnell-Pamment Y, Lind C, Palmberg C, et al. Determination of site-specificity of S-glutathionylated cellular proteins. Biochemical and Biophysical Research Communications, 2005, 332 (2): 362–369. DOI:10.1016/j.bbrc.2005.04.130 |
| [41] | 邹朝霞, 周宏博, 高旭. 谷胱甘肽化修饰与氧化还原信号转导. 生命的化学, 2007, 27(5): 410–413. |
| [42] | Sevilla F, Camejo D, Ortiz-Espín A, et al. The thioredoxin/peroxiredoxin/sulfiredoxin system: current overview on its redox function in plants and regulation by reactive oxygen and nitrogen species. Journal of Experimental Botany, 2015, 66 (10): 2945–2955. DOI:10.1093/jxb/erv146 |
| [43] | Biteau B, Labarre J, Toledano MB. ATP-dependent reduction of cysteine-sulphinic acid by S. cerevisiae sulphiredoxin. Nature, 2003, 425 (6961): 980–984. DOI:10.1038/nature02075 |
| [44] | 李奎, 白洁. 硫氧还蛋白的共价修饰. 生命的化学, 2010, 30(1): 54–58. |
| [45] | 王珊, 王辉, 陈小燕. 硫氧还蛋白-过氧化物氧还蛋白与过氧化氢构成环路参与肿瘤的发生与发展. 中国生物化学与分子生物学报, 2015, 31(4): 360–366. |
| [46] | 郑琼, 马旭俊, 杨传平. 硫氧还蛋白 (Trx) 的研究进展. 分子植物育种, 2006, 4(6S): 78–82. |
| [47] | Lee K, Lee J, Kim Y, et al. Defining the plant disulfide proteome. Electrophoresis, 2004, 25 (3): 532–541. DOI:10.1002/(ISSN)1522-2683 |
| [48] | Yano H, Wong JH, Lee YM, et al. A strategy for the identification of proteins targeted by thioredoxin. Proceedings of the National Academy of Sciences, 2001, 98 (8): 4794–4799. DOI:10.1073/pnas.071041998 |
| [49] | Le Moan N, Clement G, Le Maout S, et al. The Saccharomyces cerevisiae proteome of oxidized protein thiols contrasted functions for the thioredoxin and glutathione pathways. Journal of Biological Chemistry, 2006, 281 (15): 10420–10430. DOI:10.1074/jbc.M513346200 |
| [50] | Allen EMG, Mieyal JJ. Protein-thiol oxidation and cell death: regulatory role of glutaredoxins. Antioxidants & Redox Signaling, 2012, 17 (12): 1748–1763. |
| [51] | Kim G, Weiss SJ, Levine RL. Methionine oxidation and reduction in proteins. Biochimica et Biophysica Acta (BBA)-General Subjects, 2014, 1840 (2): 901–905. DOI:10.1016/j.bbagen.2013.04.038 |
| [52] | Valley CC, Cembran A, Perlmutter JD, et al. The methionine-aromatic motif plays a unique role in stabilizing protein structure. Journal of Biological Chemistry, 2012, 287 (42): 34979–34991. DOI:10.1074/jbc.M112.374504 |
| [53] | Gustavsson N, Kokke B, Härndahl U, et al. A peptide methionine sulfoxide reductase highly expressed in photosynthetic tissue in Arabidopsis thaliana can protect the chaperone-like activity of a chloroplast-localized small heat shock protein. The Plant Journal, 2002, 29 (5): 545–553. DOI:10.1046/j.1365-313X.2002.029005545.x |
| [54] | Bechtold U, Murphy DJ, Mullineaux PM. Arabidopsis peptide methionine sulfoxide reductase2 prevents cellular oxidative damage in long nights. The Plant Cell, 2004, 16 (4): 908–919. DOI:10.1105/tpc.015818 |
| [55] | Li CW, Lee SH, Chieh PS, et al. Arabidopsis root-abundant cytosolic methionine sulfoxide reductase B genes MsrB7 and MsrB8 are involved in tolerance to oxidtive stress. Plant and Cell Physiology, 2012, 53 (10): 1707–1719. DOI:10.1093/pcp/pcs114 |
| [56] | Hrabak EM, Chan CWM, Gribskov M, et al. The Arabidopsis CDPK-SnRK superfamily of protein kinases. Plant Physiology, 2003, 132 (2): 666–680. DOI:10.1104/pp.102.011999 |
| [57] | Tarafdar S, Rusan NM, Levine RL. Site specific oxidation of calm-odulin by methionine sulfoxide reductase a in Drosophila. The FASEB Journal, 2016, 30 (1 Supplement): 652–654. |
| [58] | Hardin SC, Larue CT, Oh MH, et al. Coupling oxidative signals to protein phosphorylation via methionine oxidation in Arabidopsis. Biochemical Journal, 2009, 422 (2): 305–312. DOI:10.1042/BJ20090764 |
| [59] | Ciorba MA, Heinemann SH, Weissbach H, et al. Regulation of voltage-dependent K+ channels by methionine oxidation: effect of nitric oxide and vitamin C. FEBS Letters, 1999, 442 (1): 48–52. DOI:10.1016/S0014-5793(98)01616-0 |
| [60] | Erickson JR, Mei-Ling AJ, Guan X, et al. A dynamic pathway for calcium-independent activation of CaMKⅡ by methionine oxidation. Cell, 2008, 133 (3): 462–474. DOI:10.1016/j.cell.2008.02.048 |
| [61] | Liu XP, Liu XY, Zhang J, et al. Molecular and functional characterization of sulfiredoxin homologs from higher plants. Cell Research, 2006, 16 (3): 287–296. DOI:10.1038/sj.cr.7310036 |
| [62] | Cerveau D, Ouahrani D, Marok MA, et al. Physiological relevance of plant 2-Cys peroxiredoxin overoxidation level and oligomerization status. Plant, Cell & Environment, 2016, 39 (1): 103–119. |
| [63] | Waszczak C, Akter S, Jacques S, et al. Oxidative post-translational modifications of cysteine residues in plant signal transduction. Journal of Experimental Botany, 2015, 66 (10): 2923–2934. DOI:10.1093/jxb/erv084 |
| [64] | Gupta R, Luan S. Redox control of protein tyrosine phosphatases and mitogen-activated protein kinases in plants. Plant Physiology, 2003, 132 (3): 1149–1152. DOI:10.1104/pp.103.020792 |
| [65] | Jacques S, Ghesquire B, Van Breusegem F, et al. Plant proteins under oxidative attack. Proteomics, 2013, 13 (6): 932–940. DOI:10.1002/pmic.201200237 |
| [66] | Walton A, Tsiatsiani L, Jacques S, et al. Diagonal chromatography to study plant protein modifications. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2016, 1864 (8): 945–951. DOI:10.1016/j.bbapap.2016.01.001 |
| [67] | Gevaert K, Van Damme J, Goethals M, et al. Chromatographic isolation of methionine-containing peptides for gel-free proteome analysis: identification of more than 800 Escherichia coli proteins. Molecular & Cellular Proteomics, 2002, 1 (11): 896–903. |
| [68] | MacCoss MJ, McDonald WH, Saraf A, et al. Shotgun identification of protein modifications from protein complexes and lens tissue. Proceedings of the National Academy of Sciences, 2002, 99 (12): 7900–7905. DOI:10.1073/pnas.122231399 |
| [69] | Jaffrey SR, Erdjument-Bromage H, Ferris CD, et al. Protein S-nitrosylation: a physiological signal for neuronal nitric oxide. Nature Cell Biology, 2001, 3 (2): 193–197. DOI:10.1038/35055104 |
| [70] | Hao G, Derakhshan B, Shi L, et al. SNOSID, a proteomic method for identification of cysteine S-nitrosylation sites in complex protein mixtures. Proceedings of the National Academy of Sciences, 2006, 103 (4): 1012–1017. DOI:10.1073/pnas.0508412103 |
| [71] | Rhee KY, Erdjument-Bromage H, Tempst P, et al. S-nitroso proteome of Mycobacterium tuberculosis: Enzymes of intermediary metabolism and antioxidant defense. Proceedings of the National Academy of Sciences, 2005, 102 (2): 467–472. DOI:10.1073/pnas.0406133102 |
| [72] | Cheng G, Ikeda Y, Iuchi Y, et al. Detection of S-glutathionylated proteins by glutathione S-transferase overlay. Archives of Biochemistry and Biophysics, 2005, 435 (1): 42–49. DOI:10.1016/j.abb.2004.12.016 |
| [73] | Klatt P, Molina EP, Pérez-Sala D, et al. Novel application of S-nitrosoglutathione-Sepharose to identify proteins that are potential targets for S-nitrosoglutathione-induced mixed-disulphide formation. Biochemical Journal, 2000, 349 (2): 567–578. |




