2. 解放军第422中心医院,湛江 524023
2. Central Hospital of PLA, Zhanjiang 524023
目前基因编辑技术主要包括锌指核酸酶技术(zinc finger nucleases,ZFNs)、类转录激活因子效应物核酸酶技术(transcription activator-like effector nucleases,TALENs)和CRISPR(clustered regularly interspaced short palindromic repeats,成簇的规律间隔的短回文重复序列)/Cas9(CRISPR-associated protein 9,CRISPR相关蛋白)技术。ZFNs技术和TALENs技术分别借助锌指蛋白和类转录激活因子效应物识别特异DNA,再利用FokⅠ核酸内切酶切割靶目标。CRISPR/Cas9技术依靠sgRNA识别序列,Cas9蛋白切割序列。虽然ZFNs技术和TALENs技术在基因、序列、细胞、物种限制和特异性方面比CRISPR/Cas9技术有优势,但随着CRISPR/Cas9技术的日趋完善,其优势逐渐凸显:(1)操作简单,设计灵活,价格低廉;(2)能实现多点编辑[1];(3)可扩展性更强,可与piggyBac(PB)转座子系统[2]或Cre/LoxP重组酶系统[3]等联用。本文介绍CRISPR/Cas9技术及其作用机制,主要针对该技术目前存在的问题及相应的改进措施进行综述,为合理使用或改进该技术提供思路。
1 CRISPR/Cas9简介CRISPR/Cas系统是原核生物特有的一种天然免疫系统,用于抵抗病毒或外源性质粒的侵害[4]。根据Cas蛋白的特点,可将CRISPR/Cas系统分为Ⅰ、Ⅱ、Ⅲ型,Ⅰ型和Ⅲ型系统需要借助复杂的蛋白复合体发挥作用,而Ⅱ型系统仅借助Cas9蛋白和导向RNA(guide RNA,gRNA)即可对靶目标进行编辑,结构更简单且易于操作,因此目前基因编辑中使用的主要是Ⅱ型化脓性链球菌(Streptococcus pyogenes,Sp)CRISPR/Cas9系统(图 1)。
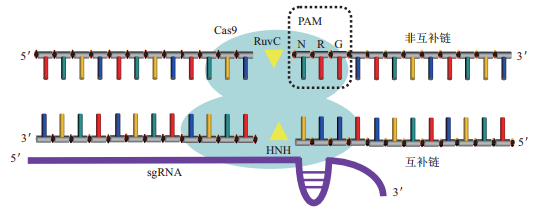
|
| 图 1 CRISPR/Cas9技术原理图 Cas9蛋白与sgRNA结合后,在识别PAM序列(NRG)的基础上匹配DNA序列,若能匹配,则RuvC结构域切割非互补链,HNH结构域切割互补链 |
CRISPR/Cas9主要由两部分组成:gRNA和Cas9蛋白。gRNA由CRISPR RNA(crRNA)与反式激活crRNA(trans-activating crRNA,tracrRNA)组成,crRNA的一部分序列能与tracrRNA配对,形成双链RNA结构;一部分序列与靶目标互补区域,以此识别靶序列。tracr RNA不仅可稳定crRNA,还可参与Cas9蛋白与DNA的结合与切割[5]。基因编辑时可使用单独的crRNA和tracr RNA,也可使用二者嵌合而成的单链向导RNA(single guide RNA,sgRNA),单独的Cas9蛋白是一个二叶片结构,其中一个叶片为核酸酶叶片,包括HNH结构域、RuvC结构域和C端结构域;另一个叶片由α螺旋结构域组成[6]。Cas9蛋白先与gRNA形成复合体,使Cas9蛋白构象变化,产生一条通道,使其更易与DNA结合[7]。该复合体随机碰撞DNA,若未识别到PAM序列,则会快速离开DNA,发生下一次碰撞[8];若识别PAM序列,Cas9蛋白的Arg1333和Arg1335会与二核苷酸鸟嘌呤结合,此时,Ser1109和Lys1107形成磷酸锁(phosphate lock)结构,与PAM序列旁的+1位核苷酸发生相互作用,促使双链DNA解旋,利用RNA-DNA碱基互补配对原则,核对序列与crRNA的互补情况[9],若在种子序列出现较多错配,则序列识别过程终止,从而确保Cas9在正确的目标处发挥作用。若完成对靶序列的识别,则可激活HNH结构域,使其发生构象改变,目标链进入其活性位点。蛋白和核酸的相互作用,使非目标链进入RuvC结构域的活性位点中[10],使Cas9蛋白对目标链和非目标链进行切割作用,从而完成基因编辑。
3 CRISPR/Cas9存在的问题及其改进方法 3.1 精确修复比例低CRISPR/Cas9切断基因组双链DNA后,断裂处的修复方式主要是非同源末端连接(non-homologous End Joining,NHEJ)[11],这是一种错误率极高的修复方式,读框移码的插入或删除,导致基因功能丧失;而能在目标位点精确插入期望序列的修复方式-同源介导的双链DNA修复(homology directed repair,HDR)所占比例常不足10%[12-14](图 2)。CRISPR/Cas9技术若要成为基因治疗的手段之一,必须实现精确定点编辑。研究表明,可通过抑制NHEJ修复或者增强HDR修复来提高精确修复的效率。
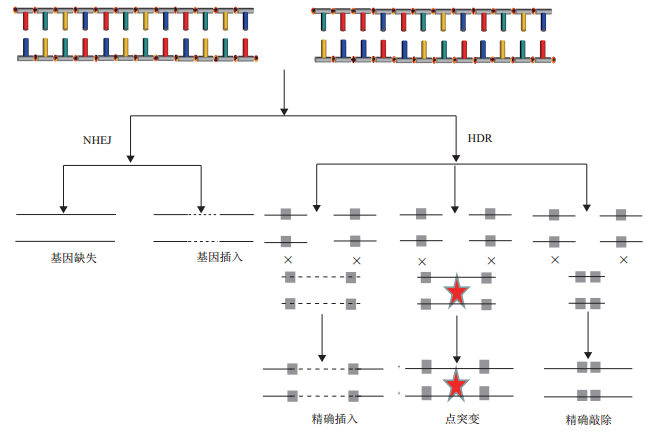
|
| 图 2 双链断裂DNA修复原理图 CRIPSR/Cas9系统产生双链断裂DNA,其修复方式主要有NHEJ和HDR。NHEJ使断裂的DNA两端直接相连或随机引入一段序列。HDR以同源序列为模板,在断裂的DNA之间精确引入、替换、敲除序列 |
NHEJ修复主要借助Ku蛋白和DNA连接酶Ⅳ实现,而Scr7以DNA连接酶Ⅳ的DNA结合域为靶标,降低其与双链断裂的结合能力,从而抑制NHEJ修复。研究表明,将Scr7与CRISPR/Cas9系统共同作用于哺乳动物细胞和小鼠,可大幅降低NHEJ修复,将精确编辑的效率提高数倍,甚至十几倍[15-17]。
而直接增强HDR修复以提高精确编辑的方法主要有调控细胞周期、引入外源性DNA、化学修饰sgRNA或小分子激活HDR等。NHEJ修复虽然可贯穿整个细胞生长周期,但其在G1期占主要地位,而HDR主要在S期和G2期发挥作用。因此对阻滞在G2/M期的细胞进行基因编辑,可大幅提高精确编辑的效率。研究表明,利用药物将细胞周期同步化在G2/M期,可将精确编辑的数量增加数倍[18, 19]。Cas9从双链断裂DNA上解离的过程是缓慢的,在完全解离之前,Cas9先释放非目标链的3'端,这使得非目标链更容易接受与其互补的外源性单链DNA,从而激活HDR修复。研究者通过导入一条与先释放的非目标链互补的单链DNA,可将HDR修复的比例提高至60%[20]。对sgRNA进行化学修饰也是一种有效可行的方法。研究表明,以2'-O-methyl(M)、2'-O-methyl 3'phosphorothioate(MS)或2'-O-methyl 3'thioPACE(MSP)修饰sgRNA,可增大HDR修复比例,若进一步增加修饰sgRNA的数量,精确编辑效率最高可达83.3%[21]。这一现象可能是由于这些化学基团可增加sgRNA在血清中的稳定性和改变核苷酸的免疫刺激能力。还有研究者在对小分子进行高通量扫描后,筛选出能提高HDR比例的2种小分子-β3肾上腺素受体激动剂L755507和胞内蛋白转运抑制剂布雷非德菌素A[22]。
此外,利用胞嘧啶核苷脱氨酶[23]或碱基修饰酶APOBEC1[24]修饰Cas9蛋白,可实现对单个核苷酸的精确编辑。虽然实现单个核苷酸编辑使CRIS-PR/Cas9技术的精确编辑发生质的飞跃,但是目前仅限于将胞嘧啶更换为胸腺嘧啶。遗传性疾病的基因突变种类繁多,仅一种核苷酸的替换尚不能满足其要求,在日后的研究中,可继续改造CRISPR/Cas9系统,以期能实现对核苷酸的精确随意替换。
3.2 PAM识别序列的限制前间区序列邻近基序(protospacer adjacent motif,PAM)是一个位于靶DNA 3'端的短核苷酸基序,可被Cas9蛋白特异性识别。化脓性链球菌可识别标准PAM NGG和非标准PAM NAG。理论上,生物基因组中NGG每隔8 bp碱基出现一次;NRG则每4 bp碱基出现一次。但由于不同基因序列上的差异,并非所有的基因都能有合适的PAM。分析玉米的全基因组,在有注释的外显子中仅30%能找到特异性切割位点[25]。这表明,PAM在一定程度上限制了Cas9剪切位点的选择。为了减弱PAM对Cas9蛋白的限制,可对Cas9蛋白进行改造和使用Cas9直系同源酶。
有学者主张改造Cas9蛋白,扩大其PAM的识别范围。化脓性链球菌Cas9突变体VQR(D1135V/R1335Q/T1337R)识别的PAM为NGAN和NGCG,突变体VRER(D1135V/G1218R/R1335E/T1337R)识别的PAM为NGC,突变体D1135E特异性识别NGG,突变体QQR1特异性识别NAAG[26]。这些变更了识别PAM的突变体,无疑扩大靶位点的选择。而研究也证实,这些突变体可以在野生型SpCas9无法找到编辑序列的斑马鱼和人类内源性基因位点中,找到合适的编辑位点[27]。
也有学者主张利用Cas9直系同源酶减弱PAM对Cas9蛋白靶位点的限制。目前通过序列比对等方法发现Cas9的直系同源酶已超过1 000种[28],但已确定PAM序列的Cas9直系同源酶仅有弗朗西丝菌(PAM为NGG)[29]、金黄色葡萄球菌(PAM为NGGRRT)[30]、脑膜炎双球菌(PAM为NNNNGATT)[31]、嗜热链球菌CRISPR1(PAM为NAAGAAW)[32]、嗜热链球菌CRISPR3(PAM为NGGNG)[33]等少数几种。这意味着Cas9直系同源酶是一个巨大的侯选库,可以用于克服化脓性链球菌Cas9在一些物种应用上的局限性。为了充分利用Cas9直系同源酶,研究者不仅利用生物信息学与PAM文库结合的方法确定更多Cas9直系同源酶的PAM序列[34],而且对已知PAM序列的Cas9直系同源酶加以改造。研究表明,弗朗西丝菌和金黄色葡萄球菌的突变体能将靶向范围扩大2-4倍[35, 36]。
此外,还有学者发现了靶向RNA的C2c2蛋白[37],这无疑将进一步扩大CRISPR/Cas技术的应用范围。
3.3 脱靶现象CRISPR/Cas9基因编辑系统依靠sgRNA的识别序列与靶向序列特异性结合。由于基因组极为复杂,sgRNA可能与其他非靶向序列局部匹配,这种局部匹配也会激活Cas9内切酶活性,从而产生脱靶(图 3)。此外,Cas9不仅识别标准PAM,也可识别非标准PAM,设计靶向序列时忽略了非标准PAM,这也可能会引起一定程度的脱靶。脱靶可能影响正常基因的功能表达,甚至激活致癌因子、抑制抑癌基因,造成安全隐患。目前主要通过改造Cas9蛋白和优化sgRNA两方面降低脱靶现象。

|
| 图 3 脱靶机制 A:sgRNA识别目标序列依赖20个碱基,1-12碱基区域为种子区域,决定了sgRNA的识别能力,13-20号碱基为非种子区域。B:由于基因组DNA的复杂性,sgRNA不仅可与目标序列结合;也可能与非目标序列结合,激活Cas9切割活性,从而切割非目标序列,产生脱靶。off-target 1是sgRNA与DNA序列在非种子区域出现3个碱基错配,off-target 2是sgRNA与DNA序列之间存在单个凸起碱基的错配。(图中箭头所指为sgRNA与DNA错配的碱基) |
改造Cas9蛋白以提高特异性的方法主要分为以下几类:(1)使Cas9蛋白的核酸内切酶活性降低或丧失。无论是使用降低HNH或RuvC结构域功能的突变体H840A和D10A[38],或是构建使Cas9蛋白丧失核酸内切酶活性,仅保留其核酸识别能力,借助其他核酸内切酶实现基因编辑的CRISPRi[39]和FokⅠ-dCas9[40, 41],这两种方法均根据Cas9蛋白能被一定错配范围内的非靶向序列激活,从而发挥切割作用这一特点设计,使其核酸内切酶活性降低或丧失,一定程度上可提高激活Cas9所需要的序列匹配度,从而大幅提高特异性。(2)降低Cas9蛋白结合DNA的能力。对Cas9蛋白进行定点突变形成eSpCas9[42]和SpCas9-HF1[43],突变体减弱了Cas9蛋白与DNA的非特异性结合的能力,增强靶向序列与Cas9蛋白结合的竞争力。(3)调控Cas9蛋白在细胞中的表达量。通过使用诱导型启动子[44]、插入4-HT依赖的内含肽[45]等方法构建诱导型Cas9蛋白,减少基因组暴露于Cas9与sgRNA复合体的时间,即Cas9与非靶向序列的结合概率,进而降低脱靶率。(4)增强Cas9蛋白对标准PAM序列的识别能力。突变体D1135E[27]对标准PAM的识别更加特异,从而降低因识别非标准PAM序列而引起脱靶。
而通过优化sgRNA降低脱靶效应的策略主要有(1)改变sgRNA的长度,截断5'端的2-3个碱基[46]或在识别序列前加2个鸟嘌呤[47],切割活性不变,脱靶率降低5 000倍。(2)增加sgRNA的稳定性。如在gRNA中增加一对A-U碱基配对等方法。此外,有研究表明Cas9直系同源酶对某些位点的特异性较SpCas9高[48, 49],这无疑为提高特异性提供了一条新思路。通过对CRISPR/Cas9系统的改进,脱靶现象大幅降低,但离完全无脱靶还有一定距离。
3.4 马赛克现象(Mosacism)基因编辑技术日趋完善,研究者期望借助基因编辑来阐述基因在早期发育过程中的作用,以及修复胚胎中的突变基因,以避免疾病的发生。目前,将Cas9和sgRNA导入受精卵多是通过电转化[50]或者显微镜下注射[51]的方式,而且多是转入Cas9 mRNA。但这些方式获得的突变体多存在马赛克现象(图 4)。马赛克现象是指将CRISPR/Cas9系统应用于多细胞生物的受精卵基因编辑时,受精卵分裂成不同的卵裂球,而Cas9蛋白对不同卵裂球的编辑能力和修复方式可能不同,从而出现带有不同编辑类型的细胞的嵌合个体,或是同时带有编辑细胞与未编辑细胞的嵌合个体。马赛克现象出现的原因可能是从受精成功到基因组第一次复制的时间极为有限,而Cas9 mRNA翻译为蛋白质需要时间长[52],错过了在单细胞受精卵发生编辑的时机,且Cas9蛋白和sgRNA半衰期较长[53],能对一定时间范围内新分裂生成的细胞进行不同的编辑。不论是观察基因的作用还是避免遗传病的发生,都要求生成非镶嵌的突变体。为避免镶嵌现象出现,必须使Cas9蛋白在细胞基因组第一次复制之前发挥作用,且之后不再发挥作用。虽然近期研究表明将Cas9蛋白和sgRNA以电转的方式导入体外受精的早期受精卵中,可大大降低携带镶嵌基因个体的出现[54]。但使该技术完全避免镶嵌现象,尚有一定难度。

|
| 图 4 马赛克现象 CRISPR/Cas9技术作用于受精卵后,未编辑细胞与编辑细胞共存,且编辑细胞的编辑位点可能不同,从而导致同一个体带有不同基因型细胞。(未编辑细胞以灰色表示,编辑细胞以白色表示,纯白细胞与白底黑点细胞表示两种不同的编辑细胞,三角形代表CRISPR/Cas9的编辑位点) |
虽然CRISPR/Cas9基因编辑技术尚存在一些问题,但是研究者一直致力于寻找解决方法,通过改造修饰Cas9蛋白、使用Cas9直系同源酶、化学物质辅助等一系列措施,已然提高了CRISPR/Cas9基因编辑技术精确修复断裂DNA能力,降低其脱靶率,减弱PAM导致的靶位点限制,在一定程度上控制了镶嵌现象的发生。研究者在寻找弥补不足的方法的同时,也在不断拓展该技术的应用领域。在器官移植领域,利用该技术已成功去除多种猪器官中的猪内源性逆转录病毒[55],使猪器官移植成为可能。在治疗病毒感染领域,体外实验和动物实验显示将该技术已能成功去除HIV[56]、乙型肝炎病毒[57]等多种病毒,为这些疾病的治疗翻开新篇章。在植物育种[58]、遗传性疾病治疗[59]等多个领域CRISPR/Cas9技术均取得重要成果。CRISPR/Cas9技术还在不断发展,期待它能在更多的领域发挥作用。
| [1] | Gao SL, Tong YY, Wen ZQ, et al. Multiplex gene editing of the Yarrowia lipolytica genome using the CRISPR-Cas9 system. J Ind Microbiol Biotechnol, 2016, 43 (8): 1085–1093. DOI:10.1007/s10295-016-1789-8 |
| [2] | Cheng M, Jin XB, Mu L, et al. Combination of the clustered regularly interspaced short palindromic repeats (CRISPR)-associated 9 technique with the piggybac transposon system for mouse in utero electroporation to study cortical development. J Neurosci Res, 2016, 94 (9): 814–824. DOI:10.1002/jnr.23776 |
| [3] | Bi YZ, Hua ZD, Liu XM, et al. Isozygous and selectable marker-free MSTN knockout cloned pigs generated by the combined use of CRISPR/Cas9 and Cre/LoxP. Sci Rep, 2016, 6 : 31729. DOI:10.1038/srep31729 |
| [4] | Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31 (3): 233–239. DOI:10.1038/nbt.2508 |
| [5] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337 (6096): 816–821. DOI:10.1126/science.1225829 |
| [6] | Jinek M, Jiang F, Taylor DW, et al. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 2014, 343 (6176): 1247997. DOI:10.1126/science.1247997 |
| [7] | Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 2014, 156 (5): 935–949. DOI:10.1016/j.cell.2014.02.001 |
| [8] | Sternberg SH, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507 (7490): 62–67. DOI:10.1038/nature13011 |
| [9] | Anders C, Niewoehner O, Duerst A, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 2014, 513 (7519): 569–573. DOI:10.1038/nature13579 |
| [10] | Jiang FG, Taylor DW, Chen JS, et al. Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science, 2016, 351 (6275): 867–871. DOI:10.1126/science.aad8282 |
| [11] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 6 : 1262–1278. |
| [12] | Gratz SJ, Ukken FP, Rubinstein CD, et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics, 2014, 196 (4): 961–971. DOI:10.1534/genetics.113.160713 |
| [13] | Miyaoka Y, Berman JR, Cooper SB, et al. Systematic quantification of HDR and NHEJ reveals effects of locus, nuclease, and cell type on genome-editing. Sci Rep, 2016, 6 : 23549. DOI:10.1038/srep23549 |
| [14] | Paquet D, Kwart D, Chen A, et al. Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9. Nature, 2016, 533 (7601): 125–129. DOI:10.1038/nature17664 |
| [15] | Ma YW, Chen W, Zhang X, et al. Increasing the efficiency of CRISPR/Cas9-mediated precise genome editing in rats by inhibiting NHEJ and using Cas9 protein. RNA Biology, 2016, 13 (7): 605–612. DOI:10.1080/15476286.2016.1185591 |
| [16] | Lin CL, Li HH, Hao MR, et al. Increasing the efficiency of CRISPR/Cas9-mediated precise genome editing of HSV-1 virus in human cells. Sci Rep, 2016, 6 : 34531. DOI:10.1038/srep34531 |
| [17] | Chu VT, Weber T, Wefers B, et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-inducedprecise gene editing in mammalian cells. Nat Biotechnol, 2015, 33 (5): 543–548. DOI:10.1038/nbt.3198 |
| [18] | Lin S, Staahl BT, Alla RK, et al. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife, 2014, 3 : e04766. |
| [19] | Yang D, Scavuzzo MA, Chmielowiec J, et al. Enrichment of G2/M cell cyclephase in human pluripotent stemcells enhances HDR-mediated gene repair with customizable endonucleases. Scientific Reports, 2016, 6 : 21264. DOI:10.1038/srep21264 |
| [20] | Richardson CD, Ray GJ, DeWitt MA, et al. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology, 2016, 34 (3): 339–344. DOI:10.1038/nbt.3481 |
| [21] | Hendel A, Bak RO, Clark JT, et al. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells. Nature Biotechnology, 2015, 33 (9): 985–989. DOI:10.1038/nbt.3290 |
| [22] | Yu C, Liu YX, Ma TH, et al. Small molecules enhance CRISPR genome editing in pluripotent stem cells. Cell Stem Cell, 2015, 16 (2): 142–147. DOI:10.1016/j.stem.2015.01.003 |
| [23] | Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science, 2016, 353 (6305): 1248. |
| [24] | Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533 (7603): 420–424. DOI:10.1038/nature17946 |
| [25] | Xie KB, et al. Genome-wide prediction of highly specific guide RNA spacers for CRISPR-Cas9-mediated genome editing in model plants and major crops. Mol Plant, 2014, 5 : 923–926. |
| [26] | Anders C, Bargsten K, Jinek M, et al. Structural plasticity of PAM recognition by engineered variants of the RNA-guided endonuclease Cas9. Mol Cell, 2016, 61 (6): 895–902. DOI:10.1016/j.molcel.2016.02.020 |
| [27] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 2015, 523 (7561): 481–485. DOI:10.1038/nature14592 |
| [28] | Chylinski K, Makarova KS, Charpentier E, et al. Classification and evolutionof type Ⅱ CRISPR-Cas systems. Nucleic Acids Res, 2014, 42 (10): 6091–6105. DOI:10.1093/nar/gku241 |
| [29] | Hirano H, Gootenberg JS, Horii T, et al. Structure and Engineering of Francisella novicida Cas9. Cell, 2016, 5 : 950–961. |
| [30] | Feng Y, Chen C, Han YX, et al. Expanding CRISPR/Cas9 genome editing capacity in zebrafish using saCas9. G3(Bethesda), 2016, 6 (8): 2517–2521. |
| [31] | Lee CM, Cradick TJ, Bao G, et al. The Neisseria meningitidis CR-ISPR-Cas9 system enables specific genome editing in mammalian cells. Mol Ther, 2016, 24 (3): 645–54. DOI:10.1038/mt.2016.8 |
| [32] | Li W, Bian X, et al. Comparative analysis of clustered regularly interspaced short palindromic repeats (CRISPR) of Streptococcus thermophilus St-Ⅰ and its bacteriophage-insensitive mutants (BIM) derivatives. Curr Microbiol, 2016, 73 (3): 393–400. DOI:10.1007/s00284-016-1076-y |
| [33] | Glemzaite M, Balciunaite E, Karvelis T, et al. Targeted gene editing by transfection of in vitro reconstituted Streptococcus thermophilus Cas9 nuclease complex. RNA Biol, 2015, 12 (1): 1–4. DOI:10.1080/15476286.2015.1017209 |
| [34] | Fonfara I, Le Rhun A, Chylinski K, et al. Phylogeny of Cas9 determines functional exchangeability of dual-RNA and Cas9 among orthologous type Ⅱ CRISPR-Cas systems. Nucleic Acids Res, 2014, 42 (4): 2577–2590. DOI:10.1093/nar/gkt1074 |
| [35] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nat Biotechnol, 2015, 33 (12): 1293–1298. DOI:10.1038/nbt.3404 |
| [36] | Hirano H, Gootenberg JS, Horii T, et al. Structure and engineering of Francisella novicida Cas9. Cell, 2016, 5 : 950–961. |
| [37] | Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353 (6299): 557. |
| [38] | Chiang TW, Ie Sage C, Larrieu D, et al. CRISPR-Cas9(D10A) nickase-based genotypic and phenotypic screening to enhance genome editing. Sci Rep, 2016, 6 : 24356. DOI:10.1038/srep24356 |
| [39] | Zhang B, Liu ZQ, Liu C, et al. Application of CRISPRi in Corynebacterium glutamicum for shikimic acid production. Biotechnol Lett, 2016, 38 (12): 2153–2161. DOI:10.1007/s10529-016-2207-z |
| [40] | Pan Y, Shen N, Jung-Klawitter S, et al. CRISPR RNA-guided FokI nucleases repair a PAH variant in a phenylketonuria model. Sci Rep, 2016, 6 : 35794. DOI:10.1038/srep35794 |
| [41] | Terao M, Tamano M, Hara S, et al. Utilization of the CRISPR/Cas9 system for the efficient production of mutant mice using crRNA/tracrRNA with Cas9 nickase and FokI-dCas9. Exp Anim, 2016, 65 (3): 275–283. DOI:10.1538/expanim.15-0116 |
| [42] | Slaymaker IM, et al. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351 (6288): 84–88. |
| [43] | Kleinstiver BP, Pattanayak V, Prew MS, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016, 529 (7587): 490–495. DOI:10.1038/nature16526 |
| [44] | Cao J, Wu LZ, Zhang SM, et al. An easy and efficient inducible CRISPR/Cas9 platform with improved specificity for multiple gene targeting. Nucleic Acids Res, 2016, 44 (19): e149. |
| [45] | Davis KM, Pattanayak V, Thompson DB, et al. Small molecule-triggered Cas9 protein with improved genome-editing specificity. Nat Chem Biol, 2015, 11 (5): 316–318. DOI:10.1038/nchembio.1793 |
| [46] | Fu YF, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology, 2014, 32 (3): 279–284. DOI:10.1038/nbt.2808 |
| [47] | Cho SW, Kim S, Kim Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res, 2014, 24 (1): 132–141. DOI:10.1101/gr.162339.113 |
| [48] | Lee CM, Cradick TJ, Bao G. The Neisseria meningitidis CRISPR-Cas9 system enables specific genome editing in mammalian cells. Mol Ther, 2016, 24 (3): 645–654. DOI:10.1038/mt.2016.8 |
| [49] | Müller M, Lee CM, Gasiunas G, et al. Streptococcus thermophilus CRISPR-Cas9 systems enable specific editing of the human genome. Mol Ther, 2016, 24 (3): 636–644. DOI:10.1038/mt.2015.218 |
| [50] | Kalebic N, Taverna E, Tavano S, et al. CRISPR/Cas9-induced disruption of gene expression in mouse embryonic brain and single neural stem cells in vivo. EMBO Rep, 2016, 3 : 338–348. |
| [51] | Hara S, et al. Microinjection-based generation of mutant mice with a double mutation and a 0.5 Mb deletion in their genome by the CR-ISPR/Cas9 system. J Reprod Dev, 2016, 62 (5): 531–536. DOI:10.1262/jrd.2016-058 |
| [52] | Kim S, Kim D, Cho SW, et al. Highly efficient RNA guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res, 2014, 6 : 1012–1019. |
| [53] | Liang PP, Xu YW, Zhang XY, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell, 2015, 6 (5): 363–372. DOI:10.1007/s13238-015-0153-5 |
| [54] | Hashimoto M, Yamashita Y, Takemoto T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Dev Biol, 2016, 418 (1): 1–9. DOI:10.1016/j.ydbio.2016.07.017 |
| [55] | Yang LH, Güell M, Niu D, et al. Genome-wide inactivation of porcine endogenous retroviruses (PERVs). Science, 2015, 350 (6264): 1101–1104. DOI:10.1126/science.aad1191 |
| [56] | Bialek JK, Dunay GA, Voges M, et al. Targeted HIV-1 Latency Reversal Using CRISPR/Cas9-Derived Transcriptional Activator Systems. PLoS One, 2016, 11 (6): e0158294. DOI:10.1371/journal.pone.0158294 |
| [57] | Zhu W, Xie K, Xu YJ, et al. CRISPR/Cas9 produces anti-hepatitis B virus effect in hepatoma cells and transgenic mouse. Virus Res, 2016, 217 : 125–132. DOI:10.1016/j.virusres.2016.04.003 |
| [58] | Khatodia S, Bhatotia K, Passricha N, et al. The CRISPR/Cas genome-editing tool: application in improvement of crops. Front Plant Sci, 2016, 7 : 506. |
| [59] | Bassuk AG, Zheng A, Li Y, et al. Precision Medicine: Genetic Repair of Retinitis Pigmentosa in Patient-Derived Stem Cells. Sci Rep, 2016, 6 : 19969. DOI:10.1038/srep19969 |




