2. 中国科学院烟台海岸带研究所海岸带生物学与生物资源利用重点实验室,烟台 264003;
3. 中国科学院大学,北京 100049
2. Key Laboratory of Coastal Biology and Biological Resources Utilization, Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003;
3. University of Chinese Academy of Sciences, Beijing 100049
真核微藻是一类能够进行光合作用的真核生物,起源于初级内共生事件,即异养宿主细胞内吞蓝藻,并将蓝藻进化成自身质体,主要包括灰藻、绿藻和红藻[1],以及在绿藻和红藻的基础上,进行次级或更高级别内共生事件后产生的其他真核微藻。由此,本文将红藻门和由红藻基础上进行次级或更高级别内共生事件后产生的微藻,统称为红藻体系,主要包括红藻门(Rhodophyta)以及红藻来源的隐藻门(Cryptophyta)、等鞭藻门(Haptophyta)、甲藻门(Dinophyta)和异鞭藻门(Heterokontophyta)。异鞭藻门包括金藻纲(Chrysophyceae)、硅藻纲(Bacillariophyceae)、硅鞭藻纲(Dictyochophyceae)、褐藻纲(Phaeophyceae)、褐枝藻纲(Phaeothanmiophyceae)和黄藻纲(Xanthophyceae)等[2]。同时,将绿藻门和绿藻来源的裸藻门(Euglenophyta)统称为绿藻体系。在与地球共进化的漫长历程中,为适应外界复杂逆境,真核微藻形成了一套极其精细而又复杂的信号转导系统,使其成为逆境生理研究的理想实验材料。
光是环境中重要的信号因子之一,对真核微藻的生长发育具有重要的调控作用。它不仅提供光合作用所需要的能量,自身也蕴含光质、光强和光周期等信息。真核微藻通过光受体(Photoreceptor)感知光信息,并通过复杂的接收和转导系统,调控多种生理过程,包括生物钟、生物节律、趋光性及次级代谢产物的合成与积累等过程。按照光信号的波段,目前光受体主要包括红光/远红光受体(光敏色素)、蓝光受体(隐花色素、向光素及Aureochrome)、绿光受体(视紫红质)及紫外光受体(紫外抗性蛋白)。近年来,无论从基础的微藻生态学研究,还是应用微藻生物学的发展,光信号引发的微藻光响应机制已经成为微藻光生物学研究领域的重点和热点之一。
蓝光受体(隐花色素、向光素及Aureochrome)能感受蓝光和近紫外光波段(320-400 nm),在蓝光调控植物体的多种生理过程(向光性、抑制幼茎伸长、叶绿体迁移、刺激气孔张开及调节基因表达)中发挥重要的作用[3]。蓝光对多种类型真核微藻的生理过程具有重要的影响,研究表明,与其他光质相比,同等强度的蓝光可有效诱导雨生红球藻虾青素的合成与积累[4],硅藻中的蓝光受体(Aureochrome)是一类重要的转录因子,可调控多种生理过程[5]。蓝光可调控莱茵衣藻的细胞周期循环、节律调节、叶绿素及类胡萝卜素合成等相关生理过程[6]。伴随着藻类光生物学研究的不断深入,微藻蓝光受体研究取得了长足发展。目前已知真核微藻至少存在3类蓝光受体,主要包括隐花色素(cryptochrome,Cry)、向光素(phototrophin,Phot)和AUREO(Aureoch-rome,Aureo)。本文重点综述了真核微藻中蓝光受体的研究进展,包括受体种类、分布、基因克隆、光化学特性、生理功能及光信号转导等方面,并对真核微藻蓝光受体下一步的研究重点进行了展望,以期为真核微藻蓝光受体的研究提供参考。
1 隐花色素(cryptochrome,CRY)CRY是一类非常重要的蓝光受体,几乎分布于所有物种[7],可介导植物、细菌、真菌及动物体内的光信号通路。CRY属于隐花色素/光解酶家族(cryptochrome/photolyase family,CPF),在氨基酸序列水平上与光解酶(Photolyase,PHO)存在极高相似性,尤其是N端光解酶同源区域(Photolyase-homologous region,PHR),该区域负责同源二聚化和发色团的结合,所有CRYs可结合黄素腺嘌呤二核甘酸(Flavin adenine dinucleotide,FAD),部分CRYs需结合MTHF(Methenyltetrahydrofolate)或8-HDF(8-hydroxy-7,8-didemethyl-5-deazariboflavin)。其中FAD的氧化还原状态决定CRYs的功能。
Chaves等[7]在拟南芥中发现了两个植物类型CRY(CRY1和CRY2)、一个动物类型CRY[6]和一个DASH(Drosophila,Arabidopsis,Synechocystis,and Homo)类型CRY[8]。3种类型CRYs的细胞定位、表达模式、功能及作用机制都不完全相同[7]。研究表明,拟南芥中CRY1蛋白可以在蓝光和黑暗条件发生自磷酸化,随着蓝光强度增加而磷酸化程度增高[9, 10]。但CRY2仅在蓝光下发生自磷酸化[11]。CRY介导的信号通路需要互作蛋白的参与,进而调控多种生理过程(图 1)。研究表明CRY1和CRY2可与COP1(Constitutive photomorphogenesis 1)和SPA(Suppressor of phytochrome A1)相互作用共同调控光形态建成[11-14]。除此之外,CRY2可与光敏色素PHYB(Phytochrome B)、转录因子PIF3(phytochrome interacting factors)和CIB1(CRY-interacting bHLH1)相互作用,进而调控某些光控基因的表达和植物开花过程[15, 16]。CRY1的C端结构域可与光敏色素PHYA相互作用[17],上述研究结果暗示PHY信号通路在CRY介导的信号转导中起重要作用。

|
| 图 1 高等植物来源CRY的结构域和预测的光信号转导模型 |
通常认为CRY起源于PHO,在进化过程不同的选择压力下,失去DNA修复能力而获得具有光感受和信号转导作用的新功能。CRY与PHO的一个重要区别特征是前者存在一个长度和相似性比较低的C端CCE(Cry c-terminal extension)区域,但这个特征仅存在于植物和动物类型CRYs。近年来,越来越多的研究结果对该观点提出质疑,在真菌[18, 19]、绿藻[20]及硅藻[20]中发现了具有调控基因表达作用的PHOs,同时新发现的DASH-CRY具有修复DNA的功能[21],尽管其信号转导功能还不清楚[22]。
1.1 绿藻体系中的CRY目前,真核微藻来源CRY的相关研究见表 1,主要包括植物(pCRY)、动物(aCRY)和DASH(dashCRY)3大类。在莱茵衣藻(C. reinhartii)和团藻(V. carteri)中分别发现了4个CRYs,即pCRY(Chlamydomonas photolyase homologue 1,CPH1)、aCRY和两个DASH类型[23, 24]。在高等植物中,编码pCRY的基因是多拷贝(2-5),但真核绿藻中是单拷贝,暗示基因复制在pCRY进化过程中发挥重要作用[25]。莱茵衣藻中pCRY在黑暗条件下积累,而在蓝光和红光条件下通过光依赖性蛋白酶迅速降解[26]。在团藻中,pCRY在体细胞中大量表达,而在生殖细胞中很少表达。体细胞的光合作用能力下降,还要经历细胞程序性死亡过程,因此我们推测pCRY是细胞程序性死亡信号途径中的重要成员[23],但该基因如何参与上述过程还没有报道。莱茵衣藻中pCRY(CPH1)对蓝光响应的光谱特征已经在体外进行验证[23, 24, 27]。
尽管aCRY最初仅局限于动物中,但也有该类型的基因在植物和真核微藻中发现。莱茵衣藻中aCRY的光谱特征和对蓝光和红光的响应特征也被验证[28]。比较有趣的是,aCRY不但可以感受蓝光信号,而且可以感受部分红光信号,这一功能可能弥补莱茵衣藻中缺少红光受体PHY所带来的缺陷[28]。通过构建aCRY的突变株,研究发现细胞周期循环、节律调节、叶绿素及类胡萝卜素合成相关基因在转录水平上受到其调控[28]。除此之外,在海洋来源一株绿藻(Ostreococcus tauri)中发现了aCRY(OtCPF1),同时具有修复DNA的能力,这对传统区分光解酶和隐花色素的规则提出了质疑[29]。
最近,DASH类型CRY也在绿藻中发现,证实绿藻中存在3种不同类型的CRYs[6]。对一株海洋绿藻(Ostreococcus tauri)来源的dashCRY(OtCPF2)的研究证实,其具有识别结合CPR类型DNA和修复能力[20]。同样,在另外一株海洋绿藻(Ostreococcus lucimarinus)中也发现了5个编码CPF的基因,其中CPF1属于aCRY,CPF2属于dashCRY。其他(CPF3-5)属于Ⅱ型CPD,比较奇怪的是,在这两株绿藻中都没有发现pCRY[20]。进一步研究表明,CPF1和CPF2都可非共价结合FAD作为发色团,除此之外CPF2还可结合MTHF作为第二个发色团。两者在体外都具有修复DNA的功能,但结合的DNA类型不同,CPF1可结合(6-4)类型,而CPF2可结合CPD类型。
上述研究发现,通过鉴定是否具有DNA修复功能而区分CRY和PHO是不准确的。同时也说明,在进化过程中,具有感知和信号转导功能是成为CRY的必要条件,但修复DNA的功能是否保留并不重要。同时有待进一步研究的问题如下:其一,与高等植物pCRY的多拷贝形式不同,真核微藻pCRY都是单拷贝,与高等植物多拷贝形式的功能有何异同;其二,目前海洋来源绿藻中没有发现pCRY,确实不存在还是未被发现,我们倾向于认为存在但不完全一样(未发表数据);其三,真核绿藻来源CRY的光化学特性与高等植物来源是否完全一致,是否存在藻类特异性;其四,真核绿藻来源DASH类型是否具有光信号转导功能。
1.2 红藻体系中的CRY在红藻体系中也发现了不同类型的CRYs(表 1)。pCRY首次在红藻(Cyanidioschyzon merolae)中发现[30],但后续研究表明,在甲藻和等鞭藻也发现了pCRY[31, 32]。但是,作为光合作用的生物,在硅藻(P. tricornutum)中仅存在aCRY和dashCRY类型,并不存在pCRY。取而代之是CYPD-CRY,这一类型CRY在基因序列上与传统的PHO存在区别,包括C端延伸区域,该延伸区域与高等植物来源pCRY中的DAS结构域不同,是否具有信号转导作用不清楚[34]。存在aCRY可解释为硅藻进化过程中的特殊性,其动物属性远高于植物属性[33]。最近,有研究者从三角褐指藻中发现了一个新型的CRY(CRYP),尽管与拟南芥来源的pCRY在N端500个氨基酸序列相似性上仅仅达到30%,但是他们认定为植物类型[31]。
aCRY存在于硅藻[35]、褐藻[36]和不等鞭藻门[37]。三角褐指藻来源aCRY(PtCPF1)与海洋绿藻来源aCRY一样,同时具有CRY功能和PHO功能,能够修复体内DNA的损伤。同时在光控基因表达调控过程中也发挥重要作用。构建三角褐指藻PtCPF1的过表达突变株,研究表明aCRY可调控光诱导下多种基因的表达,包括类胡萝卜素和四吡咯合成途径、碳氮代谢过程、遗传信息加工及捕光复合物的生成[7]。通过重组表达方式,该aCRY的功能在哺乳动物体内也进行了实验验证[35]。
DASH类型CRY广泛分布于红藻、硅藻、褐藻、甲藻及不等鞭藻门[6, 7, 35, 37]。其中动物类型的CRY定位于细胞核[35],而DASH类型定位于质体[38]。在红藻(C. merolae)中发现了3个编码DASH类型CRY的基因(CmPHR2,CmPHR5和CmPHR6)[39],其中CmPHR2和CmPHR5属于植物中的DASH类型,具有修复DNA的功能。与之相反,CmPHR6不具有修复任何损伤类型DNA的功能。但令人惊奇的是,在大肠杆菌中重组表达CmPHR6,与野生菌株相比,工程菌株具有更强耐受紫外光照的能力,暗示这个基因在紫外保护DNA损伤机制中发挥重要作用[39]。
红藻体系CRYs需要进一步研究的问题如下:首先,红藻体系中是否具有pCRY;其次,aCRY广泛存在于红藻体系微藻中,但具体功能和作用机制需进一步澄清;最后,广泛分布该体系的微藻中的DASH类型是否具有光受体的功能还不确定。
综上所述CRY广泛分布于所有真核微藻中,不同物种来源CRYs的系统进化分析结果表明[32],CRY基因起源于PHO基因家族,在进化过程的不同选择压力下,部分丧失修复DNA功能及获得信号转导新功能而形成。同时也说明不同种类CRYs的形成早于初级内共生事件。但真核微藻中为什么存在不同种类的CRYs,功能有何异同,作用的分子机制如何,这些是下一步真核微藻隐花色素光受体研究的热点及难点。由于CRY和PHO在序列水平上存在较大相似性,通过序列比对和结构比较预测其生物学功能是非常困难的,因此鉴定光谱特性和光响应机制是区分二者的必要手段。
2 向光素PHOT和AUREOPHOT(Phototropins)和AUREO(Aureochrome)同属于光-氧-电(light-oxygen-voltage,LOV)蓝光受体蛋白家族,该家族成员还包括植物类型PHOT、植物类型ZTL/FKF1/LKP2(Zeitlupe/Flavin-binding,kelch repeat,F-box 1/Lov kelch protein 2)及真菌WC-1(white-collar 1)蛋白[40]。LOV结构域大约包括110个氨基酸,能够结合黄素单核苷酸FMN(flavin mononucleotide)作为发色团[41],进而通过折叠形成PAS(Per-Arndt-Sim)结构域[42]。研究证实LOV蛋白在古菌、细菌和真核生物中都有发现,所有真核生物的LOV蛋白都是共同的细菌起源[40]。
拟南芥有两个向光素受体(PHOT1和PHOT2),PHOT蛋白包括N端光感知结构域和C端丝氨酸/苏氨酸激酶(STK)结构域(图 2-A),其中前者包括两个非常相似的光-氧-电LOV(light-oxygen-voltage)结构域(LOV1和LOV2),负责感知蓝光信号、结合发色团黄素单核苷酸(flavin mononucleotide,FMN)及光依赖自磷酸化,而后者负责蓝光信号转导[43-45]。蓝光可激发PHOT在体内[46]和体外[47]进行自磷酸化,但发生在分子内还是分子间目前还不清楚。PHOT光化学特性研究发现在黑暗情况下,LOV蛋白不能与FMN共价结合,从而在447 nm具有最大吸收值,形成没有活性的LOV447[43, 48],在光照情况下,FMN与LOV蛋白中保守的半胱氨酸残基进行共价结合,从而在390 nm具有最大吸收值,形成具有活性的LOV390[43, 49],两种不同的状态可发生互转。而在紫外光的照射下,可将具有活性的LOV390转化成黑暗状态下存在的无活性的LOV447,但具体的分子机制还不清楚[50]。

|
| 图 2 高等植物和绿藻来源PHOT的结构域和预测的光信号转导模型 |
PHOT的N端LOV2和C端STK结构域之间存在一个大约20个氨基酸的保守Jα螺旋区,LOV2经Jα螺旋和STK的耦合是蓝光诱导PHOT发生自磷酸化所必需的条件[43]。PHOT中已鉴定大量的磷酸化位点和互作蛋白[51-55]。自磷酸化之后的PHOT蛋白通过构象改变激活激酶活性,进而与互作蛋白共同调控多种生理反应(图 2-B)。拟南芥两个PHOTs(PHOT1和PHOT2)蛋白在功能上有重叠,但也有部分功能是特异的,如高光诱导下,PHOT2在防止光氧化损伤和叶绿体的避光反应过程中发挥重要作用。不同类型LOV蛋白在不同生物中的分布不完全一样,如PHOT仅仅发现在绿藻和高等植物中存在,而AUREO是红藻次级内共生形成的进行光合作用异鞭藻门的所特有。
2.1 绿藻体系特有PHOTPHOT蛋白仅分布高等植物和真核绿藻,目前研究的绿藻来源PHOTs,见表 2。在莱茵衣藻仅发现了一个PHOT,通过RNA干扰实验验证,其体内PHOT参与调控有性生活周期[56, 57],并能调控系列靶基因在转录水平的表达[58],包括捕光复合物、叶绿素和类胡萝卜素的生物合成相关基因[59]。其功能在拟南芥PHOT1和PHOT2的双突变株中进行回复验证,说明不同生物来源的PHOT的作用机制比较保守[60]。通过构建PHOT的突变株[61],进一步研究表明PHOT在控制莱茵衣藻眼点大小变化过程中发挥重要作用。在有光条件下,phot突变株的眼点变大,正常株眼点大小不变;而在黑暗条件下,正常株眼点变大,而突变株不变大,功能互补之后,眼点大小变大[62]。
在另外一株模式绿藻团藻中也发现了一个编码PHOT的基因,该基因的表达与细胞的发育相关,换句话说基因高表达抑制细胞增长(直径小于8 μm),最终形成体细胞;反之低表达促进细胞增长(直径大于8 μm),最终形成生殖细胞[63]。除此之外,在其他绿藻中也陆续发现了编码PHOT的基因,但对功能的研究较少,如在海洋来源绿藻(Ostreococcus tauri)中同时发现了编码PHOT的基因[64]。研究表明,在真核绿藻(Ostreococcus tauri)中同样存在PHOT介导的信号通路,但作用机制和调控的生理过程与拟南芥存在差异[53]。在其他绿藻中都存在编码PHOT的基因(未发表数据),都是单拷贝形式。关于真核微藻中LOV蛋白的系统进化表明,绿藻来源的PHOT的LOV结构域与褐藻来源的AUREO中的LOV结构域聚在一起,而不是与绿藻自身来源的LOV-HKs中的LOV结构域,说明这两种类型的LOV结构域在进化上是独立事件[65-67]。
上述研究表明,高等植物中编码PHOT的基因是多拷贝形式,而真核绿藻来源都是单拷贝形式,它们之间的功能能否进行互补,也就是说真核绿藻来源PHOT是否具有高等植物来源不同形式所具备的全部功能;真核绿藻来源的PHOT在拟南芥PHOT的双突变株中进行部分功能互补,PHOT在真核绿藻体内的功能到底如何;作用机制与高等植物是否存在差异。与拟南芥相比,真核绿藻中的PHOT参与的生物学过程和具体作用机制知之甚少,这将是下一步工作研究的重点。
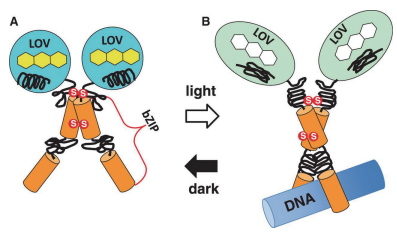
2.2 红藻体系特有AUREOAUREO是红藻体系中通过次级内共生形成的异鞭藻门中进行光合作用的藻类特有的蓝光受体,属于LOV光受体蛋白家族[40, 5],与这些藻类的生境密切相光,即生长环境中存在大量的蓝光光质。AUREO首先在黄藻纲(Vaucheria frigida)[5]、褐藻纲(Fucus distichus和Saccharina japonica)[5, 68]及硅藻纲(T. pseudonana)[5]中发现,属于蓝光受体,同时具有转录因子功能。与其他类型蓝光受体(CRY和PHOT)基于光依赖型磷酸化和自磷酸化进行信号转导不同,AUREO通过蛋白构象改变形成二聚体从而进行光感应和信号转导(图 3),详细阐述请参考文献[69-72]。
AUREO蛋白结构与LOV家族其他成员存在较大差异,主要表现为LOV存在C端,可结合发色团黄素单核苷酸FMN,而N端含有转录因子(bZIP)结构域(图 3),可结合TGACGT基因序列[5]。蓝光可促进bZIP结构域与DNA的结合度[5],说明AUREO是一类蓝光依赖型转录因子。无隔藻(Vaucheria frigida)中存在两个AUREO同源基因,分别标记VfAUREO1和VfAUREO2,在体内具有不同的功能。通过RNAi技术构建两个基因的单突变和双突变株,证实VfAUREO1能介导蓝光诱导的分枝现象,但不是必须的,同时是性器官原基形成必须的;VfAUREO2在前者的基础上进一步促进分枝形成,但不起决定性作用[5, 73]。
通过LOV结构域同源比对的方法,已经极大丰富了真核微藻中AUREO的成员(表 3),在两株硅藻(Thalassiosira pseudonana和Phaeodactylum tricornutum)中分别发现了4个不同编码AUREOs的基因[34]。来源三角褐指藻(P. tricornutum)的AUREO1a可通过细胞周期蛋白CYC2调控细胞分裂[74]。来源伪矮海链藻(T. pseudonan)的AUREO1a在提高藻体耐受紫外光照射的过程中起重要作用[73]。在其他真核微藻中也陆续发现了编码AUREO的基因,如硅藻(Ectocarpus siliculosus,Fucus distichus)、针胞藻(Chattonella antiqua)、微拟球藻(Nannochloropsis gaditana CCMP526)、棕鞭藻(Ochromonas danica)[75, 76]。但AUREO并不存在于所有异鞭藻门的藻株中,如隐藻和等鞭藻[75],进一步研究发现AUREO在非进行光合作用的鞭毛藻类中都不存在[75],这些藻株中的蓝光感知机制还有待于进一步研究。
上述研究表明,AUREO是一类非常特殊的蓝光受体,首先,AUREO分布比较特殊,仅仅是红藻体系中部分藻株存在,是否由他们的生境所决定,目前未知;其次,AUREO不仅作为蓝光受体,同时是一类非常重要的转录因子,可与特定的DNA序列相结合,进而直接调控目的基因的表达。其他类型的光受体必须首先与转录因子蛋白互作,然后才能调控基因的表达。在氨基酸序列水平上,AUREO蛋白中同样存在LOV的结构域,这与PHOT中的LOV结构域一样,说明它们具有共同的起源和进化路径。基于上述分析,我们提出AUREO可能的两种起源假说:其一,LOV和bZIP结构域的结合是形成AUREO的重要事件,这一过程的发生应该晚于红藻的次级内共生事件,仅在某些藻株中形成;其二,这一过程伴随着红藻次级内共生事件同时发生,但在后续的进化过程中发生了基因丢失现象。但上述假说需要进一步的论证。红藻体系中的AUREO作为转录因子的功能机制尚不清楚,筛选AUREO蛋白特异性结合的靶基因也是下一步研究的重点。同时组合真核绿藻PHOT中的LOV结构域和真核红藻AUREO中的bZIP结构域能否创造具有功能的AUREO也将是一个创新性的思路,该思路可为验证上述假说提供强有力的支持。
3 展望植物光受体介导的信号转导和调控生理过程的分子机制是当前国际植物学界研究的前沿和热点,取得了许多新的突破性进展。在高等植物光受体研究中,光敏色素、隐花色素、向光素和紫外光受体感知光信号、传递、转导信号及调控分子机制基本廓清。基本过程如下:生物体通过光受体感知光信号,光受体发生二聚化、磷酸化及构象改变,进一步与调控因子互作,作用于目的基因,调控基因表达,最终影响多种生理过程。尽管如此,随着对光信号转导网络的深入了解,认为光信号转导网络是异常复杂的。当前的工作应致力于寻找光信号转导途径中的重要组合或关键基因,并研究它们与转录因子之间的相互作用以及如何调控下游靶基因。另外,不同的光受体之间的相互作用和信息流的交叉也是下一步研究的热点和难点。与之相比,在真核微藻中关于光受体的研究刚刚起步,目前仅有部分微藻来源光受体的基因克隆、光化学特性鉴定及功能研究,本研究认为下一步微藻中蓝光受体的研究,应重点关注微藻蓝光受体的进化起源、功能解析及信号转导等方面,为阐明微藻蓝光受体调控微藻生理过程的分子机制等奠定科学基础。
| [1] | Gould SB, Waller RF, Mcfadden GI. Plastid evolution. Annual Review of Plant Biology, 2008, 59 : 491–517. DOI:10.1146/annurev.arplant.59.032607.092915 |
| [2] | Keeling PJ. The number, speed, and impact of plastid endosymbioses in eukaryotic evolution. Annual Review of Plant Biology, 2013, 64 (4): 583–607. |
| [3] | 范玉琴, 李德红. 植物的蓝光受体及其信号转导. 激光生物学报, 2004, 13(4): 314–320. |
| [4] | Cui HL, Yu XN, Wang Y, et al. Evolutionary origins, molecular cloning and expression of carotenoid hydroxylases in eukaryotic photosynthetic algae. Bmc Genomics, 2013, 14 (1): 135–143. DOI:10.1186/1471-2164-14-135 |
| [5] | Fumio T, Daisuke Y, Mié I, et al. Aureochrome, a photoreceptor required for photomorphogenesis in stramenopiles. Proceedings of the National Academy of Science, 2007, 104 (49): 19625–19630. DOI:10.1073/pnas.0707692104 |
| [6] | Kianianmomeni A, Hallmann A. Algal photoreceptors: in vivo functions and potential applications. Planta, 2014, 239 (1): 1–26. DOI:10.1007/s00425-013-1962-5 |
| [7] | Chaves I, Pokorny R, Byrdin M, et al. The cryptochromes: blue light photoreceptors in plants and animals. Annual Review of Plant Biology, 2011, 62 : 335. DOI:10.1146/annurev-arplant-042110-103759 |
| [8] | 孙燕, 许志茹. 植物的蓝光受体. 植物生理学报, 2008, 44(01): 144–150. |
| [9] | Jean-Pierre B, Baldissera G, Armin D, et al. Novel ATP-binding and autophosphorylation activity associated with Arabidopsis and human cryptochrome-1. European Journal of Biochemistry, 2003, 270 (14): 2921–2928. DOI:10.1046/j.1432-1033.2003.03691.x |
| [10] | Yu X, Klejnot J, Zhao X, et al. Arabidopsis cryptochrome 2 completes its posttranslational life cycle in the nucleus. Plant Cell, 2007, 19 (10): 3146–3156. DOI:10.1105/tpc.107.053017 |
| [11] | 常立, 文国琴. 植物蓝光受体研究进展. 生物技术通讯, 2004, 15(2): 169–171. |
| [12] | Wang H, Ma LG, Li JM, et al. Direct interaction of Arabidopsis cryptochromes with COP1 in light control development. Behavior Genetics, 2001, 294 (5540): 154–158. |
| [13] | Liu B, Zuo ZC, Liu HT, et al. Arabidopsis cryptochrome 1 interacts with SPA1 to suppress COP1 activity in response to blue light. Genes & Development, 2011, 25 (10): 1029–1034. |
| [14] | Zuo ZC, Liu HT, Liu B, et al. Blue light-dependent interaction of CRY2 with SPA1 regulates COP1 activity and floral initiation in Arabidopsis. Current Biology, 2011, 21 (10): 841–847. DOI:10.1016/j.cub.2011.03.048 |
| [15] | 李旭. 拟南芥蓝光受体隐花素CRY2光激发机理和功能的生化分析[D]. 长沙: 湖南大学, 2012. |
| [16] | Ahmad M, Jarillo JA, Smirnova O, et al. The CRY1 blue light photoreceptor of Arabidopsis interacts with phytochrome A in vitro. Molecular Cell, 1998, 1 (7): 939–948. DOI:10.1016/S1097-2765(00)80094-5 |
| [17] | 张云云. 拟南芥蓝光受体CRY2互作蛋白激酶的筛选及验证[D]. 长春: 吉林大学, 2015. |
| [18] | Berrocal-Tito GM, Esquivel-Naranjo EU, Horwitz BA, et al. Trichoderma atroviride PHR1, a fungal photolyase responsible for DNA repair, autoregulates its own photoinduction. Eukaryotic Cell, 2007, 6 (9): 1682–1692. DOI:10.1128/EC.00208-06 |
| [19] | Bayram O, Biesemann C, Krappmann S, et al. More than a repair enzyme: Aspergillus nidulans photolyase-like CryA is a regulator of sexual development. Molecular Biology of the Cell, 2008, 19 (8): 3254–3262. DOI:10.1091/mbc.E08-01-0061 |
| [20] | Heijde M, Zabulon G, Corellou F, et al. Characterization of two members of the cryptochrome/photolyase family from Ostreococcus tauri provides insights into the origin and evolution of cryptochromes. Plant Cell & Environment, 2010, 33 (10): 1614–1626. |
| [21] | Yihua H, Richard B, Smith BS, et al. Crystal structure of cryptochrome 3 from Arabidopsis thaliana and its implications for photolyase activity. Proceedings of the National Academy of Sciences, 2006, 103 (47): 17701–17706. DOI:10.1073/pnas.0608554103 |
| [22] | Brudler R, Hitomi K, Daiyasu H, et al. Identification of a new cryptochrome class, structure, function, and evolution. Molecular Cell, 2003, 11 (1): 59–67. DOI:10.1016/S1097-2765(03)00008-X |
| [23] | Dominik I, Ramona S, Joachim H, et al. Blue light induces radical formation and autophosphorylation in the light-sensitive domain of Chlamydomonas cryptochrome. Journal of Biological Chemistry, 2007, 282 (30): 21720–21728. DOI:10.1074/jbc.M700849200 |
| [24] | Dominik I, Richard P, Elena H, et al. Photoreaction of plant and DASH cryptochromes probed by infrared spectroscopy: the neutral radical state of flavoproteins. Journal of Physical Chemistry B, 2010, 114 (51): 17155–17161. DOI:10.1021/jp1076388 |
| [25] | Lariguet P, Dunand C. Plant photoreceptors: phylogenetic overview. Journal of Molecular Evolution, 2005, 61 (4): 559–569. DOI:10.1007/s00239-004-0294-2 |
| [26] | Reisdorph NA, Small GD. The CPH1 Gene of Chlamydomonas reinhardtii encodes two forms of cryptochrome whose levels are controlled by light-induced proteolysis. Plant Physiology, 2004, 134 (4): 1546–1554. DOI:10.1104/pp.103.031930 |
| [27] | Thomas L, Dominik I, Bernhard D, et al. Microsecond light-induced proton transfer to flavin in the blue light sensor plant cryptochrome. Journal of the American Chemical Society, 2009, 131 (40): 14274–14280. DOI:10.1021/ja901628y |
| [28] | Benedikt B, Katja P, Meike S, et al. A flavin binding cryptochrome photoreceptor responds to both blue and red light in Chlamydomonas reinhardtii. Plant Cell, 2012, 24 (7): 2992–3008. DOI:10.1105/tpc.112.098947 |
| [29] | Benedikt B, Nico M, Tilman K, et al. News about cryptochrome photoreceptors in algae. Plant Signaling & Behavior, 2013, 8 (2): 1–4. |
| [30] | Asimgil H, Kavakli IH. Purification and characterization of five members of photolyase/cryptochrome family from Cyanidioschyzon merolae. Plant Science, 2012, 185-186 (4): 190–198. |
| [31] | Juhas M, Zadow A, Spexard M, et al. A novel cryptochrome in the diatom Phaeodactylumtricornutum influences the regulation of light-harvesting protein levels. Febs Journal, 2014, 281 (9): 2299–2311. DOI:10.1111/febs.12782 |
| [32] | Oliveri P, Fortunato AE, Petrone L, et al. The Cryptochrome/Photolyase Family in aquatic organisms. Marine Genomics, 2014, 14 (1): 23–37. |
| [33] | Chris B, Allen AE, Badger JH, et al. The Phaeodactylum genome reveals the evolutionary history of diatom genomes. Nature, 2008, 456 (7219): 239–244. DOI:10.1038/nature07410 |
| [34] | Depauw FA, Rogato A, Ribera d' Alcalá M, et al. Exploring the molecular basis of responses to light in marine diatoms. Journal of Experimental Botany, 2012, 63 (4): 1575–1591. DOI:10.1093/jxb/ers005 |
| [35] | Sacha C, Manuela M, Tomoko I, et al. Diatom PtCPF1 is a new cryptochrome/photolyase family member with DNA repair and transcription regulation activity. Embo Reports, 2009, 10 (6): 655–661. DOI:10.1038/embor.2009.59 |
| [36] | Cock JM, Sterck L, Rouzé P, et al. The Ectocarpus genome and the independent evolution of multicellularity in brown algae. Nature, 2010, 465 (7298): 617–621. DOI:10.1038/nature09016 |
| [37] | Vieler A, Wu G, Tsai CH, et al. Genome, functional gene annotation, and nuclear transformation of the heterokont oleaginous alga Nannochloropsis oceanica CCMP1779. PLoS Genetics, 2012, 8 (11): e1003064. DOI:10.1371/journal.pgen.1003064 |
| [38] | Brunelle SA, Starr HE, Sotka EE, et al. Characterization of a dinoflagellate cryptochrome blue-light receptor with a possible role incircadian control of the cell cycle1. Journal of Phycology, 2007, 43 (3): 509–518. DOI:10.1111/jpy.2007.43.issue-3 |
| [39] | Asimgil H, Kavakli IH. Purification and characterization of five members of photolyase/cryptochrome family from Cyanidioschyzon merolae. Plant Science, 2012, s185-186 (4): 190–198. |
| [40] | Ulrich K, Bui Quang M, Aba L, et al. Distribution and phylogeny of light-oxygen-voltage-blue-light-signaling proteins in the three kingdoms of life. Journal of Bacteriology, 2009, 191 (23): 7234–7242. DOI:10.1128/JB.00923-09 |
| [41] | Christie JM, Salomon M, Nozue K, et al. LOV (light, oxygen, or voltage) domains of the blue-light photoreceptor phototropin (nph1): binding sites for the chromophore flavin mononucleotide. Proc Natl Acad Sci USA, 1999, 1999 : 8779–8783. |
| [42] | 乔新荣, 陈琼. 植物向光素信号通路中NPH3蛋白的研究进展. 植物生理学报, 2015(6): 829–834. |
| [43] | 乔新荣, 段鸿斌, 叶兆伟. 植物向光素受体与信号转导机制研究进展. 生物技术通报, 2014(8): 1–7. |
| [44] | Harper SM, Neil LC, Gardner KH. Structural basis of a phototropin light switch. Science, 2003, 301 (5639): 1541–1545. DOI:10.1126/science.1086810 |
| [45] | Corchnoy SB, Swartz TE, Lewis JW, et al. Intramolecular proton transfers and structural changes during the photocycle of the LOV2 domain of phototropin 1. Journal of Biological Chemistry, 2003, 278 (2): 724–731. DOI:10.1074/jbc.M209119200 |
| [46] | Briggs WR, Christie JM, Salomon M. Phototropins: a new family of flavin-binding blue light receptors in plants. Antioxidants & Redox Signaling, 2001, 3 (5): 775–788. |
| [47] | Christie JM, Swartz TE, Bogomolni RA, et al. Phototropin LOV domains exhibit distinct roles in regulating photoreceptor function. Plant Journal, 2002, 32 (2): 205–219. DOI:10.1046/j.1365-313X.2002.01415.x |
| [48] | Salomon M, Christie JM, Knieb E, et al. Photochemical and mutational analysis of the FMN-binding domains of the plant bluelight receptor, phototropin. Biochemistry, 2000, 39 (31): 9401–9410. DOI:10.1021/bi000585+ |
| [49] | Hiroko K, Takeshi K, Steen C, et al. Responses of ferns to red light are mediated by an unconventional photoreceptor. Nature, 2003, 421 (6920): 287–290. DOI:10.1038/nature01310 |
| [50] | Kennis JTM, Stokkum IHM, Van Sean C, et al. The LOV2 domain of phototropin: A reversible photochromic switch. Journal of the American Chemical Society, 2004, 126 (14): 4512–4513. DOI:10.1021/ja031840r |
| [51] | Christie JM. Phototropin blue-light receptors. Annual Review of Plant Biology, 2007, 58 (1): 21–45. DOI:10.1146/annurev.arplant.58.032806.103951 |
| [52] | Yuki T, Masayoshi N, Koji O, et al. Light-induced movement of the LOV2 domain in an Asp720Asn mutant LOV2-kinase fragment of Arabidopsis phototropin 2. Biochemistry, 2011, 50 (7): 1174–1183. DOI:10.1021/bi101689b |
| [53] | Sullivan S, Thomson CE, Lamont DJ, et al. In vivo phosphorylation site mapping and functional characterization of Arabidopsis phototropin 1. Molecular Plant, 2008, 1 (1): 178–194. DOI:10.1093/mp/ssm017 |
| [54] | Shin-Ichiro I, Toshinori K, Masaki M, et al. Blue light-induced autophosphorylation of phototropin is a primary step for signaling. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105 (14): 5626–5631. DOI:10.1073/pnas.0709189105 |
| [55] | Sullivan S, Petersen J, Blackwood L, et al. Functional characterization of Ostreococcus tauri phototropin. New Phytologist, 2015 . DOI:10.1111/nph.13640 |
| [56] | Huang K, Merkle T, Beck CF. Isolation and characterization of a Chlamydomonas gene that encodes a putative blue-light photoreceptor of the phototropin family. Physiologia Plantarum, 2002, 115 (4): 613–622. DOI:10.1034/j.1399-3054.2002.1150416.x |
| [57] | Kasahara M, Swartz TE, Olney MA, et al. Photochemical properties of the flavin mononucleotide-binding domains of the phototropins from Arabidopsis, rice, and Chlamydomonas reinhardtii. Plant Physiology, 2002, 129 (2): 762–773. DOI:10.1104/pp.002410 |
| [58] | Huang K, Beck CF. Photoropin is the blue-light receptor that controls multiple steps in the sexual life cycle of the green alga Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences, 2003, 100 : 762–73. |
| [59] | Chung-Soon I, Stephan E, Huang K, et al. Phototropin involvement in the expression of genes encoding chlorophyll and carotenoid biosynthesis enzymes and LHC apoproteins in Chlamydomonas reinhardtii. Plant Journal, 2006, 48 (1): 1–16. DOI:10.1111/tpj.2006.48.issue-1 |
| [60] | Christie JM. Phototropin blue-light receptors. Annual Review of Plant Biology, 2007, 58 (4): 21–45. |
| [61] | Zorin B, Lu Y, Sizova I, et al. Nuclear gene targeting in Chlamydomonas as exemplified by disruption of the PHOT gene. Gene, 2009, 432 (s1-2): 91–96. |
| [62] | Jessica T, Andre G, Jana S, et al. Phototropin influence on eyespot development and regulation of phototactic behavior in Chlamydomonas reinhardtii. Plant Cell, 2012, 24 (11): 4687–4702. DOI:10.1105/tpc.112.103523 |
| [63] | Berthold P, Tsunoda SP, Oliver P, et al. Channelrhodopsin-1 initiates phototaxis and photophobic responses in Chlamydomonas by immediate light-induced depolarization. Plant Cell, 2008, 20 (6): 1665–1677. DOI:10.1105/tpc.108.057919 |
| [64] | El-Batoul DT, Christie JM, Sophie SF, et al. A eukaryotic LOV-histidine kinase with circadian clock function in the picoalga Ostreococcus. Plant Journal, 2011, 65 (4): 578–588. DOI:10.1111/tpj.2011.65.issue-4 |
| [65] | Losi A, Gärtner W. The evolution of flavin-binding photoreceptors: an ancient chromophore serving trendy blue-light sensors. Annual Review of Plant Biology, 2012, 63 (3): 49–72. |
| [66] | Losi A, Gärtner W. Old chromophores, new photoactivation paradi-gms, trendy applications: flavins in blue light-sensing photorece-ptors. Photochemi Photobiol, 2011, 87 (3): 491–510. DOI:10.1111/php.2011.87.issue-3 |
| [67] | Tseng TS, Frederickson MA, Briggs WR, et al. Light-activated bacterial LOV-domain histidine kinases. Methods in Enzymology, 2010, 471 : 125–134. DOI:10.1016/S0076-6879(10)71008-9 |
| [68] | Deng YY, Yao JT, Fu G, et al. Isolation, expression, and characterization of blue light receptor Aureochrome gene from Saccharina japonica(Laminariales, Phaeophyceae). Marine Biotechnology, 2013, 16 (2): 135–143. |
| [69] | Osamu H, Ken T, Kazunori Z, et al. Blue light-induced conformational changes in a light-regulated transcription factor, Aureochrome-1. Plant & Cell Physiology, 2013, 54 (1): 93–106. |
| [70] | Herman E, Sachse M, Kroth PG, et al. Blue-light-induced unfolding of the Jα helix allows for the dimerization of aureochrome-LOV from the diatom Phaeodactylum tricornutum. Biochemistry, 2013, 52 (18): 3094–3101. DOI:10.1021/bi400197u |
| [71] | Mitra D, Yang X, Moffat K. Crystal structures of aureochrome1 LOV suggest new design strategies for optogenetics. Structure, 2012, 20 (4): 698–706. DOI:10.1016/j.str.2012.02.016 |
| [72] | 黄一江. 微拟球藻NgLACS和NgAUREO1基因的克隆表达与功能分析[D]. 泰安: 山东农业大学, 2014. |
| [73] | Noriyuki S, Masamitsu W. Evolution of three LOV blue light receptor families in green plants and photosynthetic stramenopiles: phototropin, ZTL/FKF1/LKP2 and aureochrome. Plant & Cell Physiology, 2013, 54 (1): 8–23. |
| [74] | Huysman MJJ, Fortunato AE, Michiel M, et al. Aureochrome1a-mediated induction of the diatom-specific cyclin dsCYC2 controls the onset of cell division in diatoms (Phaeodactylum tricornutum). Plant Cell, 2013, 25 (1): 215–228. DOI:10.1105/tpc.112.106377 |
| [75] | Mié I, Fumio T, Hisayoshi N, et al. Distribution and phylogeny of the blue light receptors aureochromes in eukaryotes. Planta, 2009, 230 (3): 543–552. DOI:10.1007/s00425-009-0967-6 |
| [76] | Gaditana N, Radakovits O, Jinkerson RE, et al. Draft genome sequence and genetic transformation of the oleaginous alga. Nature Communications, 2012, 3 (2): 686–698. |