转座子(transposon,Tn)是真核生物基因组中普遍存在的可移动和自主复制的DNA重复序列,1950年由著名遗传学家McClintock首先在玉米中发现[1]。根据转座机制不同,转座子可分为反转座子和DNA转座子两大类型。以前认为转座子是无功能的“垃圾DNA”,但目前研究表明,转座子在植物基因组进化、基因表达调控、系统发育和遗传多样性评价方面具有重要作用。
分离、鉴定植物转座子并深入研究其基因调控功能,对利用生物技术开展作物遗传改良具有较高的应用价值。如淀粉合成酶(granule-bound starch synthase,GBSSI)基因决定植物中直链淀粉的合成,转座子通过诱发玉米GBSSI基因突变形成等位基因,从而改良了淀粉品质[2]。然而,对于转座子是如何调控植物基因表达这一问题认识还较为模糊。近年来,国内外学者在植物转座子类型和结构特征、活性及其影响因素、预测方法及标记开发等方面取得一系列成果,跟踪和分析植物转座子的最新研究进展对全面了解转座子的功能具有重要意义。为了理清植物转座子与基因表达调控关系,本文综述了植物转座子分类、起源和转座机制以及转座子与宿主基因组间的表观遗传互作,阐述了不同转座子对基因表达调控方式,并对今后研究前景进行了展望。
1 植物转座子根据转座机制不同,转座子可分为Class Ⅰ元件(反转座子)和Class Ⅱ元件(DNA转座子)两大类型。转座子转座引起插入突变、产生新的基因、产生染色体畸变、引起生物进化,从而引发许多遗传变异。
反转座子(retrotransposon)是一类以RNA为中间媒介,通过“复制-粘贴”方式转座的多拷贝、高度异质性的移动元件。根据是否含有长末端重复序列(long terminal repeat,LTR)可分为LTR-反转座子和非LTR-反转座子。其中,LTR-反转座子主要分为Ty1-copia和Ty3-gypsy两个亚家族,非LTR-反转座子则包括长散布重复序列(long interspersed repetitive element,LINE)和短散布重复序列(short interspersed repetitive element,SINE)两类,LINE负责SINE的转座。反转座子不仅可以在世代中纵向传递,也可在物种间进行水平转移,或者逆转录错误,从而产生高度异质性。在芸薹属近缘种中,转座子的扩增差异造成甘蓝基因组比白菜基因组大,但白菜基因组中转座子插入基因内部频率要高于甘蓝基因组,完整的LTR比单独的LTR具有更强的读出活性,DNA转座子更偏向于插入或保留在基因的内含子中[3]。
DNA转座子(DNA transposon)是通过DNA中间体,以“剪切-粘贴”方式进行转座的单拷贝的转座元件。根据能否自主转座,又可分为自主型DNA转座子和非自主DNA转座子,后者只有在自主型DNA转座子存在时才能进行转座,而前者自身可以编码转座酶进行转座。微小倒置重复转座元件(miniature inverted repeat transposable element,MITE)是一类具有反转座子活性的高拷贝的非自主DNA转座子,1992年,由Bureau等在玉米中发现[4],被认为是基因组进化的驱动力,其数量与植物基因组大小呈显著相关。如玉米基因组的Ac/Ds和Spm/En转座系统[5]。
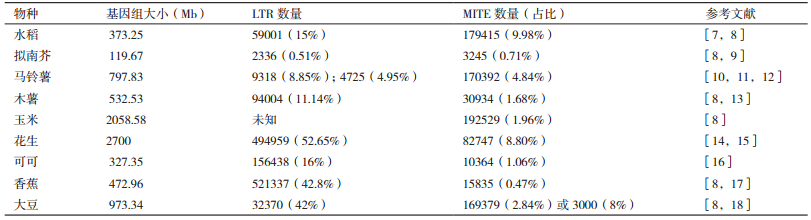
根据转座子的转座酶氨基酸序列差异,高等植物转座子超家族包括LTR反转座子、hAT、CACTA因子、Mutator和MULEs、Tc1/mariner、MITEs等[6]。此外,转座子也可分为插入序列(insertional sequence,IS)、复合转座子和复杂转座子。一些常见植物基因组大小、不同类型转座子数量,见表 1。
转座子是基因组的重要组成部分,可能起源于病毒或逆转录病毒退化[19]。一些研究表明,MITEs起源于对应的自主型转座子的部分缺失。转座子活性受发育过程和环境调节,正常情况下大多数转座子不具有活性。DNA甲基化酶可以抑制反转座子的迁移,转录后调控机制也引起反转座子转录沉默[20]。
通过在靶DNA上造成交错切口,转座子与突出的末端相连,填补缺口,这就是转座。转座子的转座机制包括复制转座、非复制转座和保守型转座。复制转座是转座子先复制一份拷贝,在解离酶将复制的拷贝解离后,通过转座酶将拷贝转座到新的位置,原先位置仍然保留原来的转座子,这种方式可引起宿主基因组扩增。非复制转座,也叫物理性运动,是在转座酶作用下,将转座子直接从原来位置转座插入新的位置并保留在插入位置上。保守型转座是一种特殊的非复制转座,只是序列的直接移动,没有核苷酸键的损失,原位点没有链断裂。
转座子有重要的生物学功能。Tn的插入为旁侧基因引入表观遗传学标记,Tn的表达和转座均影响基因表达调控。Tn和基因的表观遗传学互作在转座子-宿主基因组的共同进化中扮演关键较色,即转座子通过表观遗传学机制发生作用。图 1表明,Tn首先可能通过水平转移或基因组杂交进入宿主基因组;在共进化过程中,Tn可能被转录激活、转座和再次沉默;最终一些Tn可能通过自我选择性删除或重组从宿主基因组中被去除。宿主则通过转录水平的基因沉默(transcriptional gene silencing,TGS)和转录后水平的基因沉默(posttranscriptional gene silencing,PTGS)机制对Tn发挥表观遗传调控作用[21]。环境刺激和硝普钠、茉莉酸甲酯诱导等可以激活转座子,而自身DNA甲基化抑制反转录转座子转座[22, 23]。小RNA是一种重要的基因表达调控因子,在正常发育过程中转座元件表达产生小RNA。不同的转座子会产生不同的小RNA,用来调控不同的基因表达[24]。piRNA途径是一种抑制转座子活性的机制,转座子和piRNA途径广泛影响染色质结构和基因表达[25]。与基因组其他非编码部分相比,Tn特别适合基因调控功能。尽管新Tn插入效应对个体有害,但对群体总体来说是有利的[26]。

|
| 图 1 转座子与宿主基因组间的表观遗传互作 |
反转座子是植物中最不稳定的遗传元件,可以调控基因表达。一个长10 422 bp的Ty3-gypsy反转座子插入红皮葡萄mybA1基因启动子,导致该基因发生突变不能合成花青苷,从而形成白皮葡萄[27]。高注量的N+辐照虽然不增加小麦反转座子Wis2-1A在基因组上的相对拷贝数,但通过提高Wis2-1A的转录水平使其邻近基因发生沉默[28]。LTR反转座子Rider介导控制番茄果实形状基因Sun发生重组,基因拷贝数产生差异,从而造成野生种LA1589和栽培种Sun1642果实形状发生改变[29]。反转座子插入到控制成花的拟南芥LEAFY基因中,导致第8代中有些个体无雄性花器官[20]。Copia-like反转座子插入血橙RUBY基因上游,调控该基因表达,促进果实变红(表 2)[30]。
转座子对宿主基因表达调控作用与转座子插入位置有关,相关研究主要集中在DNA转座子。
当插入到基因的启动子序列时可能导致基因沉默,如某些MITE可作为顺式作用元件影响基因表达[31]。水稻IR24品系ubiquitin2(rubq2)启动子包含两个MITE元件(Kiddo和MDM1),而T309品系rubq2启动子缺失Kiddo,由于Kiddo对基因转录的增强效应被特异序列的表观遗传修饰减弱,导致两者rubq2转录水平没有差异[32]。由于拟南芥精氨酸脱羧酶ADC1启动子有一个含有许多转录因子结合基序的MITE元件,表现出与同源基因ADC2不同的表达模式[33]。5'端的Tourist C元件与过氧化氢酶(catalase)基因在水稻花器官中表达密切相关[34]。插入野生大麦Hsdr4启动子中的MITE使该基因参与脱水胁迫耐性[35]。
当插入到基因3'和5'非翻译区或内含子时可能会影响基因的转录、转录后加工和翻译,有时还会影响基因表达的发育阶段性和组织特异性。Hbr元件插入玉米抗病基因hm的3'端,影响mRNA的稳定性使该基因失活[36]。一个长185 bp的MITE插入玉米fie2基因3'端,从而影响该基因的正常表达[37]。一个长205 bp MITE插入花生FAD2B基因编码区3'端,降低ahFAD2B基因转录水平,这可能是产生高油酸花生突变体Mycogen-Flavo和M2-225的原因[38]。一个长11.1 kb的类转座子元件插入小麦MATE1B基因3'端,增强该基因在根尖表达,促进柠檬酸分泌提高小麦耐铝性[39]。一个长276 bp的类Tourist插入小麦HSP16.9-3A基因的3'端,增强了该基因的转录从而提高小麦耐热性[40, 41]。
当插入到基因的编码序列时可能导致基因沉默。MITE插入玉米过氧化酶Pox3基因第二个外显子,引起编码区剪接位置发生改变,影响了该基因的正常表达[42]。MATE是一个负责高粱耐铝位点Alt(SB)的一个铝激活柠檬酸转运子,其调节区的多态性可能有助于大规模等位基因效应,通过诱导根尖分泌柠檬酸增加耐铝性[43]。在油菜FLC.A10基因上游MITE插入或删除引起的等位基因多样性是冬油菜和春油菜基因分型的主要原因[44]。
核基质亲和性也与转座子对基因表达影响有关,具有核基质附着区的mPing插入水稻Rurm1基因导致颖壳变小[45]。
MITEs可以上调附近基因,也能形成dsRNA产生小RNA下调基因表达。有研究表明,在拟南芥中,转座子的变异可能影响基因表达进而调控基因的功能和表型[46]。怎么验证转座子序列的变异引起了表型的变异,这是一个挑战。
4 结语转座子是植物产生变异的重要来源,不同的转座子通过不同的转座机制插入宿主基因组,影响基因表达和功能,在基因组进化研究方面具有重要理论意义。此外,因在基因组中分布广泛、重复性好、高多态性和共显性等优点,转座子也可应用于生物多样性及遗传连锁分析、功能基因鉴定和植物性状改良等方面。反转座子可用于高通量分子标记技术开发,如特异序列扩增多态性(sequence-specific amplification polymorphism,S-SAP)、反向反转座子扩增多态性(inter-retrotransposon amplified polymorphism,IRAP)[47]、反转座子-微卫星扩增多态性(retrotransposon-microsatellite amplified polymorphism,REMAP)[48]和基于反转座子的插入多态性(retrotransposon-based insertion polymorphism,RBIP),在植物遗传多样性、系统进化、品种鉴定、遗传连锁图谱构建和基因定位等研究领域发挥重大作用。利用转座子标签体系还可以鉴定功能基因,即通过转座子插入得到的突变体,经分子分析证实后,可用转座子作探针,通过杂交从基因库中克隆到被标签的基因。通过应用于功能基因组学研究,转座子标签法已成为研究植物基因功能的重要技术平台。
转座子的自身调控机制是多方面的包括转座子系统本身介导的调控、宿主介导的调控、重复诱导的基因沉默和环境影响,这说明它的调控是相当复杂的,尚需进一步深入研究。金鱼草转座子Tam3转座依赖于低温,Tam3的甲基化水平受其转座酶活性控制[49]。植物是怎么控制转座子的激活;与动物一样,植物中是否也存在抑制转座子活性的piRNA途径;不同转座子间是否存在互作;高通量测序技术特异性好、灵敏度和精确性高,目前已被广泛应用于转座子研究,在揭示转座子转座机制和生物学功能等方面取得了很大的发展[50],也必将推动这些问题的解答。
| [1] | McClintock B. The origin and behavior of mutable loci in maize. Proc Natl Acad Sci USA, 1950, 36 : 344–355. DOI:10.1073/pnas.36.6.344 |
| [2] | 姚新灵, 白桦. 玉米转座子、GBSSI基因及表达研究进展. 生物技术, 2000, 10(4): 36–39. |
| [3] | 赵美霞, 张彪, 刘胜毅, 等. 白菜和甘蓝基因组转座子表达及其对基因调控的潜在影响. 遗传, 2013, 35(8): 1014–1022. |
| [4] | Bureau TE, Wessler SR. Tourist: a large family of small inverted repeat elements frequently associated with maize genes. Plant Cell, 1992, 4 (10): 1283–1294. DOI:10.1105/tpc.4.10.1283 |
| [5] | 温小杰, 张学勇, 郝晨阳, 等. MITE转座元件在植物中的研究进展. 中国农业科学, 2008, 41(8): 2219–2226. |
| [6] | 李宏, 黄晓天. 高等植物转座子的超家族及其应用. 亚热带植物科学, 2010, 39(3): 93–96. |
| [7] | 高东迎, 何冰, 孙立华. 水稻转座子研究进展. 植物学通报, 2007, 24(5): 667–676. |
| [8] | Chen J, Hu Q, Zhang Y, et al. P-MITE: a database for plant miniature inverted-repeat transposable elements. Nucleic Acids Res, 2014, 42 : D1176–D1181. DOI:10.1093/nar/gkt1000 |
| [9] | Wang X, Weigel D, Smith LM. Transposon variants and their effects on gene expression in Arabidopsis. PLoS Genetics, 2013, 9 (2): e1003255. DOI:10.1371/journal.pgen.1003255 |
| [10] | Momose M, Abe Y, Ozeki Y. Miniature inverted-repeat transposable elements of Stowaway are active in potato. Genetics, 2010, 186 : 59–66. DOI:10.1534/genetics.110.117606 |
| [11] | 许莹修, 杜建厂. 马铃薯 (Solanum tuberosum L.) 全基因组水平上LTR-逆转座子的鉴定与进化分析. 基因组学与应用生物学, 2013, 32(6): 734–742. |
| [12] | 李智菲, 李卫涛, 押辉远. 马铃薯全基因组LTR反转录转座子分析. 湖北农业科学, 2013, 52(17): 4235–4237. DOI:10.3969/j.issn.0439-8114.2013.17.061 |
| [13] | Wang W, Feng B, Xiao J, et al. Cassava genome from a wild ance-stor to cultivated varieties. Nature Communications, 2014, 5 : 5110. DOI:10.1038/ncomms6110 |
| [14] | Shirasawa K, Hirakawa H, Tabata S, et al. Characterization of active miniature inverted-repeat transposable elements in the peanut genome. Theor Appl Genet, 2012, 124 : 1429–1438. DOI:10.1007/s00122-012-1798-6 |
| [15] | Bertioli DJ, Cannon SB, Froenicke L, et al. The genome sequences of Arachis duranensis and Arachis ipaensis, the diploid ancestors of cultivated peanut. Nature Genetics, 2016, 48 (4): 436–446. |
| [16] | Sveinsson S, Gill N, Kane NC, et al. Transposon fingerprinting using low coverage whole genome shotgun sequencing in Cacao (Theobroma cacao L.) and related species. BMC Genomics, 2013, 14 : 502. DOI:10.1186/1471-2164-14-502 |
| [17] | D'Hont A, Denoeud F, Aury JM, et al. The banana (Musa acuminata) genome and the evolution of monocotyledonous plants. Nature, 2012, 488 : 213–219. DOI:10.1038/nature11241 |
| [18] | Du J, Grant D, Tian Z, et al. SoyTEdb: A comprehensive database of transposable elements in the soybean genome. BioMed Central Genomics, 2010, 11 : 113. |
| [19] | 李宏. 转座子的起源及其和物种进化的关系. 渝州大学学报, 1993, 25(1): 47–56. |
| [20] | Mirouze M, Reinders J, Bucher E, et al. Selective epigenetic control of retrotransposition in Arabidopsis. Nature, 2009, 461 (7262): 427–430. DOI:10.1038/nature08328 |
| [21] | Bucher E, Reinders J, Mirouze M. Epigenetic control of transposon transcription and mobility in Arabidopsis. Curr Opin Plant Biol, 2012, 15 (5): 503–510. DOI:10.1016/j.pbi.2012.08.006 |
| [22] | 梁楠松, 曾凡锁, 李博, 等. 硝普钠 (SNP) 和茉莉酸甲酯 (MeJA) 诱导的白桦Ty1-copia类转座子的克隆及分析. 东北林业大学学报, 2013, 41(10): 69–74. DOI:10.3969/j.issn.1000-5382.2013.10.015 |
| [23] | 蒋爽, 滕文元, 宗宇, 等. 植物LTR反转录转座子的研究进展. 西北植物学报, 2013, 33(11): 2354–2360. |
| [24] | Martinez G, Slotkin RK. Developmental relaxation of transposable element silencing in plants: functional or byproduct?. Curr Opin Plant Biol, 2012, 15 (5): 496–502. DOI:10.1016/j.pbi.2012.09.001 |
| [25] | Sienski G, Donertas D, Brennecke J. Transcriptional silencing of transposons by Piwi and maelstrom and its impact on chromatin state and gene expression. Cell, 2012, 151 (1): 964–980. |
| [26] | Rebollo R, Romanish MT, Mager DL. Transposable elements: an abundant and natural source of regulatory sequences for host genes. Annu Rev Genet, 2012, 46 : 21–42. DOI:10.1146/annurev-genet-110711-155621 |
| [27] | Kobayashi S, Goto-Yamamoto N, Hirochika H. Retrotransposon-induced mutations in grape skin color. Science, 2004, 304 (5673): 982. DOI:10.1126/science.1095011 |
| [28] | 押辉远, 谷运红, 王卫东, 等. 低能N+辐照提高小麦反转录转座子Wis2-1A的转录并影响其邻近基因表达. 核农学报, 2008, 22(5): 595–599. |
| [29] | Han X, Ning J, Erin S, et al. Retrotransposon-mediated gene duplication underlies morphological variation of tomato fruit. Science, 2008, 319 : 1527–1530. DOI:10.1126/science.1153040 |
| [30] | Butelli E, Licciardello C, Zhang Y, et al. Retrotransposons control fruit-specific, cold-dependent accumulation of anthocyanins in blood organges. Plant Cell, 2012, 24 (3): 1242–1255. DOI:10.1105/tpc.111.095232 |
| [31] | Yang G, Dong J, Chandrasekharan MB, et al. Kiddo, a new transposable element family closely associated with rice genes. Mol Genet Genomics, 2001, 266 (3): 417–424. DOI:10.1007/s004380100530 |
| [32] | Yang GJ, Lee YH, Jiang YM, et al. A two-edged role for the transposable element kiddo in the rice ubiquitin2 promoter. Plant Cell, 2005, 17 (5): 1559–1568. DOI:10.1105/tpc.104.030528 |
| [33] | El Amrani, Marie L, Ainouche A, et al. Genome-wide distribution and potential regularoty functions of AtATE, a novel family of miniature inverted-repeat transposable elements in Arabidopsis thaliana. Mol Genet Genomics, 2002, 267 : 459–471. DOI:10.1007/s00438-002-0675-4 |
| [34] | Iwamoto M, Higo K. Tourist C transposable elements are closely associated with genes expressed in flowers of rice (Oryza sativa). Mol Genet Genomics, 2003, 268 : 771–778. |
| [35] | Suprunova T, Krugman T, Distelfeld A, et al. Identification of a novel gene (Hsdr4) involved in water-stress tolerance in wild barley. Plant Mol Biol, 2007, 64 : 17–34. DOI:10.1007/s11103-006-9131-x |
| [36] | Tu Z. Three novel families of miniature inverted-repeat transposable elements are associated with genes of the yellow fever mosquito, Aedes aegypti. Proc Natl Acad Sci USA, 1997, 94 (14): 7475–7480. DOI:10.1073/pnas.94.14.7475 |
| [37] | Danilevskaya ON, Hermon P, Hantke S, et al. Duplicated fie genes in maize: expression pattern and imprinting suggest distinct functions. Plant Cell, 2003, 15 : 425–438. DOI:10.1105/tpc.006759 |
| [38] | Patel M, Jung S, Moore K, et al. High-oleate peanut mutants result from a MITE insertion into the FAD2 gene. Theor App Genet, 2004, 108 (8): 1492–1502. DOI:10.1007/s00122-004-1590-3 |
| [39] | Tovkach A, Ryan PR, Richardson AE, et al. Transposon-mediated alteration of TaMATE1B expression in wheat confers constitutive citrate efflux from root apices. Plant Physiol, 2013, 161 : 880–892. DOI:10.1104/pp.112.207142 |
| [40] | Li JT, Wang ZZ, Peng HR, et al. A MITE insertion into the 3'-UTR regulates the transcription of TaHSP16.9 in common wheat. The Crop J, 2014, 2 (6): 381–387. DOI:10.1016/j.cj.2014.07.001 |
| [41] | 李静婷, 王健胜, 杨风岭. 小麦MITE转座子对sHSP基因的表达调控研究. 江苏农业科学, 2015, 43(12): 29–32. |
| [42] | Guillet-Claude C, Birolleau-Touchard C, Manicacci D, et al. Nucleotide diversity of the Zmpox3 maize peroxidase gene: relationships between a MITE insertion in exon 2 and variation in forage maize digestibility. Biomed Centr Genet, 2004, 5 (19): 1–11. |
| [43] | Magalhaes JV, Liu J, Guimaraes CT, et al. A gene in the multidrug and toxic compound extrusion (MATE) family confers aluminum tolerance in sorghum. Nature Gene, 2007, 39 (9): 1156–1161. DOI:10.1038/ng2074 |
| [44] | Hou J, Long Y, Raman H, et al. A tourist-like MITE insertion in the upstream region of the BnFLC. A10 gene is associated with vernalization requirement in rapeseed (Brassica napus L.). BMC Plant Biol, 2012, 12 (1): 238–250. DOI:10.1186/1471-2229-12-238 |
| [45] | Kikuchi K, Terauchi K, Wada M, et al. The plant MITE mPING is mobilized in anther culture. Nature, 2003, 421 : 167–170. DOI:10.1038/nature01218 |
| [46] | Wang X, Weigel D, Smith LM. Transposon variants and their effects on gene expression in Arabidopsis. PLoS Genet, 2013, 9 (2): e1003255. DOI:10.1371/journal.pgen.1003255 |
| [47] | Nair AS, Teo CH, Schwarzacher T, et al. Genome classification of banana cultivars from South India using IRAP markers. Euphytica, 2005, 144 : 285–290. DOI:10.1007/s10681-005-7321-2 |
| [48] | Branco CJS, Vieira EA, Malone G, et al. IRAP and REMAP assessments of genetic similarity in rice. J Appl Genet, 2007, 48 (2): 107–113. DOI:10.1007/BF03194667 |
| [49] | Hashida SN, Uchiyama T, Martin C, et al. The temperature-dependent change in methylation of the Antirrhinum transposon Tam3 is controlled by the activity of its transposase. Plant Cell, 2006, 18 : 104–118. DOI:10.1105/tpc.105.037655 |
| [50] | 刘振, 徐建红. 高通量测序技术在转座子研究中的应用. 遗传, 2015, 37(9): 885–898. |