2. 中国人民解放军南京军区军事医学研究所,南京 210002
2. Institute of Military Medical Sciences, Nanjing Command, Chinese People's Liberation Aarmy, Nanjing 210002
猪链球菌2型(S. suis 2)是猪链球菌35种血清型中致病性最强、分布最广的烈性人畜共患病原菌。近几十年,S. suis 2已在欧洲、美洲、亚洲的几十个国家陆续引发了200多例散发的人群恶性感染事件[1]。尤其在2005年的中国四川省人群中暴发了规模巨大的S. suis 2疫情,大量患者出现了链球菌高侵袭、深部组织感染的罕见毒性休克综合征(STSS),病死率高达62%-82%[2]。北京中科院通过流行病学调查、动物模型、多重PCR及基因生物信息学分析等方法,确认了S. suis 2是四川省猪链疫情暴发和STSS出现的原因[3]。
大量研究表明暴露在病原菌细胞壁外的蛋白不仅参与细菌的免疫防御机制,也涉及细菌侵染宿主过程中一系列环节,如黏附、侵袭、定植及穿梭等[4, 5]。近年报道一些在细菌体内参与基础代谢的蛋白,大都缺乏信号肽序列和细胞壁锚定结构,但也能出现在细胞表面[6, 7]。烯醇化酶(Enolase,ENO)似乎就是如此,在细菌细胞质中是一个参与糖代谢的金属酶,也能在许多病原菌胞壁外参与感染过程[10]。本研究在前期已完成S. suis 2 05ZYH33(四川省资阳2005年疫区分离菌株)全基因组的测定,在此基础上发现了CDS SSU1503基因序列与多种细菌Enolase编码基因高度同源,故推断为Enolase疑似编码序列(eno),进而本研究对eno基因进行序列信息分析和克隆表达,检测eno基因编码蛋白Enolase在菌内的酶活性和菌外的定位,并对蛋白引发的免疫抑制作用进行探究。
1 材料与方法 1.1 材料本研究所用实验材料及其来源如表 1所示。
利用BLAST(ExPASy提供)进行Enolase蛋白质序列相似度比对,在蛋白序列数据库中利用PSIBLAST程序搜索序列显著相似的Enolase同源序列。再利用Clustal X软件开展多重序列比对以及分子遗传分析软件生成Enolase的系统进化树。利用软件SignalP 4.1和TMHMM 2.0分别预测S. suis 2 05ZYH33中Enolase的信号肽及跨膜区结构。
1.2.2 Enolase分子克隆根据S. suis 2 05ZYH33 Enolase序列设计合成引物(DNAStar软件设计,上海芃硕生物科技有限公司合成),进行PCR扩增。上游引物为5' -GCGGATCC ATGTCAATTATTACTG-3' (前端带有BamH Ⅰ切割位点);下游引物为5' -GCCTCGA GTATGGATTTACCTGTTA G-3' (前端带有Xho Ⅰ切割位点)。PCR程序:95℃预变性2 min;循环30次:94℃变性40 s,55℃复性35 s,72℃延伸1 min;72℃延伸7 min。PCR产物经1%琼脂糖凝胶电泳和试剂盒回收回收后与pMD-18T载体连接,并转化进入感受态E. coli DH5α。菌液PCR阳性者提取质粒进行酶切鉴定。用BamH Ⅰ和Xho Ⅰ分别对pMD-18T∷eno和pET32a进行双酶切,用T4 DNA连接酶连接酶切后的目的片段,构建成表达质粒pET32a∷eno,再次转化进入感受态E. coli DH5α,菌液PCR阳性者提取重组质粒,单酶切(BamH Ⅰ)和双酶切(BamH Ⅰ和Xho Ⅰ)处理后,琼脂糖电泳后经凝胶成像仪鉴定阳性者由上海伯豪生物技术公司测序。
1.2.3 Enolase表达和纯化提取重组表达载体pET32a∷eno转化进入感受态E. coli BL21,IPTG(异丙基-β-D-半乳糖苷)诱导后,用无菌PBS重悬离心后菌体沉淀,进行超声裂解细菌,离心收集上清,经12% SDS-PAGE电泳、染色和脱色后,鉴定Enolase是否表达。大量培养重组菌E. coli BL21(含pET32a∷eno)并诱导表达,超声破碎菌液离心取上清与His标签纯化树脂孵育过夜。次日将孵育物装进层析柱中,PBS平衡后,用5 mmol/L咪唑洗脱非特异性蛋白,用100 mmol/L咪唑洗脱Enolase目的蛋白。目的蛋白经快速蛋白液相层析(FPLC)法进一步得到纯化,再次用SDS-PAGE电泳分析。
1.2.4 Enolase酶活性测定酶催化体系为:重组Enolase(5 μg),2-PGE(2-磷酸甘油酸盐,以0.25-12 mmol/L的浓度逐渐增加),HEPES(100 mmol/L,pH 7.0),MgCl2(10 mmol/L),KCl(7.7 mmol/L)。每个浓度37℃条件下反应5 min,测定OD240nm。以2-PGE浓度为横坐标,以OD值为纵坐标,绘出米氏方程曲线,再根据Lineweaver-Burk双倒数法计算Enolase的米氏常数Km。
1.2.5 流式细胞术定位分析将培养的05ZYH33菌液调整到108 CFU/mL,取1 mL菌液离心后用0.01 mol/L PBS洗沉淀3次,并重悬于100 μL PBS中,细菌总数约为108个。设实验组和对照组,实验组加入100倍稀释的大鼠抗Enolase蛋白多抗血清(对照组只加入大鼠阴性血清),4℃孵育1 h,离心后用0.01 mol/L PBS洗沉淀3次再重悬于100 μL PBS中,加入50倍稀释的FITC-兔抗大鼠IgG,4℃孵育1 h,离心后用PBS洗涤沉淀4次并重悬,最后用4%多聚甲醛进行固定。用荧光激活细胞分选(FACS)技术检测。
1.2.6 外周血单核细胞MTT测试通过密度梯度离心法从健康人的全血中分离出外周血单核细胞(PBMCs),洗涤后,用细胞培养液RPMI 1640(添加抗生素和10%胎牛血清)将PBMCs浓度调整到105/ mL,并铺于96孔圆底组织培养板中(每孔200 μL),细胞培养箱37℃孵育,至细胞长满单层。设实验组和阴性对照组,实验组设3个浓度,每孔加入重组Enolase 10 μg、5 μg和0.25 μg(100 μL,各10个复孔),阴性对照组只加入RPMI 1640(100 μL,10个复孔),细胞培养箱37℃孵育72 h,实验组和对照组每孔分别加入5 mg/mL噻唑蓝溶液(MTT),继续培养4 h,酶标仪570 nm处测定OD值(计算复孔平均值)。
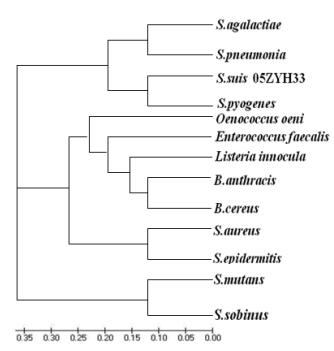
2 结果 2.1 eno基因生物信息学分析BLAST比对分析发现,S. suis 2 05ZYH33 CDSS-SU1503基因编码序列与S. pyogene、S. pneumoniae、S. sobrinu、S. agalactiae、S.mutans核酸序列相似性分别都达到88%以上,氨基酸序列则更加保守,相似性达到91%-96%之间,故将S. suis 2 05ZYH33中CDSSSU1503基因序列推断为Enolase基因疑似序列。分子遗传软件生成S. suis 2 05ZYH33 Enolase蛋白进化树(图 1,图 2)。
|
| 图 1 S. suis 2 05ZYH33 Enolase蛋白进化树 |

|
| 图 2 S. suis 05ZYH33 Enolase与其他相似序列的比对 |
SignalP 4.1和TMHMM 2.0分析发现05ZYH33 Enolase既没有信号肽序列(图 3-A),也没有跨膜区结构(图 3-B)。

|
| 图 3 SignalP 4.1和TMHMM 2.0对Enolase信号肽(A)和跨膜区(B)的预测 |
重组载体pMD18T∷eno和pET32a∷eno经单酶切(BamH Ⅰ)和双酶切(BamH Ⅰ和Xho Ⅰ)后,核酸凝胶电泳显示(图 4)eno疑似片段,生物公司测序后确定eno长1 308 bp。

|
| 图 4 质粒pMD-18T∷eno(A)和质粒pET32a∷eno(B)的酶切鉴定 1:10 000 bp DNA marker;2:质粒pMD-18T∷eno(A)和pET32a∷eno(B):3;BamH Ⅰ单酶切;4:BamH Ⅰ和Xho Ⅰ双酶切 |
重组菌E. coli BL21(含pET32a∷eno)诱导表达,进行SDS-PAGE电泳,与蛋白标记比对后显示在75 kD处有一条明显增多的蛋白条带(图 5-A)。目的蛋白通过Expasy估算的分子量为54 kD,加上融合标签(Trx Tag+S Tag+His Tag)分子量约为21 kD,故认为融合蛋白的分子量与预测一致。利用FPLC技术进行蛋白纯化,每个收集管经电泳后显示出特异条带(图 5-B,5-C)。

|
| 图 5 Enolase蛋白的SDS-PAG电泳和FPLC鉴定 A:SDS-PAGE电泳检测Enolase蛋白表达(1:蛋白marker,2:pET32a / E. coli BL21,3:未经诱导的pET32a∷eno / E. coli BL21,4:IPTG诱导的pET32a∷eno / E. coli BL21);B:FPLC技术纯化蛋白图谱;C:Enolase纯化蛋白SDS-PAGE电泳鉴定 |
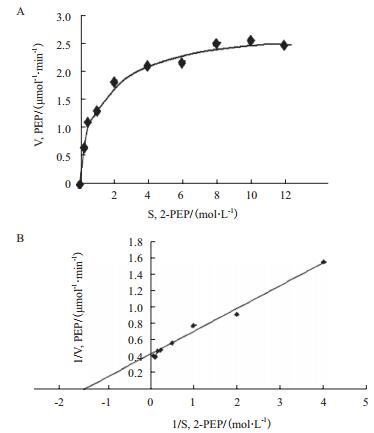
米氏方程曲线可以看出,在Enolase催化下,随着底物2-PEG的增加,产物磷酸烯醇丙酮酸盐(PEP)含量随之增加(图 6-A),表明Enolase的确能够将2-PEG转化为PEP。双倒数作图法得出,横坐标轴截距为-1/Km(1.4308),纵坐标轴截距为1/Vmax(0.405 2)(图 6-B)。由此算出,Vmax=1/0.405 2,Km=1/1.430 8(即0.7 mmol/L)。
|
| 图 6 纯化Enolase酶活测定 |
流式细胞术采集图中,FACS前向角(FCS)表示S. suis 2 05ZYH33体积大小,侧向角(SSC)表示S. suis 2 05ZYH33数量多少(图 7-A)。大鼠阴性血清标记FITC峰图显示对照组荧光含量较低,为1.06%(图 7-B),大鼠抗Enolase血清标记FITC峰图显示实验组荧光含量较高,峰图明显右移,为4.33%(图 7-C)。表明本室制备的鼠抗Enolase能够与05ZYH33细菌表面的Enolase结合,验证了S. suis 2 05ZYH33细菌表面确实存在Enolase。

|
| 图 7 荧光激活细胞分拣器对S. suis 2 Enolase的胞外定位检测 A:S. suis 2采集图;B:大鼠阴性血清标记FITC峰图;C:大鼠抗Enolase血清标记FITC峰图 |
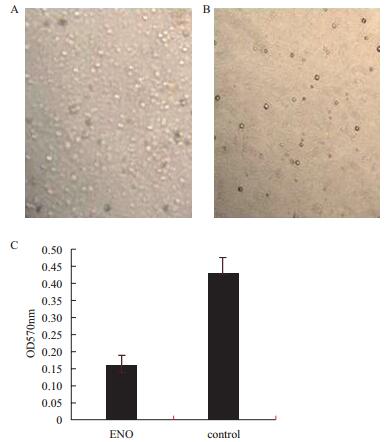
电子显微镜观察发现,不加Enolase的阴性对照组中PBMCs圆形、饱满、透明、贴壁生长(图 8-A),而加Enolase孵育的实验组中PBMCs数量明显减少,脱落漂浮、透光性差(图 8-B)。酶标仪OD570nm结果显示,实验组OD值明显低于对照组(图 8-C),P < 0.05(实验组中Enolase不同浓度的OD值变化不大,未图示),暗示Enolase与PBMCs活性之间存在联系。
|
| 图 8 Enolase引起PBMCs凋亡 A:加Enolase孵育的PBMCs电子显微镜;B:不加Enolase孵育的PBMCs电子显微镜图;C:实验组和对照组在酶标仪OD570nm测定结果 |
本研究通过BLAST蛋白序列相似度比对、Clustal多重序列比对及分子遗传分析发现,S. suis 2 05ZYH33 CDS SSU1503基因序列及氨基酸序列与多种细菌中Enolase序列存在高同源性,故将S. suis 2 05ZYH33中CDSSSU1503基因序列推断为Enolase基因疑似序列,同时暗示Enolase基因编码序列在进化中十分保守[8]。本研究对2005年四川疫区分离获得的强毒株S. suis 2 05ZYH33中Enolase编码基因进行分子克隆和蛋白表达,发现Enolase以可溶形式存在于重组菌中,经His标签亲和层析法和快速蛋白液相层析技术纯化后,获得了75 kD的Enolase重组蛋白,与预测相一致。酶催化实验显示纯化的重组Enolase具有酶活性,能将底物2-PEG转化为产物PEP,说明Enolase在S. suis 2体内确实能参与基础糖分解代谢。研究资料显示,Enolase同时也能催化糖生成逆向反应[9]。Enolase有两个Mg2+离子结合位点,当结合一个Mg2+后能引起Enolase活性位点构型变化从而有利于结合第2个Mg2+和底物[10, 11]。生物信息学分析显示,S. suis 2 Enolase既没有信号肽序列,也没有细胞膜锚定结构。然而,荧光激活细胞分选(FACS)技术检测结果却显示Enolase确实在S. suis 2细胞壁表面存在。关于细胞质中的Enolase是如何穿过细胞膜在细胞壁表面存在的机制尚未明确,可能Enolase是通过非共价键结合了其他细菌表面蛋白。细菌吸附宿主细胞是侵染致病的第一步,推测Enolase确实可能参与了感染过程。
在金黄色葡萄球菌、链球菌(A、C、G群)、淋病奈瑟菌、沙门氏菌、幽门螺杆菌、流感嗜血杆菌及伯氏疏螺旋体等多种病原菌细胞壁表面都发现存在Enolase,促进细菌穿越基底膜,参与感染过程[12-14]。研究发现,S. pyogenes胞外的Enolase依靠C末端的赖氨酸残基结合血纤溶酶原,赖氨酸基因敲除株明显表现出侵袭细胞外基质屏障能力的减弱[15]。S. mutans胞外的Enolase能结合宿主口腔中的唾液粘液素MG2,有助于细菌定植于口腔组织,并进一步发生迁移[16]。近年来发现,在哺乳动物的神经组织、肌肉组织及肿瘤细胞和某些免疫细胞表面也存在Enolase。当S. suis 2感染宿主时,宿主产生的抗细菌Enolase抗体也能够结合自身的Enolase,从而引发交叉反应,导致很多自身免疫性疾病的发生[17]。如急性风湿性发热、系统性红斑狼疮、子宫内膜异位及免疫紊乱患者血清中都检测出升高Enolase抗体[18]。
单核细胞具有吞噬、清除损伤或衰老细胞、产生抗体等作用,能够有效抵御或清除入侵的病原生物,是机体免疫防御系统的重要组成部分[19]。本研究发现外周血单核细胞在Enolase作用下,出现数量减少、脱落漂浮、透光性差等现象,表明细胞受到了损伤或发生了凋亡。MTT比色法,是检测细胞活性的常用技术,活细胞线粒体中的琥珀酸脱氢酶(SDH)能将MTT还原成蓝紫色结晶沉淀,死细胞则不能[20],因此MTT结晶量越大,则OD值越大,表明细胞活性越强。外周血单核细胞MTT测试显示PBMCs在Enolase作用下,OD值明显下降,表明PBMCs活细胞数量的减少。前期溶血空斑试验发现Enolase能够引发小鼠体内免疫抑制状态出现[21],此结果与溶血空斑实验结果相符。推测细菌表面Enolase可能通过直接或间接损伤单核细胞或诱发单核细胞凋亡,从而影响单核细胞的免疫防御机制。
4 结论同源性分析发现S. suis 2 05ZYH33 CDSSSU1503基因序列及氨基酸序列与多种细菌中Enolase序列高度同源,故推断此序列为S. suis 2 05ZYH33 Enolase基因疑似序列。SignalP和TMHMM分析发现Enolase没有信号肽也没有跨膜区。eno分子克隆并测序显示长度为1 308 bp,pET32a∷eno重组表达质粒经诱导表达并纯化后,获得75 kD的Enolase蛋白。FCM定位分析显示Enolase也存在于细菌表面。MTT试验检测表明Enolase能够引发PBMCs活性的下降。
致谢 本课题的实施得到了中国科学院微生物研究所高福实验室猪链球菌课题组的支持和帮助,在此表示衷心的感谢。| [1] | Zhang YY, Ding DD, Liu ML, et al. Effect of the glycosyltransferases on the capsular polysaccharide synthesis of Streptococcus suis serotype 2. Microbiological Research, 2016, 24 (1): 185–189. |
| [2] | Tang JQ, Wang CJ, Feng YJ, et al. Streptococcal toxic shock syndrome caused by Streptococcus suis serotype 2. PLoS Med, 2006, 3 (5): 151–155. DOI:10.1371/journal.pmed.0030151 |
| [3] | Chen C, Tang JQ, Dong W, et al. A Glimpse of Streptococcal toxic shock syndrome from comparative genomics of S. suis 2 Chinese isolates. PLoS ONE, 2007, 2 (7): 5298–5305. |
| [4] | Zhang CP, Zhang ZQ, Li S. Antimicrobial resistance profile and genotypic characteristics of Streptococcus suis capsular type 2 isolated from clinical carrier sows and diseased pigs in China. BioMed Research International, 2015, 20 (22): 3649–3657. |
| [5] | Pian YY, Li XQ, Zheng YL, et al. Binding of human fibrinogen to MRP enhances Streptococcus suis survival in host blood in a αXβ2 integrin-dependent manner. Scientific Reports, 2016, 6 (4): 26966–26969. |
| [6] | JF Mariscotti, JJ Quereda, MG Pucciarelli. Contribution of sortase A to the regulation of Listeria monocytogenes LPXTG s. International Microbiology, 2012, 15 (1): 43–51. |
| [7] | Michon C, Langella P, Eijsink VGH, et al. Display of recombinant proteins at the surface of lactic acid bacteria:strategies and applications. Microbial Cell Factories, 2016, 15 (1): 1–16. DOI:10.1186/s12934-015-0402-6 |
| [8] | Wongsawan K, Gottschalk M, Tharavichitkul P, et al. Serotype-and virulence-associated gene profile of Streptococcus suis isolates from pig carcasses in Chiang Mai Province, Northern Thailand. Journal of Veterinary Medical Science, 2014, 77 (2): 233–236. |
| [9] | Wu CH, Kuo YH, Hong RL, et al. α-Enolase-binding peptide enhances drug delivery efficiency and therapeutic efficacy against colorectal cancer. Science Translational Medicine, 2015, 7 (290): 1250–1268. |
| [10] | Zhong Z, Peng N, Ying Q, et al. An electrochemical immunosensor for simultaneous multiplexed detection of neuron-specific enolase and pro-gastrin-releasing peptide using liposomes as enhancer. Electrochimica Acta, 2011, 56 (16): 5624–5629. DOI:10.1016/j.electacta.2011.04.012 |
| [11] | Guillou C, Derambure C, Fréret M, et al. Prophylactic injection of recombinant alpha-Enolase reduces arthritis severity in the collagen-induced arthritis mice model. Plos One, 2014, 10 (8): 4478–4488. |
| [12] | Ebner P, Prax M, Nega M, et al. Excretion of cytoplasmic proteins (ECP) in Staphylococcus aureuss. Molecular Microbiology, 2015, 97 (4): 775–789. DOI:10.1111/mmi.2015.97.issue-4 |
| [13] | Wang J, Wang K, Chen D, et al. Cloning and characterization of surface-localized α-Enolase of Streptococcus iniae, an effective protective antigen in mice. International Journal of Molecular Sciences, 2015, 16 (7): 14490–14510. DOI:10.3390/ijms160714490 |
| [14] | M Ween, J Ahern, A Carroll, et al. A small volume technique to examine and compare alveolar macrophage phagocytosis of apoptotic cells and non typeable Haemophilus influenzae(NTHi). Journal of Immunological Methods, 2016, 429 (11): 7–14. |
| [15] | Balhara V, Deshmukh SS, Kalman L, et al. The interaction of streptococcal enolase with canine plasminogen:the role of surfaces in complex formation. PLoS One, 2014, 9 (2): 88395–88402. DOI:10.1371/journal.pone.0088395 |
| [16] | Mitsuhata C, Puteri MM, Ohara Y, et al. Possible involvement of enolase in fluoride resistance in Streptococcus mutans. Pediatric Dental Journal, 2014, 24 (1): 12–16. DOI:10.1016/j.pdj.2013.10.002 |
| [17] | Ge JP, Catt DM, Gregory RL, et al. Streptococcus mutans Surface α-Enolase Binds Salivary Mucin MG2 and human plasminogen. Infection & Immunity, 2004, 72 (11): 6748–6752. |
| [18] | Patricia AF, Pancholi V, Marcelo MN. Antibodies to streptococcal surface enolase react with human α-enolase:implications in poststreptococcal sequelae. The Journal of Infectious Diseases, 2000, 182 (1): 1712–1721. |
| [19] | G Ahangari, SE Koochak, LM Amirabad, et al. Investigation of 5-HT2A gene expression in PBMCs of patients with allergic asthma. Nature, 2015, 14 (1): 529–532. |
| [20] | Nazarpour R, Zabihi E, Alijanpour E, et al. Optimization of human peripheral blood mononuclear cells (PBMCs) cryopreservation. International Journal of Molecular & Cellular, 2012, 1 (2): 88–93. |
| [21] | 孙雯. 猪链球菌2型的表面蛋白烯醇化酶在细菌黏附和引发免疫下调中的作用. 微生物学免疫学进展, 2010, 38(2): 28–31. |





