木质素是植物体中仅次于纤维素的一种重要大分子有机物质,其总量占生物圈中有机碳含量的30%,占木本植物细胞壁干重的16%-35%[1]。陆生植物的木质素合成是其适应陆地生态环境的重要进化特征之一。细胞内合成的木质素单体渗入到一些特化的细胞壁中(如木质部的导管、厚壁组织和韧皮部纤维等)形成木质素的聚合体,对植物体维持完整的细胞结构、疏导水分和养料、防御病原菌侵害等方面有重要作用。由此可见,木质素在植物的生长发育和抗性方面具有重要的生物学功能[2, 3]。
普遍认为,4-香豆酸:辅酶A连接酶(4-coum-arate:coenzymeA ligase,4CL)作用于苯丙烷类代谢途径的第3个步骤,是联系木质素前体和各个分支途径的纽带,在木质素单体合成过程中发挥了重要的调控作用[4, 5]。4CL催化肉桂酸及其羟基或甲氧基衍生物生成相应的辅酶A酯,这些中间产物随后进入苯丙烷类衍生物支路合成途径[6]。其中以p-香豆酸、阿魏酸和芥子酸为底物生成的香豆酰CoA、阿魏酰CoA、芥子酰CoA,被进一步转化为肉桂醇衍生物,最终通过脱氢还原反应生成木质素单体[7]。由于木质素重要的生物学功能及生产价值,近年来对4CL酶基因的研究成为热点。本文对植物4CL基因家族、4CL蛋白酶学、4CL蛋白晶体学、4CL调控木质素生物合成等方面进展展开综述如下,旨为今后对该基因的研究提供参考。
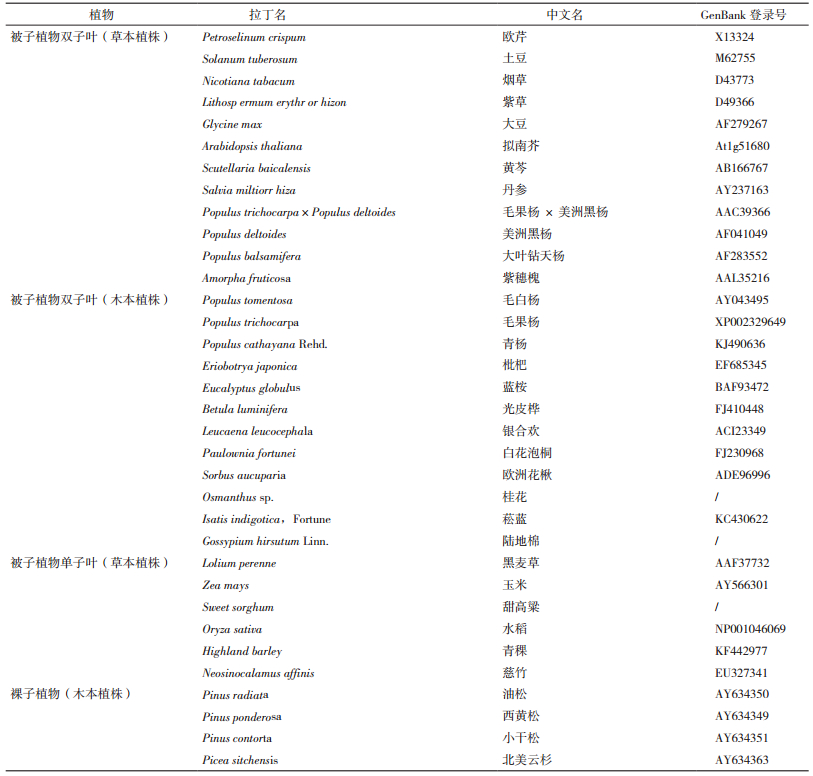
1 植物4CL基因家族近30年来,植物4CL基因序列被相继克隆出来。我们从NCBI(national centerfor biotechnology information)了解到,4CL基因的DNA或cDNA序列已从欧芹(Petroselinum crispum)[8]、水稻(Oryza.sativa)[9]、拟南芥(Arabidopsis thaliana)[1]、烟草(Nicotiana tabacum)[10]、和毛白杨(populus tomentosa carr)[11]等约40多种[12-21]植物中克隆出来(表 1)。黄胜雄[22]等对已有的4CL序列进行系统进化分析,发现植物4CL基因主要聚为2类:第1类包括拟南芥、大豆、烟草、杨树等大部分双子叶植物的4CL基因;第2类包括水稻、玉米、黑麦草这几种单子叶植物和裸子植物的4CL基因。这表明在4CL基因与植物的进化过程密切相关。同时,也表明单子叶植物和双子叶植物分化之前,4CL基因在植物中已经存在,而且4CL基因在植物中的进化时间早于单子叶和双子叶植物的分化时间。
随着植物中分离鉴定到越来越多的4CL基因,研究人员发现4CL具有被多基因所编码的特点,表明4CL基因在植物的进化过程中,随着植物基因组的倍增产生了基因的多个拷贝,形成了同源的家族基因。目前已知,拟南芥[23, 24]、大豆(Glycine max)[25]、覆盆子(Rubus idaeus L.)[26]、香鳞毛蕨(Dryopteris fragrans)[16]、美洲山杨(Populus tremuloides)[7]中存在2-4个4CL家族成员,这些4CL家族基因的出现,与植物进化过程中一些新功能的产生有着密切的联系,因此各个家族成员在基因结构和功能上的相似度不尽相同。
尽管目前已从许多植物中分离鉴定了4CL基因,4CL基因家族的大小依然未知,甚至在拟南芥、杨树等模式植物中,虽然已知全部基因组序列,4CL基因家族的真正范围仍然未知[27]。造成这种现象的原因可能是因为4CL基因具有特异性的表达模式,在特定的时空低丰度表达某些家族成员,从而阻碍了基因家族的鉴定[28-30]。相信随着功能基因组学和比较基因组学的不断深入,研究人员将逐渐确定不同植物基因组中4CL基因的数目。
2 4CL酶学特性在木质素生物合成过程中,4CL基因对羟基肉桂酸衍生物的偏好性及其表达模式有很大差异。例如,拟南芥中At4CLl和At4CL2编码的同工酶与木质素单体合成密切相关[31]。而At4CL3主要在花中表达,激活p-香豆酸作为查耳酮合成酶的底物,参与类黄酮的生物合成;At4CL4在只在特定条件下低水平表达[32],其发育和胁迫诱导的表达模式机理至今未知。大豆[33]中Gm4CL1和Gm4CL2参与植物生长和发育(包括木质化过程)。毛白杨[34]中,Ptc4CL1参与木质部的木质素生物合成,而Ptc4CL2参与表皮细胞中酚类化合物(如类黄酮)的生物合成。以上结果表明,同一物种的4CL基因的不同成员mRNA的表达表现出器官、组织的特异性,这可能与不同成员参与不同的分支途径有关。
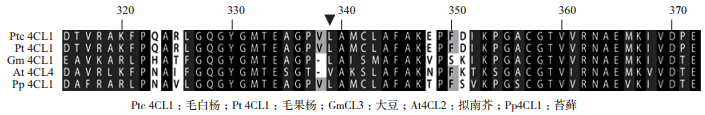
植物4CL对芥子酸没有活性是一个普遍现象。例如,毛白杨4CL1[35]对p-香豆酸、咖啡酸、阿魏酸有很高的亲和力,它们的Km值在40-60 mol/L之间,而对肉桂酸的亲和力较低,对芥子酸几乎无活性;拟南芥中At4CL2的最适底物为咖啡酸,对肉桂酸的转化能力也很低,对阿魏酸和芥子酸没有活性。大豆中GmCL2、GmCL3和GmCL4均不能催化芥子酸,而At4CL4和Gm4CLl能特异的催化芥子酸。对已有的4CL氨基酸序列比对分析表明,在所有已知的4CL氨基酸序列中存在保守的肽基序(motif):AMP结合功能域(Motif Ⅰ)和Box Ⅱ(Motif Ⅱ)。Motif Ⅰ作为底物结合区域(SBP)参与了底物的识别。SBP的空间大小决定了4CL能否与羟基肉桂酸衍生物结合。Motif Ⅱ直接参与催化反应,同时与稳定蛋白质的活性和空间构象有关[36]。用blast比对不具有芥子酸催化活性的4CL基因序列与At4CL4和Gm4CLl(图 1)发现,SBP中的氨基酸338Val在At4CL4和Gm4CLl中特异性缺失[37]。这个研究结果有助于人们更好地理解苯丙烷类代谢途径中关键酶的催化行为,并设汁具有新的底物特异性的4CL蛋白。但是,并未从根本上揭示4CL同工酶底物特异性的分子机制。要阐明4CL蛋白结构与功能的关系,最佳的方法就是获得4CL蛋白的晶体,采集晶体结构数据,解析4CL蛋白的原子结构和催化机制。
3 4CL蛋白晶体学研究早在20世纪70年代初,4CL蛋白的研究就已经开始进行,但是4CL蛋白的三维结构解析进展缓慢,其蛋白结晶体的获得成为当前4CL研究中的一个难点。2010年,胡永林等[38]获得了高质量的毛白杨4CL1蛋白质结晶体,并解析出毛白杨4CL1晶体结构(图 2-A)。毛白杨4CL1基因包含536个氨基酸残基,4CL1蛋白由两个有机结合的球状结构域组成。这两个结构域分别为N-domain和C-domain。其中N-domain较大,由434个氨基酸残基组成,是底物容纳口袋。N-domain又可以分成3个子结构,这3个子结构分别为N1、N2和N3。C-domain较小,由第435-536位的氨基酸残基组成,包含了催化位点。根据对4CL-APP复合体的晶体结构研究分析(图 2-B)发现,C-domain相对于N-domain旋转了818度,N-domain中每355到391个原子的长度为1.9-2.2Å,而C-domain的每71-103个原子的长度为0.9-1.7Å。4CL具有催化活性的氨基酸残基为Lys-438、Gln-443和Lys-523,底物识别活性的残基为Tyr-236、Gly-306、Gly-331、Pro-337和Val-338。底物识别口袋的大小决定了4CL的底物特异性。
毛白杨4CL1母体蛋白晶体和衍射单晶的获得,使得从分子水平上阐明其催化反应和底物偏好选择机理提供了可能,并为4CL蛋白参与的生物代谢途径的改造提供了理论依据,同时也为植物中木质素生物合成和黄酮类的生物合成机制提供了直接的理论依据,在苯丙烷类化合物的生物合成中具有极大的应用价值。
4 4CL基因的表达与调控植物次生代谢途径中绝大多数基因属于调控基因,4CL基因也不例外,其在植物不同组织中的差异表达主要受发育和环境因子的双重调控。4CL受发育调控的研究例证很多,如Lee等[39-41]发现拟南芥4CL基因在苗期阶段即被活化,其活性与子叶和根部木质素形成有密切相关。赵艳玲等[42, 43]发现,毛白杨4CL的表达水平与早材和晚材的形成阶段相符合,在一个生长季节中呈现“双峰”模式。Reinold等[44]通过原位杂交研究烟草花发育过程中4CL的时空表达规律,结果表明在花发育的不同阶段,转基因烟草的心皮、花药、花瓣及萼片中4CL的表达量存在差异。
植物4CL基因在不同发育阶段表达受到启动子的调节。研究人员最早从欧芹中克隆到4CL启动子,全长1 530 bp,在木质部特性定位表达[45]。丹参[11]4CL启动子驱动GUS在幼嫩拟南芥子叶的顶端和下胚轴,以及生长40 d的拟南芥叶片的边缘、萼片、花药中表达。潘翔等[34]分离克隆了毛白杨中4个4CL基因家族成员的启动子,并在转基因烟草中分析4个启动子的组织特异性表达模式。研究表明,4个启动子驱动GUS的表达部位和表达模式有差异,其中Pto4CL4p和Pto4CL5p调控的表达模式比较接近,Pto4CL3p驱动的组织特异性表达同时具有发育阶段特异性。
除发育调控外,4CL基因在植物中也被伤害、病原体感染、紫外线辐射等外界刺激所激活而表达。目前已证实茉莉酸处理、伤害胁迫处理欧芹的成熟叶片,4CL mRNA均瞬间高丰度增加[46]。Uhlmann等[33]用晚疫病菌(Phytophthora infestans)感染马铃薯叶片,4CL转录活性2 h后达到最大值,接种12 h后,4CL酶活性增加2倍。Ehlting等[23]用植物霜霉病的致病寄生真菌(Peronos poraparasitica)的孢子感染拟南芥子叶,发现该致病因素强烈诱导At4CL1和At4CL2 mRNA的表达,而At4CL3 mRNA则不被诱导;紫外线照射拟南芥6 h后,At4CLl和At4CL2转录活性达到最高;含致病基因avrB的十字花科(Cruciferae)黑斑病假单胞菌(Pseudomonas syringae pv. maculicola)侵染拟南芥6 h后,4CL均被激活表达,并产生不协调的交互作用。At4CL基因不同程度的胁迫反应也说明植物体内4CL家族各成员可能参与了不同的生物代谢途径。
随着分子生物学尤其是基因测序技术的不断发展,更多不同来源的苯丙烷类代谢途径基因被分离和鉴定,通过与已知的序列进行比对分析,发现4CL、PAL和C4H基因的启动子区域都有保守的“AC”顺式作用元件,包括boxP、boxA、boxL。他们是决定以上3个基因在诱导因子、紫外线和伤害等胁迫条件下特异性表达的关键元件[47]。Logemann等[48]发现,含有紫外线的白光、真菌诱导子和机械伤害能诱导欧芹PAL、C4H和4CL基因mRNA同时瞬时表达。诱导子处理燕麦叶片后,PAL、C4H和4CL酶活性同时达到最高水平。以上研究结果表明,PAL、C4H和4CL基因的表达具有转录水平上的协同调控性。由于PAL、4CL和C4H是苯丙烷代谢途径的关键基因,对木质素生物合成具有重要调控作用。因此,以上研究成果,有利于研究者探究苯丙烷类代谢途径和木质素生物合成途径中各个酶系之间的相互作用,也为实现多基因的协同表达调控提供了更多的信息。
5 4CL基因在基因工程中的应用把木质素生物合成途径中的关键基因定位为靶基因,构建该基因的反义、正义或干涉结构的表达载体并进行遗传转化,利用获得的转基因植株,进行基因表达和调控研究,是对木质素合成途径进行系统研究的有力手段之一。4CL处于苯丙烷类代谢途径形成不同类型产物的转折点上,催化各种羟基肉桂酸生成相应的硫酯,这些硫酯同时也是苯丙烷类代谢途径和各种末端产物生成途径的分支点。因此,通过基因工程手段上下调节4CL酶活性,是对其进行功能分析的重要途径。目前已获得大量4CL转基因植株,这些转基因植株在4CL表达、木质素含量、组成和植株发育等方面存在很大差异[27]。
Lee[41]采用反义手段分别抑制拟南芥和烟草中的4CL活性,结果发现转基因植株中木质素总量明显下降,G型木质素降低,S型木质素不发生变化,从而导致S/G比值升高,同时转基因植株的生长发育没有受到影响,导管也未发生形变。Xu等[49]通过RNA干涉方式抑制柳枝稷(Panicum virgatum)中4CL的表达,研究结果中,转基因柳枝稷4CL酶活性降低80%,木质素总量、G型木质素含量都显著降低,而转基因植株的生物量,纤维水解能力显著提高,同时导管形态未发生变化。Hu等[7]利用反义RNA技术抑制山杨(Populus davidiana Dode)4CL1基因表达,4CL1活性下降90%以上的转基因株系中木质素含量下降达45%,且根、茎、叶生长势增强,纤维素含量明显增加,转基因植株中木质素与纤维素的含量存在互补调控作用。Voelker等[50]运用反义RNA技术抑制杂交杨(Poplus tremula populusalba)中4CL1表达,结果发现,木质素的总量与4CL1 mRNA的表达量呈明显正相关关系,4CL mRNA表达量降低50%,导致木质素含量下降超过10%,木材S/G比值也降低,转基因植株出现矮灌化,导管出现塌陷形变,但是木质素的降低并没有引起糖化作用的提高。Wagner等[51]采用RNA干涉手段抑制油松(Pinus tabulaeformis)4CL的活性,转基因油松的木质素总量降低28%,G型木质素降低,H型木质素提高,H/G比对照高4倍,同时木材细胞壁中多糖含量明显增加,虽然转基因植株表型未发生变化,但是管胞的木质化程度极大地降低,仅仅只有S1层中的管胞的木质化程度正常。
我国科研工作者对4CL基因调控木质素的生物合成也进行了系统的研究。李金花[27]以毛果杨×美洲黑杨杂种H11为材料,获得了杨树Ptd4CL3的全长cDNA序列,并通过Northem杂交证实,Ptd4CL3在木质部中表达量最高,将Ptd4CL3正义、反义基因分别导入白杨派杂种无性系717中,获得转基因杨树,结果表明,正义和反义4CL基因调控均引起转基因杨树4CL酶活性和木质素总量的降低,并且反义调控比正义调控对基因表达的抑制作用更明显,且所有转基因植株的形态和生长发育未见异常。李桢[52]通过反义抑制4CL基因表达获得了转基因毛白杨,显微切片观察发现,转基因植株中存在极少数轻度形变或发育不良的导管,但转基因杨生长状态良好。贾彩虹[53, 54]发现反义抑制的4CL转基因烟草木质素含量平均下降10.3%,且开花期存在花瓣开裂现象。陆海等[55]将组成型启动子CaMV 35S及形成层定位启动子GRPl.8分别和反义毛白杨4CLI基因融合,并分别导入烟草中,通过对转基因烟草纤维素和木质素含量的对比分析,发现CaMV35S启动的反义4CL1转基因烟草纤维素含量比对照提高了11.4%,而木质素含量降低了19.1%;GRP1.8启动的反义4CLl转基因烟草木质素含量较对照平均下降了13.7%,而纤维素含量升高了15.6%。赵艳玲等[43]将GRP1.8启动子和CaMV35S启动子分别与反义毛白杨4CL1基因结合,并分别导人741毛白杨中,获得转基因741毛白杨,分析发现,GRP1.8启动的反义4CL1酶活性在茎和根中的降低量高于叶片;另外还发现,由CaMV35S启动的4CL1 RNA干涉转基因毛白杨,4CL基因的表达量、酶活性、木质素含量都显著降低,并且,RNA干扰技术阻碍基因表达能力要优于反义RNA技术,这可能是由于4CL基因有许多同源的基因家族成员,RNA干涉更有效的降低了同源基因的表达。目前为止,温室中的转基因幼龄杨的生理特性已经得到了详细研究,而野外生长的转基因杨的生理生化特性没有足够认识,而且调控木质素合成的转基因植物也缺乏整个生长季的观察和检测分析,植物在野外生长过程中会遇到许多在温室中不存在的因素的影响(如风、雨和霜等),因此考察在温室中培育的植物在野外继续生长过程中能否保持其特征是一项很基础的研究。田晓明等[56]对1年生和5年生转基因杨树分别从生理生化、基因表达、细胞壁组成、细胞壁解剖结构和木材品质等角度进行研究。结果表明,调控4CL的基因表达,将对木质素单体合成途径相关基因表达产生协同调控作用,最终也对木质素单体组成产生影响。
6 展望4-香豆酰-CoA连接酶(4CL)是苯丙烷类代谢途径下游分支途径上处于终端位置的酶,是连接苯丙烷代谢途径与木质素生物途径的关键酶,在木质素单体合成中起关键调节作用。但生物某个性状通常是由多个基因共同控制,在林业生产上,由于单个转基因的信号强度较弱,很难获得理想实验的植株,多基因共同转化将有待解决这一难题。随着分子生物学尤其是“基因组计划”的发展,拟南芥、水稻、毛果杨等植物全基因组测序已经完成。使得研究人员不仅可以研究单一酶基因的结构和功能,还可以从基因家族、基因簇等层面研究代谢途径中多个酶基因的协同表达与调控,这为研究4CL的结构和功能提供了新的方向。
已完成的拟南芥全基因组测序中鉴定出4种4CL基因的功能,但在拟南芥基因组中还存在许多4CL相似的基因,这些4CL相似基因的功能尚不得而知。毛果杨等木本植物中有多少4CL基因,其功能如何尚在研究中。相信“基因组计划”的发展,会分离并鉴定出更多植物的4CL基因。
由于木本植物生长周期较长,个体基因型的不同会在不同程度上影响到植株再生和转化效率。因此,调控4CL基因影响植株生长的作用机制尚不清楚,关于转4CL基因木本植物的细胞壁化学组成、木材品质和解剖特性等材性研究尚不多见。田晓明等[56]对转4CL基因毛白杨进行细胞壁组成、细胞壁解剖结构和木材品质等分析发现,4CL1基因对生长素含量具有间接调控作用,转基因毛白杨木质部导管形态变化,导致木质部传导性降低,干扰植物的正常生理代谢,从而对植株生长产生影响。
| [1] | Hamberger B, Hahlbrock K. The 4-coumarate: CoA ligase gene family in Arabidopsis thaliana comprises one rare, sinapate-activating and three commonly occurring isoenzymes. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101 (7): 2209–2214. DOI:10.1073/pnas.0307307101 |
| [2] | Pan X, Li H, Wei H, et al. Analysis of the spatial and temporal expression pattern directed by the Populus tomentosa 4-coumarate: CoA ligase Pto4CL2 promoter in transgenic tobacco. Molecular Biology Reports, 2013, 40 (3): 2309–2317. DOI:10.1007/s11033-012-2312-6 |
| [3] | Endler A, Martens S, Wellmann F, et al. Unusually divergent 4-coumarate: CoA-ligases from Ruta graveolens L. Plant molecular biology, 2008, 67 (4): 335–346. DOI:10.1007/s11103-008-9323-7 |
| [4] | Allina SM, Pri-hadash A, Theilmann DA, et al. 4-Coumarate: coenzyme A ligase in hybrid poplar. Properties of native enzymes, cDNA cloning, and analysis of recombinant enzymes. Plant Physiol, 1998, 116 (2): 743–754. DOI:10.1104/pp.116.2.743 |
| [5] | Gui J, Shen J, Li L. Functional characterization of evolutionarily divergent 4-coumarate: coenzyme a ligases in rice. Plant Physiol, 2011, 157 (2): 574–586. DOI:10.1104/pp.111.178301 |
| [6] | Alberstein M, Eisenstein M, Abeliovich H. Removing allosteric feedback inhibition of tomato 4-coumarate: CoA ligase by directed evolution. The Plant Journal, 2012, 69 (1): 57–69. DOI:10.1111/tpj.2011.69.issue-1 |
| [7] | Hu WJ, Kawaoka A, Tsai CJ, et al. Compartmentalized expression of two structurally and functionally distinct 4-coumarate: CoA ligase genes in aspen (Populus tremuloides). Proceedings of the National Academy of Sciences of the United States of America, 1998, 95 (9): 5407–5412. DOI:10.1073/pnas.95.9.5407 |
| [8] | Lozoya E, Hoffmann H, Douglas C, et al. Primary structures and catalytic properties of isoenzymes encoded by the two 4-coumarate: CoA ligase genes in parsley. European Journal of Biochemistry / FEBS, 1988, 176 (3): 661–667. DOI:10.1111/ejb.1988.176.issue-3 |
| [9] | Zhao Y, Kung SD, Dube SK. Nucleotide sequence of rice 4-coumarate: CoA ligase gene, 4-CL. 1. Nucleic Acids Research, 1990, 18 (20): 6144. DOI:10.1093/nar/18.20.6144 |
| [10] | Hai L, Zeng Q, Zhao YL, et al. Xylem-specific expression of a GRP1. 8 promoter: : 4CL gene construct in transgenic tobacco. Plant Growth Regulation, 2003, 41 (3): 279–286. DOI:10.1023/B:GROW.0000007507.98276.1d |
| [11] | 范丙友, 陆海, 蒋湘宁. 维管植物4-香豆酸:辅酶A连接酶 (4CL) 研究进展. 林业科学, 2007(2): 96–103. |
| [12] | 韩欣. 马尾松木质素合成途径中4CL基因克隆及RNA干扰载体构建研究[D]. 长沙: 中南林业科技大学, 2012. |
| [13] | 侯思宇, 赵盖超, 刘荣华, 等. 苦荞Ft4CL基因克隆、生物信息学及分子进化分析. 山西农业大学学报:自然科学版, 2015(1): 411–415. |
| [14] | 胡尚连, 曹颖, 黄胜雄, 等. 慈竹4CL基因的克隆及其生物信息学分析. 西北农林科技大学学报:自然科学版, 2009(8): 204–210. |
| [15] | 霍松, 陈慧, 朱琼华, 等. 象草4CL基因片段的克隆及RNAi表达载体构建. 草业学报, 2012(1): 296–301. DOI:10.11686/cyxb20120139 |
| [16] | 李珊珊. 香鳞毛蕨4CL基因家族的克隆及功能验证[D]. 哈尔滨: 东北农业大学, 2015. |
| [17] | 刘文哲. 紫穗槐UGPase和4CL基因的克隆及在植物中的转化与表达[D]. 大连: 大连理工大学, 2002. |
| [18] | 龙松华, 李翔, 陈信波, 等. 亚麻4CL基因克隆及RNAi遗传转化. 西北植物学报, 2014(12): 2405–2411. |
| [19] | 母洪娜, 孙陶泽, 徐晨, 等. 桂花 (Osmanthus fragrans Lour.)4-香豆酸辅酶A连接酶 (4CL) 基因克隆与表达分析. 分子植物育种, 2016(3): 536–541. |
| [20] | 倪志勇, 王娟, 吕萌, 等. 棉花4-香豆酸辅酶A连接酶基因克隆及原核表达. 西北植物学报, 2010(3): 429–436. |
| [21] | 杨冬梅, 王学敏, 高洪文, 等. 东方山羊豆4香豆酸:辅酶A连接酶 (4CL) 基因的克隆和荧光定量表达分析. 草地学报, 2010(4): 533–538. DOI:10.11733/j.issn.1007-0435.2010.04.010 |
| [22] | 黄胜雄, 胡尚连, 孙霞, 等. 木质素生物合成酶4CL基因的遗传进化分析. 西北农林科技大学学报:自然科学版, 2008(10): 199–206. |
| [23] | Ehlting J, Buttner D, Wang Q, et al. Three 4-coumarate: coenzyme A ligases in Arabidopsis thaliana represent two evolutionarily divergent classes in angiosperms. The Plant Journal, 1999, 19 (1): 9–20. DOI:10.1046/j.1365-313X.1999.00491.x |
| [24] | Ehlting J, Shin JJK, Douglas CJ. Identification of 4-coumarate: coenzyme A ligase (4CL) substrate recognition domains. Plant J, 2001, 27 (5): 455–465. DOI:10.1046/j.1365-313X.2001.01122.x |
| [25] | Lindermayr C, Mollers B, Fliegmann J, et al. Divergent members of a soybean (Glycine max L.)4-coumarate: coenzyme A ligase gene family. European Journal of Biochemistry / FEBS, 2002, 269 (4): 1304–1315. DOI:10.1046/j.1432-1033.2002.02775.x |
| [26] | Kumar A, Ellis b E. 4-coumarate: CoA ligase gene family in Rubus idaeus: cDNA structures, evolution, and expression. Plant Molecular Biology, 2003, 51 (3): 327–340. DOI:10.1023/A:1022004923982 |
| [27] | 李金花. 杨树4CL基因调控木质素生物合成的研究[D]. 北京: 中国林业科学研究院, 2005. |
| [28] | Jung JH, Kannan B, Dermawan H, et al. Precision breeding for RNAi suppression of a major 4-coumarate: coenzyme A ligase gene improves cell wall saccharification from field grown sugarcane. Plant Mol Biol, 2016, 92 (4-5): 505–517. DOI:10.1007/s11103-016-0527-y |
| [29] | Zhang E, Guo X, Meng Z, et al. Construction, expression, and characterization of Arabidopsis thaliana 4CL and Arachis hypogaea RS fusion gene 4CL: : RS in Escherichia coli. World Journal of Microbiology & Biotechnology, 2015, 35 (4): 1–7. |
| [30] | Wen SC, Wang YQ, Luo J, et al. Cloning and tissue expression of 4-coumarate coenzyme A ligase gene in Angelica sinensis. Plant Molecular Biology, 2015, 40 (24): 4824–4829. |
| [31] | Cukovic D, Ehlting J, Vanziffle JA, et al. Structure and evolution of 4-coumarate: coenzyme A ligase (4CL) gene families. Biological Chemistry, 2001, 382 (4): 645–654. |
| [32] | Li Y, Kim JI, Pysh L, et al. Four isoforms of Arabidopsis thaliana 4-coumarate: CoA ligase (4CL) have overlapping yet distinct roles in phenylpropanoid metabolism. Plant Physiology, 2015, 169 (4): 2409–2421. |
| [33] | Uhlmann A, Ebel J. Molecular cloning and expression of 4-coumarate: coenzyme A ligase, an enzyme involved in the resistance response of soybean (Glycine max L.) against pathogen attack. Plant Physiol, 1993, 102 (4): 1147–1156. DOI:10.1104/pp.102.4.1147 |
| [34] | 潘翔. 毛白杨4CL基因启动子及APX的结构与功能研究[D]. 北京: 北京林业大学, 2013. |
| [35] | 邹恩强. 转4CL基因对植物木质素代谢的影响[D]. 济南: 山东师范大学, 2011. |
| [36] | Stuible H, Buttner D, Ehlting J, et al. Mutational analysis of 4-coumarate: CoA ligase identifies functionally important amino acids and verifies its close relationship to other adenylate-forming enzymes. FEBS Letters, 2000, 467 (1): 117–122. DOI:10.1016/S0014-5793(00)01133-9 |
| [37] | 饶国栋, 张永卓, 魏弘宜, 等. 毛白杨4-香豆酸:辅酶A连接酶 (4CL3) 的酶学特征研究. 林业科学研究, 2013, 26(5): 542–547. |
| [38] | Hu Y, Gai Y, Yin L, et al. Crystal structures of a Populus tomentosa 4-coumarate: CoA ligase shed light on its enzymatic mechanisms. The Plant Cell, 2010, 22 (9): 3093–3104. DOI:10.1105/tpc.109.072652 |
| [39] | Lee D, Ellard M, Wanner LA, et al. The Arabidopsis thaliana 4-cou-marate: CoA ligase (4CL) gene: stress and developmentally regulated expression and nucleotide sequence of its cDNA. Plant Molecular Biology, 1995, 28 (5): 871–884. DOI:10.1007/BF00042072 |
| [40] | Lee D, Douglas CJ. Two divergent members of a tobacco 4-coumarate: coenzyme A ligase (4CL) gene family. cDNA structure, gene inheritance and expression, and properties of recombinant proteins. Plant Physiol, 1996 (1): 193–205. |
| [41] | Lee D, Meyer K, Chapple C, et al. Antisense suppression of 4-cou-marate: coenzyme A ligase activity in Arabidopsis leads to altered lignin subunit composition. The Plant Cell, 1997, 9 (11): 1985–1998. DOI:10.1105/tpc.9.11.1985 |
| [42] | 赵艳玲, 陆海, 蒋湘宁. GRP1. 8融合4CL1基因调控杨树木质素生物合成. 成都大学学报:自然科学版, 2012(1): 99–102. |
| [43] | 赵艳玲, 陆海, 陶霞娟, 等. GRP1. 8融合反义4CL1基因调控烟草木质素生物合成. 北京林业大学学报, 2003(4): 16–20. |
| [44] | Reinold S, Hauffe KD, Douglas CJ. Tobacco and Parsley 4-Coumarate: coenzyme A ligase genes are temporally and spatially regulated in a cell type-specific manner during tobacco flower development. Plant Physiol, 1993, 101 (2): 373–383. DOI:10.1104/pp.101.2.373 |
| [45] | Hauffe KD, Paszkowski U, Schulze-lefert P, et al. A parsley 4CL-1 promoter fragment specifies complex expression patterns in transgenic tobacco. The Plant Cell, 1991, 3 (5): 435–443. DOI:10.1105/tpc.3.5.435 |
| [46] | Lois R, Hahlbrock K. Differential wound activation of members of the phenylalanine ammonia-lyase and 4-coumarate: CoA ligase gene families in various organs of parsley plants. Z Naturforsch C, 1992, 47 (1-2): 90–94. |
| [47] | Li ZB, Li CF, Li J, et al. Molecular cloning and functional characterization of two divergent 4-coumarate: coenzyme A ligases from Kudzu (Pueraria lobata). Biological & Pharmaceutical Bulletin, 2014, 37 (1): 113–122. |
| [48] | Logemann E, Parniske M, Hahlbrock K. Modes of expression and common structural features of the complete phenylalanine ammonia-lyase gene family in parsley. Proc Natl Acad Sci USA, 1995, 92 (13): 5905–5909. DOI:10.1073/pnas.92.13.5905 |
| [49] | Xu B, Escamilla-trevino LL, Sathitsuksanoh N, et al. Silencing of 4-coumarate: coenzyme A ligase in switchgrass leads to reduced lignin content and improved fermentable sugar yields for biofuel production. The New Phytologist, 2011, 192 (3): 611–625. DOI:10.1111/nph.2011.192.issue-3 |
| [50] | Voelker SL, Lachenbruch B, Meinzer FC, et al. Antisense down-regulation of 4CL expression alters lignification, tree growth, and saccharification potential of field-grown poplar. Plant Physiol, 2010, 154 (2): 874–886. DOI:10.1104/pp.110.159269 |
| [51] | Wang DD, Bai H, Chen WQ, et al. Identifying a cinnamoyl coenzy-me a reductase (CCR) activity with 4-coumaric acid: coenzyme A ligase (4CL) reaction products in Populus tomentosa. J of Plant Biol, 2009, 52 (5): 482–491. DOI:10.1007/s12374-009-9062-6 |
| [52] | 李桢, 王宏芝, 李瑞芬, 等. 植物木质素合成调控与生物质能源利用. 植物学报, 2009(3): 262–272. |
| [53] | 贾彩红. 低木质素含量的转基因毛白杨 (Populus tomentosa) 的获得与毛白杨4CL基因启动子的克隆[D]. 保定: 河北农业大学, 2004. |
| [54] | 贾彩红, 王宏芝, 杜克久, 等. 抑制4CL基因表达的转基因毛白杨中木质素含量与茎杆颜色的关系. 农业生物技术学报, 2004(6): 621–624. |
| [55] | Lu H, Zhao YL, Jiang XN. Stable and specific expression of 4-coumarate: coenzyme A ligase gene (4CL1) driven by the xylem-specific Pto4CL1 promoter in the transgenic tobacco. Biotechnology Letters, 2004, 26 (14): 1147–1152. DOI:10.1023/B:BILE.0000035487.91628.9e |
| [56] | Tian XM, Xie J, Zhao Y, et al. Sense-, antisense-and RNAi-4CL1 regulate soluble phenolic acids, cell wall components and growth in transgenic Populus tomentosa Carr. Plant Physiol Biochem, 2013, 65 : 111–119. DOI:10.1016/j.plaphy.2013.01.010 |