随着分子生物学的发展,基因表达分析在发现和预测新基因及其功能、探究基因表达调控的复杂网络等研究中日趋重要。实时荧光定量PCR(real-time quantitative PCR,qPCR)是在传统PCR技术基础上发展起来的一种新的核酸定量技术,具有重复性好、灵敏度高、特异性强、高通量等特点,已成为分子生物学研究中检测基因表达水平的一种重要方法[1]。在qPCR分析过程中,RNA的质量、cDNA的质量与起始浓度、扩增效率等都会对结果的准确性产生影响,因此,必须选择合适的内参基因进行校正和标准化[2]。理想内参基因应该在特定试验条件下的所有细胞或组织中均相对稳定地表达,不会受到任何内源性或外源性因素的影响。常用的内参基因主要有肌动蛋白基因(ACT)、转录延伸因子基因(EF1α)、α/β微管蛋白基因(TUA/TUB)、18S/25S核糖体RNA基因(18S/25S rRNA)、甘油醛-3-磷酸脱氢酶基因(GAPDH)等管家基因。近年来随着基因芯片技术和转录组测序等研究的深入,新的内参基因也不断被挖掘,如网格蛋白衔接蛋白复合物基因(CAC)、SAND蛋白家族基因(SAND)、PPR蛋白家族基因(PPR)、F-box蛋白家族基因(F-box)、ABC转运因子基因等[3-5]。但是,大量研究表明,随着研究对象的不同或者试验条件的变化,没有任何一种内参基因的表达是始终稳定的,即内参基因的选择不存在通用性[6]。因此,在研究目标基因的表达情况时,研究者必须首先在具体试验条件下对内参基因进行筛选与验证,然后再选择合适的内参基因用于校准,这是保证qPCR分析结果准确性的重要前提。
珙桐(Davidia involucrata Baill.),又名鸽子树,为珙桐科珙桐属落叶乔木,是我国特有的单型属珍稀孑遗植物,属于国家Ⅰ级保护植物[7]。在我国,珙桐主要分布于四川、重庆、湖北、贵州、湖南、云南、甘肃等省市。珙桐是著名观赏树种,具有一些独特的生物学性状,如圆形的头状花序、对生的白色叶状苞片等。对珙桐独特的生物学表型展开研究,从分子生物学水平上阐述其特异器官发育的调控规律,寻找关键调控基因,是珙桐分子生物学研究的重要方向。目前,有关珙桐中关键基因的克隆,基因的表达模式分析及功能研究等相对较少,而在已有的少量报道中,均选择常见管家基因(如ACT、18S rRNA等)作为内参基因,但所选用基因是否适合作为该研究中的内参基因,尚缺乏一定理论依据[8, 9]。因此,在珙桐分子生物学研究中,需要发掘更多具有较高表达稳定性的内参基因,这对于提高珙桐基因表达研究的准确性至关重要。
CAC(Clathrin adaptor complexes),即AP2M(Adaptor protein-2 Mu-adaptin),是细胞中的一类衔接蛋白(Adaptor protein,AP)。衔接蛋白指在细胞内信号传递过程中,在蛋白质间起连接作用的一类蛋白质,进化上比较保守。植物中共有5种类型的AP复合体,即AP-1、AP-2、AP-3、AP-4、AP-5,每个AP复合体均由2个大亚基(α和β)、一个中亚基(μ)和一个小亚基(σ)组成。其中,AP-2复合体存在于质膜上,在网格蛋白介导的质膜内吞过程中发挥重要作用[10, 11]。植物中对CAC基因的研究还不够深入,有关其具体生物学功能的报道也较少。但近年来有研究表明,在一些植物中,CAC可作为特定条件下的内参基因应用于基因表达分析中。例如,Czechowski等[3]利用拟南芥Affymetrix ATH1全基因组基因芯片数据,筛选出了在拟南芥不同组织以及非生物胁迫下均能稳定表达的22个基因,并结合5个管家基因(ACT2、EF-1α、GAPDH、UBQ10、UBC),对它们的表达稳定性进行评价,结果发现,筛选出的新内参基因的表达稳定性普遍优于管家基因,并且新内参基因CAC(AT5G46630)具有较高的表达稳定性。随后,在番茄、荞麦、竹子、鹰嘴豆、西瓜等植物中的相关研究也表明,CAC可作为内参基因使用[12-16]。
目前,很多植物的基因表达分析中都选用了CAC作为内参基因,而有关珙桐CAC基因的研究尚未见报道。为此,本研究克隆珙桐CAC基因,并对其进行相关生物信息学分析。同时,结合5个管家基因(ACT7、EF1a、GAPDH、β-TUB、18S rRNA),利用半定量PCR、qPCR以及内参基因稳定性分析软件探讨DiCAC在珙桐不同组织的表达稳定性。同时,白色苞片是珙桐代表性状,苞片的发育机制研究是本课题组今后的研究重点,因此也对DiCAC在不同发育阶段苞片中的表达稳定性进行分析,旨在为珙桐分子生物学研究提供新的、稳定的内参基因资源,为进一步研究珙桐的基因表达模式,揭示珙桐苞片发育的分子机制奠定理论基础。
1 材料与方法 1.1 材料本研究供试材料为珙桐,采集地位于湖南省张家界市桑植县八大公山国家级自然保护区,采集时间为2016年4月中旬至7月下旬。廖春林对采集样品进行了鉴定,确定采集材料均为珙桐样品,所有采集的珙桐样品均保存于本实验室。本研究中,主要选择保护区内树龄20年左右,能稳定开花的珙桐植株作为采集对象,分别采集以下两组样品,第一组,不同组织样品,共8种:根、茎、芽、叶片、苞片(中期)、雄蕊、果肉、种子;第二组,不同发育阶段苞片样品,参照Vekemans等[8]的分类方法具体分为:幼期苞片(绿色)、前期苞片(浅绿色)、中期苞片(纯白色)、后期苞片(浅黄色)、晚期苞片(黄色待脱落)。每份样品包括3个生物学重复,所有样品采集后立即置于液氮中速冻保存,然后置于-70℃超低温冰箱中贮藏备用。
1.2 方法 1.2.1 总RNA提取与cDNA合成分别称取适量冻存珙桐样品,于盛有液氮的研钵中迅速研磨至粉末,按照E.Z.N.A.TM Plant RNA Protocol Ⅱ(Omega)法提取各样品的总RNA,并采用RQ1 RNase-Free DNase(Promega)去除基因组DNA。随后,按照5×PrimeScript RT Master Mix(PerfeCt Real Time)试剂盒(TaKaRa)操作说明反转录合成cDNA第一链,紫外分光光度计检测所得cDNA的浓度,并将各样品的cDNA浓度统一至100 ng/μL,分别用于目标基因克隆、半定量PCR和qPCR的表达分析。
1.2.2 目标基因克隆及序列分析将TAIR(The Arabidopsis Information Resource)数据库中拟南芥CAC基因(登录号为AT5G46630)的核酸序列与本研究小组前期构建的珙桐转录组数据库[17]中的基因序列进行本地BLAST比对,获得一条同源性较高的片段c44712.graph_c0。根据其ORF序列信息设计基因特异性扩增引物DiCAC-F1(5'-GGAAAGATGCCGGTGGCTGCT-3')和DiCAC-R1(5'-CTAGGACCTAATCTCATAT GAACCA-3')。以珙桐苞片cDNA为模板,PCR扩增程序如下:94℃预变性5 min;94℃变性45 s,51℃退火45 s,72℃延伸2 min,35个循环;72℃延10 min。扩增产物经琼脂糖凝胶电泳检测后,切胶回收纯化,与pMD18-T载体(TaKaRa)连接,转化至大肠杆菌DH5α感受态细胞,挑选阳性克隆送往铂尚生物技术有限公司测序。
利用ProtParam、ProtScale、Psort等在线软件对该基因编码的氨基酸序列进行组成成分、理化性质、亚细胞定位预测等初步分析;利用DNAMAN软件进行氨基酸序列的比对及同源性分析;利用MEGA 5.0软件Neighbor joining(NJ)算法进行聚类分析。
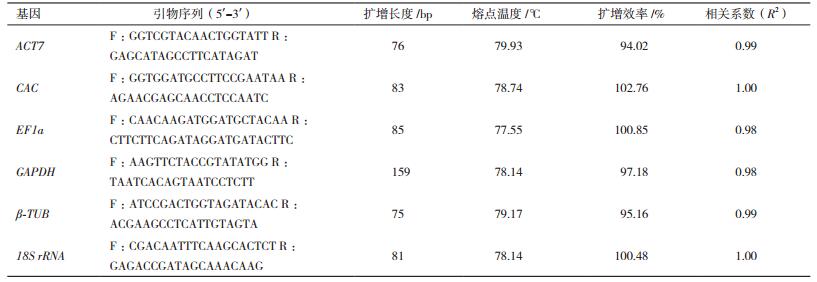
1.2.3 目标基因及5种管家基因作为内参基因的稳定性评价从珙桐转录组数据库中获得c44712. graph_c0与5个管家基因(ACT7、EF1a、GAPDH、β-TUB、18S rRNA)的序列信息,根据qPCR引物设计原则,通过网站IDTdna(http://eu.idtdna.com/primerquest)设计各内参基因的qPCR引物(表 1),引物序列由上海生工公司合成。qPCR反应体系为10 μL,包括SYBR Green Master Mix(High ROX)(Biotool)5 μL、2 μmol/L Forward Primer 2 μL、2 μmol/L Reverse Primer 2 μL、100 ng/μL cDNA 1 μL;反应程序为:95℃ 5 min,95℃ 15 s,60℃ 40 s,40 cycles。
将所得qPCR数据进行整理,利用在线分析软件ReFinder(http://fulxie.0fees.us/?type=reference&i=1)分别对此6个内参基因在珙桐不同组织以及不同发育阶段苞片中的表达稳定性进行评价。ReFinder整合了4种常用于内参基因稳定性评价的方法:即geNorm、Normfinder、BestKeeper和ΔCt值分析法[18-21]。ReFinder程序首先分别用上述4种方法对所有内参基因的表达稳定性进行评价并给出排名,然后计算各基因由不同方法所得的排名的几何平均值,以此作为综合评价指数,指数数值越小,表明基因表达越稳定[22]。
1.2.4 目标基因表达分析在珙桐不同组织中,以EF1a(ReFinder软件分析结果中EF1a的表达稳定性综合排名位于最末)作为对照,进行DiCAC的半定量PCR及qPCR表达分析;在不同发育阶段苞片中,以GAPDH(ReFinder软件分析结果中GAPDH的表达稳定性综合排名位于最末)作为对照,进行DiCAC的半定量PCR及qPCR表达分析。qPCR表达分析计算公式为:Q(相对表达量)=2ΔCt,ΔCt=Ct(叶芽)-Ct(其它组织)或ΔCt=Ct(幼期苞片)-Ct(其它苞片),应用SPSS 17.0软件对基因的相对表达量进行差异显著性分析。半定量PCR的基因引物序列及扩增程序,见表 2。
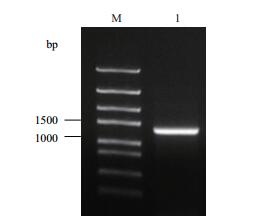
以珙桐叶片cDNA为模板,对珙桐CAC基因的ORF序列进行扩增,扩增产物经琼脂糖凝胶电泳检测,发现在接近1 500 bp处有一条清晰亮带(图 1)。经测序,该序列长度为1 317 bp,编码438个氨基酸,编码产物与拟南芥CAC基因同源性达94%,确认其为一个珙桐的CAC基因,将其命名为DiCAC,已在GenBank上注册,登录号为KX268525。
|
| 图 1 DiCAC基因PCR扩增琼脂糖凝胶电泳图 M:分子量标准;1:DiCAC基因PCR产物 |
对DiCAC编码的氨基酸序列进行初步的理化性质分析。该氨基酸的相对分子量大小约为49.20 kD,理论等电点为9.34。氨基酸组成中,含量最多的3种氨基酸依次为缬氨酸(10.3%)、亮氨酸(8.2%)、赖氨酸(8.0%),且碱性氨基酸(赖氨酸8.0%,精氨酸5.3%、组氨酸0.2%)含量略多于酸性氨基酸(天冬氨酸3.9%、谷氨酸6.2%)。总平均亲水系数为-0.039,其中,第23位的酪氨酸亲水性最强,第70位的异亮氨酸疏水性最强。亚细胞定位预测其有56.5%的几率定位于细胞质内。
2.2 珙桐DiCAC基因的同源性分析与聚类分析将DiCAC基因序列在NCBI网站上进行Blast X同源性比对分析,结果表明,DiCAC氨基酸序列与葡萄(Vitis vinifera)、枣(Ziziphus jujuba Mill.)、甜橙(Citrus sinensis)、可可(Theobroma cacao)、蓖麻(Ricinus communis)、莲(Nelumbo nucifera)的CAC氨基酸序列相似度较高,同源性达到97%,其次,与胡杨(Populus euphratica)、无油樟(Amborella trichopoda)、苹果(Malus x domestica)、烟草(Nicotiana tabacum Linn.)、大豆(Glycine max)的同源性达到96%,与巨桉(Eucalyptus grandis)、番茄(Solanum lycopersicum)的同源性达到96%,与拟南芥(Arabidopsis thaliana)的同源性达到94%。说明本研究中克隆到的基因为珙桐中CAC基因,且该基因在木本植物中较为保守。
将DiCAC氨基酸序列与筛选出的同源性较高的10种植物CAC氨基酸序列进行多重比较,结果(图 2)表明,不同植物的CAC氨基酸序列只在个别位置有突变发生,其中,珙桐DiCAC基因与其它10种植物CAC基因编码的氨基酸共有8个位点的差异,分别是37位的M(甲硫氨酸)/I(异亮氨酸)、38位的H(组氨酸)/N(天冬酰胺)、80位的C(半胱氨酸)/S(丝氨酸)、308位的V(缬氨酸)/I(异亮氨酸)、388位的T(苏氨酸)/N(天冬酰胺)0、389位的R(精氨酸)/K(赖氨酸)、431位的A(丙氨酸)/G(甘氨酸)、438位的C(半胱氨酸)/S(丝氨酸)。

|
| 图 2 珙桐CAC氨基酸序列与其它植物CAC氨基酸序列的多重比对分析 Nt:Nicotiana tabacum Linn(XM_016637430);Sl:Solanum lycopersicum(XM_004241917);Zj:Ziziphus jujuba Mill.(XM_016010516);Cs:Citrus sinensis(XM_015532479);Vv:Vitis vinifera(XM_002281261);Eg:Eucalyptus grandis(XM_010057029);Tc:Theobroma cacao(XM_007039461);Md:Malus x domestica(XM_008342858);Di:Davidia involucrata Baill.(KX268525);Pe:Populus euphratica(XM_011029892);At:Arabidopsis thaliana(NM_124033) |
为了进一步探讨珙桐DiCAC基因与其它植物CAC基因之间的进化关系,选取一致性较高的16种植物的CAC基因的编码产物氨基酸序列进行聚类分析,结果(图 3)表明,DiCAC与葡萄(Vitis vinifera)CAC蛋白的亲缘关系最近,其次与可可(Theobroma cacao)CAC蛋白具有相对较近的亲缘关系,而与巨桉(Eucalyptus grandis)、烟草(Nicotiana tabacum Linn.)、番茄(Solanum lycopersicum)等其它植物CAC蛋白组成的小进化群体则亲缘关系较远。

|
| 图 3 珙桐CAC氨基酸序列与其它植物CAC氨基酸序列的聚类分析 |
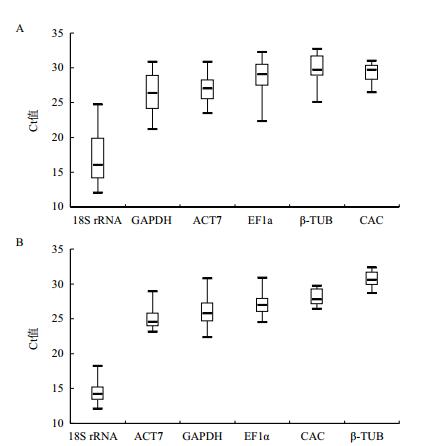
表 1中各基因引物的扩增效率大于94%,相关系数R2>0.98,可知各基因引物的扩增效率良好,能用于qPCR分析。基因的Ct值与表达丰度成反比,Ct值越小,基因的表达丰度越高,反之亦然。首先通过qPCR检测6个内参基因在珙桐样品中的的Ct值,然后对它们的表达丰度进行比较。结果表明,在不同组织中,6个基因当中18S rRNA的Ct值在12-24之间,表达丰度最高,CAC的Ct值在26-30之间,表达丰度最低(图 4-A);在不同发育阶段的苞片中,6个基因当中18S rRNA的Ct值在12-18之间,表达丰度最高,β-TUB的Ct值在28-32之间,表达丰度最低,CAC的Ct值在27-29之间,表达丰度仅略高于β-TUB(图 4-B)。
|
| 图 4 6个内参基因在珙桐不同样品中的Ct值分布 A:6个内参基因在珙桐不同组织中的Ct值分布;B:6个内参基因在珙桐不同发育阶段苞片中的Ct值分布。箱体代表Ct值的集中范围,箱体中央横线代表中位数,箱体上、下边分别代表上四分位数和下四分位数,箱体上、下端箱须分别代表最大值与最小值;6个内参基因根据中位数值由低到高依次排列 |
此外,通过比较发现,在珙桐两组样品中,CAC的Ct值在不同样品间的变化范围均相对较小,数据较为集中,尤其是在不同发育阶段的苞片样品中,Ct值的波动范围更小;而5个管家基因的Ct值在不同样品间变化范围相对较大,数据较为分散。初步判断可知,珙桐CAC基因在不同组织与不同发育阶段苞片中的的表达均较为稳定(图 4)。
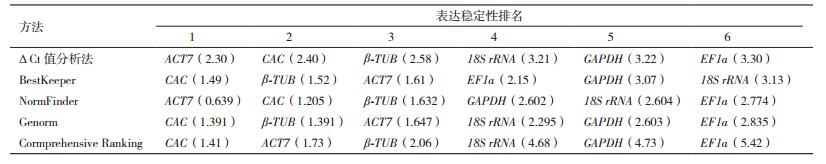
2.4 珙桐DiCAC基因及5种管家基因作为内参基因的稳定性评价利用ReFinder软件,对6个内参基因在珙桐不同组织中的表达稳定性进行评价,结果显示(表 3),在ΔCt值分析法与NormFinder算法结果中,稳定性排名第一的均为ACT7,排名第六的均为EF1a;在BestKeeper与Genorm算法结果中,稳定性排名第一的均为CAC,排名第六的分别为18S rRNA 和EF1a;不同算法结果中,6个内参基因的稳定性排名并不完全一致,经采用几何平均值法综合分析后,基因的表达稳定性由高到低依次为:CAC>ACT7>β-TUB>18S rRNA>GAPDH>EF1a。
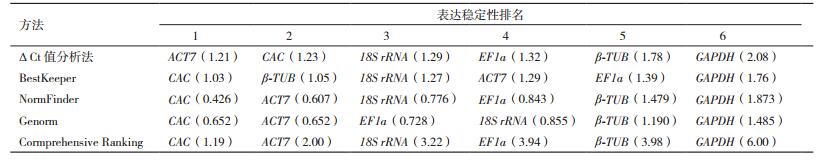
利用ReFinder软件,对6个内参基因在珙桐不同发育阶段苞片中的表达稳定性进行评价,结果显示(表 4),除在ΔCt值分析法算法结果中稳定性排名第一的为ACT7外,在其余的BestKeeper、NormFinder、Genorm 3种算法结果中,稳定性排名第一的均为CAC;另外,在ΔCt值分析法、BestKeeper、NormFinder、Genorm 4种算法结果中,稳定性排名第六的均为GAPDH;同样,4种算法结果中,6个内参基因的稳定性排名并完全一致,经采用几何平均值法综合分析后,基因的表达稳定性由高到低依次为:CAC>ACT7>18S rRNA> EF1a>β-TUB>GAPDH。对6个内参基因分别在珙桐两组样品中的表达稳定性综合排名进行比较,分析可知,在此两组样品中,基因表达稳定性排名居于首位的均为CAC,而稳定性排名最末位的为不同基因,在不同组织中为EF1a,在不同发育阶段苞片中为GAPDH。
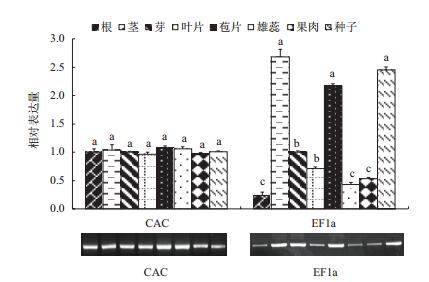
CAC和EF1a基因在洪洞不同组织中的半定量PCR电泳结果表明(图 5):对照基因EF1a在珙桐茎、芽、苞片(中期)、种子中的表达量较高,而在根、叶片、雄蕊、果肉中的表达量较低;不同于EF1a,DiCAC在8个组织中的表达量基本一致。在qPCR中,以芽的表达量为1计算,相对定量结果表明:对照基因EF1a在珙桐茎、苞片、种子中的表达量较高,2.5左右,芽和叶片中次之,而在根、雄蕊、果肉中表达量较低,0.5左右;相比于EF1a,DiCAC在珙桐各组织中的表达量基本一致,均为1左右,差异不显著。
|
| 图 5 CAC和EF1a在珙桐不同组织中的表达模式 直方柱上小写字母相同表示差异不显著(P ≥ 0.05);直方柱上小写字母不同表示差异显著(P ≤ 0.05) |
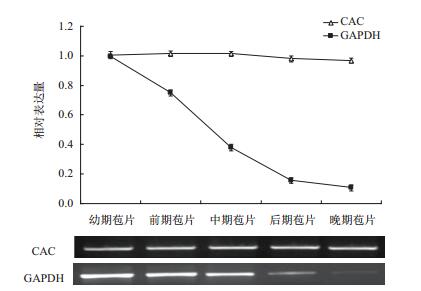
DiCAC和GAPDH基因在珙桐不同发育阶段苞片中半定量PCR和qPCR表达分析。半定量PCR电泳结果表明(图 6):对照基因GAPDH在发育幼期、前期、中期的苞片中表达量较高,而在发育后期、晚期的苞片中表达量较低;与GAPDH不同,DiCAC在各发育阶段的苞片中表达量无明显差异。在qPCR中,以芽的表达量为1计算,相对定量结果表明:GAPDH在发育幼期的苞片中表达量较高,此后,随着苞片发育不断成熟,表达量逐渐降低,晚期苞片中表达量约降至0.1;不同于GAPDH,DiCAC在不同发育阶段苞片中的表达量基本保持一致,均在1左右。综上可知,DiCAC在珙桐不同组织和不同发育阶段苞片中均呈稳定表达模式。
|
| 图 6 DiCAC和GAPDH在珙桐不同发育阶段苞片中的表达模式 |
CAC蛋白参与植物中许多重要的生命活动,其中,最主要的功能即是在网格蛋白介导的质膜内吞过程中负责货物蛋白的分拣、网络蛋白和其他辅助蛋白的招募等,此外,也参与细胞生长发育、细胞信号传导以及应对外界环境的刺激等生命活动过程[23, 24]。近年来的一些研究表明,CAC氨基酸序列高度保守,且表达量在不同细胞或组织中变化较小,可以作为某些植物基因表达分析中的内参基因。例如,CAC在拟南芥、西红柿与荞麦的各组织器官、竹子的不同发育时期、西瓜果实发育的不同阶段、鹰嘴豆非生物胁迫处理下表达都很稳定,可作为内参基因用于对目标基因进行校准[3, 12-16]。但目前有关珙桐CAC基因的研究尚未见报道。
本研究从珙桐中克隆到一个CAC基因,利用生物信息学手段分析了该基因氨基酸序列的组成成分以及一些理化性质,将其与拟南芥、葡萄、大豆、桃、芹菜等其它植物的CAC氨基酸序列特征进行比较[25],结果发现不同植物来源CAC氨基酸序列的组成成分及各类理化性质较为相似,如氨基酸残基数均为438个,相对分子质量为49.20-49.40 kD,等电点为9.20-9.40,碱性氨基酸含量略多于酸性氨基酸,蛋白质疏水性较强等。进一步将珙桐DiCAC基因与葡萄、枣、胡杨、巨桉、拟南芥等其它植物CAC基因进行同源性分析与聚类分析,结果表明,珙桐DiCAC基因编码的氨基酸序列与其它植物CAC氨基酸序列虽有个别位点的差异,但整体上氨基酸序列相似度达到了94%以上,可见珙桐DiCAC基因与其它植物,尤其是木本植物CAC基因的同源性较高。其中,与葡萄CAC氨基酸序列相似度为97%,同源性最高,亲缘关系最近。综上所述,基因序列和结构较高的相似性说明本研究中克隆到的珙桐DiCAC基因准确可靠。同时,不同植物的CAC基因编码的氨基酸序列具有高度同源性,进一步表明植物CAC基因高度保守,这与它参与植物的多种重要生命活动密切相关。
研究表明,不同物种间同源内参基因的表达存在差异,即在某物种中表达稳定性高的内参基因在另一物种中的表达稳定性并不一定高,甚至可能较差。例如,CAC基因在拟南芥与番茄的各组织中表达稳定,可以作为内参基因,但其在侧柏、葡萄、柚木各组织中的表达稳定性却较差,不适合作为内参基因[3, 12, 26-28]。因此,虽然不同植物间CAC基因具有较高的同源性,并且有报道表明CAC可作为拟南芥、番茄等植物中的内参基因,但是,并不能就此认为DiCAC同样适合在珙桐中作为内参基因使用。为此,本研究通过对DiCAC基因在珙桐不同组织以及不同发育阶段苞片中的表达特性进行分析,以判断DiCAC作为内参基因的可行性。在珙桐两组样品中,结合5个常用管家基因ACT7、EF1a、GAPDH、β-TUB、18S rRNA,运用多种方法对包括DiCAC在内的6个基因的表达稳定性进行评价。经分析可知,虽然由于各方法的运算原理和判断标准不完全相同,导致4种分析方法中内参基因的稳定性排名存在一定差异,但是,整体排名结果基本一致,最终,综合排名结果表明,DiCAC在珙桐不同组织与不同发育阶段苞片中的表达稳定性排名均居于首位,这说明相对于其它5个管家基因,DiCAC在珙桐不同样品间表达更为稳定。为了进一步检验DiCAC的表达稳定性,本研究结合半定量PCR与qPCR两种方法对DiCAC分别在珙桐两组样品中的表达量进行了分析,结果发现DiCAC在两组样品中的表达量均基本一致,相反,分别作为对照的EF1a与GAPDH的表达量却波动较大,此结果与ReFinder程序的分析结果基本吻合。因此,推断DiCAC在珙桐不同组织以及不同发育阶段苞片中均呈组成型表达,作为此特定条件下的内参基因是可行的。此外,在基因表达分析中,理想的内参基因不仅表现为表达水平稳定,而且要求其稳定的表达量与目标基因的表达量相近[29]。本研究发现,DiCAC在珙桐不同组织与不同发育阶段苞片中的表达丰度均较低,因此,推断其更适合用于对表达丰度较低的目标基因进行校准。
综上所述,本研究首次克隆了珙桐CAC基因,并且对其在珙桐不同组织以及不同发育阶段苞片中作为内参基因的可行性进行了评价。DiCAC的克隆不仅有效填补了珙桐中CAC基因研究的空白,丰富了植物CAC基因核酸数据库,而且为深入研究珙桐相关基因的表达分析提供了参照序列和参考依据,这将有助于提高基因表达分析结果的精确性。但是,DiCAC作为内参基因是否同样适用于珙桐的其它试验条件中还有待于进一步研究。
4 结论本研究从珙桐中克隆到一个编码网格蛋白衔接蛋白复合物的基因DiCAC(GenBank登录号为KX268525),该基因开放阅读框全长1 317 bp,编码438个氨基酸,与其他植物的CAC氨基酸序列相似度达到94%以上,其中与葡萄CAC氨基酸序列的同源性最高。半定量PCR与实时荧光定量PCR分析结果表明,DiCAC在珙桐不同组织以及不同发育阶段苞片中表达均相对稳定,且表达稳定性优于常见管家基因,可以作为珙桐相关基因表达研究中的内参基因。
| [1] | Bustin SA, Benes V, Nolan T, et al. Quantitative real-time RT-PCR a perspective. J Mol Endocrinol, 2015, 34 (3): 597–601. |
| [2] | VanGuilder HD, Vrana KE, Freeman WM. Twenty-five years of quantitative PCR for gene expression analysis. Biotechniques, 2008, 44 (5): 619. |
| [3] | Czechowski T, Stitt M, Altmann T, et al. Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis. Plant Physiol, 2005, 139 (1): 5–17. DOI:10.1104/pp.105.063743 |
| [4] | Libault M, Thibivilliers S, Bilgin DD, et al. Identification of four soybean reference genes for gene expression normalization. The Plant Genome, 2008, 1 (1): 44–54. DOI:10.3835/plantgenome2008.02.0091 |
| [5] | Yang H, Liu J, Huang S, et al. Selection and evaluation of novel reference genes for quantitative reverse transcription PCR based on genome and transcriptome data in Brassica napus L. Gene, 2014, 538 (1): 113–122. DOI:10.1016/j.gene.2013.12.057 |
| [6] | 袁伟, 万红建, 杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择. 植物学报, 2012, 4: 427–436. |
| [7] | 中国植被编委会. 中国植被[M]. 北京: 科学出版社, 1980: 298-299. |
| [8] | Vekemans D, Viaene T, Caris P, et al. Transference of function shapes organ identity in the dove tree inflorescence. New Phytol, 2012, 193 (1): 216–228. DOI:10.1111/j.1469-8137.2011.03915.x |
| [9] | Lei N, Peng S, Niu B, et al. Molecular cloning and characterization of a novel microsomal oleate desaturase gene DiFAD2 from Davidia involucrata Baill. Biol Plantarum, 2010, 54 (1): 41–46. DOI:10.1007/s10535-010-0006-2 |
| [10] | Motley AM, Berg N, Taylor MJ, et al. Functional analysis of AP-2 α and μ2 subunits. Molecular Biology of the Cell, 2006, 17 (12): 5298–5308. DOI:10.1091/mbc.E06-05-0452 |
| [11] | Jackson LP, Kelly BT, McCoy AJ, et al. A large-scale conformational change couples membrane recruitment to cargo binding in the AP2 clathrin adaptor complex. Cell, 2010, 141 (7): 1220–1229. DOI:10.1016/j.cell.2010.05.006 |
| [12] | Rodriguez EM, Borges AA, Borges-Perez A, et al. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process. BMC Plant Biol, 2008, 8 (1): 131. DOI:10.1186/1471-2229-8-131 |
| [13] | Demidenko NV, Logacheva MD, Penin AA. Selection and validation of reference genes for quantitative real-time PCR in buckwheat (Fagopyrum esculentum) based on transcriptome sequence data. PLoS One, 2011, 6 (5): e19434. DOI:10.1371/journal.pone.0019434 |
| [14] | Fan C, Ma J, Guo Q, et al. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis). PLoS One, 2013, 8 (2): e56573. DOI:10.1371/journal.pone.0056573 |
| [15] | Reddy DS, Bhatnagar-Mathur P, Reddy PS, et al. Identification and validation of reference genes and their impact on normalized gene expression studies across cultivated and wild cicer species. PLoS One, 2016, 11 (2): e0148451. DOI:10.1371/journal.pone.0148451 |
| [16] | Kong Q, Yuan J, Gao L, et al. Evaluation of appropriate reference genes for gene expression normalization during watermelon fruit development. PLoS one, 2015, 10 (6): e0130865. DOI:10.1371/journal.pone.0130865 |
| [17] | Li M, Dong XJ, Peng JQ, et al. De novo transcriptome sequencing and gene expression analysis reveal potential mechanisms of seed abortion in dove tree (Davidia involucrata Baill.). BMC Plant Biol, 2016, 16 : 82. DOI:10.1186/s12870-016-0772-x |
| [18] | Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol, 2002, 3 (7): RESEARCH0034. |
| [19] | Andersen CL, Jensen JL, Ørntoft TF. Normalization of realtime quantitative reverse transcription-PCR data:a modelbased variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res, 2004, 64 (15): 5245–5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [20] | Pfaffl MW, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity:BestKeeper Excel-based tool using pair-wise correlations. Biotechnol Lett, 2004, 26 (6): 509–515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [21] | Silver N, Best S, Jiang J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR. BMC Mol Biol, 2006, 7 (1): 33. DOI:10.1186/1471-2199-7-33 |
| [22] | Chen D, Pan X, Xiao P, et al. Evaluation and identification of reliable reference genes for pharmacogenomics, toxicogenomics and small RNA expression analysis. J Cell Physiol, 2011, 226 (10): 2469–2477. DOI:10.1002/jcp.v226.10 |
| [23] | Lacruz RS, Brookes SJ, Wen X, et al. Adaptor protein complex2-mediated, clathrin-dependent endocytosis, and related gene activities, are a prominent feature during maturation stage amelogenesis. Journal of Bone and Mineral Research, 2013, 28 (3): 672–687. DOI:10.1002/jbmr.1779 |
| [24] | Di Rubbo S, Irani NG, Kim SY, et al. The clathrin adaptor complex AP-2 mediates endocytosis of brassinosteroid insensitive1 in Arabidopsis. The Plant Cell, 2013, 25 (8): 2986–2997. DOI:10.1105/tpc.113.114058 |
| [25] | 王广龙, 王枫, 徐志胜, 等. 芹菜衔接蛋白基因AgMu2的克隆与表达分析. 园艺学报, 2014, 7: 1369–1378. |
| [26] | Chang E, Shi S, Liu J, et al. Selection of reference genes for quantitative gene expression studies in Platycladus orientalis (Cupressaceae) using real-time PCR. PLoS One, 2012, 7 (3): e33278. DOI:10.1371/journal.pone.0033278 |
| [27] | Reid KE, Olsson N, Schlosser J, et al. An optimized grapevine RNA isolation procedure and statistical determination of reference genes for real-time RT-PCR during berry development. BMC Plant Biology, 2006, 6 : 27. DOI:10.1186/1471-2229-6-27 |
| [28] | Galeano E, Vasconcelos TS, Ramiro DA, et al. Identification and validation of quantitative real-time reverse transcription PCR reference genes for gene expression analysis in teak (Tectona grandis L. f.). BMC Research Notes, 2014, 7 (464). |
| [29] | Gimeno J, Eattock N, Van Deynze A, et al. Selection and validation of reference genes for gene expression analysis in switchgrass (Panicum virgatum) using quantitative real-time RT-PCR. PLoS One, 2014, 9 (3): e91474. DOI:10.1371/journal.pone.0091474 |