甜菊糖苷(SGs)是菊科植物甜叶菊次生代谢产生的一类四环二萜类物质,因其高甜度、低能量且无毒副作用等优势逐渐成为替代蔗糖的理想甜味剂[1, 2]。随着人们对甜菊糖苷的关注,甜菊糖苷生物合成途径的研究也日益成为科研工作的热点[3]。甜菊糖苷主要在甜叶菊的绿色组织中合成[4],整个生物合成途径共包括16个酶促反应[5]。初始的七步反应用来合成异戊烯焦磷酸(IPP),IPP与甲基赤藓糖醇途径(MEP)合成的二甲烯丙基焦磷酸(DMAPP)经过接下来的4个酶促反应生成内根-贝壳杉烯酸[6],这四步反应在甜叶菊体内与赤霉素的生物合成共享[6]。最后5个酶促反应则是甜菊糖苷生物合成所特有[6],其中关键节点酶促反应由不同的内根-贝壳杉烯酸脱氢酶(KAH)控制,在13位碳位置脱氢就生成为不同甜菊糖苷的基本骨架的甜菊醇,若在7位碳位置脱氢则合成赤霉素[7, 8]。因此不同的KAH编码基因调控植物体内赤霉素和甜菊糖苷的合成。
甜菊糖苷生物合成途径中所特有的酶促反应中的酶已经被成功纯化并验证其催化活性[9, 10, 11],编码这些酶的基因也逐渐被克隆。目前4种糖基转移酶的编码基因均已被成功克隆,其中SrUGT85C2、SrUGT74G1和SrUGT76G1不仅在异源酵母表达[10, 12]中成功验证相对应的糖基转移酶活性,并且用农杆菌介导转录水平沉默技术在甜叶菊叶片中也成功证明其对应的糖基转移酶活性[5];Wang等[13]首次克隆到具有将甜菊双糖苷转化为甜菊单糖苷活性的糖基转移酶基因SrUGT91d2,并成功通过异源体系大肠杆菌验证了其编码活性。关键分支点的内根-贝壳杉烯酸脱氢酶的编码基因目前已有的研究中共克隆到8个,包括已验证无催化酶活的KAH_ AEH65420、KAH_AEH65421和KAH_AEH65423共3个基因[1],未验证催化活性的FJ494665[14],已验证具有催化活性的包括来源于甜叶菊的KAHn2、KAH_ACD93722、DQ398871.3和来源于拟南芥并经过优化的CYP714A2[13]。通过农杆菌介导技术瞬时沉默DQ398871.3会显著降低所有甜菊糖苷的含量[5];KAHn2和KAH_ACD93722在异源体系大肠杆菌中内根-贝壳杉烯酸转化活性极低;来源于拟南芥的CYP714A2最早在异源酵母体系中发现具有贝壳杉烯酸脱氢酶活性,通过将其氮端进行不同的改造优化,最终可转化甜菊醇浓度最高达到15.47 mg/L[13]。
综上所述,甜菊糖苷生物合成途径的关键节点酶-内根-贝壳杉烯酸脱氢酶(KAH)的编码基因虽然克隆成功,但真正具有高效转化活性的內源基因并没有,即使是改造后的外源CYP714A2活性也远低于预期值。因此本研究从多个甜叶菊材料的基因库中扩增KAH编码基因,并结合生物信息学等手段对获得的不同KAH基因进行分析、生物功能预测和表达分析,为筛选高效转化活性的新KAH编码基因、进一步基因改造及异源高效表达等提供更多內源KAH候选基因,同时也进一步揭示甜菊糖苷积累的分子调控机制。获得内源性的具有高效转化活性的KAH基因将进一步完善甜菊糖苷生物合成的分子调控机制并为高效异源合成甜菊糖苷提供候选基因,对甜菊糖苷生物合成机制的研究和工业生产都具有重要的意义。
1 材料与方法 1.1 供试材料植物材料:江甜1号、江甜3号、鑫丰3号、谱星1号、守田3号;本实验室甜叶菊新品系SR1、SR2和SR3。载体:pMD-19T。
1.2 方法 1.2.1 基因序列的获得 1.2.1.1 不同KAH基因核酸序列的获得从NCBI数据库中下载已报道的编码内根-贝壳杉烯酸脱氢酶的SrKA13H的核酸序列,根据其保守区序列设计扩增不同甜叶菊中该酶的编码基因,引物序列KAH-F(5'-3'):TAGGTA CCGAATGATTCAAGTTCTAACACCGAT;KAH-R(5'-3')):GCGAGCTCCACATAATCTGTA ACTACCAATAA。采集田间不同品种甜叶菊开花前的叶片,用OMEGA公司的RNA提取试剂盒提取不同甜叶菊品种的总RNA,并用TAKARA公司的反转录试剂盒反转录得到不同品种的总cDNA。分别用不同品种的cDNA做模版,以上述引物扩增不同品种的KAH编码基因。PCR扩增到的不同序列分别酶连到克隆载体pMD-19T后转化入大肠杆菌(EH5α)中,摇菌并菌液PCR验证酶连成功后送至Invitrogen公司测序获得核酸序列。选择来源于总糖苷含量最高的SR2和相对含量较低的守田3号的多个菌落进行测序,其他品种仅进行单菌落测序。
1.2.1.2 拟南芥P450家族基因序列的获得拟南芥中细胞色素P450家族基因的核酸和氨基酸序列均从P450数据库(http://drnelson.uthsc.edu/CytochromeP450.html)下载获得。
1.2.2 不同KAH基因的生物信息学分析不同KAH基因的核苷酸和氨基酸序列差异分析由软件DNAMAN完成,基因的开放阅读框(ORF)预测由在线软件ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)和DNAMAN完成,核苷酸翻译为氨基酸序列由Primer premier 5.0完成,核苷酸序列的保守区域分析在NCBI在线分析完成(http://www.ncbi.nlm.nih.gov/guide/domains-structures/),不同KAH基因和拟南芥中P450家族成员的进化树分析由MEGA5.1软件完成。
1.2.3 定量PCR检测SrKAHs表达分别采集收获期江甜3号、守田3号和谱星1号的叶片,苗期守田3号植株顶部叶片(顶叶)、中间部位叶片(中叶)、植株近基部叶片(底叶)、茎和根五个不同组织部位的植物样品,采用OMEGA公司针对多糖多酚样品的RNA提取方法获得其RNA并通过TAKARA公司的定量专用反转录试剂盒获得cDNA。利用ABI Primer Express设计SrKAHs特异扩增定量引物(F:ACCCATCGCGGTTTGAAG;R:CCCCTCCAAATGGAACATATGT),采用ABI 7500定量PCR仪检测SrKAHs在甜叶菊不同品种和不同组织部位中的表达。
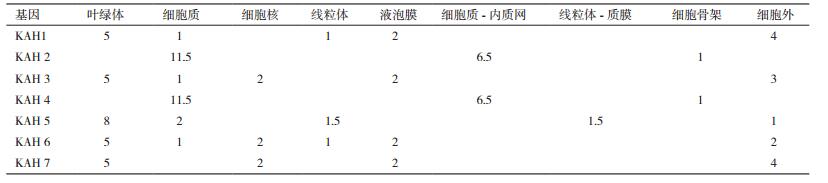
2 结果 2.1 不同品种甜叶菊中KAH编码序列分析 2.1.1 不同KAH基因的核苷酸序列分析从不同甜叶菊品种中克隆到的KAH核苷酸序列经DNAMAN软件分析结果(图 1)表明,共扩增到6组7个不同KAH编码序列。守田3号中扩增到两个不同的KAH核酸序列,其中一条KAH核酸序列与来源于SR1、SR3、鑫丰3号、谱星1号KAH核酸序列完全一致,长度为1 501 bp,经分析后存在两个开放阅读框分别命名为KAH1和KAH2,另一个KAH核酸序列与来源于江甜1号的KAH序列完全一致,长度为1 502 bp,命名为KAH3;江甜3号中扩增到一个不同于其他品种但同样长为1 502 bp的KAH核酸序列,命名为KAH4;本实验室甜叶菊新品系SR2中共扩增到3个长度不一的KAH核酸序列,较短的一条为1 498 bp,两条较长的为1 503 bp,分别命名为KAH5、KAH6和KAH7(表 1)。通过比对结果可知,这6组不同的核酸序列高度同源,由于开放阅读框的位置差异,7个不同KAH基因的编码序列在3'端高度保守,核酸序列差异均是个别碱基的缺失或改变。
|
| 图 1 不同KAH基因的编码核酸序列同源性比对 KAH1和KAH2分别代表来源于守田3号的两个基因的编码核酸序列;KAH3是来源于江甜1号中的KAH编码核酸序列;KAH4是来源于江甜3号KAH编码核酸序列;KAH5、KAH6和KAH7是来源于甜叶菊新品系SR2中的3个不同KAH的编码核酸序列 |
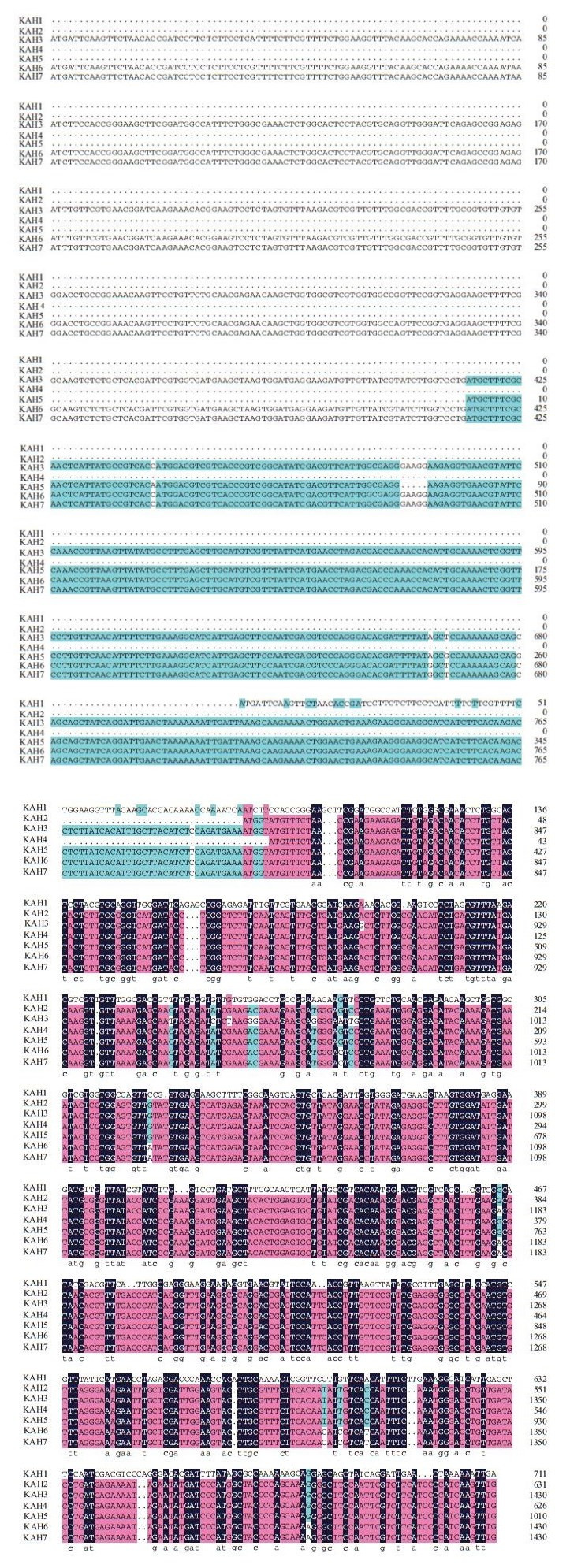
从8个甜叶菊品种中共扩增到6组不同的KAH核酸序列,经过ORF Finder和DNAMAN分析预测后共获得7个不同ORF。来源于守田3号等品种的KAH1和KAH2分别编码236和208个氨基酸;从江甜1号扩增获得的KAH3核酸序列翻译成476个氨基酸;江甜3号的KAH核酸序列预测得到3个ORF但经分析只有编码208个氨基酸的最长的ORF具有保守序列命名为KAH4;来源于SR2的3个KAH核KAH5编码336个氨基酸,KAH6和KAH7均编码476个氨基酸(表 1)。核酸序列的个别碱基改变导致了ORF的位置和大小的变化,因此氨基酸同源性比对结果显示7个氨基酸序列差异较大(图 2-A)。根据同源性可以分为3组,第1组的KAH2、KAH4虽然长度比KAH5短,但氨基酸序列均与KAH5的部分氨基酸序列完全匹配;第2组KAH3、KAH6和KAH7虽然均为476个氨基酸,但同源性为98%;这两组KAH序列的同源度为96%;第3组的KAH1与其他6个基因的同源度仅为14%(图 2-B)。
|
| 图 2 不同KAH基因的氨基酸序列比对 (A)不同KAH基因氨基酸序列的差异。(B)不同KAH基因氨基酸序列的同源度。KAH1和KAH2分别代表守田3号中的KAH核酸序列预测的2个ORF编码的氨基酸序列;KAH3是江甜1号中的KAH核酸序列编码的氨基酸序列;KAH4是江甜3号核酸序列预测的第3个开放阅读框编码的氨基酸序列;KAH5、KAH6和KAH7是甜叶菊新品系SR2中的3个不同KAH核酸序列编码的氨基酸序列 |
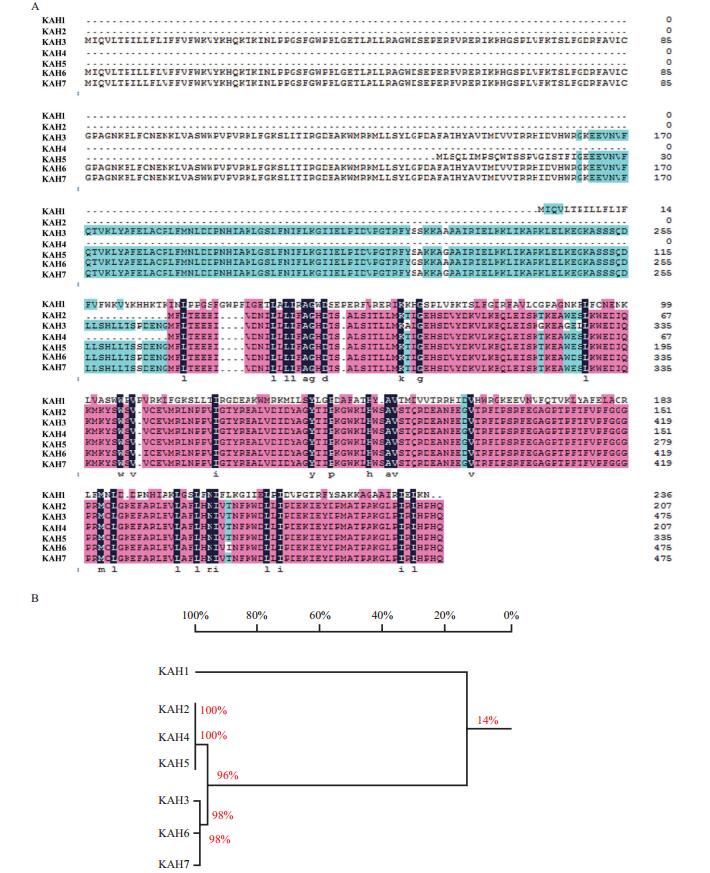
根据不同的KAH编码序列的比对结果和定量引物设计优先选择检测的位置,7个不同的KAH基因在mRNA水平无法区分,因此在不同SrKAHs保守区域设计定量引物分别检测在收获期不同品种和苗期守田3号不同组织部位中所有KAH编码基因的同时表达的水平。结果(图 3-A)显示SrKAHs在江甜3号叶片中表达最高,其次是守田3号,谱星1号叶片中表达最低。不同组织部位表达检测结果(图 3-B)显示,SrKAHs在叶片中表达高于根系,茎中表达最低;SrKAHs在不同冠层叶片中表达差异也非常显著,中叶中表达最高,底叶其次,顶叶表达最低。
|
| 图 3 SrKAHs表达模式 A:SrKAHs在不同品种甜叶菊叶片中表达;B:SrKAHs在甜叶菊不同组织中表达 |
通过NCBI在线分析7个不同KAH基因的保守区域发现在它们的氨基酸序列中均包含细胞色素P450家族的保守区。将7个KAH基因的氨基酸序列与拟南芥中的P450家族基因核酸序列进行进化分析,结果(图 4)表明这7个KAH基因均与拟南芥中CYP716A亚家族进化关系最近。拟南芥中CYP716A亚家族已报道的共有两个基因CYP716A1和CYP716A2,通过DNAMAN软件分析确定7个KAH按照编号顺序与CYP716A1的同源性依次为44.50%、52.46%、46.67%、52.46%、48.42%、46.44%和46.90%,与CYP716A2的同源性依次为44.27%、55.74%、47.71%、55.74%、50.18%、47.48%和47.94%(表 1)。

|
| 图 4 甜叶菊不同KAH基因和拟南芥P450家族成员进化树 A:不同KAH基因和拟南芥中P450家族进化树;B:不同KAH序列与拟南芥中CYP716、CYP718家族进化分析。KAH1和KAH2分别代表守田3号核酸预测的两个开放阅读框;KAH3是江甜1号的KAH;KAH4是江甜3号核酸序列预测的第3个开放阅读框;KAH5、KAH6和KAH7是甜叶菊新品系SR2中扩增到的3个不同KAH基因。 |
通过在线软件WoLF PSORT(http://wolfpsort.org/)对7个KAH基因的亚细胞定位分析,表格中数字越大代表基因定位到此细胞结构的可能性越大。结果(表 2)显示KAH3、KAH5、KAH6定位在叶绿体中;KAH2和KAH4定位在细胞质中;KAH1和KAH7可能定位在叶绿体也有可能定位在细胞外。
贝壳杉烯酸脱氢酶(KAH)是甜菊糖苷和赤霉素生物合成途径上的关键分支节点酶,不同KAH的活性决定下游系列产物的合成和积累[8, 9]。自然进化与人工选育法获得高糖苷积累甜叶菊品种的过程会造成基因信息的改变。因此本研究选择不同甜叶菊品种分别克隆KAH编码基因,然后通过研究分析这些序列结构特征、表达模式等预测其功能,试图筛选具有高效底物转化活性KAH的编码基因并进一步揭示糖苷的积累差异的分子机制。通过对获得不同SrKAHs核酸序列比对分析发现,这些序列高度同源,差异仅为不连续的个别碱基,说明这些品种虽然选育时间差异较大但或许来源同一原始种系。其中总糖苷含量最高的SR2叶片中扩增到3个高度同源的KAH编码基因,总糖苷含量相对较低的守田3号中扩增到两个高度同源的KAH编码基因,总糖苷含量最低的谱星1号中仅存在一个KAH的编码基因。经过氨基酸序列分析预测发现,这些核酸序列虽然高度同源但由于突变碱基的位置造成了ORF的位置和大小的改变。SR2的3个SrKAHs和守田3号的其中一个SrKAH均只有1个ORF编码476个氨基酸序列,守田3号的另1个SrKAH基因存在两个ORF分别编码236和208个氨基酸。已有文献报道收获期的江甜3号总糖苷含量略高于守田3号,谱星1号总糖苷含量显著低于这两个品种[15],定量PCR检测3个品种甜叶菊中SrKAHs的表达水平发现总糖苷含量较高的江甜3号中SrKAHs表达也最高,总糖苷含量最低的谱星1号中其表达水平最低;但总糖苷含量与江甜3号无显著差异的守田3号中SrKAHs的表达也显著低于江甜3号。另外,甜菊糖苷主要积累在甜叶菊叶片中且苗期越靠近植株顶端的叶片中总糖苷积累越多[4],不同组织中SrKAHs的表达结果显示其在叶片组织中的表达也显著高于茎和根系;但SrKAHs在中叶中的表达显著高于底叶。综上所述,不同甜叶菊品种中KAH编码基因的数目和其编码蛋白大小存在显著差异;SrKAHs的表达水平与基因数目不存在正相关关系,但不同甜叶菊品种和同一植株不同组织之间,其表达水平可用于指示总糖苷的积累水平。因此我们推断甜叶菊中存在多个KAH编码基因且其编码蛋白的大小导致其生物功能的差异,这可能是造成甜叶菊中总糖苷积累差异的原因之一。
基因结构决定基因的功能,对不同SrKAHs基因保守结构域的分析结果表明7个SrKAHs均含有细胞色素P450的结构域,与之前克隆成功的具有底物转化活性的 SrKA13H 同属于P450家族[14],且有研究报道P450家族中存在参与萜类合成的亚家族基因[16]。细胞色素P450家族庞大,基因众多,参与植物体多种生物途径包括激素、色素等物质的生物合成过程[17, 18]。为进一步明确这些基因的具体功能,将这7个KAH编码基因与研究较为透彻的拟南芥P450家族进行进化分析,发现这7个SrKAHs均与其CYP716家族进化关系最近。分别与CYP716家族的两个基因进行同源性分析并根据P450家族的分类命名规则[19, 20]可知,KAH2和KAH4与CYP716A家族基因同源性高于55%,因此同属于CYP716A亚家族;KAH1、KAH3、KAH5、KAH6和KAH7均高于40%低于55%,因此属于CYP716的新的亚家族。已有的研究报道中还未有关于CYP716家族基因的功能描述,但此家族基因目前仅在拟南芥中克隆发现[21],说明此基因家族不是赤霉素生物合成的专有家族,那么很可能此家族基因在甜叶菊中参与甜菊糖苷的生物合成。最早研究报道KAH的是从甜叶菊叶绿体中纯化得到的并验证了其转化活性[7],但后续研究者根据其上游的催化酶KO的定位对之前结论提出质疑且认为KAH应定位在内质网或者细胞质中起催化作用[8];而7个不同KAHase编码基因的亚细胞定位分析结果显示KAH2和KAH4定位在细胞质中,其他基因均定位在叶绿体中。由此推测叶绿体和细胞质中均可产生甜菊醇,且由不同的SrKAHs编码不同的KAHase进行底物转化。
新的KAH编码基因的发现和功能分析预测进一步完善了甜菊糖苷生物合成机制的研究,这些基因均具备KAH编码基因的保守结构域。但其实际底物转化活性还需进一步将这些候选基因转化入大肠杆菌、酵母等异源体系或者直接转化入甜叶菊內源体系,通过检测其对甜菊醇转化为甜菊糖苷的活性等方法验证。但这些新基因的克隆为获得更高底物转化活性基因的获得和后续实现异源合成甜菊糖苷提供了候选基因。
4 结论通过对不同甜叶菊材料中KAH编码序列的克隆、序列及表达模式分析可知,既存在多个甜叶菊材料拥有一个相同的KAH编码序列的情况,也存在一个甜叶菊材料拥有多个KAH编码序列的情况,且这些编码序列编码氨基酸的长度差异可以导致其功能上的差异。SrKAHs的表达与编码基因的数目和序列差异不存在相关性但可以用来指示甜叶菊糖苷积累水平。对这些存在差异的氨基酸序列分析可知虽同属于P450家族但分属于不同亚家族且在亚细胞结构中定位不同。综上所述,在不同甜叶菊材料中KAH编码序列的数目、长度和亚细胞定位等差异或许是造成基因表达和功能差异继而导致总糖苷积累差异的原因之一。
| [1] | Ceunen S, Geuns JMC. Steviol glycosides:chemical diversity, metabolism, and function. J Nat Prod, 2013, 76 (6): 1201–1228. DOI:10.1021/np400203b |
| [2] | Shafii B, Vismeh R, Beaudry R, et al. Large-scale profiling of diterpenoid glycosides from Stevia rebaudiana using ultrahigh performance liquid chromatography/tandem mass spectrometry. Anal Bioanal Chemi, 2012, 403 (9): 2683–2690. DOI:10.1007/s00216-012-6071-y |
| [3] | Yadav SK, Guleria P. Steviol glycosides from Stevia:biosynthesis pathway review and their application in foods and medicine. Crit Rev Food Sci Nutr, 2012, 52 (11): 988–998. DOI:10.1080/10408398.2010.519447 |
| [4] | Bondarev NI, Sukhanova MA, Reshetnyak OV, et al. Steviol glycoside content in different organs of Stevia rebaudiana and its dynamics during ontogeny. Biologia Plantarum, 2003, 47 (2): 261–264. |
| [5] | Guleria P, Yadav SK. Agrobacterium mediated transient gene silencing (AMTS) in Stevia rebaudiana:insights into steviol glycoside biosynthesis pathway. PLoS One, 2013, 8 (9): e74731. DOI:10.1371/journal.pone.0074731 |
| [6] | Wanke M, Skorupinska-Tudek K, Swiezewska E. Isoprenoid biosynthesis via 1-deoxy-D-xylulose 5-phosphate/2-C-methylD-erythritol 4-phosphate (DOXP/MEP) pathway. Acta Biochimica Polonica-English Edition, 2001, 48 (3): 663–672. |
| [7] | Kim KK, Sawa Y, Shibata H. Hydroxylation ofent-Kaurenoic Acid to Steviol in Stevia rebaudiana Bertoni—Purification and Partial Characterization of the Enzyme. Archives of Biochemistry and Biophysics, 1996, 332 (2): 223–230. DOI:10.1006/abbi.1996.0336 |
| [8] | Brandle JE, Telmer PG. Steviol glycoside biosynthesis. Phytochemistry, 2007, 68 (14): 1855–1863. DOI:10.1016/j.phytochem.2007.02.010 |
| [9] | Brandle JE, Richman A, Swanson AK, et al. Leaf ESTs from Stevia rebaudiana:a resource for gene discovery in diterpene synthesis. Plant Mol Biol, 2002, 50 (4-5): 613–622. |
| [10] | Richman A, Swanson A, Humphrey T, et al. Functional genomics uncovers three glucosyltransferases involved in the synthesis of the major sweet glucosides of Stevia rebaudiana. The Plant Journal, 2005, 41 (1): 56–67. |
| [11] | Kishore GM, Motion M, Hicks PM, et al. Production of steviol glycosides in microorganisms:U. S, 13/701, 406[P]. 2011-6-2. |
| [12] | Humphrey TV, Richman AS, Menassa R, et al. Spatial organisation of four enzymes from Stevia rebaudiana that are involved in steviol glycoside synthesis. Plant Mol Biol, 2006, 61 (1-2): 47–62. DOI:10.1007/s11103-005-5966-9 |
| [13] | Wang J, Li S, Xiong Z, et al. Pathway mining-based integration of critical enzyme parts for de novo biosynthesis of steviolglycosides sweetener in Escherichia coli. Cell Res, 2015, 26 : 258–261. |
| [14] | 郭书巧, 束红梅, 郑卿, 等. 甜菊醇糖苷生物合成途径关键酶基因KA13H的克隆及序列分析. 基因组学与应用生物学, 2010, 29(5): 911–918. |
| [15] | 胡秀英, 王仲伟, 黄苏珍. 甜菊糖苷含量跃变的理化特性分析. 中国糖料, 2016, 38(4): 19–23. |
| [16] | Collu G, Unver N, Peltenburg-Looman AMG, et al. Geraniol 10-hydroxylase1, a cytochrome P450 enzyme involved in terpenoid indole alkaloid biosynthesis. FEBS Lett, 2001, 2 : 215–220. |
| [17] | Mizutani M, Ohta D. Diversification of P450 genes during land plant evolution. Ann Rev Plant Biol, 2010, 61 : 291–315. DOI:10.1146/annurev-arplant-042809-112305 |
| [18] | Durst F, Ο'Keefe DP. Plant cytochromes P450:an overview. Drug Metabolism and Drug Interactions, 1995, 3-4 : 171–188. |
| [19] | 余小林, 曹家树, 崔辉梅, 等. 植物细胞色素P450. 中国细胞生物学学报, 2004, 26: 561–566. DOI:10.3969/j.issn.1674-7666.2004.06.004 |
| [20] | 贺丽虹, 赵淑娟, 胡之璧. 植物色素P450基因与功能研究进展. 药物生物技术, 2008, 15(2): 142–147. |
| [21] | Fukushima EO, Seki H, Ohyama K, et al. CYP716A subfamily members are multifunctional oxidases in triterpenoid biosynthesis. Plant and Cell Physiology, 2011, 52 (12): 2050–2061. DOI:10.1093/pcp/pcr146 |