2. 国家食用菌加工技术研发分中心,武汉 430064
2. National R & D Center for Edible Fungi Processing, Wuhan 430064
真菌是真核生物中一支庞大而种类繁多的物种,目前已知的约10多万种[1]。自从1996年酿酒酵母(Saccharomyces cerevisiae)基因组被测定以来[2],越来越多的真菌基因组被解析和公布。截至目前,测序完成且公开的真菌基因组达800多个(NCBI Genome数据库)。测序获得了大量功能未知的真菌DNA序列,鉴定这些基因的功能,解析这些序列的生物学特性己成为后基因组时代的研究热点。目前,人们开发了多种基因功能研究方法,如转座子标签法[3]、T-DNA插入法[4]、RNA干扰[5]、超表达[6]和microRNA[7]等技术。这些技术虽然都能对相关基因的表达进行调控,但是不能对目标基因进行定点修饰,因而还不能称为基因编辑技术[8]。最初,人们主要采用同源重组介导的基因打靶技术对模式生物的基因组进行编辑,但因筛选标记物有限且效率不高,严重限制了该技术的应用[9]。近年来,人们发明了锌指核酸酶(zinc finger nucleases,ZFN)[10]、类转录激活因子效应物核酸酶(transcri-ption activator-like effector nucleases,TALEN)[11]及CRISPR/Cas(clustered regularly interspaced short palindromic repeat/CRISPR-associated proteins)[12]基因编辑技术。目前,CRISPR/Cas9系统以其设计简单、操作方便、精确、效率高等优点广泛应用于动物、植物和微生物等基因组的定点编辑,正在给功能基因组学带来一场革命性的改变。本文综述了CRISPR/Cas系统的结构、作用机理、分类情况及其在酿酒酵母和其他丝状真菌中的应用,并对该技术可能出现的问题及应用前景进行了展望,以期为真菌基因编辑研究提供参考。
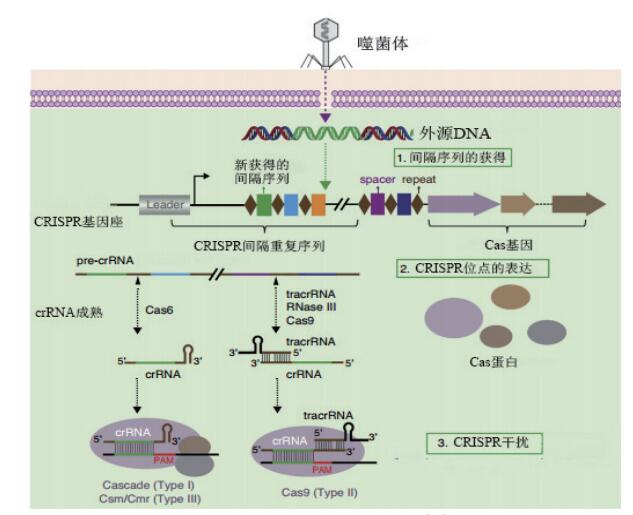
1 CRISPR/Cas系统的结构一个典型的CRISPR/Cas基因座由一个编码Cas蛋白的操纵子和一个CRISPR重复间隔序列组成,大部分生物体含有1-2个CRISPR/Cas基因座。典型的CRISPR重复间隔序列由一系列短的高度保守的正向重复序列(repeats)和长度相似的间隔序列(spacers)按一定顺序排列组成[13](图 1)。其中,重复序列是一类长度约21-48 bp的具有回文结构或者发夹样二级结构的序列,按相似性不同被分为12个簇。间隔序列来源于外源基因片段,长度一般为26-72 bp,间隔序列与重复序列依次串联排列,可以拥有2-375个重复[14]。CRISPR重复间隔序列5' 端连接着一段富含A/T碱基并且包含启动子的前导序列(leader sequence),长度约20-534 bp[15]。Cas基因是一类保守的基因家族,主要编码核酸酶、DNA解旋酶、聚合酶等与切割、修饰核酸相关的蛋白。一个CRISPR/Cas基因座通常包含4-20个不同的Cas基因,这些基因可以位于CRISPR重复间隔序列的上游或者下游[15, 16]。
2 CRISPR/Cas系统的作用原理CRISPR/Cas系统作用机理可以大致分为获得、表达和干扰3个步骤[17](图 2)。(1)CRISPR间隔序列的获得(spacer acquisition):当外源噬菌体或质粒等的遗传物质进入细菌细胞后,Cas1和Cas2蛋白及其复合体对外源DNA序列中的PAM(protosp-acer adjacent motif)序列进行识别。然后特定的Cas蛋白将PAM序列旁的原型间隔序列(proto-spacer)加工成为间隔序列并整合插入到前导序列与间隔重复序列之间[18]。因此,CRISPR基因座中的间隔序列从5' -3' 的排列也记录了外源遗传物质入侵的时间顺序。此外,PAM序列长2-5 bp,一般与proto-spacer相隔1-4 bp,被认为是CRISPR系统对自身序列和外源序列正确识别的关键机制[19]。(2)CRISPR位点的表达:CRISPR重复间隔序列被转录为pre-crRNA后,特异的核酸内切酶对pre-crRNA进行切割,形成小的crRNAs(由一个间隔序列与部分重复序列组成),也被称为guide RNAs[18]。研究发现,在没有受到外界压力时,CRISPR基因座表达水平较低;当外源的质粒或噬菌体入侵宿主菌时,CRISPR的表达很快被诱导上调[20]。(3)CRISPR/Cas系统对外源遗传物质的识别与降解,即免疫干扰:当宿主细胞再次受到相同的外源DNA入侵时,crRNAs和Cas蛋白等形成的复合物体会在靶序列上寻找PAM序列,随后crRNAs通过碱基配对识别靶序列上相应的互补序列,外源DNA在配对的特定位置被具有核酸酶活性的Cas蛋白切割[21]。DNA损伤会触发细胞自身的DNA修复机制,同源定向修复(homology-directed repair,HDR)以外源供体DNA(donor DNA)为模板,修复后会导致目标基因的失活或外源片段的插入[22];非同源末端连接(non-homologous end joining,NHEJ),无需修复模板,可能出错)修复会在双链断裂(double-stranded break,DSB)位置随机插入或删除部分碱基对,导致目标基因移码突变或关键区域被破坏[23]。
3 CRISPR/Cas系统的分类根据所含特征基因(signature genes)及作用机制的不同,CRISPR/Cas系统可分为Typle Ⅰ、Typle Ⅱ和Typle Ⅲ 3个大的类型,每一类型又分为许多亚型[24](图 3)。随着研究的深入,人们还发现了其他类型的CRISPR/Cas系统,但是其具体作用机制尚不清楚[24, 25]。Ⅰ型CRISPR/Cas系统的特征基因是Cas3基因,该基因编码的蛋白质具有核酸酶和DNA解旋酶活性[26]。当CRISPR重复间隔序列被转录为pre-crRNA后,具有核酸内切酶活性的Cas6蛋白在间隔序列上游8 bp位置对重复序列进行切割,形成两头是部分重复序列、中间是间隔序列的crRNAs,随后crRNAs和其他Cas蛋白形成防御复合体Cascade(CRISPR-associated complex for antiviral defense)。Cascade中crRNAs识别外源DNA后,召集Cas3蛋白对靶序列进行切割降解[27]。Ⅱ型CRISPR/Cas系统的特征基因是Cas9基因,编码的蛋白具有核酸内切酶活性并且受RNA的引导[28]。在表达阶段,CRISPR/Cas基因座上游的转录活化RNA(Trans-activating crRNA,tracrRNA)与Cas9结合形成复合体,该复合体通过tracrRNA与pre-crRNA中重复序列互补配对紧密结合,随后RNase Ⅲ将含有crRNA的复合体切下。由tracrRNA、crRNA和Cas9蛋白组成的复合体识别外源DNA并对其切割降解[28, 29]。Ⅲ型CRISPR/Cas系统比较复杂,该系统由特征基因Cas10以及重复相关可疑蛋白(repeat-associated mysterious protein,RAMP)Csm/Cmr组成[30]。与Ⅰ型CRISPR/Cas系统类似,pre-crRNA首先被Cas6蛋白切割成小的crRNAs,随后Csm/Cmr-Cas10蛋白复合体与crRNAs结合并切除crRNAs 3’末端的重复序列形成成熟crRNAs。随后该复合体对外源DNA及其转录的RNA进行识别切割[31]。
4 CRISPR/Cas9基因编辑系统在真菌中的应用作为一种功能强大的基因组编辑工具,CRISPR/Cas9基因编辑系统已成功应用于人类细胞、线虫、斑马鱼、果蝇、水稻、拟南芥等模式生物[32]。2013年,DiCarlo等[33]首次将CRISPR/Cas9基因编辑系统应用于真菌酿酒酵母(Saccharomyces cerevisiae),成功敲除了细胞膜精氨酸透性酶CAN1基因,目前该系统已广泛应用于多种真菌基因功能研究。
4.1 在酵母菌中的应用酿酒酵母是代谢工程研究最受关注的模式种,也是功能基因组学、蛋白质相互作用、细胞周期、细胞衰老凋亡等研究的优选模式生物[34]。近年来,CRISPR/Cas9基因编辑技术的诞生为酵母基因功能、代谢调控等研究提供了简单、快速和高效的方法。
DiCarlo等[33]首先将CRISPR/Cas9基因编辑系统引入酿酒酵母,随后人们针对酵母菌自身特点,在载体构建、筛选程序等方面进行了大量改进,如Laughery等[35]利用Bcl Ⅰ和Swa Ⅰ限制性内切酶特性,使得20 bp crRNA插入sgRNA表达盒变得简单高效;Zhang等[36]首先将Cas9基因导入酵母菌基因组获得Cas9蛋白表达工程菌,然后构建含有sgRNA载体和供体DNA转化上述工程菌,降低了载体构建的难度;Jakočiūnas等[37]在此基础上利用USER克隆技术,将多个sgRNA串联,一次性敲除了多个基因,大大提高了敲除效率;Tsarmpopoulos等[38]利用CRISPR/Cas9系统进行基因敲除时,设计了90 bp的供体DNA,该DNA与靶标基因上下游同源,转化后直接利用PCR筛选转化子,简化了筛选程序;Bao等[39]将供体DNA(100 bp)与20 bp crRNA嵌合连接片段、tracrRNA与Cas9基因分别进行表达,有效避免了脱靶的问题;2016年,Smith等[40]利用dCas9蛋白特性,设计了一种诱导型单链CRISPR干涉(CRISPRi)系统,大大扩展了CRISPR系统在酵母中的应用。
除了基因敲除,利用供体DNA与目标基因的同源性,CRISPR/Cas9系统还能介导基因插入。Ryan等[41]将Sp Cas9基因和自我裂解型肝炎病毒(HDV)核酸酶与sgRNA嵌合片段克隆至pCAS质粒骨架中,构建了多重CRISPR系统(Multiplex CRISPR,CRISPRm),利用CRISPRm系统将粗糙脉孢菌(Neurospora crassa)纤维糊精转运子(cdt-1)和胞内β-葡糖苷酶基因(gh1-1)分别插入酵母菌ura3和lyp1基因位点,通过CRISPRm系统对cdt-1和gh1-1基因进行定点突变,使得酵母菌利用纤维二糖的能力提高了10倍。Ronda等[42]将含有3个sgRNA串联的载体和3个分别含有β-胡萝卜素合成基因BTS1、crtYB、crtI的供体DNA一起转化已经获得Cas9基因插入的酵母工程菌,使得酵母工程菌具有合成β-胡萝卜素能力。类似的,Tsai等[43]利用CRISPR/Cas9基因编辑系统将毕赤酵母(Scheffersomyces stipitis)木糖还原酶(XYL1)、木糖醇脱氢酶(XYL2)和木酮糖激酶(XYL3)基因分别插入酵母PHO13和ALD6基因位点,获得了具有分解木糖能力的酵母工程菌;Mans等[44]利用CRISPR/Cas9基因编辑系统构建了具有粪肠球菌(Enterococcus faecalis)丙酮酸脱氢酶复合体合成能力的酵母工程菌。此外,Horwitz等[45]和Shi等[46]分别利用CRISPR/Cas9基因编辑系统构建酵母工程菌时均实现了大片段的插入,插入的片段达到24 kb。更令人惊奇的是,EauClaire及其团队[47]利用CRISPR/Cas9基因编辑系统在酵母菌细胞内构建了一条β-胡萝卜素生物合成途径,该生物合成途径包含的关键基因多达17个。除了模式种酿酒酵母,目前CRISPR/Cas9基因编辑系统也应用到其他酵母菌,如布拉酵母(Saccharomyces boulardii)[48]、裂殖酵母(Schizosaccharomyces pombe)[49]、致病菌白色假丝酵母(Candida albicans)[50]等的研究。
4.2 在丝状真菌中的应用丝状真菌广泛分布于自然界,与人类的生产、生活密切相关。目前,许多丝状真菌基因组已经测序完毕,对丝状真菌的研究已步入后基因组时代。2015年,Liu及其团队[51]首先将CRISPR/Cas9基因编辑系统应用于里氏木霉(Trichoderma reesei)的研究。该团队首先构建了优化密码子的Cas9蛋白表达载体,利用根癌农杆菌(Agrobacterium tumefaciens)AGL-1介导转化里氏木霉菌丝体,形成里氏木霉Cas9表达平台(Cas9-expressing chassis)。然后构建sgRNA载体,对乳酸核糖转移酶ura5基因进行了敲除,敲除率几乎达到100%。但是同时对甲基转移酶lae1基因、葡萄糖信号相关基因vib1和转录调控因子clr2进行敲除时,敲除率只有4.2%。此外,该团队还构建了具有同源臂的供体DNA,将斜卧青霉菌(Penicillium decumbens)乳酸核糖转移酶poura5基因插入到自身甲基转移酶lae1基因位点,并且发现同源臂≥600 bp,重组率几乎达到100%;同源臂长200 bp时重组率也可以达到93%。
随后,Matsu-ura等[52]在研究粗糙脉孢菌(Neurospora crassa)时,构建了以构巢曲霉(Aspergillus nidulans)tripC启动子驱动Cas9蛋白基因、酿酒酵母SNR52启动子驱动sgRNA的CRISPR/Cas9系统,成功将β-tubulin启动子与转录因子clr-2融合片段、糖原合酶1启动子与荧光素酶基因融合片段分别插入粗糙脉孢菌clr-2和clr-1基因位点,结果表明强启动子的插入使得clr-2基因表达量提高了200倍;而荧光素酶基因的插入使得粗糙脉孢菌转化子的筛选更简单。与此同时Nødvig团队[53]也将其利用CRISPR/Cas9系统对丝状真菌进行基因编辑的研究结果发表在PLoS One杂志上。该团队采用单质粒构建策略将sgRNA和Cas9蛋白基因置于一个表达质粒上,其中sgRNA由构巢曲霉gpdA启动子驱动,Cas9基因由构巢曲霉tef1启动子驱动。利用该载体成功敲除了构巢曲霉yA基因、棘孢曲霉(Aspergillus aculeatus)albA和pyrG基因、黑曲霉(Aspergillus niger)、炭黑曲霉(Aspergillus carbonarius)、泡盛曲霉(Aspergillus luchuensis)及巴西曲霉(Aspergillus brasiliensis)albA基因。该研究结果表明启动子对sgRNA和Cas9蛋白表达影响较大,为提高基因编辑效率最好选用同源强启动子。
类似的,Katayama等[54]以pUNA质粒为骨架,采用单质粒构建策略成功构建了CRISPR/Cas9基因编辑系统,其中amyB启动子与Cas9基因连接,U6启动子与sgRNA连接。利用该系统成功敲除了米曲霉(Aspergillus oryzae)wA,yA,和pryG基因,但是敲除率不高,只有10%-20%。Fuller等[55]和Zhang等[56]两个团队分别建立了烟曲霉(Aspergillus fumigatus)CRISPR/Cas9基因编辑系统,前者对聚酮合酶基因pksP进行了敲除,但是敲除效率只有25%-53%;后者对pksP基因及钙调磷酸酶催化亚基cnaA分别进行了敲除,敲除效率为63%-97%,远远高于Fuller等研究结果。究其原因可能是载体构建时选择的启动子以及选择的策略不同,Zhang等[56]研究结果表明双质粒系统比单质粒系统编辑效率要高,另外使用gpdA或niiA等强启动子驱动Cas9的表达也能有效提高敲除效率。Arazoe等[57]采用单质粒策略构建稻瘟病菌(Pyricularia oryzae)CRISPR/Cas9基因编辑系统,成功敲除了小柱孢酮脱水酶SDH基因。研究发现采用U6启动子,敲除效率为36.1-83.6%;采用tripC启动子时,敲除效率为9.8%-27.1%,这表明驱动sgRNA表达的启动子不同,敲除效率也不同,在构建CRISPR/Cas9基因编辑系统时要选择合适的启动子。
Schuster等[58]在另外一种病原真菌玉米黑粉菌(Ustilago maydis)中采用单质粒策略构建了CRISPR/Cas9基因编辑系统,利用该系统成功敲除了成丝和致病相关基因bE1和bW2,敲除效率高达70%-100%。为检验CRISPR/Cas9基因编辑系统的脱靶现象,对两个敲除株进行了基因组重测序,结果发现两个敲除株基因组中分别有84和60处特定变异,突变率是人类细胞的4倍,Schuster等[58]认为脱靶率较高的原因可能是原生质体制备时采用了混合的破壁酶。此外,2016年4月,Science杂志刊文介绍了宾夕法尼亚州立大学Yang Yinong团队利用CRISPR/Cas9基因编辑系统敲除了双孢蘑菇(Agar-icus bisporus)基因组中6个多酚氧化酶(PPO)基因中的一个,使得PPO酶活降低30%,有效减缓了双孢蘑菇的褐变现象[59]。虽然该文没有公开其具体操作过程,但是这是第一个利用CRISPR/Cas9系统对大型真菌基因组进行编辑的报道。
5 展望随着大规模基因组测序工作的开展,越来越多的真菌基因组信息被披露,阐明基因功能、解析代谢途径必将成为真菌后基因组时代的重大课题。简单、高效、操作性强的基因功能研究方法无疑会给真菌基因功能组学研究带来一场巨变。
相比于ZFNs和TALENs系统,CRISPR/Cas9系统有着无可比拟的优势:首先,CRISPR/Cas9系统应用广泛:该系统选择靶位点的要求是具有PAM序列,这一序列在基因组中广泛存在,在任意基因中都能找到数个靶位点,因此几乎可以对所有基因进行编辑[14];其次,CRISPR/Cas9系统组成简单,载体组装方便:sgRNA和Cas9蛋白是该系统的两大组件,根据需要可将二者置于一个转化载体上,也可将二者置于不同的转化载体上,具有分子生物学背景的人员可快速完成[18];第三,CRISPR/Cas9系统可以同时对多个基因进行编辑;可以将针对多个靶点的sgRNA与Cas9克隆于同一个转化载体中,一次转化就可能获得多个位点突变的突变体,大大提高了基因编辑效率[28]。
当然,CRISPR/Cas9系统也存在一定局限性。(1)CRISPR/Cas9技术的靶向编辑的效率有待提高:不同来源的Cas9基因,在不同细胞内表达水平和活性是不同的,造成突变效率也不同。研究表明,Cas9基因表达水平与启动子强度、转化的材料及转化方法有关[53, 57]。在真菌中,人们大多采用真菌偏好的密码子对Cas9酶进行优化,并用gpdA、β-tubulin、tripC、amyB和U6等强启动子驱动Cas9基因的表达,而在转化材料上大多采用原生质体,以此来提高CRISPR/Cas9系统的编辑效率。(2)存在一定的脱靶效应:CRISPR/Cas9基因编辑系统的特异性取决于sgRNA上的识别序列。然而,在复杂的生物基因组中,sgRNA的识别序列可能会与非靶点DNA发生局部匹配[60]。为降低CRISPR/Cas9系统的脱靶效应,研究者开发了许多专业软件用于寻找CRISPR/Cas9靶位点,如Cas-OFFinder、CRISPR Design等[61]。这些在线软件可以辅助科研人员快速筛选特定基因序列所包含的所有CRISPR/Cas9靶点,有针对性的避开可能脱靶的位点、突变效率较高的位点,从而降低甚至消除脱靶现象。
2016年,河北科技大学的韩春雨研究小组以CRISPR/Cas9系统为蓝本,设计了NgAgo-gDNA基因编辑系统,该系统包含格氏嗜盐碱杆菌(Natronobacterium gregoryi)Argonaute蛋白和一段长24 bp的5’磷酸化的单链引导DNA(gDNA)。在人类细胞中,其切割效率与CRISPR/Cas9系统相当,但是该系统gDNA容错度更低,1个碱基错配会影响Argonaute蛋白的切割效率,3个碱基的错配将会终止Argonaute蛋白的切割[62]。该系统可有效解决脱靶效应,为基因编辑技术的推广和应用奠定了基础。
目前,CRISPR/Cas9基因编辑系统仅在模式种酿酒酵母和少量真菌中得到应用。随着CRISPR/Cas9技术的改进和完善,以及NgAgo-gDNA基因编辑系统的出现,可以预见真菌基因组的编辑将会越来越简单,这将为真菌功能基因挖掘、品种定向改良、发育相关基因定点突变以及创制新的种质资源带来突破性进展。
| [1] | Tani S, Kawaguchi T, Kobayashi T. Complex regulation of hydrolytic enzyme genes for cellulosic biomass degradation in filamentous fungi. Appl Microbiol Biotechnol, 2014, 98 (11): 4829–4837. DOI:10.1007/s00253-014-5707-6 |
| [2] | Goffeau A, Barrell BG, Bussey H, et al. Life with 6000 genes. Science, 1996, 274 (5287): 546, 563–567. |
| [3] | Villalba F, Lebrun MH, Hua-Van A, et al. Transposon impala, a novel tool for gene tagging in the rice blast fungus Magnaporthe grisea. Mol Plant Microbe Interact, 2001, 14 (3): 308–315. DOI:10.1094/MPMI.2001.14.3.308 |
| [4] | Krysan PJ, Young JC, Sussman MR. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell, 1999, 12 : 2283–2290. |
| [5] | Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391 (6669): 806–811. DOI:10.1038/35888 |
| [6] | Yin C, Zheng L, Zhu J, et al. Enhancing stress tolerance by overexpression of a methionine sulfoxide reductase A (MsrA) gene in Pleurotus ostreatus. Applied Microbiology and Biotechnology, 2015, 99 (7): 3115–3126. DOI:10.1007/s00253-014-6365-4 |
| [7] | Jones-Rhoades MW, Bartel DP, Bartel B. MicroRNAs and their regulatory roles in plants. Annual Review of Plant Biology, 2006, 57 : 19–53. DOI:10.1146/annurev.arplant.57.032905.105218 |
| [8] | 曾秀英, 侯学文. CRISPR/Cas9基因组编辑技术在植物基因功能研究及植物改良中的应用. 植物生理学报, 2015, 51(9): 1351–1358. |
| [9] | Shalem O, Sanjana NE, Zhang F. High-throughput functional genomics using CRISPR/Cas9. Nature Reviews Genetics, 2015, 16 (5): 299–311. DOI:10.1038/nrg3899 |
| [10] | Urnov FD, Rebar EJ, Holmes MC, et al. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics, 2010, 11 (9): 636–646. DOI:10.1038/nrg2842 |
| [11] | Bedell VM, Wang Y, Campbell JM, et al. In vivo genome editing using a high-efficiency TALEN system. Nature, 2012, 491 (7422): 114–118. DOI:10.1038/nature11537 |
| [12] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 6121 : 823–826. |
| [13] | Amitai G, Sorek R. CRISPR-Cas adaptation:insights into the mechanism of action. Nat Rev Microbiol, 2016, 2 : 67–76. |
| [14] | Barrangou R, Dudley EG. CRISPR-based typing and next-generation tracking technologies. Annual Review of Food Science and Technology, 2016, 7 : 395–411. DOI:10.1146/annurev-food-022814-015729 |
| [15] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology, 2010, 64 : 475–493. DOI:10.1146/annurev.micro.112408.134123 |
| [16] | Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR/Cas9. Science, 2014, 346 (6213): 1258096. DOI:10.1126/science.1258096 |
| [17] | Jiang F, Doudna JA. The structural biology of CRISPR-Cas systems. Curr Opin Struct Biol, 2015, 30 : 100–111. DOI:10.1016/j.sbi.2015.02.002 |
| [18] | Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation. Annu Rev Genet, 2011, 45 : 273–297. DOI:10.1146/annurev-genet-110410-132430 |
| [19] | Plagens A, Richter H, Charpentier E, et al. DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes. FEMS Microbiol Rev, 2015, 3 : 442–463. |
| [20] | Agari Y, Sakamoto K, Tamakoshi M, et al. Transcription profile of Thermus thermophilus CRISPR systems after phage infection. Journal of Molecular Biology, 2010, 395 (2): 270–281. DOI:10.1016/j.jmb.2009.10.057 |
| [21] | Tsui TK, Li H. Structure principles of CRISPR-Cas surveillance and effector complexes. Ann Rev Biophy, 2015, 44 : 229–255. DOI:10.1146/annurev-biophys-060414-033939 |
| [22] | Wang H, La Russa M, Qi LS. CRISPR/Cas9 in genome editing and beyond. Ann Rev Biochem, 2016, 85 : 227–264. DOI:10.1146/annurev-biochem-060815-014607 |
| [23] | Lieber MR. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry, 2010, 79 : 181–211. DOI:10.1146/annurev.biochem.052308.093131 |
| [24] | Wright AV, Nuñez JK1, Doudna JA. Biology and applications of CRISPR systems:harnessing nature' s toolbox for genome engineering. Cell, 2016, 164 (1-2): 29–44. DOI:10.1016/j.cell.2015.12.035 |
| [25] | Sternberg SH, Richter H, Charpentier E, et al. Adaptation in CRISPR-Cas systems. Molecular Cell, 2016, 6 : 797–808. |
| [26] | Gong B, Shin M, Sun J, et al. Molecular insights into DNA interference by CRISPR-associated nuclease-helicase Cas3. Proc Natl Acad Sci USA, 2014, 111 (46): 16359–16364. DOI:10.1073/pnas.1410806111 |
| [27] | Huo Y, Nam KH, Ding F, et al. Structures of CRISPR Cas3 offer mechanistic insights into Cascade-activated DNA unwinding and degradation. Nat Struct Mol Biol, 2014, 21 (9): 771–777. DOI:10.1038/nsmb.2875 |
| [28] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337 (6096): 816–821. DOI:10.1126/science.1225829 |
| [29] | Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Research, 2011, 39 (21): 9275–9282. DOI:10.1093/nar/gkr606 |
| [30] | Hale CR, Zhao P, et al. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex. Cell, 2009, 139 (5): 945–956. DOI:10.1016/j.cell.2009.07.040 |
| [31] | Samai P, Pyenson N, Jiang W, et al. Co-transcriptional DNA and RNA cleavage during type Ⅲ CRISPR-Cas immunity. Cell, 2015, 161 (5): 1164–1174. DOI:10.1016/j.cell.2015.04.027 |
| [32] | Dominguez AA, Lim WA, Qi LS. Beyond editing:repurposing CRISPR/Cas9 for precision genome regulation and interrogation. Nature Reviews Molecular Cell Biology, 2016, 17 (1): 5–15. |
| [33] | DiCarlo JE, Norville JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucl-eic Acids Research, 2013, 41 (7): 4336–4343. DOI:10.1093/nar/gkt135 |
| [34] | Jakočiūnas T, Jensen MK, Keasling JD. CRISPR/Cas9 advances engineering of microbial cell factories. Metabolic Engineering, 2016, 34 : 44–59. DOI:10.1016/j.ymben.2015.12.003 |
| [35] | Laughery MF, Hunter T, Brown A, et al. New vectors for simple and streamlined CRISPR/Cas9 genome editing in Saccharomyces cerevisiae. Yeast, 2015, 32 (12): 711–720. DOI:10.1002/yea.3098 |
| [36] | Zhang GC, Kong II, Kim H, et al. Construction of a quadruple auxotrophic mutant of an industrial polyploid saccharomyces cerevisiae strain by using RNA-guided Cas9 nuclease. Appl Environm Microbiol, 2014, 80 (24): 7694–7701. DOI:10.1128/AEM.02310-14 |
| [37] | Jakočiūnas T, Bonde I, Herrgård M, et al. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metabolic Engineering, 2015, 28 : 213–222. DOI:10.1016/j.ymben.2015.01.008 |
| [38] | Tsarmpopoulos I, Gourgues G, Blanchard A, et al. In-yeast engineering of a bacterial genome using CRISPR/Cas9. ACS Synthetic Biology, 2016, 5 (1): 104–109. DOI:10.1021/acssynbio.5b00196 |
| [39] | Bao Z, Xiao H, Liang J, et al. Homology-integrated CRISPR-Cas (HI-CRISPR) system for one-step multigene disruption in Sacch-aromyces cerevisiae. ACS Synth Biol, 2015, 5 : 585–594. |
| [40] | Smith JD, Suresh S, et al. Quantitative CRISPR interference screens in yeast identify chemical-genetic interactions and new rules for guide RNA design. Genome Biology, 2016, 17 : 45. DOI:10.1186/s13059-016-0900-9 |
| [41] | Ryan OW, et al. Selection of chromosomal DNA libraries using a multiplex CRISPR system. eLife, 2014, 3 : e03703. |
| [42] | Ronda C, Maury J, Jakočiunas T, et al. CrEdit:CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae. Microbial Cell Factories, 2015, 14 : 97. DOI:10.1186/s12934-015-0288-3 |
| [43] | Tsai CS, Kong II, Lesmana A, et al. Rapid and marker-free Cas9/CRISPR refactoring yields equivalent xylose-utilization performa-nce in yeast. Biotechnol Bioeng, 2015, 111 : 2406–2411. |
| [44] | Mans R, van Rossum HM, Wijsman M, et al. CRISPR/Cas9:a molecular Swiss army knife for simultaneous introduction of multiple genetic modifications in Saccharomyces cerevisiae[J]. FEMS Yeast Research, 2015, 15(2). pii:fov004. |
| [45] | Horwitz AA, Walter JM, Schubert MG, et al. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-Cas. Cell Systems, 2015, 1 (1): 88–96. DOI:10.1016/j.cels.2015.02.001 |
| [46] | Shi S, Liang Y, Zhang MM, et al. A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways in Saccharomyces cerevisiae. Metabolic Engineering, 2016, 33 : 19–27. DOI:10.1016/j.ymben.2015.10.011 |
| [47] | EauClaire SF, Zhang J, Rivera CG, et al. Combinatorial metabolic pathway assembly in the yeast genome with RNA-guided Cas9. J Ind Microbiol Biotechnol, 2016, 43 (7): 1001–1015. DOI:10.1007/s10295-016-1776-0 |
| [48] | Liu JJ, et al. Metabolic engineering of probiotic Saccharomyces boulardii. Appl Environ Microbiol, 2016, 8 : 2280–2287. |
| [49] | Jacobs JZ, Ciccaglione KM, Tournier V, et al. Implementation of the CRISPR/Cas9 system in fission yeast. Nature Communications, 2014, 5 : 5344. DOI:10.1038/ncomms6344 |
| [50] | Vyas VK, Barrasa MI, Fink GR. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances, 2015, 1 (3): e1500248. DOI:10.1126/sciadv.1500248 |
| [51] | Liu R, Chen L, Jiang YP, et al. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system. Cell Discovery, 2015, 1 : 15007. DOI:10.1038/celldisc.2015.7 |
| [52] | Matsu-ura T, Baek M, Kwon JG, et al. Efficient gene editing in Neurospora crassa with CRISPR technology. Fungal Biology and Biotechnology, 2015, 2 : 4. DOI:10.1186/s40694-015-0015-1 |
| [53] | Nødvig CS, et al. A CRISPR/Cas9 system for genetic engineering of filamentous fungi. PLoS One, 2015, 10 (7): e0133085. DOI:10.1371/journal.pone.0133085 |
| [54] | Katayama T, Tanaka Y, Okabe T, et al. Development of a genome editing technique using the CRISPR/Cas9 system in the industrial filamentous fungus Aspergillus oryzae. Biotechnology Letters, 2016, 38 (4): 637–642. DOI:10.1007/s10529-015-2015-x |
| [55] | Fuller KK, Chen S, Loros JJ, et al. Development of the CRISPR/Cas9 System for Targeted Gene Disruption in Aspergillus fumigatus. Eukaryot Cell, 2015, 14 (11): 1073–1080. DOI:10.1128/EC.00107-15 |
| [56] | Zhang C, Meng X, Wei X, et al. Highly efficient CRISPR mutagenesis by microhomology-mediated end joining in Aspergillus fumigatus. Fungal Genetics and Biology, 2016, 86 : 47–57. DOI:10.1016/j.fgb.2015.12.007 |
| [57] | Arazoe T, Miyoshi K, Yamato T, et al. Tailor-made CRISPR/Cas system for highly efficient targeted gene replacement in the rice blast fungus. Biotechnol Bioeng, 2015, 12 : 2543–2549. |
| [58] | Schuster M, Schweizer G, Reissmann S, et al. Genome editing in Ustilago maydis using the CRISPR-Cas system. Fungal Genetics and Biology, 2016, 89 : 3–9. DOI:10.1016/j.fgb.2015.09.001 |
| [59] | Waltz E. Gene-edited CRISPR mushroom escapes US regulation. Nature, 2016, 532 (7599): 293. DOI:10.1038/nature.2016.19754 |
| [60] | Quétier F. The CRISPR/Cas9 technology:Closer to the ultimate toolkit for targeted genome editing. Plant Science, 2016, 242 : 65–76. DOI:10.1016/j.plantsci.2015.09.003 |
| [61] | Peng R, Lin G, Li J. Potential pitfalls of CRISPR/Cas9-mediated genome editing. FEBS Journal, 2016, 283 (7): 1218–1231. DOI:10.1111/febs.2016.283.issue-7 |
| [62] | Gao F, Shen XZ, Jiang F, et al. DNA-guided genome editing using the Natronobacterium gregoryi Argonaute[J]. Nature Biotechnology, 2016, doi:10.1038/nbt.3547. |