2. 中国食品发酵工业研究院,北京 100015;
3. 中国科学院生态环境研究中心,北京 100085;
4. 中粮长城桑干酒庄(怀来)有限公司,张家口 075400
2. China National Research Institute of Food & Fermentation Industries, Beijing 100015;
3. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085;
4. Chateau SunGod GreatWall, Zhangjiakou 075400
葡萄酒酿造过程的本质是从葡萄收集、破碎成汁、葡萄酒一次、二次发酵直到包装、储藏整个工艺中多种微生物参与代谢的过程。新鲜采摘酿酒葡萄上的微生物数量大约为107-108 CFU/g(葡萄鲜重),这些微生物会对后续的葡萄酒发酵过程产生重要影响[1]。Leveau等[2]通过焦磷酸测序方法研究美国加利福尼亚Clarksburg地区的酿酒葡萄发现假单胞杆菌属(Pseudomonas)是霞多丽葡萄主要的细菌类群。Bokulich等[3]通过Illumina Miseq高通量测序方法研究了美国加利福尼亚两个不同地区霞多丽、赤霞珠、仙粉黛酿酒葡萄汁中的微生物群落,发现其中的葡糖杆菌属(Gluconobacter)等在葡萄酒酿造过程中能够促进酯类物质的生成,改善葡萄酒的感官,而乳杆菌属(Lactorbacillus)则会导致葡萄酒产生异味。Martin等[4]通过T-RFLP技术研究了法国Lussac St. Emilion地区不同葡萄园中美乐葡萄上的附生细菌,发现其优势菌群为假单胞杆菌属(Pseudomonas)和鞘脂单胞菌属(Sphingomonas),其中的假单胞杆菌属(Pseudomonas)是葡萄中普遍存在的细菌类群;同时该研究还发现酿酒葡萄园土壤中的微生物和葡萄上的微生物存在联系,因此推测植物不同器官上的附生菌可能有着共同的来源。迄今为止,虽已有少量有关酿酒葡萄微生物的报道,但对其群落结构特点、对后续葡萄酒发酵的影响等仍然知之甚少。因此,探明不同品种酿酒葡萄微生物群落结构,从源头上了解葡萄酒酿造原料的品质,对于保证葡萄酒产业健康发展具有重要意义。
沙城地区多丘陵山地,土壤为褐土,光照充足,昼夜温差大,干燥少雨,是酿酒葡萄种植最为理想的黄金地带。目前,沙城地区种植着几十个品种的酿酒葡萄,几乎涵盖了我国酿酒葡萄的全部品种,是我国优质葡萄酒的主要产区之一。本研究应用Illumina平台的Hiseq 2500高通量测序技术,分析了沙城地区不同品种酿酒葡萄表面的细菌、真菌群落组成、结构以及多样性,旨在为研究不同品种酿酒葡萄微生物对酿酒品质的影响提供理论依据。
1 材料与方法 1.1 材料 1.1.1 样地概况采样样地位于河北省怀来县沙城镇某酿酒葡萄园内,该地区位于北纬40°4' -40°35' ,东经115°16' -115°58' ,属于温带大陆性季风气候,雨热同期,光照充足,日照率68%,年均气温9.1℃,白天太阳辐射强,夜间地面散热快,昼夜温差大,5-9月是农作物生长季节,期间昼夜温差可达11℃-12.4℃。样地属于规模化酿酒葡萄园,葡萄树龄最短的为10年,最长的为37年。
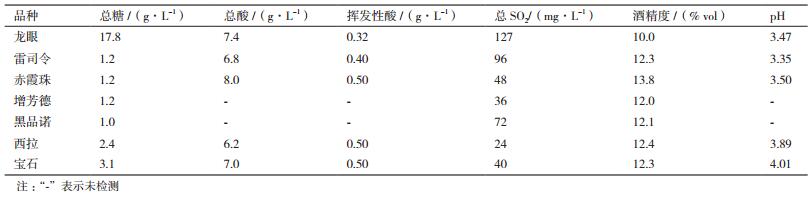
1.1.2 样品采集分别采集霞多丽(Chardonnay,Cha)、雷司令(Riesling,Rie)、黑品诺(Pinot nior,Pin)、宝石(Gem,Gem)、赤霞珠(Cabernet Sauvignon,Cab)、增芳德(Zinfandel,Zin)、龙眼(Longan,Lon)、西拉(Syrah,Syr)、美乐(Merlot,Mer)共9个品种的酿酒葡萄果实。酿酒葡萄样品用经75%酒精消毒后的剪刀采集新鲜、饱满、无病害的果实,置于无菌自封袋中,再将装有葡萄样品的自封袋置于低温采样箱中,带回实验室,-20℃冷冻备用。每个品种酿酒葡萄样品采集均按照“五点取样法”收集5个植株的果实,混合后用于后续实验。部分品种酿酒葡萄发酵制成葡萄酒的理化指标如表 1所示,均采用GB/15038-2006方法测定。其中龙眼是我国特有的葡萄品种,和其他酿酒葡萄品种相比,其含糖量较高,既可作为酿酒葡萄,酿造半甜型葡萄酒,也可作为鲜食葡萄。
每个酿酒葡萄品种果实样品称取约30 g,加入100 mL PBS(0.1 mol/L,pH7.0)缓冲液,180 r/min涡旋震荡30 min,超声15 min,0.22 μm微孔滤膜(Millipore,USA)抽滤,滤膜剪碎放入灭菌离心管中,用DNA提取试剂盒(FastDNA SPIN Kit for Soil,MP)提取总DNA,用核酸蛋白测定仪(Nano Drop,Thermo fisher)检测其浓度和纯度。
1.2.2 PCR扩增和测序采用细菌16S rRNA基因V5-V7区引物799F(5' -AACGGATTAGATACCG-3' )和1193R(5' -ACGTCATCCCCACCTTCC-3' )[5],以及真菌ITS1区引物(5' -CTTGGTCATTTAGAGGAAG-TAA-3' )和(5' -GCTGCGTTCTTCATCGAT GC-3' )[4]进行PCR扩增。50 μL反应体系如下:DNA模板1 μL,正反向引物(10 μmol/L)各2 μL,dNTPs(2.5 μmol/L)4 μL,10×Pyrobest Buffer 5 μL,Pyrobest DNA Polymerase(2.5 U/μL)0.3 μL,ddH2O 35.7 μL。扩增程序如下:94℃预变性5 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min,28个循环;最后72℃延伸10 min。每个样品3个重复,将同一样品3个重复的PCR产物混匀后用1.2%的琼脂糖凝胶电泳进行检测,再用AxyPrep DNA凝胶回收试剂盒(AXYGEN,USA)切胶回收。将回收的PCR产物用Qubit(Life Technology,USA)进行定量化和均一化,用Illumina建库试剂盒进行文库构建,并用Agilent 2100(Agilent,USA)检测文库片段分布,将构建好的合格文库送至北京新科开源基因有限公司进行测序(Hiseq 2500,PE250)。
1.2.3 数据分析 1.2.3.1 数据质控及OTU划分将下机原始数据进行质控,删除含有连续 > 5 bp的Q值 < 15的低质量碱基及含有接头序列的测序数据。然后,根据barcode标签提取每个样本序列数据,用软件Mothur(version 1.36.1)[6]进行数据清洗及操作分类单元(Operational taxonomic unit,OTU)划分,OTU序列相似度设为97%,统计每个样品含有OTU的数目以及每个OTU含有序列的条数,以每个OTU中重复条数最多的序列作为每个OTU的代表序列[7]。
1.2.3.2 丰富度和多样性分析选择在序列97%相似度阈值下绘制稀释曲线(rarefaction curve),稀释曲线反映了样品的取样深度,可以用来评价测序数量是否足以覆盖所有类群。计算丰富度指数Chao 1和ACE,二者反映样品的丰富度,数值越高说明群落物种的丰富度越高。计算多样性指数Shannon和Simpson,二者反映样品的多样性程度,Shannon数值越高说明群落物种的多样性越高,Simpson则相反,数值越高说明群落物种的多样性越低[8-10]。
1.2.3.3 物种信息注释为得到每个OTU对应的物种分类信息,采用RDP算法对97%阈值的OTU代表序列进行物种注释。用数据库Silva(Release115http://www.arb-silva.de)和Unite(https://unite.ut.ee/)分别对细菌和真菌进行比对,在门(Phylum)和属(Genus)的水平上统计每个样品的群落组成,为了得到主要菌群信息,规定细菌门的水平上选取相对丰度大于0.5%的门,属的水平上选取相对丰度大于1%的属;真菌门的水平上菌群分类较少,不设定阈值,属的水平上选取相对丰度大于1%的属。提取序列数占序列总数1%以上的OTU为中心OTU,在NCBI上进行Blast序列比对。其他数据分析及统计均采用软件R 3.0和Origin 9.0。
2 结果 2.1 有效序列及OTU统计对9个酿酒葡萄品种果实表面的细菌16S rRNA基因V5-V7区以及真菌ITS1区进行PCR扩增和测序,过滤掉原始数据中的低和霞多丽的真菌测序量不足,舍弃,其余8个细菌样品共得到154 612条有效序列,在质量序列,去除线粒体DNA、叶绿体DNA以及嵌合体等,其中葡萄品种西拉的细菌测序量97%相似度下划分OTU,共得到1 897个OTU;8个真菌样品共得到714 707条有效序列,在97%相似度下将其聚为不同的OTU,共得到1 313个OTU。
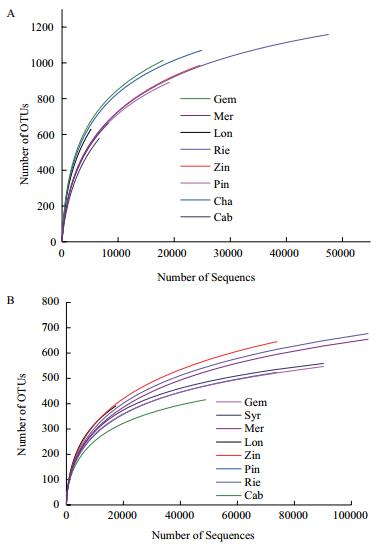
2.2 丰富度和多样性分析如图 1所示,相似度在97%条件下不同品种酿酒葡萄表面细菌16S rRNA基因V5-V7区和真菌ITS1区的稀释曲线,当细菌OTU数量达到600个左右,真菌OTU数量达到300个左右时,曲线开始趋于平缓,说明本研究中样品的取样深度足够,测序数量足以覆盖所有类群。
|
| 图 1 97%相似度条件下的稀释曲线 A:细菌16S rRNA基因V5-V7区稀释曲线;B:真菌ITS1区稀释曲线 |
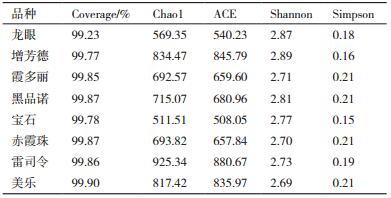
如表 2所示,不同品种酿酒葡萄表面细菌16S rRNA基因V5-V7区样本的测序深度指数coverage均较高(最低的为龙眼94.87%),说明此次测序能够覆盖样本中绝大多数的微生物信息,可以代表样本的真实情况。不同品种酿酒葡萄表面细菌的丰富度指数Chao1和ACE最大的为雷司令,最小的为赤霞珠,说明这8种酿酒葡萄表面雷司令细菌群落最丰富,赤霞珠则最稀少。细菌的多样性指数Shannon最大的为霞多丽,最小的为赤霞珠,Simpson指数则基本相反,说明这8种酿酒葡萄表面霞多丽细菌群落多样性最高,赤霞珠最低。
如表 3所示,不同品种酿酒葡萄表面真菌ITS1区测序深度指数coverage更高,即使是最低的龙眼也可达99.23%,表明此次测序数据量可以代表样本的真实情况。不同品种酿酒葡萄表面真菌的丰富度指数Chao 1和ACE最大的均为雷司令,Chao1指数最小的为龙眼,ACE指数最小的为宝石,龙眼也较小,Chao1和ACE基本一致,说明这8种酿酒葡萄表面雷司令真菌群落最丰富,龙眼和宝石则最稀少。真菌的多样性指数Shannon最大的为增芳德,最小的为美乐,Simpson指数则基本相反,说明这8种酿酒葡萄表面增芳德真菌群落多样性最高,美乐最低。
在门的分类水平上,不同品种酿酒葡萄表面细菌除未被分类(unclassfied)类群外,主要分布在8个已知的门(图 2-A):变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、芽单胞菌门(Gemmatimonadetes)、醋杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)和Thermi。在这8个门的细菌中,占比最高的是变形菌门(Proteobacteria),可占霞多丽总细菌的75.30%,占比最低的黑品诺也可达31.30%。在属的分类水平上,不同品种酿酒葡萄表面细菌主要分布在6个属(图 2-B):欧文氏菌属(Erwinia)、假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)、丙酸杆菌属(Propionibacterium)、Kaistobacter和小双孢菌属(Microbispora)。在这6个属的细菌中,占比最高的是欧文氏菌属(Erwinia),可占赤霞珠总细菌的46.20%,占比最低的黑品诺则仅为4.20%。

|
| 图 2 不同品种酿酒葡萄表面微生物在门和属水平上的比较 A:细菌门水平;B:细菌属水平;C:真菌门水平;D:真菌属水平 |
如图 2-C所示,不同品种酿酒葡萄表面真菌多样性较少,仅分布在3个门:子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota)。在这3个门的真菌中,占比最高的是子囊菌门(Ascomycota),可占雷司令总真菌的92.50%,占比最低的宝石也可达89.38%。占比最低的是接合菌门(Zygomycota),仅占龙眼总真菌的1.43%,增芳德则低达0.63%。如图 2-D所示,不同品种酿酒葡萄表面真菌主要分布在9个属(排序不分先后):链格孢菌属(Alternaria)、枝孢属(Cladosporium)、茎点霉属(Phoma)、镰孢属(Fusarium)、耐冷酵母属(Guehomyces)、孢霉属(Mortierella)、隐球菌属(Cryptococcus)、黑附球菌属(Epicoccum)和漆斑霉属(Myrothecium)。在这9个属的真菌中,占比最高的是链格孢菌属(Alternaria),可占黑品诺总真菌的46.24%,占比最低的霞多丽也可达20.57%。
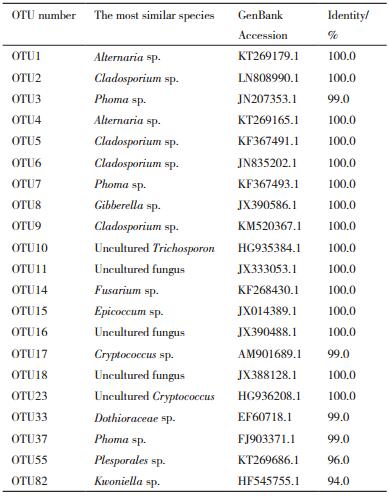
2.4 中心OTU及最相似序列分析如表 4所示,对于不同品种酿酒葡萄表面细菌提取序列数占序列总数1%以上的OTU为中心OTU,得到16个相对丰度较高的OTU。细菌中心OTU所含序列数占序列总数比例最高的是赤霞珠(69.02%),最低的是霞多丽(43.13%)。将这些OTU的代表序列在NCBI上进行Blast序列比对得到的最相似菌种分别为节杆菌(Arthrobacter sp.,OTU4、OTU7和OTU9)、假单胞菌(Pseudomonas sp.,OT-U1和OTU10)、芽孢杆菌(Bacillus sp.,OTU2)、动性球菌(Planococcus sp.,OTU5)、泛生菌(Pantoea sp.,OTU6)、短小杆菌(Curtobacterium sp.,OTU8)、纤维菌(Cellulomonas sp.,OTU11)、柄杆菌(Caul-obacter sp.,OTU12)、迪茨氏菌(Dietzia sp.,OTU-21)、Uncultured malikia sp.(OTU22)、甲基杆菌(Methylobacterium sp.,OTU37)、根瘤菌(Mesorhizo-bium sp.,OTU47)和未知种属的放线菌(OTU23)。
如表 5所示,对于不同品种酿酒葡萄表面真菌提取序列数占序列总数1%以上的OTU为中心OTU,得到21个相对丰度较高的真菌OTU,真菌中心OTU所含序列数占序列总数的比例均在80%以上。将这些OTU的代表序列在NCBI上进行Blast序列比对得到最相似菌种分别为枝孢菌(Cladosporium sp.,OTU2、OTU5、OTU6和OTU9)、茎点霉(Phoma sp.,OTU3、OTU7和OTU37)、链格孢菌(Alternaria sp.,OTU1和OTU4)、Gibberia sp.(OTU8)、镰孢菌(Fusarium sp.,OTU14)、黑附球菌(Epicoccum sp.,OTU15)、隐球菌(Cryptococcus sp.,OTU17)、小穴壳菌(Dothioraceae sp.,OTU33)、Plesporales sp.(OTU55)、Kwoniella sp.(OTU82)和未知种属的OTU(OTU10、OTU11、OTU16、OTU18和OTU23)。
随着分子生物学技术的发展,新一代高通量测序技术凭借其通量高、速度快、成本低等特点为研究微生物多样性提供了新的技术平台。本研究采用Illumina平台的Hiseq 2500高通量测序技术对不同品种酿酒葡萄表面细菌16S rRNA基因V5-V7区和真菌ITS1区进行了扩增和测序,结果显示8个细菌样品共得到154 612条有效序列,最少的为龙眼,有5 191条,最多的为霞多丽,有24 995条;8个真菌样品共得到714 707条有效序列,最少的为龙眼,有17 466条,最多的为梅璐哲,有152 569条。采用细菌16S rRNA基因V5-V7区引物(799F和1193R)是为了在扩增过程中避免线粒体DNA和叶绿体DNA的干扰,这样虽然避免了植物来源物种信息的干扰,但是也无法捕捉到蓝细菌等的物种信息;而且,实际上,引物799F仅能和RDP数据库中62%的序列匹配[11],因此,本研究中细菌的有效序列条数(154 612条)较真菌(714 707条)低。在97%相似度下将其聚为不同的OTU,细菌共得到1 897个OTU,真菌共得到1 313个OTU。各样品的覆盖度(coverage)结果显示在相似度为97%的条件下,OTU涵盖了酿酒葡萄表面94%以上的细菌和真菌。以上结果说明此次测序能够覆盖样本中绝大多数的微生物信息,可以代表样本的真实情况。因此,高通量测序技术是分析酿酒葡萄微生物群落的有力工具。
高通量测序技术较传统的分子生物学手段(如聚丙烯酰胺凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)、克隆文库、末端限制性片段长度多态性(Terminal restriction fragment length polymorphism,T-RFLP)和荧光原位杂交(Fluorescence in situ hybridization,FISH)等以及磷脂脂肪酸(Phospholipid fatty acid,PLFA)等技术,具有明显的优势。例如,Redford等[12-14]通过DGGE技术研究棉白杨叶际微生物,结果发现每个样品的可视条带仅为几十条,很难反映叶际微生物群落的全貌,而用高通量测序技术每个样品能够得到上万条序列,其检测灵敏度远远超过DGGE,可以得到更为全面的微生物群落信息。T-RFLP技术可通过和已有数据库进行比对,获得所扩增片段的系统进化信息,如Berg等[15]利用此技术研究了马铃薯抵抗植物病原真菌的附生细菌的群落结构,但是该技术对微生物种属的定性判断存在一定误差。PLFA技术可通过不同微生物含有不同种类和数量的PLFAs对微生物进行分析,如Zhang等[16, 17]利用此技术研究了氯氰菊酯杀虫剂对辣椒和黄瓜叶际微生物群落的影响,但是该技术仅能检测到叶际中活体微生物群落的动态变化。可见,高通量测序技术较传统的非培养方法可以得到更为全面、准确、丰富的微生物群落信息。
高通量测序技术因其突出的优点已经在蔬菜、水果等植物表面微生物群落研究中得以应用。例如,2010年,Lopez-Velasco等[18]将焦磷酸测序技术应用于菠菜叶际细菌16S rRNA基因多样性的研究,在新鲜菠菜叶际发现了8 800个独有的序列,其中75%是以前从未报道过的。2012年,Bokulich等[19]通过焦磷酸测序技术研究了贵腐葡萄酒发酵过程中细菌16S rRNA基因V4-V5区的动态变化,从发酵结束的酒泥中分离出α-变形菌(Alphaproteobacteria)两个种的细菌,揭示了低丰度微生物群落的多样性,这些结果表明贵腐葡萄酒较之前的认识更为复杂,同时为监测葡萄酒发酵过程的动态变化奠定了基础。2013年,Leff等[20]通过焦磷酸测序技术对不同品种新鲜水果和蔬菜表面细菌16S rRNA基因V5-V6区进行了分析,结果发现苹果、桃、葡萄以及蘑菇表面主要的细菌类群为放线菌门(Actinobacteria)、芽孢杆菌门(Bacillus)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)。如今,高通量测序技术仍然在不断改进,必将推动着微生物生态学研究更加迅猛的发展。
3.2 不同品种酿酒葡萄优势菌群存在差异本研究发现酿酒葡萄表面细菌在门的水平上最优势的类群是变形菌门(Proteobacteria),放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)。Leveau等[2]通过焦磷酸测序技术发现霞多丽葡萄上53.5%的细菌为变形菌门(Proteobacteria),其他主要有厚壁菌门(Firmicutes,15.1%),拟杆菌门(Bacteroidetes,10.1%)和放线菌门(Actinobacteria,8.0%)。Martin等[4]通过T-RFLP技术发现美乐葡萄上的优势细菌也主要归属于变形菌门(Proteobacteria),本研究结果与上述结果基本一致。另外,本研究发现酿酒葡萄表面细菌在属的水平上最优势的类群是欧文氏菌属(Erwinia)、假单胞菌属(Pseudomonas)和芽孢杆菌属(Bacillus)。Leveau等[2]发现霞多丽葡萄上的优势菌属为假单胞菌属(Pseudomonas)。Martin等发现美乐葡萄上的优势菌属为假单胞菌属(Pseudomonas)和鞘脂单胞菌属(Sphingomona)。本研究结果与这些结果具有相似之处,只是本研究发现鞘脂单胞菌属(Sphingomona)并不是沙城地区美乐葡萄的优势菌属。在真菌方面,有关葡萄品种和葡萄表面真菌之间关系的研究较少[20-23],Bokulish等[4, 19]研究发现增芳德葡萄表面上的优势真菌菌属为C. zemplinina,C. zemplinina能够增强葡萄酒的感官品质,但是本研究发现增芳德主要的真菌菌属为链格孢菌属(Alternaria),这与前人研究结果差异较大。造成本研究结果与上述文献报道结果不一致的原因可能是多方面的,如葡萄产地、环境以及采样时间等,Martin等[24]的采样时间是在秋季葡萄成熟期,本研究采样的时间是在七月葡萄生长旺盛期,此时正处于葡萄浆果着色的转色期,这个时期,浆果已不再膨大,果皮中的叶绿素大量分解,红葡萄品种果皮开始积累色素,基本转变为红色和深蓝色,而白葡萄品种透明,浆果含糖量直线上升,这个时期基本是大部分葡萄品种含酸量下降的转折点。
本研究还发现酿酒葡萄表面微生物的优势类群因葡萄品种不同存在较大差异。无论是细菌还是真菌在门的水平上差异相对较小,尤其是真菌,3个优势门的真菌可占雷司令总真菌的92.50%,占比最低的宝石也可达89.38%,但在属的水平上差异较大。在细菌属的水平上,霞多丽上的欧文氏菌属(Erwinia)可占细菌总数的46.20%,而雷司令仅为4.30%;雷司令上假单胞菌属(Pseudomonas)为10.17%,黑品诺仅为3.03%;黑品诺上的芽孢杆菌属(Bacillus)为19.96%,赤霞珠仅为1.43%;西拉上的丙酸杆菌属(Propionibacterium)为16.39%,赤霞珠仅为0.03%。在真菌属的水平上,龙眼上的链格孢菌属(Alternaria)可占真菌总数的39.40%,最少的霞多丽也占到20.57%;龙眼上的枝孢属(Cladosporium)为24.14%,而西拉仅为6.74%;赤霞珠上的茎点霉属(Phoma)为30.62%,而雷司令仅为0.64%;增芳德中的隐球菌属(Cryptococcus)为1.17%,霞多丽仅为0.29%。由此可见,酿酒葡萄表皮微生物群落因葡萄品种不同差异较大,葡萄品种是影响微生物群落的最重要的因素。
3.3 微生物群落及其对葡萄酒酿造的影响研究还发现,细菌中心OTU结果显示酿酒葡萄表面假单胞菌(Pseudomonas sp.)和芽孢杆菌(Bacillus sp.)含量较多,这两种细菌是自然生态系统中广泛存在的类群,其中的假单胞菌能够产生胞外多糖,有利于微生物膜(biofilm)的形成,对于微生物在葡萄表面定殖有着重要的作用。细菌中心OTU结果还显示葡萄表面还有较多短小杆菌(Curtobacterium sp.)和根瘤菌(Mesorhizobium sp.),有研究表明短小杆菌(Curtobacterium sp.)分泌物对葡萄酒品质无明显影响[24],而根瘤菌(Mesorhizobium sp.)具有固氮的独特作用,能够促进葡萄树的生长[25]。
真菌中心OTU结果显示酿酒葡萄表面含有较多链格孢菌(Alternaria sp.),有研究表明其可产生链格孢醇、链格孢醇单甲醚、链孢菌酮酸等70多种有毒的真菌代谢物从而引发葡萄黑斑病,人及动物摄入这些真菌毒素可导致急性或慢性中毒,有些还有致癌、致畸、致突变等的作用[26, 27]。链格孢菌(Alternaria sp.)在其他水果如苹果、西红柿、蓝莓以及谷物微生物研究中也有报道[28]。真菌中心OTU结果还显示葡萄表面还有较多枝孢菌(Cladosporium sp.)、茎点霉(Phoma sp.)和隐球菌(Cryptococcus sp.),枝孢菌(Cladosporium sp.)是环境中广泛存在的丝状真菌,茎点霉(Phoma sp.)和隐球菌(Cryptococcus sp.)通常主要来源于葡萄园。枝孢菌(Cladosporium sp.)和茎点霉(Phoma sp.)可使葡萄腐烂而产生毒素,会造成葡萄中化学成分改变并且在葡萄酒发酵过程中可影响酿酒酵母的生长繁殖。隐球菌(Cryptococcus sp.)可产生果胶酶,提高葡萄酒酿造过程中的出汁率[29-31]。葡萄酒发酵过程中的微生物在没有外源接种的情况下通常认为源自酿酒葡萄本身[32]。生长在葡萄上的霉菌产生的多种代谢产物(如真菌毒素赭曲霉素A)会干扰葡萄的微生物生态环境,从而影响葡萄酒发酵过程中酿酒酵母的生长,改变酒的风味[33, 34]。真菌还能够在葡萄表面创造一种有利于醋酸菌生长的环境,产生酸性物质(如醋酸等),从而改变葡萄汁和葡萄酒的pH,并且产生酸味,对葡萄酒的感官、生化和微生物的稳定性有直接影响[35-37],甚至会导致酒的腐败变质[38]。
4 结论不同品种酿酒葡萄表皮微生物群落结构差异明显,微生物丰度最大的为雷司令,细菌主要分布在变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)等8个门,优势细菌主要分布在欧文氏菌属(Erwinia)、假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)等6个属。真菌仅分布在子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota)3个门,链格孢菌属(Alternaria)、枝孢属(Cladosporium)、茎点霉属(Phoma)、镰孢属(Fusarium)等9个属。
| [1] | 孙炜宁, 张巧格, 李兴兴, 等. 葡萄酒酿造过程中微生物多样性的研究现状. 食品研究与开发, 2014, 35(18): 365–368. |
| [2] | Leveau JHJ, Tech JJ. Grapevine microbiomics:bacterial diversity on grape leaves and berries revealed by high-throughput sequence analysis of 16S rRNA amplicons[C]. International Symposium on Biological Control of Postharvest Diseases:Challenges and Opportunities 905, 2010:31-42. |
| [3] | Bokulich NA, Thorngate JH, Richardson PM, et al. Microbial biogeography of wine grapes is conditioned by cultivar, vintage, and climate. Proceedings of the National Academy of Sciences, 2014, 111 (1): E139–E148. DOI:10.1073/pnas.1317377110 |
| [4] | Martins G, Lauga B, Miot-Sertier C, et al. Characterization of epiphytic bacterial communities from grapes, leaves, bark and soil of grapevine plants grown, and their relations. PLoS One, 2013, 8 (8): e73013. DOI:10.1371/journal.pone.0073013 |
| [5] | Chelius MK, Triplett EW. The diversity of archaea and bacteria in association with the Roots of Zea mays L. Microbial Ecology, 2001, 41 (3): 252–263. DOI:10.1007/s002480000087 |
| [6] | Schloss PD, Westcott SL, Ryabin T, et al. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75 (23): 7537–7541. DOI:10.1128/AEM.01541-09 |
| [7] | Zarraonaindia I, Owens SM, Weisenhorn P, et al. The soil microbiome influences grapevine-associated microbiota. MBio, 2015, 6 (2): e02527–14. |
| [8] | Chao A. Nonparametric estimation of the number of classes in a population. Scandinavian Journal of Statistics, 1984, 11 (4): 265–270. |
| [9] | 郭正刚, 王根绪, 沈禹颖, 等. 青藏高原北部多年冻土区草地植物多样性. 生态学报, 2004, 24(1): 149–155. |
| [10] | Gupta C, Dubey R, Maheshwari D. Plant growth enhancement and suppression of Macrophomina phaseolina causing charcoal rot of peanut by fluorescent Pseudomonas. Biology and Fertility of Soils, 2002, 35 (6): 399–405. DOI:10.1007/s00374-002-0486-0 |
| [11] | Redford AJ, Bowers RM, Knight R, et al. The ecology of the phyllosphere:geographic and phylogenetic variability in the distribution of bacteria on tree leaves. Environmental Microbiology, 2010, 12 (11): 2885–2893. DOI:10.1111/emi.2010.12.issue-11 |
| [12] | Bodenhausen N, Horton MW, Bergelson J. Bacterial communities associated with the leaves and the roots of Arabidopsis thaliana. PLoS One, 2013, 8 (2): e56329. DOI:10.1371/journal.pone.0056329 |
| [13] | Redford AJ, Fierer N. Bacterial succession on the leaf surface:a novel system for studying successional dynamics. Microbial Ecology, 2009, 58 (1): 189–198. DOI:10.1007/s00248-009-9495-y |
| [14] | 夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价. 微生物学报, 2014, 54(12): 1489–1499. |
| [15] | Berg G, Krechel A, Ditz M, et al. Endophytic and ectophytic potato-associated bacterial communities differ in structure and antagonistic function against plant pathogenic fungi. FEMS Microbiology Ecology, 2005, 51 (2): 215–229. DOI:10.1016/j.femsec.2004.08.006 |
| [16] | Zhang B, Bai Z, Hoefel D, et al. The impacts of cypermethrin pesticide application on the non-target microbial community of the pepper plant phyllosphere. Science of the Total Environment, 2009, 407 (6): 1915–1922. DOI:10.1016/j.scitotenv.2008.11.049 |
| [17] | Zhang B, Zhang H, Bo Jin, et al. Effect of cypermethrin insecticide on the microbial community in cucumber phyllosphere. Journal of Environmental Sciences, 2008, 20 (11): 1356–1362. DOI:10.1016/S1001-0742(08)62233-0 |
| [18] | Lopez-Velasco G, Welbaum GE, Boyer RR, et al. Changes in spinach phylloepiphytic bacteria communities following minimal processing and refrigerated storage described using pyrosequencing of 16S rRNA amplicons. Journal of Applied Microbiology, 2011, 110 (5): 1203–1214. DOI:10.1111/jam.2011.110.issue-5 |
| [19] | Bokulich NA, Joseph CML, Allen G, et al. Next-generation sequencing reveals significant bacterial diversity of botrytized wine. PLoS One, 2012, 7 (5): e36357. DOI:10.1371/journal.pone.0036357 |
| [20] | Leff JW, Fierer N. Bacterial communities associated with the surfaces of fresh fruits and vegetables. PLoS One, 2013, 8 (3): e59310. DOI:10.1371/journal.pone.0059310 |
| [21] | Sabate J, Cano J, Esteve-Zarzoso B, et al. Isolation and identification of yeasts associated with vineyard and winery by RFLP analysis of ribosomal genes and mitochondrial DNA. Microbiological Research, 2002, 157 (4): 267–274. DOI:10.1078/0944-5013-00163 |
| [22] | Cordero-Bueso G, Arroyo T, Serrano A, et al. Influence of the farming system and vine variety on yeast communities associated with grape berries. International Journal of Food Microbiology, 2011, 145 (1): 132–139. DOI:10.1016/j.ijfoodmicro.2010.11.040 |
| [23] | Tofalo R, Schirone M, Torriani S, et al. Diversity of Candida zemplinina strains from grapes and Italian wines. Food Microbiology, 2012, 29 (1): 18–26. DOI:10.1016/j.fm.2011.08.014 |
| [24] | Martins G, Miot-Sertier C, Lauga B, et al. Grape berry bacterial microbiota:impact of the ripening process and the farming system. International Journal of Food Microbiology, 2012, 158 (2): 93–100. DOI:10.1016/j.ijfoodmicro.2012.06.013 |
| [25] | 姜怡, 杨颖, 陈华红, 等. 植物内生菌资源. 微生物学通报, 2005, 32(6): 146–147. |
| [26] | da Motta S, Valente Soares L M. Survey of Brazilian tomato products for alternariol, alternariol monomethyl ether, tenuazonic acid and cyclopiazonic acid. Food Additives & Contaminants, 2001, 18 (7): 630–634. |
| [27] | 李凤琴. 链格孢毒素及其食品卫生问题. 中国食品卫生杂志, 2001, 13(6): 45–49. |
| [28] | Chulze S, Torres A, Dalcero A, et al. Production of alternarioi and alternariol monomethyl ether in natural substrates in comparison with semisynthetic culture medium. Mycotoxin Research, 1994, 10 (2): 79–84. DOI:10.1007/BF03192256 |
| [29] | Pretorius IS, Van der Westhuizen TJ, Augustyn OPH. Yeast biodiversity in vineyards and wineries and its importance to the South African wine industry. A review. South African Journal of Enology and Viticulture, 1999, 20 (2): 61–68. |
| [30] | Barata A, Malfeito-Ferreira M, Loureiro V. The microbial ecology of wine grape berries. International Journal of Food Microbiology, 2012, 153 (3): 243–259. DOI:10.1016/j.ijfoodmicro.2011.11.025 |
| [31] | 李华. 现代葡萄酒工艺学[M]. 西安: 陕西人民出版社, 2000. |
| [32] | Bokulich NA, Ohta M, Richardson PM, et al. Monitoring seasonal changes in winery-resident microbiota. PLoS One, 2013, 8 (6): e66437. DOI:10.1371/journal.pone.0066437 |
| [33] | Stander MA, Steyn PS. Survey of ochratoxin A in South African wines. S Afr J Enol. Vitic, 2002, 23 (1): 9–13. |
| [34] | Jackson RS. Wine science:principles and applications[M]. Academic Press, 2008. |
| [35] | Boulton RB, Singleton VL, Bisson LF, et al. Principles and practices of winemaking. Springer Science & Business Media, 2013 . |
| [36] | Swiegers JH, Bartowsky EJ, Henschke PA, et al. Yeast and bacterial modulation of wine aroma and flavour. Australian Journal of Grape and Wine Research, 2005, 11 (2): 139–173. DOI:10.1111/j.1755-0238.2005.tb00285.x |
| [37] | Innerebner G, Knief C, Vorholt JA. Protection of Arabidopsis thaliana against leaf-pathogenic Pseudomonas syringae by Sphingomonas strains in a controlled model system. Applied and Environmental Microbiology, 2011, 77 (10): 3202–3210. DOI:10.1128/AEM.00133-11 |
| [38] | Fowles GWA. Acids in grapes and wines:A review. Journal of Wine Research, 1992, 3 (1): 25–41. DOI:10.1080/09571269208717912 |