海藻糖[1]由两个葡萄糖分子结合而成的非还原性双糖,因其特殊的生物学功能和其独特的应用价值,广泛应用于食品、药品农牧业等行业,其发展前景非常广阔,但是其生产上的不足导致其应用受到一定程度的限制。经过科学家不断地探索研究表明,当前倍受认可的海藻糖生产工艺是利用海藻糖合酶(trehalose synthase)[2, 3]直接麦芽糖生成海藻糖,但海藻糖合酶的获取受到限制,影响海藻糖的生产。Chen和Li等[4, 5]利用大肠杆菌表达系统成功实现了海藻糖合成酶稳定表达,因大肠杆菌自身具有产胞内内毒素的缺点,致使海藻糖以及安全性在食品、药品农牧业等行业备受质疑。为诱导表达外源蛋白,常用的诱导型启动子系统有Pgrac、Pxyl和PsacB系统[6-8],这3个启动子系统在大肠杆菌和枯草杆菌研究工作中应用较广。Wang等[9]就通过IPTG诱导Pgrac启动子表达系统成功将海藻糖合成酶在枯草芽孢杆菌中表达,但是它们各自的成本高、有毒性、表达量低的缺点使得它们在工业生产中有一定的局限性。
因此,为实现海藻糖合成酶的安全高效分泌表达系统,安全的宿主菌和安全的转录表达系统的选择是关键因素。枯草芽孢杆菌(Bacillus subtilis)[10, 11]作为一种革兰氏阳性细菌,由于其自身的非致病特性和分泌外源蛋白能力强的特性,已经成为在工业化和相关食品药学蛋白生产中重要的生物技术工具,是公认的一种安全菌株。枯草芽孢杆菌有多种不同的转录表达系统,最近有文献[12]报道枯草芽孢杆菌的一种潜在的麦芽糖诱导型转录系统,Pglv启动子是一个可调控启动子,该操纵元由编码6-磷酸-α-葡萄糖苷酶(GlvA)的基因,调控蛋白(GlvR)基因和磷酸烯醇丙酮酸-糖磷酸转移系统透性酶(GlvC)基因以及它们的调控元件组成。麦芽糖在被GlvC转运到枯草芽孢杆菌胞质的同时被磷酸化成为6-磷酸麦芽糖,GlvR与6-磷酸麦芽糖结合后被激活,从而与Pglv启动子结合促进了其转录强度。而葡萄糖的代谢产物调节蛋白CcpA[13]与在麦芽糖启动子区域的cre序列结合,对glv启动子起到反馈抑制作用。该表达系统所用到的诱导物麦芽糖具有成本低、无毒性的特点,其安全和有效性在制备食品级的酶制剂具有广泛的应用前景。
本实验选择在枯草芽孢杆菌中遗传稳定较好的载体PHT01,围绕以cre序列定点突变优化的麦芽糖启动子调控元件构建海藻糖合成酶表达载体,同时通过单交叉互换法敲除α-淀粉酶基因来优化宿主菌株,实现重组质粒在枯草芽孢杆菌中高效表达,旨在获得麦芽糖诱导海藻糖合成酶的安全诱导表达系统,为枯草芽孢杆菌工程菌安全高效表达系统的研究提供了有力的工具。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒实验所用的菌株有大肠杆菌DH5α(E.coli DH5α)(作为克隆宿主)购自艾德莱生物科技有限公司,恶臭假单胞杆菌KT2440(Pseudomonas putida KT2440)、枯草芽孢杆菌168(B.subtilis 168)和枯草芽孢杆菌WB800n(B.subtilis WB800n)菌株以及质粒pPIC9K和穿梭表达质粒PHT01购于杭州宝赛生物科技有限公司。
1.1.2 生化试剂T4 DNA连接酶和限制酶购于宝生物工程(大连)有限公司,高纯质粒小量快速提取试剂盒、细菌基因组提取试剂盒、零背景pTOPO-TA克隆试剂盒、pZERO-Blunt零背景平末端快速连接试剂盒、2×Taq PCR MasterMix和2×HiFi-PCR master购于艾德莱生物科技有限公司,SanPrep柱式DNA胶回收试剂盒和DNA纯化回收试剂盒购于生工生物工程(上海)股份有限公司。
1.1.3 引物及核苷酸序列本实验采用的引物通过Oligo7.0软件设计,引物合成由上海生工生物工程公司合成,其引物序列见表 1。测序引物为T3和T7启动子的通用引物。1.1.4培养基LB培养基:蛋白胨10 g/L、酵母浸粉5 g/L、氯化钠10 g/L,pH 7.0-7.4;GM增殖培养基:蛋白胨10 g/L、酵母浸粉5 g/L、氯化钠10 g/L、0.5 mol/L山梨醇RM复苏培养基:蛋白胨10 g/L、酵母浸粉5 g/L、氯化钠10 g/L、0.5 mol/L山梨醇、0.38 mol/L甘露醇;ETM电转缓冲液:0.5 mol/L山梨醇、0.5 mol/L甘露醇、0.5 mol/L海藻糖、10 %甘油。
麦芽糖启动子Pglv含有一个cre序列GTAAACGTTATCA,该序列与葡萄糖代谢蛋白CcpA结合抑制启动子的表达,Yang等[14]将序列的CG碱基突变为AT碱基可大大减弱葡糖糖的抑制。因此为了进一步提高表达效果,本研究通过利用引物定点突变的方式对麦芽糖启动子Pglv的中间的cre序列进行定点突变,以B.subtilis 168的基因组为模板,用引物Pglv-1-F和Pglv-1-R-C(含有将CG碱基突变为AT)、引物Pglv-2-F-C和Pglv-2-R-C分别PCR扩增269 bp的Pglv-1片段和121 bp的Pglv-2片段,分别在片段Pglv-1的5' 端引入BamH Ⅰ酶切位点,在片段Pglv-2的3' 端引入GlvA基因的57 bp片段、TAA终止密码子和来源于B.subtilis 168的GlvC基因的SD序列,通过重叠PCR技术[15, 16]将Pglv-1和Pglv-2两个片段连接得到Pglv-1+2基因片段。以Pseudomonas putida KT24-40的基因组为模板,用引物TreS-F-C和TreS-R PCR扩增出2 067 bp的海藻糖合成酶基因片段TreS,在TreS片段的3' 端引入Aat Ⅱ酶切位点,通过重叠PCR技术Pglv-1+2和TreS两个片段连接得到Pglv-TreS基因片段。将Pglv-TreS片段连接到pTOPO-TA粘性末端载体,将载体转化到大肠杆菌DH5α感受态细胞及提取质粒。用BamH Ⅰ和Aat Ⅱ两个限制酶消化pTOPO-T-Pglv-TreS-1,胶回收Pglv-TreS-1基因片段,克隆到PHT01载体的相应酶切位点上,得到表达载体Pglv-PHT01-TreS(图 1)。
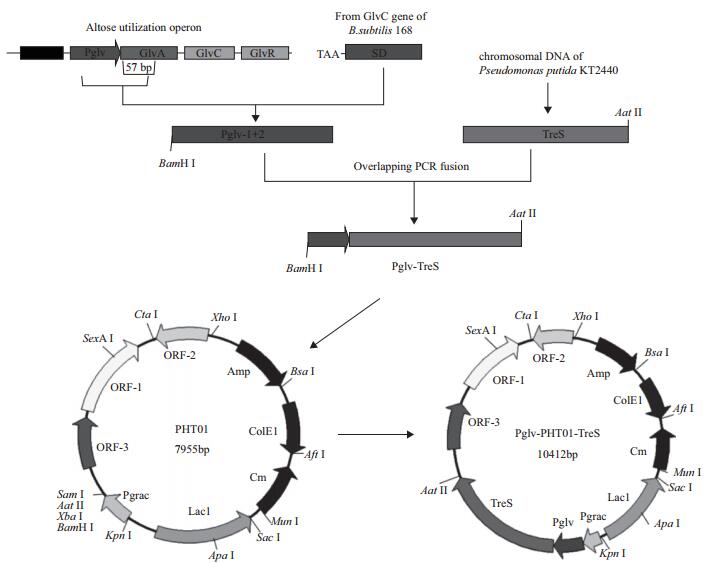
|
| 图 1 质粒载体Pglv-PHT01-TreS构建流程图 |
挑取新鲜的LB固体培养基表面的B.subtilis WB800n单菌落,接种于5 mL的LB培养基中,37℃、200 r/min培养过夜;取500 μL的上述菌液转接到50 mL的增殖培养基GM中,接种后OD600值达0.15,37℃、200 r/min培养至OD600=1.0;将菌液转移至已灭过菌的100 mL离心管,冰水浴10 min后,5 000 r/min、4℃离心8 min,收集菌体;离心后的菌体用预冷的电转缓冲液ETM洗涤3-4次,将洗涤后的菌体重悬于500 μL的ETM中,即为枯草芽孢杆菌电转化感受态细胞[17, 18];将制备好的感受态细胞分装60 μL每管,-80℃保存备用。
1.2.3 构建重组菌B.subtilisWB800n(Pglv-PHT01-TreS)从-80℃冰箱取出一管60 μL的感受态,加入6 μL的质粒Pglv-PHT01-TreS混合均匀,预冷5 min后将混合的菌液加入2 mm的电转杯中,用Eppendorf电转仪在1 500 V、5 ms条件下电击一次。电转完毕后,迅速加入1 mL复苏培养基RM,37℃、180 r/min复苏4 h后,离心重悬后涂布在含氯霉素(10 μg/mL)固体LB培养基,倒置放在37℃恒温培养箱中培养1-2 d,筛选抗氯霉素的菌株。
1.2.4 阳性克隆菌株的鉴定挑取上述抗氯霉素的菌株的单菌落,接种于含氯霉素抗性的LB培养基中,在37℃、200 r/min条件下培养10 h后离心收集菌体,利用艾德莱生物科技有限公司提供的高纯质粒小量快速提取试剂盒提取其质粒。然后以质粒为模板,通过引物TreS-F-C/TreS-R来进行PCR扩增,扩增产物利用1%琼脂糖凝胶电泳进行验证,最终获得阳性克隆菌株WB800n(Pglv-PHT01-TreS)。
1.2.5 利用单交叉互换敲除法敲除枯草芽孢杆菌α-淀粉酶基因以枯草芽孢杆菌B.subtilis168基因组为模板,通过引物amyE-1-F/amyE-1-R扩增α-淀粉酶基因amyE的前500 bp长度的基因,得到基因片段amyE-1,该片段作为同源片段可与基因组发生同源交叉互换;以质粒pPIC9K为模板,通过引物kan-F/kan-R扩增得到卡那霉素(Kanamycin)抗性基因kan;通过重叠PCR技术将两个片段连接得到片段amyE-1-kan;在基因片段amyE-1-kan的两端分别引入BamH Ⅰ酶切位点和相应的保护碱基;将PCR扩增回收的片段amyE-1-kan通过BamH Ⅰ酶切消化后,通过与上述制备的感受态细胞WB800n混合,加入2 mm的电转杯,1 500 V、5 ms条件下电击一次。加入复苏培养基RM后,37℃、180 r/min复苏4 h,离心重悬后涂布在含卡那霉素(15 μg/mL)固体LB培养基,在37℃的恒温培养箱中培养1-2 d,挑取单菌落培养过夜后,提取基因组并通过引物kan-F/kan-R进行PCR验证卡那霉素抗性基因片段,筛选抗卡那霉素的α-淀粉酶基因缺失菌株WB800n(ΔamyE)。
1.2.6 构建重组菌株B.subtilisWB800n(ΔamyE,Pglv-PHT01-TreS)根据上述制备枯草芽孢杆菌WB800n(ΔamyE)的感受态细胞,将构建的麦芽糖诱导型质粒Pglv-PHT01-TreS与感受态细胞混合后,通过电转化方法电击一次,迅速加入复苏培养基RM,37℃、180 r/min复苏4 h后涂布在含氯霉素的10 μg/mL)固体LB培养基,倒置放在37℃恒温培养箱中培养1-2 d,挑取单菌落培养过夜后,提取质粒并PCR验证海藻糖合成酶基因片段,筛选抗氯霉素的菌株WB800n(ΔamyE,Pglv-PHT01-TreS)。
1.3 麦芽糖诱导表达试验 1.3.1 不同麦芽糖质量分数的诱导效果分别挑取保藏的B.subtilis WB800n(Pglv-PHT01-TreS)和B.subtilis WB800n(ΔamyE,Pglv-PHT01 -TreS)单菌落,接种于50 mL的LB液体培养基,37℃、200 r/min摇床培养10 h后,按比例1:50的接种量转接于100 mL新鲜的LB液体培养基,分别培养7瓶并标号,分别添加麦芽糖至终质量分数为0%、1.5%、3%、4.5%、6%、7.5%和10%,37℃、200 r/min条件下培养14 h。取样测定海藻糖合成酶酶活力。
1.3.2 麦芽糖诱导时机的影响选择不同的诱导时机,诱导效果也不同,为了确定最佳的诱导时机,在菌种活化后,按比例1:50的接种量转接于100 mL新鲜的LB液体培养基,分别培养6瓶并标号,通过分光光度计测定其OD600值,当菌体培养到OD600为0.1、0.8、1.2、2.1、2.8和3.2时,分别添加最佳质量分数的诱导剂麦芽糖,37℃、200 r/min条件下培养14 h。取样测定海藻糖合成酶酶活力。
1.3.3 麦芽糖诱导时间的影响在最恰当诱导时机条件下,添加最佳质量分数的麦芽糖诱导48 h,每隔4 h取样,以不添加加麦芽糖诱导剂作为对照组,测定海藻糖合成酶酶活性并测取其随诱导时间的增长而变化趋势,留样进行SDS-PAGE蛋白电泳分析,同时检测诱导过程中发酵液的葡萄糖含量随时间的变化。
1.3.4 海藻糖合成酶活性测定以质量分数为30%的麦芽糖为底物进行测定转化酶活试验。取诱导后的10 mL菌样,首先利用生物传感器SBA-40D检测不同时间取样的葡萄糖的含量,然后8 000 r/min离心10 min弃去上清得到沉淀,然后取1 mL配置好的磷酸盐缓冲溶液(pH8.0磷酸氢二钠-磷酸二氢钠缓冲液配制)重悬沉淀,加入20 μL终溶度为4 μg/mL的溶菌酶37℃水浴2 h裂解细胞壁制得约1 mL的粗酶液,然后加入1 mL配置好的质量分数为60%的麦芽糖母液混匀,在25℃振荡水浴锅中50 r/min转化20 h后煮沸10 min,通过液相色谱分析仪[19]的方法测定其转化的海藻糖的含量并计算出不同诱导时间的每毫升粗酶液的酶活。
酶活力单位定义:在酶的最佳酶反应条件下(25℃,pH 8.0),将300 g/L麦芽糖转化为海藻糖,每小时产生1 μmol海藻糖所需的酶量定义一为个酶活单位(U)。
1.3.5 SDS-PAGE检测分析参照《蛋白质技术手册》,按比例配置15%聚丙烯酰胺凝胶。对其裂解细胞后得到的粗酶液蛋白进行测定分析。
2 结果 2.1 重组菌B.subtilis WB800n(Pglv-PHT01-TreS)的构建 2.1.1 表达载体Pglv-PHT01-TreS的构建以B.su-btilis 168的基因组为模板,用引物Pglv-1-F/Pglv-1-R-C(含有将CG碱基突变为AT)、引物Pglv-2-F-C/Pglv-2-R-C分别进行PCR扩增得到基因片段Pglv-1和Pglv-2,以Pseudomonas putida KT2440的基因组为模板,用引物TreS-F-C和TreS-R PCR进行PCR扩增得到基因片段TreS,然后通过两次重叠PCR技术将3个片段连接,通过1 %琼脂糖凝胶电泳检测发现在2 500 bp左右出现特异性电泳条带(图 2-A),与理论值2 457 bp相符,表明获得了Pglv-TreS基因片段。将制得的片段克隆到表达载体PHT01相应酶切位点,通过双酶切体系BamH Ⅰ/Aat Ⅱ酶切载体,用1%琼脂糖凝胶电泳检测发现在2 500 bp左右出现特异性电泳条带(图 2-B),表明表达载体Pglv-PHT01-TreS构建成功。
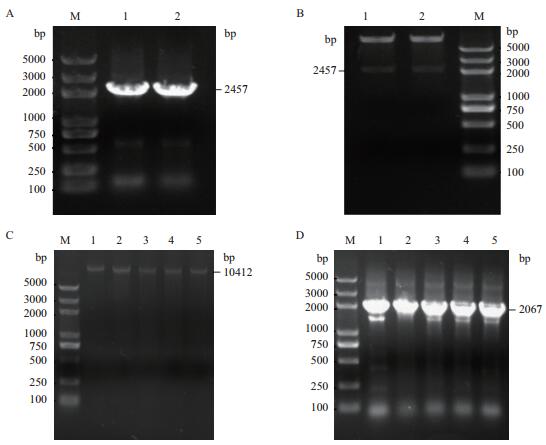
|
| 图 2 1%琼脂糖凝胶电泳图 (A)两次重叠PCR后连接得到的Pglv-TreS片段电泳图,M:maker DL5000,1-2:基因片段Pglv-TreS;(B)酶切验证Pglv-TreS基因片段电泳图,M:maker DL5000,1-2:Pglv-TreS基因片段;(C)提取重组枯草芽孢杆菌的质粒Pglv-PHT01-TreS电泳图,M:maker DL5000,1-5:质粒Pglv-PHT01-TreS片段;(D)PCR验证提取质粒Pglv-PHT01-TreS的TreS片段电泳图,M:maker DL5000,1-5:TreS基因片段 |
提取质粒Pglv-PHT01-TreS并用核酸超微量分光光度计(BioFuture MD2000)测定回收DNA浓度,结果显示DNA浓度为206.82 ng/μL。将6 μL提取的质粒与60 μL B.subtilis WB800n感受态细胞混合并进行电转化,对阳性克隆子进行提取质粒并通过引物TreS-F-C/TreS-R进行PCR验证,结果(图 2-C、2-D),表明质粒载体Pglv-PHT01-TreS成功转化到B.subtilis WB800n中,并获得了阳性克隆菌株。
2.2 α-淀粉酶基因缺失菌株B.subtilis WB800n(ΔamyE)的构建以B.subtilis 168的基因组为模板,用引物amyE-1-F/amyE-1-R和kan-F/kan-R分别扩增α-淀粉酶基因amyE的前500 bp长度的基因片段amyE-1,以及扩增得到卡那霉素(Kanamycin)抗性基因片段kan;利用重叠PCR技术将两个片段连接,通过1 %琼脂糖凝胶电泳检测发现在2 000 bp左右出现特异性电泳条带(图 3-A),与理论值2 002 bp大小相符,即两个片段连接成amyE-1-kan片段。胶回收片段,BamH Ⅰ限制酶酶切后,通过上述电转化方法将片段酶切后的amyE-1-kan转化到B.subtilis WB800n中,涂板,挑取阳性克隆子,PCR验证kan基因片段,通过1 %琼脂糖凝胶电泳检测发现在1 500 bp左右出现特异性电泳条带(图 3-B),与理论值1 502 bp大小相符,说明单交叉互换方式成功将α-淀粉酶基因amyE从B.subtilis WB800n基因组中敲除。
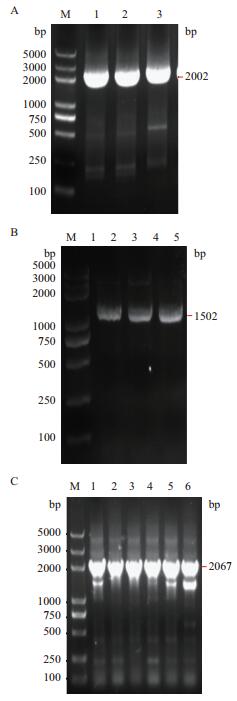
|
| 图 3 1%琼脂糖凝胶电泳图 (A)重叠PCR连接得到的片段amy-1-kan电泳图,M:maker DL5000,1-3:基因片段amy-1-kan;(B)PCR验证α-淀粉酶基因缺失重组菌的卡那霉素抗性基因kan电泳图,M:maker DL5000,1-5:kan基因片段;(C)PCR验证重组菌B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)的TreS片段电泳图,M:maker DL5000,1-6:为TreS基因片段 |
根据上述的枯草芽孢杆菌感受态的制备方法,制备B.subtilis WB800n(ΔamyE)感受态,将质粒Pglv-PHT01-TreS与感受态混合后,通过电转化方法转化复苏后,涂板,挑取阳性克隆子,通过引物TreS-F-C/TreS-R进行PCR验证海藻糖合成酶基因片段,通过1 %琼脂糖凝胶电泳检测发现在2 000 bp左右同样出现特异性电泳条带(图 3-C),证明质粒Pglv-PHT01-TreS成功转化到B.subtilis WB800n(Δa-myE),得到了重组菌株B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)。
2.4 麦芽糖诱导表达分析 2.4.1 不同麦芽糖质量分数的诱导表达分析通过添加至终质量分数为0%、1.5%、3%、4.5%、6%、7.5%和10%的麦芽糖进行诱导表达试验,从图 4可以看出,麦芽糖添加量到终质量分数为4.5%时诱导海藻糖合成酶酶活性最高。诱导初期海藻糖合成酶的酶活增长明显,但是随着麦芽糖诱导质量分数的增加,酶活逐渐下降。产生这种现象的原因,主要由于前期营养物质丰富,菌体增长迅速,海藻糖合成酶表达量高,后期由于营养物质的匮乏以及麦芽糖易作为被微生物利用的二糖,致使麦芽糖的质量分数降低,诱导能力下降。因此确定麦芽糖的最佳诱导质量分数为4.5%。

|
| 图 4 麦芽糖添加终质量分数对海藻糖合成酶酶活力的影响 |
同时还可以看出含有α-淀粉酶基因缺失的菌株B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)海藻糖合成酶酶活表达量要高于含有α-淀粉酶基因的重组菌B.subtilis WB800n(Pglv-PHT01-TreS),酶活可达26 U/mL。说明产胞外的α-淀粉酶对降解麦芽糖起到了酶解作用,致使麦芽糖的质量分数降低,使得未敲除α-淀粉酶基因的重组菌B.subtilis WB800n(Pglv-PHT01-TreS)表达量不高。
2.4.2 麦芽糖不同时期的诱导表达分析当菌体培养到OD600值为0.1、0.8、1.2、2.1、2.8和3.2时,分别添加至终质量分数为4.5%的麦芽糖诱导剂进行诱导,诱导14 h后取样测定酶活,测定结果如图 5,可以看出当菌体OD600值达到1.2时加入麦芽糖诱导的效果最好,产生的海藻糖合成酶酶活明显比其他时期诱导的效果要高,后期随着菌体的生长,酶活逐渐下降,这种现象由于培养基中营养物质的由多到少和菌体生长期变化所致。

|
| 图 5 麦芽糖诱导时机对海藻糖合成酶酶活的影响 |
同时含有α-淀粉酶基因缺失的菌株B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)海藻糖合成酶酶活要高于含有α-淀粉酶基因的重组菌B.subtilis WB800n(Pglv-PHT01-TreS),酶活最高可达28.2 U/mL。因此在菌体生长到OD600值为1.2时,添加至终质量分数为4.5%的麦芽糖诱导效果最佳。
2.4.3 海藻糖合成酶酶活随诱导时间的变化曲线测定随着麦芽糖诱导时间的增加,每隔4 h取样测定酶活,诱导48 h后酶活曲线如图 6所示,可以看出:不添加麦芽糖诱导的重组菌B.subtilis WB800n(Pglv-PHT01-TreS)和B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)海藻糖合成酶酶活始终处于低酶活状态,而添加麦芽糖至终质量分数为4.5%诱导时,海藻糖合成酶酶活表达明显,说明重组质粒Pglv-PHT01-TreS在麦芽糖诱导条件下,成功实现在B.subtilis WB800n中表达,同时其酶活表达量在发酵初期增加迅速,后期随着诱导时间的延长,酶活逐渐下降。后期营养物质的缺乏致使菌体自溶是产生酶活降低的主要原因。

|
| 图 6 海藻糖合成酶酶活随麦芽糖诱导时间变化曲线 |
在整个诱导过程中,含有α-淀粉酶基因缺失的重组菌株B.subtilis WB800n(ΔamyE,Pglv-PHT01 -TreS)的海藻糖合成酶的表达量一直高于含有α-淀粉酶基因的重组菌株B.subtilis WB800n(Pglv-PHT01-TreS)的海藻糖合成酶表达量,在发酵18 h时酶活达到最高29.2 U/mL,而重组菌株B.subtilis WB800n(Pglv-PHT01-TreS)酶活只能达到18.9 U/mL。通过测定发酵液中葡萄糖的含量(图 7)显示,菌株B.subtilis WB800n(Pglv-PHT01-TreS)发酵过程中,葡萄糖的含量呈现缓慢增加趋势,而菌株B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)的葡萄糖一直处于低含量状态,进一步证实了枯草芽孢杆菌产胞外的α-淀粉酶大量降解麦芽糖成葡萄糖,大大降低了麦芽糖的诱导效率,同时葡萄糖的增加也会反馈抑制麦芽糖启动子的转录表达,因此,该产胞外的α-淀粉酶基因的敲除可大大提高海藻糖合成酶的表达量。

|
| 图 7 发酵液中葡萄糖的含量变化曲线 |
在添加麦芽糖至终终质量分数为4.5%时诱导重组菌B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)18 h获得的海藻糖合成酶与不添加诱导剂重组菌B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)发酵18 h所获得的海藻糖合成酶作平行的(SDS -PAGE)凝胶分析比对,结果(图 8)显示,在66.4-97.2 kD之间明显有大小约75 kD的条带,与海藻糖合成酶蛋白的理论大小相符。不加诱导剂时,海藻糖合成酶表达量小,肉眼难辨(图 8泳道1);而加入麦芽糖后,目的蛋白的表达量明显,条带较粗(图 8泳道2),表明无麦芽糖诱导时海藻糖合成酶的表达量不及有麦芽糖在优化条件下进行诱导的海藻糖合成酶的蛋白表达量。

|
| 图 8 SDS-PAGE分析海藻糖合成酶 M:蛋白标准品;1:重组菌B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)在未添加麦芽糖诱导条件下发酵18 h的胞内海藻糖合成酶蛋白表达量;2:重组B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)在添加终质量分数为4.5%的麦芽糖诱导条件下发酵18 h的胞内海藻糖合成酶蛋白表达量 |
本实验首次构建了通过采用枯草芽孢杆菌麦芽糖能够诱导型调控元件来实现调控海藻糖合成酶在枯草芽孢杆菌的表达系统,并通过改变麦芽糖启动子的碳代谢调节蛋白CcpA的cre结合序列,减弱葡萄糖代谢反馈抑制[20],从而提高了目的蛋白的转录水平。通过初步优化发酵条件,制得海藻糖合成粗酶在终质量分数为4.5%麦芽糖诱导18 h时,酶活达到最高18.9 U/mL。为了提高海藻糖合成酶的表达量,通过但交叉互换的方法敲除了枯草芽孢杆菌含有的α-淀粉酶基因,使得胞外的麦芽糖不被α-淀粉酶降解成葡萄糖而诱导效果下降,同时葡萄糖的生成量的降低大大减弱了糖代谢对麦芽糖启动子的反馈抑制作用,使制得的工程菌在麦芽糖的诱导下,酶活提高到了29.2 U/mL。与大肠杆菌商用诱导表达系统[21]相比,该系统所制备的海藻糖在食品医疗行业不再受到限制,且诱导物本身的价格低廉、无毒的特点在实际生产中更具有优势。
为了保证外源蛋白酶的活性,选择破碎枯草芽孢杆菌细胞壁方式是本研究课题的关键前提,本研究是通过试验比较了超声破碎、高压细胞破碎、研磨法和溶菌酶破碎4种方式破碎效果,最终确定采用溶菌酶破碎效果最好。革兰氏细菌的细胞壁[22]主要由肽聚糖和磷壁酸组成,肽聚糖作为细胞壁的主要稳定结构,溶菌酶可以专一性地作用于肽聚糖分子的N-乙酰胞壁酸(NAM)与乙酰葡萄糖氨(NAG)之间的β-1,4糖苷键使之断裂,使得细胞壁变得松弛破裂,从而保证了破碎制得的蛋白酶酶活性验证的准确性以及试验延续性。
在验证诱导发酵后期,随着诱导时间的增加,葡萄糖的含量不断地上升。相关文章[23-25]中已经报道了枯草芽孢杆菌在发酵过程中可以产生大量分泌到胞外的α-淀粉酶。在这基础上,后续试验可将海藻糖合成酶的基因替换枯草芽孢杆菌基因组上的α-淀粉酶基因(不含信号肽),这样可以消除胞外α-淀粉酶对麦芽糖的降解,减低葡萄糖的抑制。将替换得到的海藻糖合成酶还可以通过α-淀粉酶的信号肽引导分泌到胞外,产胞外海藻糖合成酶既可为工业化生产降低了成本,最重要的是还可以直接以诱导物麦芽糖为底物直接转化为海藻糖,为制备海藻糖提供了新方法,具有重要的研究意义。
4 结论基于海藻糖合成酶在生产上的不足导致其应用受到一定程度的限制,本实验通过基因改造的手段,利用枯草芽孢杆菌的麦芽糖诱导调控元件,构建海藻糖合成酶在安全菌株枯草芽孢杆菌中的高效表达系统。通过利用基础培养基初步验证重组菌的发酵条件,当培养重组菌达到OD600为1.2时,添加终质量分数为4.5%的麦芽糖诱导发酵18 h,重组菌B.subtilis WB800n(Pglv-PHT01-TreS)海藻糖合成酶酶活达到18.9 U/mL,而对宿主菌优化后,重组菌B.subtilis WB800n(ΔamyE,Pglv-PHT01-TreS)酶活提高到了29.2 U/mL,成功实现了麦芽糖诱导表达海藻糖合成酶的高效安全表达系统。
| [1] | Elbein AD, Pan YT, Pastuszak I, et al. New insights on trehalose:a multifunctional molecule. Glycobiolgy, 2003, 13 (4): 17R–27R. DOI:10.1093/glycob/cwg047 |
| [2] | Jing S, Wang T, Ma C, et al. Homology modeling and function of trehalose synthase from Pseudomonas putida P06. Biotechnology Letters, 2014, 36 (5): 1009–1013. DOI:10.1007/s10529-013-1450-9 |
| [3] | Dong Y, Ma L, Duan Y. The effect of high pressure on the intracellular trehalose synthase activity of Thermus aquaticus. World Journal of Microbiology & Biotechnology, 2016, 32 (1): 1–6. |
| [4] | Chen PT, Chiang CJ, Chen YT, et al. Strategy for stable and high-level expression of recombinant trehalose synthase in Escherichia coli. Journal of Agricultural & Food Chemistry, 2012, 60 (23): 6063–6068. |
| [5] | Zhang Q, Wang C, Ma Y, et al. Cloning and expression of maltooligosyltrehalose synthase from Sulfolobus acidocaldarius in Escherichia coli. Journal of Shihezi University (Natural Science), 2006, 17 (3): 339–341. |
| [6] | Phan TTP, Tran LT, Schumann W, et al. Development of Pgrac 100-based expression vectors allowing high protein production levels in Bacillus subtilis, and relatively low basal expression in Escherichia coli. Microbial Cell Factories, 2015, 14 (1): 1–9. DOI:10.1186/s12934-014-0183-3 |
| [7] | Bhavsar AP, Zhao XE. Development and characterization of a xylose-dependent system forexpression of cloned genes in Bacillus subtilis:Conditional complementation of a teichoic acid mutant. Applied & Environmental Microbiology, 2001, 67 (1): 403–410. |
| [8] | Zukowski MM, Miller L. Hyperproduction of an intracellular heterologous protein in a sacUh mutant of Bacillus subtilis. Gene, 1986, 46 (2-3): 247–255. DOI:10.1016/0378-1119(86)90409-9 |
| [9] | Wang T, Dai K, Liu H, et al. Cloning and expression of a trehalose synthase from Pseudomonas putida KT2440 in Bacillus subtilis W800N for the production of trehalose. Journal of Pure & Applied Microbiology, 2014, 8 (2): 1687–1692. |
| [10] | Yu X, Tian J, Liu X, et al. Research progress of Bacillus subtilis expression system and its promoter regulatory elements. Biotechnology Bulletin, 2015, 31 (2): 35–44. |
| [11] | 王金斌, 陈大超, 李文, 等. 食品级枯草芽孢杆菌表达系统的最新研究进展. 上海农业学报, 2014, 30(01): 115–120. |
| [12] | Zhang L, Li X, Wei D, et al. Expression of plectasin in Bacillus subtilis, using SUMO technology by a maltose-inducible vector. Journal of Industrial Microbiology, 2015, 42 (10): 1369–1376. DOI:10.1007/s10295-015-1673-y |
| [13] | Yamamoto H, Serizawa M, Thompson J, et al. Regulation of the glv operon in Bacillus subtilis:YfiA (GlvR) is a positive regulator of the operon that is repressed through CcpA and cre. Journal of Bacteriology, 2001, 183 (17): 5110–5121. DOI:10.1128/JB.183.17.5110-5121.2001 |
| [14] | Yang MM, Zhang WW, Chen YL, et al. Development of a Bacillus subtilis, expression system using the improved Pglv, promoter. Microbial Cell Factories, 2010, 9 (1): 1–8. DOI:10.1186/1475-2859-9-1 |
| [15] | Qiu DS, Liu XJ, Wang J, et al. Artificial synthesis of TAT PTD-tachyplesin fusion gene by overlap extension PCR. Agricultural Biotechnology, 2013, 2 (03): 14–17. |
| [16] | Ho SN, Hunt HD, Horton RM, et al. Site-directed mutagenesis by overlap extension using the polymerase chain reaction. Gene, 1989, 77 (1): 51–59. DOI:10.1016/0378-1119(89)90358-2 |
| [17] | Zhang Z, Ding ZT, Shu D, et al. Development of an efficient electroporation method for iturin A-producing Bacillus subtilis ZK. International Journal of Molecular Sciences, 2015, 16 (4): 7334–7351. DOI:10.3390/ijms16047334 |
| [18] | Lu YP, Zhang C, Lv FX, et al. Study on the electro-transformation conditions of improving transformation efficiency for Bacillus subtilis. Letters in Applied Microbiology, 2012, 55 (1): 9–14. DOI:10.1111/lam.2012.55.issue-1 |
| [19] | Yue ME, Niu X. Determination of trehalose and glycerin in Artemias by high performance liquid chromatography. Chemical Reagents, 2009, 31 (5): 375–376. |
| [20] | Zhang Y, Yang M, Zhou H, et al. Optimization of expression of alkali-tolerant xylanase introduced by Bacillus subtilis. Acta Agriculturae Boreali-Occidentalis Sinica, 2011, 20 (1): 14–13. |
| [21] | Rao B, Fan J, Sun J, et al. An auto-inducible expression system based on the RhlI-RhlR quorum-sensing regulon for recombinant protein production in E. coli. Biotechnology & Bioprocess Engineering, 2016, 21 (1): 160–168. |
| [22] | 王静华, 赵洪涛, 汪以真. 细菌细胞壁肽聚糖的研究进展. 中国兽药杂志, 2004, 38(01): 38–40. |
| [23] | Westers H, Darmon E, Zanen G, et al. The Bacillus, secretion stress response is an indicator for α-amylase production levels. Letters in Applied Microbiology, 2004, 39 (1): 65–73. DOI:10.1111/lam.2004.39.issue-1 |
| [24] | Vitikainen M, Pummi T, Airaksinen U, et al. Quantitation of the capacity of the secretion apparatus and requirement for PrsA in growth and secretion of α-amylase in Bacillus subtilis. Journal of Bacteriology, 2001, 183 (6): 1881–1890. DOI:10.1128/JB.183.6.1881-1890.2001 |
| [25] | Demirkan E. Production, purification, and characterization of a-amylase by Bacillus subtilis and its mutant derivates. Turkish Journal of Biology, 2011, 21 (8): S70–S70. |





