2. 甘肃农业大学生命科学技术学院,兰州 730070;
3. 甘肃农业大学动物科技学院,兰州 730070
2. College of Life Science and Technology, Gansu Agriculture University, Lanzhou 730070;
3. College of Animal Science & Technology, Gansu Agriculture University, Lanzhou 730070
牦牛(Bos grunniens)是长期放牧于海拔高达3 000 m以上的高寒地区特殊牛种,属于地方类半野生型的种群,是经过长期的自然选择和人工选育而形成的高原特有畜种资源。牦牛既是我国特有的畜种资源,也是我国稀有而珍贵的遗传资源[1]。牦牛生长发育缓慢、群体规模小、生产性能低下,从而影响牦牛的经济价值和使用价值[2]。
近年来,人们已对与牦牛生长发育等相关基因进行了分子结构特征、多态性和表达分析,且有部分侯选基因的研究相对比较深入。如肌肉生长抑制素(MSTN)基因和胰岛素样生长因子家族(IGF-IR)基因的单核苷酸多态性(single nucleotide polymorphisms,SNPs)对不同年龄阶段的牦牛生产性能有显著影响[3];胰岛素样生长因子2(IGF-2)基因的SNPs对牦牛的生长发育具有显著差异[4];生长分化因子10(GDF-10)、印度刺猬因子(IHH)、黑素皮质素受体-4(MC4R)等基因的SNPs对不同牦牛种群的生产性状有显著影响[5]等。上述基因均可作为牦牛选育的候选基因。
研究发现,跨膜蛋白18(Transmembrane Proteins-18,TMEM-18)是一个末端为低聚嘧啶的基因,该基因能够在体内和体外参与神经干细胞迁移的调节[6]。Hindorff等[7]通过全基因组关联分析(Genome-wide association study,GWAS)发现TMEM-18基因与肥胖密切相关。此后研究表明TMEM-18基因主要通过大脑中的神经系统来调节脂肪细胞分化从而调控肥胖[8];而TMEM-18基因除神经系统中表达外,在不同组织中均见表达[9]。近年来,人类全基因组的研究发现,TMEM-18基因存在与人肥胖连锁的单核苷酸多态性(single nucleotide polymorphism,SNP),该基因的多个SNPs位点(rs6548238、rs2867125、rs4854344和rs756131)均可增加人体质指数(Body Mass Index,BMI)和体重[10-15]。早期研究发现TMEM-18基因可调节小鼠的生长发育,不仅在小鼠大脑中强烈表达,而且在小鼠其他组织中也有表达[16]。在TMEM-18基因的家畜动物研究中,大白猪、南阳牛、郏县红牛、草原红牛、鲁西牛和秦川牛中均检测发现TMEM-18基因的多个SNPs位点均对家畜动物的生长性能有显著影响[17, 18]。因此,我们推测TMEM-18可能与牦牛生长发育及其生产性能有密切关系。然而,牦牛TMEM-18基因的结构、功能及SNPs与生产性能关联分析的研究迄今尚未见报道。本实验拟对牦牛TMEM-18基因的SNPs进行检测,初步筛选与牦牛生产性状相关的多态位点,分析其遗传变异与生长性能的关系,从而为今后牦牛遗传改良和选育以及TMEM-18基因的功能研究奠定基础。
1 材料与方法 1.1 材料实验样本采自于甘肃省天祝县某屠宰场,选取2-3岁的牦牛群体采集192头天祝雌性牦牛血样。颈静脉采血3 mL,轻微振荡混匀后于-80℃保存备用。实地测量并记录其生长性状(体斜长、体高、胸围、管围、体重)及屠宰性能(胴体、净肉、屠宰率、净肉率)。
1.2 方法 1.2.1 基因组DNA提取利用血液基因组DNA提取试剂盒(Omega Bio-Tek,D6919-01B)提取基因组DNA,经1%的琼脂糖凝胶电泳检测DNA的完整性后使用紫外分光光度计检测DNA的浓度。从上述检测合格的DNA样本中随机抽取20 μL稀释至18-20 ng/μL,用作PCR扩增的模板,其余的母液于-80℃保存备用。
1.2.2 基因组DNA池构建选取稀释后的200份DNA样本,每个样本各取1 μL,混合均匀,构建牦牛基因组DNA混合池。
1.2.3 引物设计和PCR扩增参考NCBI数据库中牦牛的TMEM-18基因(登录号:NW_005393077.1),采用交叉重叠原理[15]运用Primer 5.0软件设计3对引物对部分内含子1和外显子5区域进行扩增(表 1),引物由上海生工生物科技有限公司合成。
PCR扩增体系(25 μL):20 ng/μL的基因组DNA 1 μL,10 pmol的上下游引物各1 μL,2×Taq PCR Master Mix(内含Taq DNA聚合酶、Mg2+、dNTPs等)(北京,百泰克)12.5 μL,灭菌超纯水9.5 μL。
PCR反应程序:95℃预变性3 min;94℃变性30 s,复性30 s(退火温度见表 1),72℃延伸30 s,35个循环;72℃延伸10 min;4℃保存。
1.2.4 DNA测序分析PCR扩增产物经琼脂糖凝胶回收试剂盒(北京,全式金)回收纯化后送至苏州金唯智生物科技有限公司进行测序,测序结果通过DNAMAN软件和Chromas软件比对分析。
1.2.5 HRM小片段法基因分型针对所发现的多态位点,利用Light Scanner Primer Design Software(Idaho公司,美国)设计出用于高分辨率熔解曲线(High Resolution melting,HRM)分析多态位点的引物(表 2),对试验群体样本进行基因型分析[19, 20]。合成2对高低温内标[21](表 3)对溶解曲线进行校正,提高分型的精确度。
HRM-PCR扩增体系(11 μL):20 ng/μL的基因组DNA 1 μL,10 pmol的上下游引物各0.2 μL,2×Taq PCR Master Mix(内含Taq DNA聚合酶、Mg2+、dNTPs等)(北京,百泰克)5 μL,灭菌超纯水3.6 μL,10×LC Green饱和染料(美国,Idaho)1 μL。
HRM-PCR反应程序:95℃预变性5 min;94℃变性20 s,Tm(℃)(表 2)复性20 s,72℃延伸20 s,35个循环;72℃延伸10 min。
高低温内标稀释方法:1 OD内标单链加400 μL水,退火体系:饱和氯化钠1 μL,内标互补双链各1 μL,补水至10 μL,95℃水浴3 min,缓慢降至室温。
HRM荧光信号采集:将稀释好的高低温内标各1 μL加入HRM-PCR产物单孔中,95℃水浴30 s;25℃水浴30 s;放入LightScanner 96(美国,Idaho)中收集荧光信号。
1.3 数据统计根据群体遗传学理论[22]计算基因型和等位基因频率,统计牦牛群体TMEM-18基因多态位点的纯和度Ho、杂合度He、有效等位基因数Ne、多态信息含量PIC[23-25],并进行Hardy-Weinberg平衡检验。采用SHEsis软件进行配对连锁不平衡分析。采用SPSS 21.0软件对TMEM-18基因多态性位点在牦牛群体中的基因型分布进行卡方检测,并对不同基因型与生产性状间的关联性进行分析。考虑到性别、环境和年龄因素的影响,采用固定模型分析基因型效应对体尺性状的影响。
固定模型:Yijk=u+Gi+Sj+Ak+Eijk
式中,Yijk:个体表型记录;u:群体均值;Gi:标记基因型效应;Sj:性别因素;Ak:年龄效应;Eijk:随机误差。
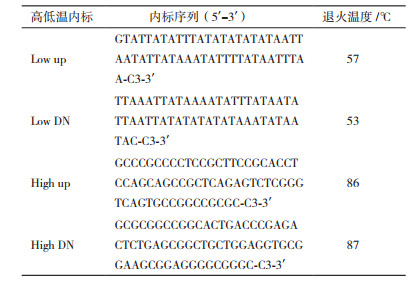
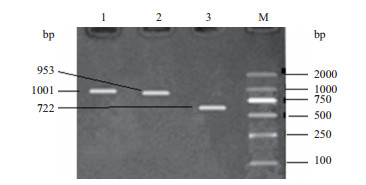
2 结果 2.1 PCR扩增产物以牦牛血液基因组DNA为模板,根据设计的引物进行PCR特异性扩增,结果见图 1。由图 1可知PCR扩增产物均为单一特异性条带。产物经过测序后长度分别为1 001、953和722 bp,与引物设计时预期长度一致。
|
| 图 1 TMEM-18基因PCR扩增电泳图 1:TMEM-18-1扩增产物;2:TMEM-18-2扩增产物;3:TMEM-18-1扩增产物;M:DNAMarker |
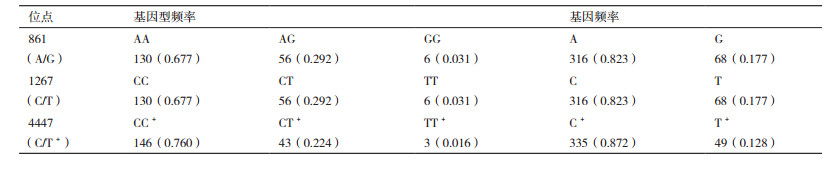
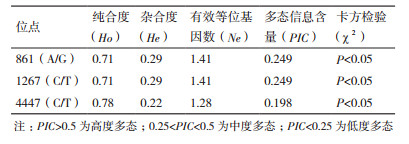
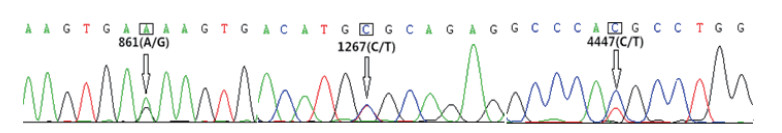
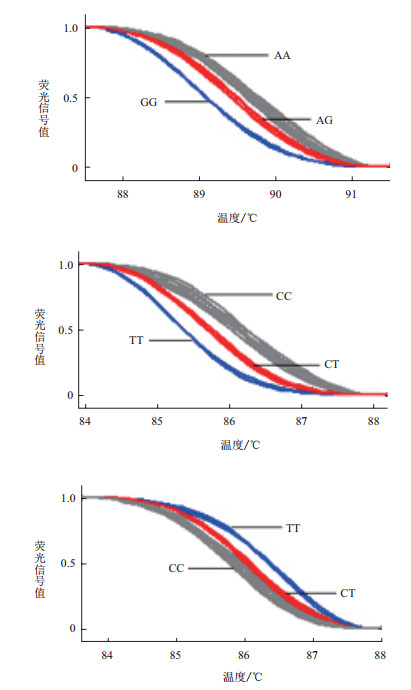
对PCR扩增产物进行测序,结果(图 2)显示,牦牛TMEM-18基因内含子1存在2个多态位点,分别是861(A/G)和1267(C/T),外显子5存在1个多态位点4447(C/T)。通过DNA混合池对牦牛TMEM-18基因测序后,应用高分辨率熔解曲线法对3个多态位点进行基因型分析(图 3)。HRM结果(图 3-A)显示,牦牛TMEM-18基因861(A/G)位点处存在3种基因型,分别是AA、AG和GG。统计分析发现,AA基因型属于优势基因型(n=130),个体数明显多于AG(n=56)和GG(n=6)型;牦牛TMEM-18基因1267(C/T)和4447(C/T)位点处均存在3种基因型,分别为CC、CT和TT型(图 3-B和图 3-C)。统计分析发现,CC基因型均属于优势基因型。
|
| 图 2 TMEM-18基因3个多态位点的测序峰图 |
|
| 图 3 TMEM-18基因3个多态性位点HRM分型曲线 A:861(A/G)位点;B:1267(C/T)位点;C:4447(C/T)位点 |
对牦牛TMEM-18基因多态位点进行基因型频率和基因频率以及相关遗传特性指标计算,结果(表 4)显示,牦牛TMEM-18基因861(A/G)的优势等位基因为A,其基因频率为0.823;牦牛TMEM-18基因1267(C/T)和4447(C/T)的优势等位基因均为C,其基因频率均大于0.8以上。3个位点均表现为低度多态(PIC < 0.25),经Hardy-Weinberg平衡检验发现,3个位点均未达到Hardy-Weinberg平衡(P < 0.05)(表 5)。配对连锁不平衡分析结果(表 6)表明,牦牛TMEM-18基因中上述位点间存在强连锁不平衡(D' > 0.75,r2 > 0.33)。多态位点的单倍型组合的分布及频率结果(表 7)显示,192头牦牛群体中存在3种单倍型,其中ACC在研究群体中个体数最多( > 80%),而GTC和GTT单倍型在群体中只存在少量的个体。
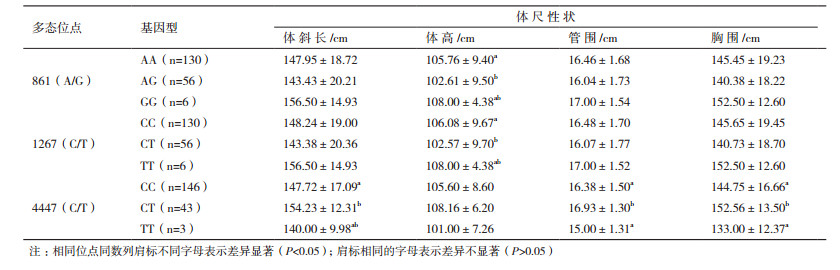
牦牛TMEM-18基因861(A/G)和1267(C/T)位点的不同基因型与不同个体的体高均存在显著差异(P < 0.05),而牦牛体斜长、管围和胸围差异均不显著(P > 0.05),其中861(A/G)位点的GG基因型的个体的生产性能均高于AA和AG基因型的个体;和1267(C/T)位点的TT基因型的个体的生产性能均高于CC、CT基因型个体;牦牛TMEM-18基因4447(C/T)位点的不同基因型个体在体斜长、管围和胸围均存在差异显著(P < 0.05),而体高差异不显著(P > 0.05),其中CT基因型的个体的生产性能均高于CC、TT基因型个体(表 8)。
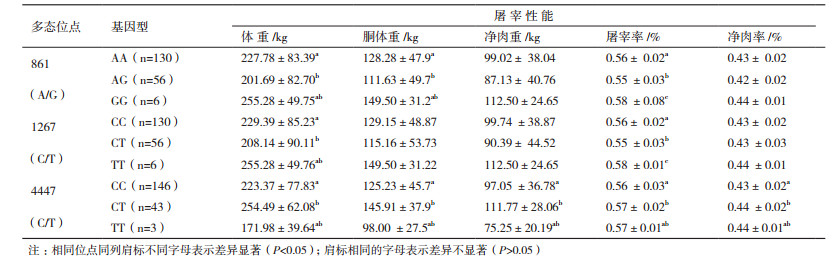
牦牛TMEM-18基因861(A/G)位点的不同基因型个体的体重、胴体重和屠宰率均存在显著差异(P < 0.05),而净肉重和净肉率差异均不显著(P > 0.05),其中GG基因型的个体的屠宰性能均高于AA和AG基因型的个体;1 267(C/T)位点的不同基因型个体的体重、屠宰率存在显著差异(P < 0.05),而胴体重和净肉重和净肉率差异均不显著(P > 0.05),其中TT基因型的个体的屠宰性能均高于CC和CT基因型的个体;4 447(C/T)位点的不同基因型个体的体重、胴体重、净肉重、净肉率和屠宰率均存在显著差异(P < 0.05),其中CT基因型的个体的屠宰性能均高于CC和TT基因型的个体(表 9)。
本实验通过DNA混合池测序得到牦牛TMEM-18基因3个SNPs(861(A/G)、1 267(C/T)和4 447(C/T)),其中1 267(C/T)和4 447(C/T)两个位点为TMEM-18基因上新发现的SNPs,其他物种均未见报道。牦牛TMEM-18基因861(A/G)位点存在3种基因型,分别是AA、GG和AG型,且等位基因A是优势等位基因;1 267(C/T)和4 447(C/T)位点均存在3种基因型,分别是CC、TT和CT型,且等位基因C是优势等位基因。这3个多态位点的优势等位基因频率均达到了0.8以上,这可能与样本的种群和自然环境有关。此外,这3个多态位点均为低度多态,遗传变异低,选择余地较小,这可能因牦牛长期生长在高海拔环境下,长年的高寒、缺氧等恶劣的自然环境下使有效等位基因和多态信息含量降低。而连锁不平衡是单倍型基因非随机分布的现象。若两个连锁基因座的两个等位基因频率分别为p1和p2,这两个等位基因组成的单体型的频率h如果为预期值(plxp2),则称这两个基因座是连锁平衡,如果h高于plxp2就存在连锁不平衡[26]。本实验中,Hardy-Weinberg平衡检验发现,3个位点均未达到Hardy-Weinberg平衡(P < 0.05),这可能与本次实验的数据结构有关,还需要进一步扩大样品进行研究,同时也表明牦牛品种的选育、迁移和遗传漂变等因素对TMEM-18基因的突变造成影响,可能导致未达到平衡。近年来在中国牛上也开始了对TMEM-18基因的序列测序和多态性研究,马伟[17]在不同品种的牛TMEM-18基因中共发现4个不同SNPs(1085(T/G)、2303(A/G)、3835(G/A)和3865(G/A)),均未达到Hardy-Weinberg平衡(P < 0.05),与本结果相似。
牦牛TMEM-18基因内含子1处存在2个多态位点,分别是861(A/G)和1267(C/T)。内含子虽为基因中无编码功能的序列,但其对基因表达调控有着决定作用[27]。一些基因中的内含子对基因表达具有正调控作用,Chang等[28]发现猪MyHC基因内含子中存在重要调控元件,可以调控转录起始来增强基因表达。而张冉[29]检测鲁西黄牛TMEM-18基因在起始密码子上游541 bp和861 bp处存在SNPs位点,且证实861 bp处的SNPs位点影响启动子活性,表现为负调控作用。由此推测,牦牛TMEM-18基因内含子861(A/G)突变也可能参与介导了该基因的转录调控。因此,本研究对了解与分析该基因遗传特征及其对重要经济性状的影响仍然有重要的意义。
关联分析表明,牦牛TMEM-18基因861(A/G)位点的不同基因型与牦牛的体高、体重、胴体重和屠宰率差异性显著(P < 0.05);1267(C/T)位点的不同基因型与牦牛的体高、体重和屠宰率差异性显著(P < 0.05),而牦牛TMEM-18基因4447(C/T)位点的不同基因型与牦牛的体斜长、管围、胸围、体重、胴体重、净肉重、净肉率和屠宰率均存在显著性差异(P < 0.05)。此外,本实验在牦牛后代群体中找到了3种单倍型组合,其中ACC单倍型组合个体数目高达80%以上,表明ACC单倍型为主要单倍型。这与目前相关文献对动物TMEM-18基因多态性及其与生产性能关联分析研究报道的相关结论一致。例如,林平[16]在大白猪TMEM-18基因中共发现了1个SNPs(91(T/G)),且与猪生长性状和腰荐结合处背膘厚性状显著关联(P < 0. 01),说明TMEM-18基因具有在选种中运用的价值。马伟[17]在不同品种的牛TMEM-18基因中共发现4个不同SNPs,对中国牛的体尺性状有显著影响,并说明在其4个新SNPs中,有3个可作为中国牛品种体尺和体重等生长性状选择分子育种的候选标记。因此本研究推测TMEM-18基因上的3个多态位点可影响牦牛的生长性状。综上所述,牦牛TMEM-18基因可作为牦牛生产性状有关的候选分子标记,对提高分子标记辅助选择的准确性,为今后牦牛分子育种提供主要的理论依据,为牦牛遗传育种提供参考。
4 结论牦牛TMEM-18基因3个多态位点与牦牛的体尺指标和屠宰性能显著相关,可以尝试作为牦牛生长性状的候选分子标记,应用于牦牛分子标记辅助育种中。
| [1] | 何梅兰, 马秀山. 天祝白牦牛产业发展现状及发展战略. 中国草食动物科学, 2015, 35(5): 71–74. |
| [2] | 马苍. 天祝白牦牛产业可持续发展思考. 家畜生态学报, 2008, 29(6): 133–135. |
| [3] | 梁春年.牦牛MSTN和IGF-IR基因的克隆及SNPs与生长性状相关性[D].兰州:甘肃农业大学, 2011. |
| [4] | 李亚兰.天祝白牦牛IGF-2基因的全长cDNA克隆, 组织表达及生产性状相关性分析[D].兰州:甘肃农业大学, 2015. |
| [5] | 李天科.牦牛GDF-10, IHH, MC4R基因的多态与生长性状的相关性研究[D].兰州:甘肃农业大学, 2014. |
| [6] | Jurvansuu J, Zhao Y, Leung DS, et al. Transmembrane protein 18 enhances the tropism of neural stem cells for glioma cells. Cancer Research, 2008, 68 (12): 4614–4622. DOI:10.1158/0008-5472.CAN-07-5291 |
| [7] | Hindorff LA, Sethupathy P, Junkins HA, et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci USA, 2009, 106 (23): 9362–9367. DOI:10.1073/pnas.0903103106 |
| [8] | Gutierrez-Aguilar R, Kim DH, Woods SC, et al. Expression of new loci associated with obesity in diet-induced obese rats: from genetics to physiology. Obesity (Silver Spring), 2011, 20 : 306–312. |
| [9] | Bernhard F, Landgraf K, Klöting N, et al. Functional relevance of genes implicated by obesity genome wide association study signals for human adipocyte biology. Diabetologia, 2013, 56 (2): 311–322. DOI:10.1007/s00125-012-2773-0 |
| [10] | Rask-Andersen M, Jacobsson JA, Moschonis G, et al. Association of TMEM18 variants with BMI and waist circumference in children and correlation of mRNA expression in the PFC with body weight in rats. European Journal of Human Genetics, 2012, 20 (2): 192–197. DOI:10.1038/ejhg.2011.176 |
| [11] | Hotta K, Nakamura M, Nakamura T, et al. Association between obesity and polymorphisms in SEC16B, TMEM18, GNPDA2, BDNF, FAIM2 and MC4R in a Japanese population. Journal of Human Genetics, 2009, 54 (12): 727–731. DOI:10.1038/jhg.2009.106 |
| [12] | Holzapfel C, Grallert H, Huth C, et al. Genes and lifestyle factors in obesity: Results from 12462 subjects from MONICA/KORA. International Journal of Obesity, 2010, 34 (10): 1538–1545. DOI:10.1038/ijo.2010.79 |
| [13] | Hong KW, Oh B. Recapitulation of genome-wide association studies on body mass index in the Korean population. International Journal of Obesity, 2011, 36 (8): 1127–1130. |
| [14] | Zhao J, Bradfield JP, Li M, et al. The role of obesity-associated loci identified in genome-wide association studies in the determination of pediatric BMI. Obesity (Silver Spring), 2009, 17 (12): 2254–2257. DOI:10.1038/oby.2009.159 |
| [15] | Almén MS, Jacobsson JA, Shaik JH, et al. The obesity gene, TMEM18, is of ancient origin, found in majority of neuronal cells in all major brain regions and associated with obesity in severely obese children. BMC Medical Genetics, 2010, 11 (1): 58. DOI:10.1186/1471-2350-11-58 |
| [16] | 林平.大白猪和通城猪肌纤维组织学特性差异和SH2B1, TEM18, IDH3B基因遗传效应分析[D].武汉:华中农业大学, 2013. |
| [17] | 马伟.牛TMEM18基因克隆, SNP检测及其与部分经济性状的关联分析[D].西安:西北农林科技大学, 2012. |
| [18] | 郭宪, 李天科, 裴杰, 阎萍, 等. 南德温杂交肉牛生长分化因子10基因多态性与体尺性状的关联分析. 中国畜牧兽医, 2014, 41(12): 197–203. |
| [19] | Michael L, Robert P, Robert P, et al. Genotyping of single-nucleotide polymorphisms by high-resolution melting of small amplicons. Molecular Diagnostics and Genetics, 2004, 50 (7): 1156–1164. |
| [20] | Ye MH, Chen JL, Zhao GP, et al. Associations of A-FABP and H-FABP markers with the content of intramuscular-lar fat in Beijing-you chicken. Animal Biotechnology, 2010, 21 : 14–24. |
| [21] | 曾凯.群体遗传学理论在检测正向选择与研究物种形成机制中的应用[D].广州:中山大学, 2007. |
| [22] | Cameron N, Steven F. Base-pair neutral homozygotes can be discriminated by calibrated high-resolution melting of small amplicons. Nucleic Acids Research, 2008, 1093 (10): 1–8. |
| [23] | Nei M, Roychoudhury AK. Sampling variances of heterqozygosity and genetic distance. Genetics, 1974, 76 (2): 379–390. |
| [24] | Nei M, Li WH. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc Natl Acad Sci USA, 1979, 76 (10): 5269–5273. DOI:10.1073/pnas.76.10.5269 |
| [25] | Yamashita R, Suzuki Y, Takeuchi N, et al. Comprehensive detection of human terminal oligo-pyrimidine (TOP) genes and analysis of their characteristics. Nucleic Acids Research, 2008, 36 (11): 3707–3715. DOI:10.1093/nar/gkn248 |
| [26] | 牛晓亮.牦牛CAPN4和CAST基因多态性及与胴体和肉质性状关联研究[D].兰州:甘肃农业大学, 2015. |
| [27] | Caws E, White SN, Riley DG, et al. Assessment of single nucleotide polymorphisms in genes residing on chromosomes 14 and 29 for association with carcass composition traits in Bos indicus cattle. Journal of Animal Science, 2005, 83 (1): 13–19. DOI:10.2527/2005.83113x |
| [28] | Chang KC. Critical regulatory domains in intron 2 of porcine sarcomeric myosin heavy chain gene. Journal of Muscle Research and Cell Motility, 2000, 21 : 451–461. DOI:10.1023/A:1005625302409 |
| [29] | 张冉.肉质性状相关基因的表达及消化道菌群多样性与动物生长发育的关系[D].济南:山东大学, 2014. |