草坪草作为地被绿化植物,在改善生态环境、美化生活中起着重要的作用[1, 2]。结缕草属(Zoysia Willd.)是一类在全球范围内广泛种植的暖季型草坪草,系禾本科结缕草属植物,它具有发达的匍匐茎和根状茎,叶片厚硬且近革质,建成的草坪具有弹性好、耐修剪、耐践踏、耐盐碱、抗高温和抗干旱等优良特性[3],因而被广泛地用于城市绿地、运动场草坪、机场草坪、水土保持、护坡护堤等诸多方面。该属植物包括11个种和部分变种、变型,主要分布在太平洋沿岸的东亚、东南亚和澳洲地区。我国是结缕草种质资源大国,分布有5个种,即日本结缕草(Zoysia japonica Steud.)、细叶结缕草(Zoysia tenuifolia Willd.ex Trin.)、大穗结缕草(Zoysia macrostachya Franch.et Sav.)、中华结缕草(Zoysia sinica Hance)和沟叶结缕草(Zoysia matrella(L.)Merr.)[3]。山东省胶州湾和辽宁省的辽东半岛是我国结缕草天然种质资源的主要分布地,也是我国结缕草种子生产的重要基地。长期以来,开发结缕草种质资源、培育优良新品种,一直是我国草坪草育种的重点内容之一。
自然条件下,干旱、低温和高盐碱等非生物逆境是影响草坪建植和养护的重要因素[4]。研究逆境胁迫响应的分子机制,培育抗逆性强的品种,一直是结缕草育种研究中的重要内容。对拟南芥、水稻以及其他多种植物的研究结果表明,植物感受逆境信号后调节多个基因的表达[5],从而进行一系列生理生化调节以应对逆境[6]。根据功能,逆境应答基因可分为两大类:一类是编码功能蛋白基因,包括一些渗透调节物合成基因、抗氧化酶基因和抗脱水蛋白基因等,主要起保护细胞免受胁迫损伤的作用;另一类是调节基因,包括转录因子、激酶基因等,它们参与应激信号转导和调节功能基因的表达[7]。转录因子由于调节多个功能基因的表达,在逆境响应中起重要作用。已报道的植物逆境响应转录因子包括AP2/EREBP类[8]、bZIP类[9]、MYB类[10]、WRKY类[11]和NAC类[12]。其中,属于AP2/EREBP类的DREB(dehydration responsive element binding)亚家族基因研究得最深入,它们参与抵抗干旱、低温和高盐胁迫应答。DREB亚家族转录因子均含有1个由57-70个左右氨基酸残基组成的保守的AP2/EREBP结构域,分为A-1-A-6六个亚组[13]。DREB1和DREB2,即A-1和A-2亚组,与逆境响应关系最密切,在林木[14]、果树[15]、禾谷类作物[16]、观赏植物[17]、草坪草[18]等种类中均有研究报道。除此以外,DREB A-4亚组的一些基因,包括拟南芥中的TINY1[19]和TINY2[20]、棉花中的GhDBP3基因[21]、玉米中的ZmDBF2基因[22]和大豆中的GmTINY1基因[23],它们的表达受低温胁迫、干旱胁迫和植物激素ABA、乙烯等不同程度的诱导。超表达TINY的拟南芥突变体表现出了植株矮小、叶厚、胚轴缩短、雌蕊和花丝缩短、花药位置低于柱头等性状[24]。转化了川桑(Morus notabilis)MnDREB4A基因的烟草叶片保水能力更强,衰老速度减慢,植株抗低温、高盐和干旱的能力也有所增强[25]。这些研究结果说明DREB亚家族A-4亚组成员也参与植物的逆境应答,具有抗逆功能。已有的研究结果为结缕草逆境响应分子调节研究提供了方向。
日本结缕草‘Meyer’转录组测序结果显示,结缕草有多个DREB转录因子基因,但是它们在抵抗逆境中的作用尚未有系统的研究[26],目前仅有一个DREB1[27]和两个DREB2[28, 29]基因序列和功能报道。根据结缕草‘Meyer’转录组的注释信息,其中有一个拼接序列可能是DREB4基因。本研究在该序列推测阅读框两端设计引物,克隆获得结缕草第一个DREB4基因,命名为ZjDREB4.1。利用生物信息学软件分析该基因的生物信息学特征,采用半定量RT-PCR技术检测低温、干旱和高盐胁迫下叶组织中ZjDREB4.1的表达状况,构建ZjDREB4.1的植物表达载体,为后续研究其功能提供理论参考和研究材料。
1 材料与方法 1.1 材料日本结缕草(Zoysia japonica Steud.)品种‘Me-yer’由中国科学院南京植物所刘建秀研究员惠赠。材料种植于培养室中,栽培基质为珍珠岩:蛭石:营养土按1:1:2比例的混合物,培养温度25℃/20℃(L/D),光周期14 h/d,正常管理。选取生长状态相近的材料,分别进行逆境处理。低温处理是将材料转移到低温生长箱中,培养温度6℃/4℃(L/D),光强6 000 lux。分别用含有20% PEG-6000和300 mmol/L NaCl的营养液浇灌根部模拟干旱和高盐胁迫。逆境处理时间梯度为2、12、24和72 h。以培养室内未处理材料为对照(0 h,CK)。取植株顶端第1-2片展开叶,液氮速冻后置于-80℃备用。
总RNA抽提试剂TRIpure Reagent、胶回收试剂盒、质粒小量制备试剂盒和pUM-T载体均购自北京百泰克生物技术有限公司。M-MLV逆转录酶和T4 DNA连接酶均购自Promega公司。大肠杆菌(Escherichia coli)DH5α购自天根生化科技(北京)有限公司。DNA限制性内切酶购自Thermo公司。实验用引物(表 1)合成及测序由生工生物工程(上海)股份有限公司完成。
采用TRIpure Rea-gent总RNA抽提试剂提取各样品中的总RNA。RNA经电泳检测完整性、Nanodrop测定浓度。以检测合格的总RNA为模板,以Oligo dT为引物,用反转录酶M-MLV反转录为cDNA,具体操作按照说明书进行。以actin1为内标基因,用cDNA特异性引物Pactin-F和Pactin-R(表 1)对cDNA进行PCR扩增,检测反转录效果。总DNA的提取采用CTAB法[30]。
1.2.2 基因的克隆分别以4℃胁迫2 h的cDNA和基因组DNA为模板,用引物P4.1-F和P4.1-R(表 1)扩增目的基因的编码区序列,反应体系为(20 μL):2×Taq PCR Master Mix 10 μL,P4.1-F和P4.1-R各2 μL(10 μmol/L),cDNA或DNA模板1 μL,ddH2O 5 μL。反应程序为:95℃ 5 min;94℃ 30 s,65℃ 30 s,72℃ 45 s,共32个循环;72℃ 7 min,4℃保存。扩增产物在1%琼脂糖凝胶中电泳检测,并回收接近700 bp的特异性产物。将回收产物与T载体(pUM-T)连接,产物转化E.coli DH5α,进行蓝白斑筛选。挑取白斑进行液体培养,取菌液进行PCR检测。随机挑取2个PCR阳性的菌液进行测序鉴定,确定克隆的基因序列完全正确。
1.2.3 基因的生物信息学分析采用生物信息学软件对获得的ZjDREB4.1序列进行相关分析,具体分析目标及选用的软件见表 2。
采用半定量RT-PCR分析叶片中ZjDREB4.1在低温、干旱以及高盐3种胁迫下的表达动态。以cDNA为模板,先用Pactin-F和Pactin-R对内参基因actin1进行PCR扩增,反应体系为(20 μL):2×Taq PCR Master Mix 10 μL,Pactin-F和Pactin-R各2 μL(10 μmol/L),cDNA模板1 μL,ddH2O 5 μL。反应程序为:95℃ 5 min;94℃ 30 s,65℃ 30 s,72℃ 45 s,共25个循环;72℃ 7 min,4℃保存。产物经1.5%琼脂糖凝胶电泳,确定条带亮度基本一致时各处理的cDNA用量。然后以该用量的cDNA为模板,用ZjDREB4.1基因特异性引物P1和P2(表 1),参照以上扩增体系进行34个循环的PCR扩增,产物经1.5%琼脂糖凝胶电泳,比较各处理条带亮度。
2 结果 2.1 ZjDREB4.1基因全长的克隆根据转录组测序信息,在预测的基因CDS两端设计引物,分别以4℃低温处理2 h的cDNA和基因组DNA为模板进行PCR扩增,结果(图 1)均获得了大小在500-750 bp之间的特异带。将该核酸片段克隆到T载体上,测序结果显示,两种模板扩增的核酸序列相同,且与转录组中的基因序列完全一致,说明该基因没有内含子。
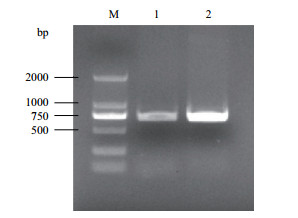
|
| 图 1 ZjDREB4.1基因扩增结果 M:标准分子量;1:模板为cDNA;2:模板为基因组DNA |
ZjDREB4.1基因CDS长651 bp,编码一条由216个氨基酸残基组成的多肽(图 2)。推测的平均蛋白质分子量为22.9 kD,理论pI为5.74。蛋白的分子式组成为C996H1556N290O319S7,其中丙氨酸(Ala)含量最高,为15.7%,其次为丝氨酸(Ser,12.0%)、脯氨酸(Pro,10.2%)和天冬酰胺(Asp,7.9%),酪氨酸(Tyr)含量最低,为0.9%。利用ExPASy分析该蛋白在酵母和大肠杆菌中的半衰期分别大于20和10 h,不稳定系数为47.80(40以上为稳定蛋白),推测为稳定蛋白。

|
| 图 2 ZjDREB4.1基因的核苷酸序列及其编码的氨基酸序列 虚线框为潜在的磷酸化位点;N端潜在的碱性核定位信号用方框标出;阴影部分表示AP2/EREBP保守结构域;*表示终止密码子 |
在NCBI网站上对ZjDREB4.1蛋白进行保守结构域预测结果表明,在50-110位氨基酸之间含有一个典型的AP2结构域(conserved AP2 domain)。cNLS Mapper分析显示,蛋白ZjDREB4.1有两个核定位信号,分别位于第32-40和第66-80位氨基酸区间(图 2);SignalP 3.0 Server软件分析确定ZjDREB4.1不具有信号肽,推测ZjDREB4.1为核定位蛋白。NetPhos软件分析显示,ZjDREB4.1蛋白存在丝氨酸和苏氨酸2个磷酸化位点,但这两个位点的磷酸化数目不同,其中丝氨酸磷酸化位点20个,苏氨酸磷酸化位点4个(图 2)。用ProtScale分析该序列的亲/疏水性,结果表明,在多肽链第38位精氨酸(Arg)有最低分值-3.333,亲水性最强;第132位赖氨酸(Lys)有最高分值2.422,疏水性最强。亲水区域大于疏水区域,故该肽链表现为亲水性。利用CFSSP预测ZjDREB4.1二级结构结果显示,该蛋白的二级结构以α-螺旋和β-折叠片为主,其中α-螺旋含量达到64.8%,还含有一部分的β-转角和无规则卷曲(图 3-A)。SWISS-MODEL server在线模型预测显示,ZjDREB4.1的第49-110位氨基酸与数据库中的14个模型相匹配,其中与3gcc.1模型相似度最高(61.4%)。以3gcc.1为模板建立的ZjDREB4.1结构模型显示,其结构域含有3个反向平行的β-sheet和一个与β-sheet几乎平行的α-helix(图 3-B)。
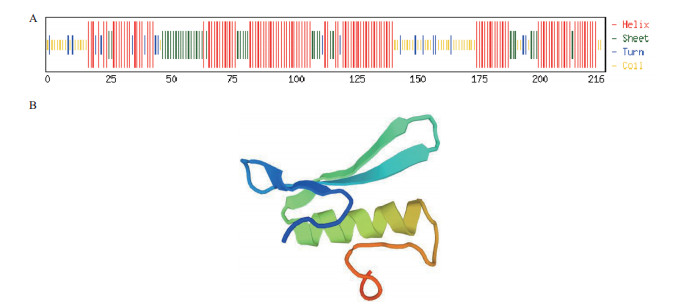
|
| 图 3 ZjDREB4.1蛋白二级结构和模型预测 A:二级结构分析(线)段由长及短分别代表Helix,Sheet,Turn,coil;B:保守结构域模型 |
利用NCBI上的BLAST对ZjDREB4.1氨基酸序列进行同源检索,结果表明,该基因与已报道的多种植物的DREB类转录因子高度相似。将ZjDREB4.1与其6个直系同源基因的氨基酸序列进行比对,结果(图 4)表明,它们的AP2结构域具有高度保守性,而在C端和N端均有较大差异性。选取模式植物拟南芥DREB A-1-A-6基因和其他6种植物的部分DREB类转录因子基因,通过DNAMAN软件对它们的氨基酸序列进行多序列比对和构建基因系统发生树,结果(图 5)显示ZjDREB4.1属于DREB亚家族的A-4亚组,与水稻的OsDREB4.4遗传距离最近。进化树中A-4亚组的10个同源基因明显地分为3个分支,水稻的4个基因分别归属到3个分支中。在第一个分支中含有单、双子叶植物的同源基因,但两类明显地分开;ZjDREB4.1与水稻的OsDREB4.1/4.4、玉米的ZmDBF2和小麦的TmCBF7同属第二分支;而水稻OsDREB4.2为一个单独的分支。说明植物DREB类转录因子A-4亚组一些成员的出现可能在单、双子叶植物分化之前。
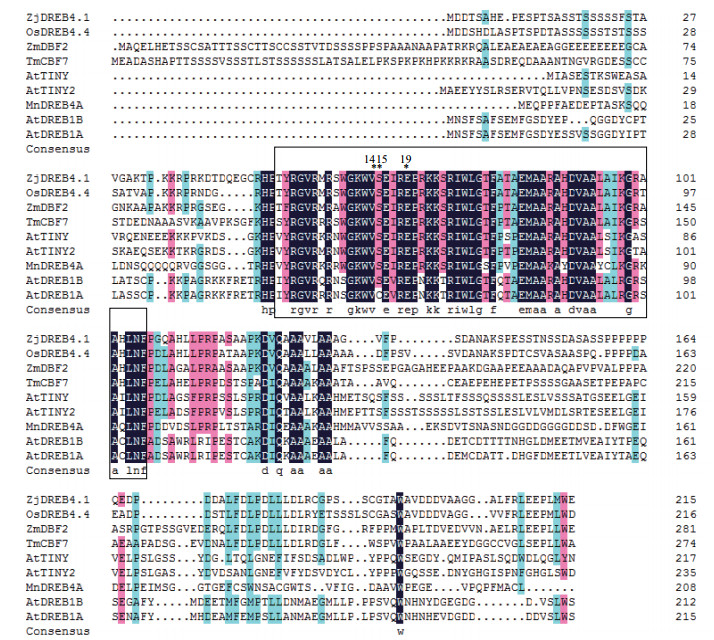
|
| 图 4 ZjDREB4.1与其他同源基因氨基酸序列比较 OsDREB4.4:水稻(Oryza.sativa,AY258282.1);AtTINY:拟南芥(Arabidopsis.thaliana,AT5G25810.1);AtTINY2:拟南芥(A.thaliana,AT5G11590.1);TmCBF7:一粒小麦(Triticum.monococcum,AAX28965.1);MnDREB4A:川桑(Morus notabilis,AHJ25978.1);ZmDBF2:玉米(Zea mays,AAM80485.1);AtDREB1B:拟南芥(A.thaliana,AT4G25490.1);AtDREB1A:拟南芥(A.thaliana,AT4G25480.1);方框内为AP2/EREBP DNA结合结构域 |
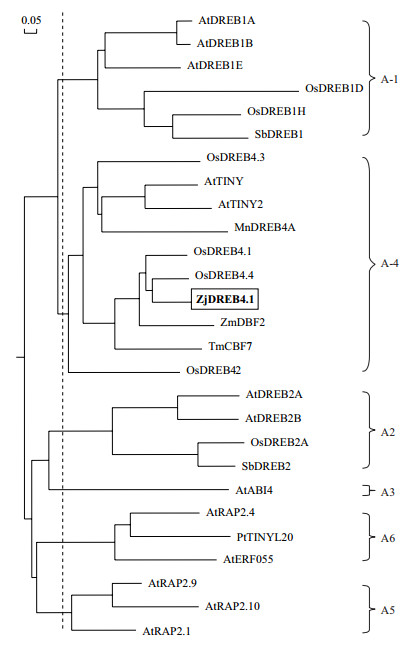
|
| 图 5 ZjDREB4.1的系统发生树 A-1-A-6:分别代表Sakuma提出的DREB家族的6个组;标尺表示分支长度的相对遗传距离;At:拟南芥(A.thaliana);Os:水稻(O.sativa);Pt:毛果杨(P.trichocarpa);Sb:高粱(S.bicolor);Tm:一粒小麦(T.monococcum);Zm:玉米(Z.mays);Mn:川桑(M.notabilis);Zj:日本结缕草(Z.japonica);方框内为日本结缕草Meyer的DREB4.1基因 |
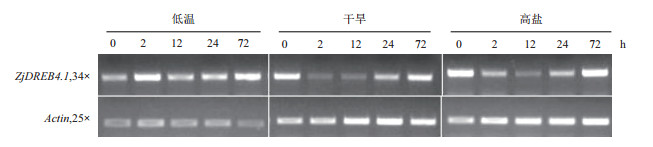
以actin1为内参,采用半定量RT-PCR技术比较ZjDREB4.1在低温、干旱和高盐胁迫下的表达量变化。结果(图 6)表明,ZjDREB4.1在正常条件下有表达;在低温胁迫2-72 h时间内上调表达,其中在2 h和72 h表达水平较高;在干旱和高盐胁迫下,2-24 h时间内均下调表达,72 h后恢复至接近正常水平,说明ZjDREB4.1是结缕草叶组织的正常表达基因,受低温胁迫诱导上调表达,在干旱和高盐胁迫72 h后保持正常表达。
|
| 图 6 不同逆境胁迫下ZjDREB4.1的表达模式 |
DREB类转录因子与植物抵抗非生物胁迫关系密切,调控多个抗逆功能基因的表达,参与植物的抗逆活性调节[31, 32]。结缕草是一种多耐逆的优质草坪草,研究其DREB基因结构、功能和表达模式可为该属植物的抗逆机制提供分子参考。转录组测序结果显示,日本结缕草中含有DREB转录因子A-1-A-6亚组多个基因,部分基因与低温胁迫应答相关[26]。Wang等[28]从结缕草‘Meyer’中克隆获得了首个DREB A-2亚组基因,过量表达该基因的拟南芥植株的抗寒能力明显增强。可祥等[29]以结缕草‘胶东青’变种为材料,克隆获得了第2个DREB A-2亚组基因ZjDREB2.2,其表达量受低温和干旱诱导上调。冯勋伟和才宏伟[27]以日本最北部原产的结缕草品系为材料,克隆获得了结缕草首个DREB A-1亚组基因ZjCBF,超表达该基因的拟南芥植株的抗寒性明显高于野生型。本研究以结缕草‘Meyer’为实验材料,克隆获得该属植物的首个DREB A-4亚组基因,命名为ZjDREB4.1。ZjDREB4.1基因编码区全长651 bp,没有内含子。已报道的川桑(Morus notabilis)[25]、毛果杨(Populus trichocarpa)[33]、水稻(Oryza sativa)[34]的DREB A-4组基因也没有内含子。说明DREB转录因子亚家族中,A-4组与A-1基因的结构相似[35],而与A-2类基因不同,后者通常含有一至多个内含子[28, 36]。
推测的ZjDREB4.1蛋白含有一个AP2结构域,其结构模型与拟南芥的AtERF1基因的AP2结构域的模型(3gcc.1)高度相似[37]。AtERF1属于乙烯响应元件结合蛋白(Ethylene-responsive element binding proteins,EREBPs),这些蛋白的AP2结构域高度保守,可以结合以GCCGCC为核心的ERE元件(简称GCC-box)[38]。拟南芥的AtTINY、AtTINY2还可以结合以CCGAC为核心序列的DRE元件。对不同DREB基因的AP2结构域进行序列比对分析发现,结构域的第14位缬氨酸(V14)和第19位谷氨酸(E19)对识别和结合DRE顺式元件至关重要[13],而第15位的丝氨酸(S15)与识别ERE元件相关[19]。ZjDREB4.1的AP2结构域具有V14、E19和S15的结构特征,其对ERE和DRE元件结合活性有待鉴定。
已报道的DREB A-4组基因中,有一些基因的表达受非生物胁迫调节。拟南芥AtTINY基因在根和叶中的表达几乎不受干旱、高盐、低温和ABA的诱导[19],但是AtTINY2基因在正常生长条件下几乎不表达,在低温、干旱和高盐胁迫下其表达量会上升[20]。水稻OsDREB4.1基因为组成型表达,OsDREB4.2和OsDREB4.3受干早和高盐诱导,但不受低温影响[34]。玉米ZmDBF2基因在低温和高盐胁迫下上调表达[22]。川桑MnDREB4A基因在叶中主要响应低温胁迫,在12-24 h时达到峰值,然后降低[25],而在根中对4种胁迫(高温、低温、高盐、干旱)几乎都有响应。本实验结果显示,结缕草叶组织中ZjDREB4.1为组成型表达,低温逆境诱导其上调表达,在干旱和高盐胁迫2-24 h时表达水平下降,72 h后恢复到正常水平。该逆境响应模式与已报道同源基因有差异,这也体现了不同植物同类基因在表达调节上有差异,这种差异可能与植物的抗逆特征相关。
4 结论本研究报道了结缕草的DREB类转录因子A-4组的一个基因ZjDREB4.1。该基因具有一个AP2结构域,与EREBP的AP2结构域的结构模型高度相似。叶组织中ZjDREB4.1为组成型表达,受低温诱导上调表达,在干旱和高盐胁迫72 h后保持正常表达。
| [1] | 任继周. 草坪业是我国全民共有、全民共建、全民共享的伟大事业--在中国草学会草坪专业委员会第七届全国代表大会暨十一届学术研讨会上的发言. 草地学报, 2008, 16(6): 545–546. |
| [2] | 单华佳, 李梦璐, 孙彦, 等. 近10年中国草坪业发展现状. 草地学报, 2003, 21(2): 222–227. |
| [3] | 董厚德, 宫莉君, 等. 中国结缕草生态学及其资源开发与应用[M]. 北京: 中国林业出版社, 2001: 105-113. |
| [4] | 王艳, 李建龙, 邓蕾, 等. 4种结缕草在南京地区的田间表现及耐寒性差异. 中国草地学报, 2009, 31(6): 34–38. |
| [5] | Barah P, Jayavelu ND, Rasmussen S, et al. Genome-scale cold stress response regulatory networks in ten Arabidopsis thaliana ecotypes. BMC Genomics, 2013, 14 : 722. DOI:10.1186/1471-2164-14-722 |
| [6] | Huang GT, Ma SL, Bai LP, et al. Signal transduction during cold, salt, and drought stresses in plants. Molecular Biology Reports, 2012, 39 (2): 969–987. DOI:10.1007/s11033-011-0823-1 |
| [7] | Nakashima K, Yamaguchi-Shinozaki K, Shinozaki K. The transcriptional regulatory network in the drought response and its crosstalk in abiotic stress responses including drought, cold, and heat. Front Plant Sci, 2014, 5 : 170. |
| [8] | Dietz KJ, Vogel MO, Viehhauser A. AP2/EREBP transcription factors are part of gene regulatory networks and integrate metabolic, hormonal and environmental signals in stress acclimation and retrograde signalling. Protoplasma, 2010, 245 (1): 3–14. |
| [9] | Hu W, Yang H, Yan Y, et al. Genome-wide characterization and analysis of bZIP transcription factor gene family related to abiotic stress in cassava. Sci Rep, 2016, 6 : 22783. DOI:10.1038/srep22783 |
| [10] | Roy S. Function of MYB domain transcription factors in abiotic stress and epigenetic control of stress response in plant genome. Plant Signal Behav, 2016, 11 (1): e1117723. DOI:10.1080/15592324.2015.1117723 |
| [11] | Chen L, Song Y, Li S, et al. The role of WRKY transcription factors in plant abiotic stresses. Biochim Biophys Acta, 2012, 1819 (2): 120–128. DOI:10.1016/j.bbagrm.2011.09.002 |
| [12] | Nakashima K, Takasaki H, Mizoi J, et al. NAC transcription factors in plant abiotic stress responses. Biochim Biophys Acta, 2011, 1819 (2): 97–103. |
| [13] | Sakuma Y, Liu Q, Dubouzet JG, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration-and cold-inducible gene expression. Biochemical and Biophysical Research Communications, 2002, 290 (3): 998–1009. DOI:10.1006/bbrc.2001.6299 |
| [14] | Chen J, Xia X, Yin W. Expression profiling and functionalcharacterization of a DREB2-type gene from Populus euphratica. Biochemical and Biophysical Research Communications, 2009, 378 (3): 483–487. DOI:10.1016/j.bbrc.2008.11.071 |
| [15] | Zhao T, Liang D, Wang P, et al. Genome-wide analysis and expression profiling of the DREB transcription factor gene family in Malus under abiotic stress. Mol Genet Genomics, 2012, 287 (5): 423–436. DOI:10.1007/s00438-012-0687-7 |
| [16] | Egawa C, Kobayashi F, Ishibashi M, et al. Differential regulation of transcript accumulation andalternative splicing of a DREB2 homolog under abiotic stress conditions in common wheat. Genes and Genetic Systems, 2006, 81 (2): 77–91. DOI:10.1266/ggs.81.77 |
| [17] | Yang Y, Wu J, Zhu K, et al. Identification and characterization of two chrysanthemum (Dendronthema × moriforlium) DREB genes, belonging to the AP2/EREBP family. Mol Biol Rep, 2009, 36 (1): 71–81. DOI:10.1007/s11033-007-9153-8 |
| [18] | Zhao H, Bughrara SS. Isolation and characterization of cold-regulated transcriptional activator LpCBF3 gene from perennial ryegrass (Lolium perenne L.). Mol Genet Genomics, 2008, 279 (6): 585–594. DOI:10.1007/s00438-008-0335-4 |
| [19] | Sun S, Yu JP, Chen F, et al. TINY, a dehydration-responsive element (DRE)-binding protein-like transcription factor connecting the DRE-and ethylene-responsive element-mediated signaling pathways in Arabidopsis. J Biol Chem, 2008, 283 (10): 6261–6271. DOI:10.1074/jbc.M706800200 |
| [20] | Wei G, Pan Y, Lei J, et al. Molecular cloning, phylogenetic analysis, expressional profiling and in vitro studies of TINY2 from Arabidopsis thaliana. J Biochem Mol Biol, 2005, 38 (4): 440–446. |
| [21] | Huang B, Liu JY. Cloning and functional analysis of the novel gene GhDBP3 encoding a DRE-binding transcription factor from Gossypium hirsutum. Biochim Biophys Acta, 2006, 1759 (6): 263–269. DOI:10.1016/j.bbaexp.2006.04.006 |
| [22] | Kizis D, Pagès M. Maize DRE-binding proteins DBF1 and DBF2 are involved in rab17 regulation through the drought-responsive element in an ABA-dependent pathway. Plant J, 2002, 30 (6): 679–689. DOI:10.1046/j.1365-313X.2002.01325.x |
| [23] | 黄方, 何慧, 迟英俊, 等. 大豆GmTINY1基因的克隆与表达分析. 作物学报, 2009, 35(12): 2174–2179. |
| [24] | Wilson K, Long D, Swinburne J, et al. A dissociation insertion causes a semidominant mutation that increases expression of TINY, an Arabidopsis gene related to APETALA2. Plant Cell, 1996, 8 (4): 659–671. DOI:10.1105/tpc.8.4.659 |
| [25] | Liu XQ, Liu CY, Guo Q, et al. Mulberry transcription factor MnDREB4A confers tolerance to multiple abiotic stresses in transgenic tobacco. PLoS One, 2015, 10 (12): e0145619. DOI:10.1371/journal.pone.0145619 |
| [26] | Wei S, Du Z, Gao F, et al. Global transcriptome profiles of 'Meyer' zoysiagrass in response to cold stress. PLoS One, 2015, 10 (6): e0131153. DOI:10.1371/journal.pone.0131153 |
| [27] | 冯勋伟, 才宏伟. 结缕草CBF基因的同源克隆及其转基因拟南芥的抗寒性验证. 作物学报, 2014, 40(9): 1572–1578. |
| [28] | Wang Z, Zhang F, Xuan JP, et al. Isolation and expression profiles of the ZjDREB1 gene encoding a DRE-binding transcription factor from zoysiagrass Zoysia japonica. Journal of Horticultural Science and Biotechnology, 2012, 87 (1): 77–83. DOI:10.1080/14620316.2012.11512834 |
| [29] | 可祥, 农钧琇, 石大林, 等. 日本结缕草'胶东青'DREB2. 2基因克隆及表达模式研究. 生物技术通报, 2016, 32(1): 115–123. |
| [30] | Murray MG, Thompson WF. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Res, 1980, 8 (19): 4321–4325. DOI:10.1093/nar/8.19.4321 |
| [31] | Lata C, Prasad M. Role of DREBs in regulation of abiotic stress responses in plants. J Exp Bot, 2011, 62 (14): 4731–4748. DOI:10.1093/jxb/err210 |
| [32] | Agarwal PK, Agarwal P, Reddy MK, et al. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants. Plant Cell Rep, 2006, 25 (12): 1263–1274. DOI:10.1007/s00299-006-0204-8 |
| [33] | Chen Y, Yang J, Wang Z, et al. Gene structures, classification, and expression models of the DREB transcription factor subfamily in Populus trichocarpa. The Scientific World Journal, 2013, 2013 : 954640. |
| [34] | Tian XH, Li XP, Zhou HL, et al. OsDREB4 genes in rice encode AP2-containing proteins that bind specifically to the dehydration-responsive element. Acta Botanica Sinica, 2005, 47 (4): 467–476. |
| [35] | Medina J, Bgarues M, Terol J, et al. The Arabidopsis CBF gene family is composed of three genes encoding AP2 domain containing proteins whose expression is regulated by low temperature but not by ABA or dehydration. Plant Physiology, 1999, 119 (2): 463–470. DOI:10.1104/pp.119.2.463 |
| [36] | 谢登雷, 崔江慧, 常金华. 高粱中SbDREB2基因的克隆与表达分析. 作物学报, 2013, 39(8): 1352–1359. |
| [37] | Allen MD, Yamasaki K, Ohme-Takagi M, et al. A novel mode of DNA recognition by a β-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA. EMBO J, 1998, 17 (18): 5484–5496. DOI:10.1093/emboj/17.18.5484 |
| [38] | Hao D, Ohme-Takagi M, Sarai A. Unique mode of GCC box recognition by the DNA-binding domain of ethylene responsive element-binding factor (ERF domain) in plants. J Biol Chem, 1998, 273 (41): 26857–26861. DOI:10.1074/jbc.273.41.26857 |






