2. 中国农业科学院研究生院,北京 100081
2. Graduate School of Chinese Academy of Agricultural Science, Beijing 100081
烟草(Nicotiana tabacum)目前是国内一种重要的经济作物。培育优质、低害、抗病抗逆、适产的烟草品种是目前烟草育种的主要目标[1]。随着分子生物学的快速发展和转基因技术的逐渐成熟,转基因技术已成为烟草育种的重要方法[2]。近年来,随着Crispr-Cas9技术的发展,在烟草基因组中定点突变一个或多个基因甚至是整个基因家族成为可能。该技术能够对目标性状进行精确改良,通过筛选得到稳定表达转基因株系,不仅能够缩短作物的育种周期,还能够解决传统育种方法不易解决的问题,如有害性状与优良性状的紧密连锁不易打破分离[3]。这给烟草的转基因育种带来了新的机遇。
为了降低假阳性的存在,在抗性培养基上长出的转基因苗还需进行进一步的检验。目前,多采用简便快捷,灵敏度高的PCR方法进行这一检测[4]。而要进行PCR检测则首先需对被检测样品进行DNA提取。目前,在众多植物种类中应用最广泛的提取DNA的方法是采用商业化的试剂盒或是基于CTAB/SDS裂解后的酚:氯仿抽提法[5, 6]或者是由此为基础衍生而来的改良法。然而,这些方法都需要在液氮或是冷冻干燥条件下将样品粉碎,紧接而来的是很多繁琐的步骤进行DNA的纯化。这些方法不仅使用了很多有害试剂和昂贵的试剂仪器,并且使用的植物组织多,步骤繁琐费时,因此当需要检测的转基因植株较多时,这些方法显得不太适用。在植物领域也有一些快速提取DNA的方法报道[7-15]。然而,所有这些建立在碱液裂解基础上的快速提取方法模板在进行PCR之前都需要进行加热/煮沸或者加入中和液(TE或者Tris-HCl)进行中和。这一些额外的步骤显然会增加样品之间的交叉污染,样品多时也会耗费大量时间。
随着烟草基因组测序的完成和众多烟草突变体库的建立,突变体的筛选以及烟草转基因的研究势必迅速发展。而这些研究则需要提取大量烟草单株的基因组进行PCR扩增来筛选目的单株。因此,本实验以前人的研究结果为基础,对NaOH碱裂解法提取烟草基因组进行改进,使其在进行大量样品的处理时更加简便、有效,旨为转基因烟草分子育种的实际应用创造良好的条件。
1 材料与方法 1.1 材料 1.1.1 植物材料烟草转基因植株采用的是通过Crispr-Cas9基因组编辑技术对烟草两个木糖基转移酶基因Ntab0067020和Ntab0104030进行编辑的T0代植株,对照植株为非转基因植株K326;该实验用到的甜高粱由本实验室保存;该实验用到的棉花、小麦、玉米和水稻材料由山东省农科院棉花研究室提供。
1.1.2 主要试剂与仪器1 mol/L,0.5 mol/L,0.25 mol/L,0.1 mol/L,0.05 mol/L,0.01 mol/L和0.001 mol/L的NaOH溶液;SDS提取方法参考Edwards(1991)[5],所用到SDS,Tris等试剂均从青岛塞恩斯试剂公司订购;DNA提取试剂盒购自北京天根生化科技有限公司;2×Taq PCR Mix购自北京全式金生物技术有限公司(2×EasyTaq PCR SuperMix+dye,AS111)、青岛擎科梓熙生物技术有限公司(2×TSINGKE Master Mix,TSE004)、Thermo Scientific公司(2×DreamTaq Green PCR Master Mix,K1081)和青岛生物科技MDBio公司(2×PCR Master Mix,P002);PCR扩增仪T100 Thermocycle(Bio-Rad);自动磨样机(Qiagen TissueLyser Ⅱ)等实验室常规仪器。
1.2 方法 1.2.1 模板制备一个直径0.25 mm的钢珠和250 μL提取液事先加入2 mL的离心管中,然后取大约10 mm2的植物叶片组织放入。在磨样机上以23 Hz频率磨样2 min,1 μL上清即可直接作为模板进行PCR反应。
1.2.2 引物设计本实验所用到的引物均用在线设计网站Oligo Calc(http://biotools.nubic.northwestern.edu/OligoCalc.html)进行设计,引物名称和序列见表 1。引物均在青岛擎科梓熙生物技术有限公司合成。
PCR扩增体系采用20 μL反应体系,包括2× PCR Mix 10 μL,前引物和后引物各6 pmol,DNA模板1 μL,用ddH2O补齐体积。PCR扩增程序如下:94℃预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸1 min,36个循环;最后72℃延伸10 min。取6 μL PCR产物在150 V电压,1.5%琼脂糖凝胶中电泳15 min检测扩增情况。凝胶用EB进行染色,凝胶成像系统拍照分析。
1.2.4 NaOH溶液浓度筛选本实验不经加热煮沸及中和等步骤,采用上清直接作为模板进行PCR扩增,因此需对碱提取液的浓度进行筛选。取烟草品种K326的叶片放入不同浓度梯度的NaOH提取液中(0.001-1 mol/L共7个浓度梯度),用磨样机打碎,取1 μL上清液作为模板,用引物Ntab0067020-SF/ Ntab0067020-SR进行PCR扩增,筛选合适的NaOH浓度。
1.2.5 适宜NaOH浓度提取液在不同品牌PCR Mix中的扩增稳定性将烟草品种K326的叶片在适宜NaOH浓度提取液中磨碎,取1 μL上清液作为模板,使用购自上述(1.1.2)4家公司的PCR Mix用引物Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR进行扩增,检验筛选到的适宜NaOH浓度提取液在不同品牌PCR Mix中的扩增稳定性。
1.2.6 本实验方法与传统方法扩增结果的比较烟草品种K326叶片分别用SDS和本实验方法进行DNA提取,各取1 μL DNA溶液作为模板,用引物Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR及另外两个半乳糖基转移酶基因Ntab0733630和Ntab0774310的特异引物Ntab0733630-SF/Ntab0733630-SR和Ntab0774310-SF/Ntab0774310-SR进行PCR扩增,进行结果比较。
1.2.7 本实验方法在鉴定烟草转基因植株上的应用通过Crispr-Cas9技术编辑的两个烟草木糖基转移酶基因Ntab0067020和Ntab0104030的阳性植株已通过PCR产物连接TA克隆载体,对单菌落进行挑菌测序得到验证。本实验采用两个基因的特异引物Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR,采用NaOH提取法获得模板对可能存在的编辑区段扩增进行PCR扩增,获得的DNA片段进行回收纯化,直接测序,进行结果比对,验证该方法在烟草转基因植株鉴定工作中的适用性。
1.2.8 本实验方法对其他作物进行PCR扩增小麦、甜高粱、烟草、棉花、水稻和玉米分别用试剂盒和本实验方法进行DNA提取,用各自的ACTIN基因引物进行PCR扩增,对扩增结果进行分析比较,验证本实验方法在其他作物上应用的可能性。所用引物,见表 1。
2 结果 2.1 适宜NaOH提取液浓度筛选如图 1所示,0.01 mol/L-0.1 mol/L之间的NaOH溶液制备的DNA模板均能扩增出条带,说明NaOH溶液粗提物可以直接作为PCR反应的模板。0.001 mol/L的NaOH溶液提取的模板虽然也能扩增出条带,但条带亮度较弱,而高于0.25 mol/L(包含0.25 mol/L)的NaOH溶液制备的DNA模板则不能扩增出条带。从扩增效果和节约试剂的角度考虑,本实验选择0.01 mol/L的NaOH溶液作为提取液进行实验。
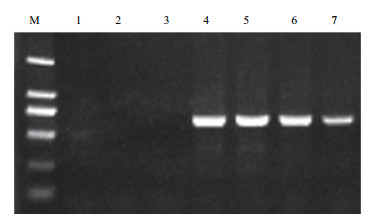
|
| 图 1 不同浓度NaOH溶液提取模板PCR结果电泳图 M:Trans2K DNA Marker;1:1 mol/L NaOH溶液;2:0.5 mol/L NaOH溶液;3:0.25 mol/L NaOH溶液;4:0.1 mol/L NaOH溶液;5:0.05 mol/L NaOH溶液;6:0.01 mol/L NaOH溶液;7:0.001 mol/L NaOH溶液 |
如图 2所示,本实验室及所内其他实验室常用的4种不同品牌PCR Mix对0.01 mol/L NaOH浓度制备的K326模板用引物Ntab0067020-SF/Ntab00670-20-S和Ntab0104030-SF/Ntab0104030-SR进行扩增,4种PCR Mix均能扩增出明亮单一的条带,扩增结果之间没有明显的差异。

|
| 图 2 NaOH提取法在不同品牌PCR Mix中扩增反应稳定性电泳图 M:Trans2K DNA Marker;1:全式金AS111 PCR Mix;2:Thermo Scientific K1081 PCR Mix;3:擎科梓熙TSE004 PCR Mix;4:MDBio P002 PCR Mix,A:引物Ntab0067020-SF/Ntab0067020-S扩增结果;B:引物Ntab0104030-SF/Ntab0104030-SR扩增结果 |
对用传统SDS方法提取的DNA作为模板和NaOH法提取的DNA模板进行PCR扩增的结果进行比较。如图 3所示,4个糖基转移酶基因特异引物Ntab0733630-SF/Ntab0733630-SR、Ntab0774310-SF/Ntab0774310-SR、Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR在SDS法和NaOH法提取的DNA模板上进行扩增均能扩增出条带清晰,明亮的条带。两种方法制备的模板PCR扩增结果没有显著差异。
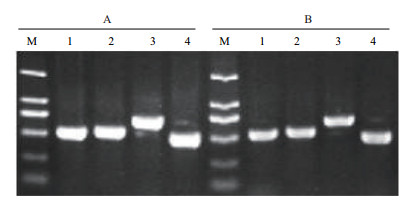
|
| 图 3 SDS方法与NaOH提取法扩增结果对比电泳图 M:Trans2K DNA Marker;1:Ntab0733630-SF/Ntab0733630-SR扩增结果;2:Ntab0774310-SF/Ntab0774310-SR扩增结果;3:Ntab0067020-SF/Ntab00-67020-SR扩增结果;4:Ntab0104030-SF/Ntab0104030-SR扩增结果,A:SDS方法提取DNA模板;B:NaOH方法提取DNA模板 |
本实验对实验室已经确认的两个通过Crispr-Cas9技术编辑的烟草木糖基转移酶基因Ntab0067020和Ntab0104030的阳性植株在编辑位点附近设计了两个基因的特异引物Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR,采用NaOH提取法获得模板进行PCR扩增、测序验证。如图 4-A、4-B所示,引物Ntab0067020-SF/Ntab0067020-SR和Ntab0104030-SF/Ntab0104030-SR在对照和转基因植株上均能扩增出明亮单一的条带。对PCR产物直接进行测序,测序结果如图 4-C、4-D所示,以A1、A3、A7、A8和B1、B3、B7和B8植株的测序结果为例,A3和B3植株为已知转入Crispr-Cas9载体但未能在基因组上成功编辑的植株,基因特异引物扩增测序结果分别与对照A1和B1一致;而A7、A8和B7、B8是在基因组上成功编辑的植株,可以看到在这4株植株上扩增的片段在各自的靶位点上都进行了编辑,存在碱基的插入/缺失,这与我们之前的鉴定结果一致。说明本实验方法提取的DNA模板可以用来对通过Crispr-Cas9技术编辑的转基因植株进行阳性苗的鉴定。
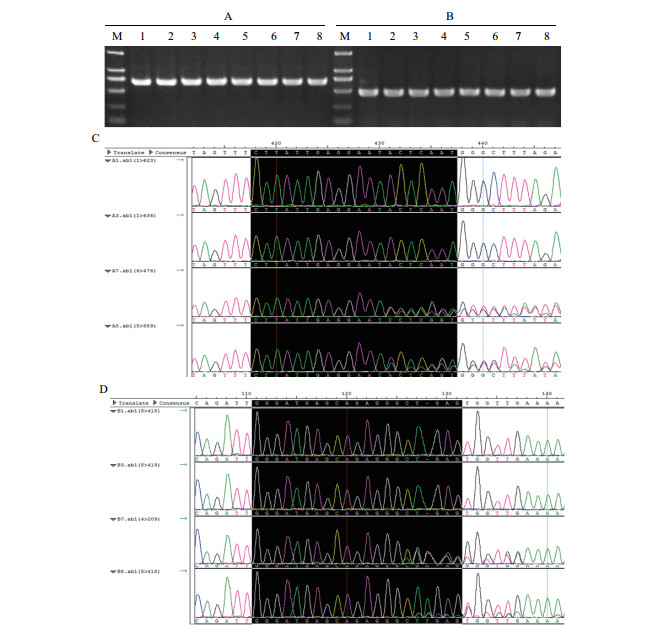
|
| 图 4 NaOH提取法在烟草转基因植株鉴定中的应用 M:Trans2K DNA Marker;1,2:非转基因K326对照;3-6:Crispr-Cas9载体阳性但gDNA未编辑植株;7,8:Crispr-Cas9载体阳性且gDNA编辑植株。A:Crispr-Cas9技术编辑Ntab0067020基因植株;B:Crispr-Cas9技术编辑Ntab0104030基因植株;C:A1、A3、A7和A8 PCR产物测序结果;D:B1、B3、B7和B8 PCR产物测序结果,黑色阴影显示的为Crispr-Cas9技术编辑的基因靶点序列 |
如图 5所示,对小麦、高粱、烟草、棉花、水稻及玉米等作物的幼叶用试剂盒及NaOH两种方法制备的模板进行ACTIN基因的扩增。结果表明,通过试剂盒方法制备的模板能在5种作物上扩出条带,而通过NaOH方法制备的模板在棉花中的扩增失败,但在其他4种作物上均能够扩增出条带,且与试剂盒扩增的效果基本一致,在烟草和水稻中甚至能扩增出长达1.5 kb的片段。

|
| 图 5 NaOH提取法在其他作物上扩增结果电泳图 M:Trans2K DNA Marker;1:小麦;2:高粱;3:烟草;4:棉花;5:水稻;6:玉米。A:试剂盒法提取DNA;B:NaOH法提取DNA |
本研究的主要目的是对碱裂解提取植物基因组的方法进行改进,并应用到烟草中以适用于大量转基因烟草阳性株的鉴定工作,节约时间成本。本研究所改进的DNA提取方法使用仪器少,样品消耗少,简单,快速,成本低。粗提上清液可以直接用来作为PCR反应的模板,能够有效的对烟草转基因植株进行鉴定。利用此方法进行DNA模板制备,一个工作人员在1 h之内就可以提取至少600个样品,提高了工作效率。另外,此方法只利用大约10 mm2的植物材料就可以提取足够200次PCR反应的模板溶液,大大减少了对植物材料的浪费。因此,筛选可以在植株生长的早期进行,加速实验进程。
碱裂解法快速提取基因组DNA的方法在拟南芥、大麦、大豆、甘蔗、水稻、玉米等植物中已经有文献报道[7, 10, 12-17]。与这些已经报道的方法相比,本方法最大的改进是上清液不经加热或中和就可以直接用来作为模板,这样尽最大的可能节省了时间降低了费用并且还能有效的避免不同样品之间的交叉污染。0.01 mol/L NaOH的pH值大约在12.93[9],我们通过对市面上4家不同公司的PCR Mix产品进行PCR扩增的稳定性实验(图 2)表明,1 μL 0.01 mol/L NaOH提取液不会对20 μL的PCR反应体系造成影响,实验结果依然稳定、可靠。但是我们注意到高于0.25 mol/L(包含0.25 mol/L)NaOH溶液制备的模板直接用来进行PCR扩增时失败(图 1),这可能是由于高浓度的NaOH溶液超出了PCR Mix中Tris-HCl缓冲液的缓冲能力,造成PCR结果失败。
虽然传统的SDS提取法和试剂盒提取的DNA量多质优,但是PCR技术对DNA质量的要求并不高,只需要一点模板的存在就可以成功扩增[7]。我们的实验结果证明了此方法提取的DNA在烟草及其他作物,如小麦、高粱、玉米中能够稳定的扩增,实验结果可靠、稳定,在烟草和水稻中甚至能扩增出长达1.5 kb的片段(图 3,图 5),并且在烟草中扩增出的PCR产物可以直接用来进行测序分析(图 4)。因此,此方法可以在这些作物中得到广泛的应用。但是用此方法制备的模板却不能在棉花中成功扩增。Xin等[16]在快速法(有碱提取液煮沸中和的步骤)提取扩增实验中也得到了类似的结果,烟草和高粱组织制备模板后在PCR混合体系中加与不加杂质抑制剂BSA和PVP都能扩增出条带,但是棉花组织却必须加入BSA和PVP才能够成功扩增。Burr等[18]报道超过1 mm2的植物材料进行粗提时,其中的叶绿素等杂质会抑制PCR反应,或许棉花叶片组织中的一些物质能够抑制Taq DNA聚合酶的活性,从而影响扩增效果。综上所述,本研究改进的烟草基因组DNA碱提取方法不仅能够对转基因烟草进行检测,对在其他作物上的应用也具有借鉴意义。
4 结论本研究改进的NaOH法制备PCR扩增模板不需要液氮、不需要昂贵的仪器、无需离心、无需更换离心管、没有加热煮沸中和等步骤,粗提物可直接用作PCR反应的模板。进行大量样品DNA模板制备时能够在最大程度上节省时间和实验花费并减少样品之间的交叉污染。此方法用来检测转基因烟草阳性株,实验结果准确,稳定,重复性好。此外,此方法的所有步骤都在室温操作并且10 mm2的叶子制备的DNA模板就足够200余次PCR反应使用,可减少对样品的浪费并在植株生长早期即可进行检测,加速实验进程。此改进方法提取的烟草基因组DNA能够用于对转基因烟草进行阳性植株鉴定。
| [1] | 尚志强. 中国烟草育种进展及发展对策. 内蒙古农业科技, 2010(5): 1–3. |
| [2] | 李彦平, 丁燕芳, 焕孙, 等. 我国烟草育种现状及思考. 河南农业科学, 2010(9): 148–156. |
| [3] | 李君, 张毅, 陈坤玲, 等. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术. 遗传, 2013, 35(11): 1265–1273. |
| [4] | 郭斌, 祁洋, 尉亚辉. 转基因植物检测技术的研究进展. 中国生物工程杂志, 2010(2): 120–126. |
| [5] | Edwards K, Johnstone C, Thompson C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Research, 1991, 19 (6): 1349. DOI:10.1093/nar/19.6.1349 |
| [6] | Murray MG, Thompson WF. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Res, 1980, 8 (19): 4321–4325. DOI:10.1093/nar/8.19.4321 |
| [7] | Wang H, Qi M, Cutler AJ. A simple method of preparing plant samples for PCR. Nucleic Acids Research, 1993, 21 (17): 4153. DOI:10.1093/nar/21.17.4153 |
| [8] | Paris M, Carter M. Cereal DNA: a rapid high-throughput extraction method for marker assisted selection. Plant Molecular Biology Reporter, 2000, 18 (4): 357–360. DOI:10.1007/BF02825063 |
| [9] | Shi SR, Datar R, Liu C, et al. DNA extraction from archival formalin-fixed, paraffin-embedded tissues: heat-induced retrieval in alkaline solution. Histochemistry and cell Biology, 2004, 122 (3): 211–218. DOI:10.1007/s00418-004-0693-x |
| [10] | von Post R, von Post L, Dayteg C, et al. A high-throughput DNA extraction method for barley seed. Euphytica, 2003, 130 (2): 255–260. DOI:10.1023/A:1022863006134 |
| [11] | Collard B, Das A, Virk P, et al. Evaluation of 'quick and dirty'DNA extraction methods for marker-assisted selection in rice (Oryza sativa L.). Plant Breeding, 2007, 126 (1): 47–50. DOI:10.1111/pbr.2007.126.issue-1 |
| [12] | 程文, 夏正俊, 冯献忠, 等. 一种快速、无损大豆种子DNA提取方法的建立和应用. 植物学报, 2016(1): 68–73. |
| [13] | 崔学强, 张树珍, 冯翠莲. 一种简易的甘蔗叶组织PCR模板制备方法. 生物技术通报, 2016(3): 58–62. |
| [14] | 桑贤春, 何光华, 张毅, 等. 水稻PCR扩增模板的快速制备. 遗传, 2003(6): 705–707. |
| [15] | 吴明生, 云晓敏, 宋歌, 等. 转基因玉米种子快速筛查方法研究与应用. 生物技术通报, 2013(1): 102–106. |
| [16] | Xin Z, Velten JP, Oliver MJ, et al. High-throughput DNA extraction method suitable for PCR. Biotechniques, 2003, 34 (4): 820–827. |
| [17] | Karakousis A, Langridge P. A high-throughput plant DNA extraction method for marker analysis. Plant Molecular Biology Reporter, 2003, 21 (1): 95–95. DOI:10.1007/BF02773402 |
| [18] | Burr K, Harper R, Linacre A. One-step isolation of plant DNA suitable for PCR amplification. Plant Molecular Biology Reporter, 2001, 19 (4): 367–371. DOI:10.1007/BF02772835 |





