2. 国家林业局资源昆虫培育与利用重点实验室,昆明 650224
2. Key Laboratory of Cultivation and Utilization of Resource Insects, State Forestry Administration, Kunming 650224
基因组编辑是指定点改造基因组,得到预期的生物体基因组序列从而发生遗传改变的技术,主要是进行DNA序列的敲除、插入、定点突变和组合编辑等,从而实现基因功能与调控元件的系统研究[1]。基因组编辑技术日益成为生物学研究的热点,目前基因组编辑技术主要包括巨核酶技术、锌指核酸酶技术、转录激活因子样效应因子核酸酶技术及CRISPR相关核酸酶技术[2]。它们在基因工程中已经得到了诸多成功的应用,然而上述传统的基因组编辑技术存在明显的不足。例如,锌指核酸梅(zinc-finger nucleases,ZFN)和转录激活因子样效应因子核酸梅(transcription activator-like effector nucleases,TALEN)核酸酶技术原理几乎是一样的,而一个新的ZFN和TALEN嵌合蛋白均需改造才能识别靶序列,这就使得两种基因组编辑技术的广泛推广应用受阻,但CRISPR相关核酸酶技术只需要一小段引导RNA(guide RNA、gRNA)就能识别特定的DNA[2],且一个新的位点只需要一个新的gRNA,极大的简化了基因组编辑的过程,扩大靶位点的选择,具有更大的潜力。CRISPR系统也可以应用于靶向基因突变和基因修正,可以实现大片段DNA的缺失以及复合基因编辑。
本文将针对CRISPR/Cas系统的结构、分类、原理以及目前具有突破性研究并迅速发展的TypeⅡ CRISPR/Cas系统和TypeⅢ CRISPR/Cas系统的突破性研究做较为详尽的综述。
1 CRISPR/Cas的基因座结构CRISPR/Cas的基因座结构相对简单(图 1),对于一个有效的CRISPR/Cas系统而言,通常包含两个部分:第一部分是位于基因组中正向重复序列和来源于外源DNA的间隔序列组成3'端CRISPR基因座,对于重复序列而言在长度和序列上几乎一致,在不同的物种之间长度范围在21-47 bp,平均在32 bp[3, 4];间隔序列在长度上是一致的,长度范围在20-72 bp,但在序列上表现出多样性[4, 5]。亲缘关系相近的物种具有相似的重复序列,但是在全部的细菌和古细菌序列中间隔序列和重复序列都表现出极大的差异[6]。第二部分是位于CRISPR基因座5'端成簇的Cas基因,主要包括一些核酸酶、解旋酶、聚合酶和RNA结合蛋白等,主要参与CRISPR/Cas系统介导的免疫过程[7]。

|
| 图 1 CRISPR/Cas基因座结构 |
目前对于CRISPR/Cas系统而言主要分为3种类型:TypeⅠ、TypeⅡ、TypeⅢ三种不同类型[8],从另一方面也反映出在自然界中CRISPR/Cas系统具有多态性。
TypeⅠCRISPR/Cas系统在细菌和古细菌中被发现,包含6种不同的Cas蛋白,其中在干涉反应中最重要和最显著的是Cas3蛋白,它包含一个HD磷酸化水解酶结构域和一个类DExH解旋酶结构域,这两个结构域在ATP和Mg2+存在的情况下能够解开dsDNA(解旋酶结构域)和切割ssDNA(HD核酸结构域)[9]。
TypeⅡ CRISPR/Cas系统仅仅在细菌的基因组中发现[9],其中Cas9是分子量很大的多功能蛋白,参与crRNAs的成熟和随后的干涉反应[10]。Cas9蛋白包含有两个亚基,在Mg2+存在条件下,其中McrA/HNH切割与crRNA互补的DNA链,而类RuvC切割非互补链[9]。
Type Ⅲ CRISPR/Cas系统主要在古细菌中发现,包括type Ⅲ-A和type Ⅲ-B两种类型,两者的差别主要是A型干扰的靶标是mRNA,B型干扰的靶标是DNA[11],其中type Ⅲ-B系统与其他1或2种CRISPR亚单位相结合[9]。
type Ⅲ-B系统特征性蛋白是Cas10蛋白,而Cas10蛋白具有RNA活性参与crRNA的成熟和剪切入侵外源DNA[9, 10]。
最近几年,CRISPR/Cas技术因其相对较为简单,而得到较为迅速的发展,如在作物育种、基因治疗以及动物建模等领域都已得到广泛的应用[12]。随着测序技术的不断发展,越来越多的细菌和古细菌的基因组信息被解密,为后续CRISPR/Cas系统的改造和应用提供更加有力的指导。
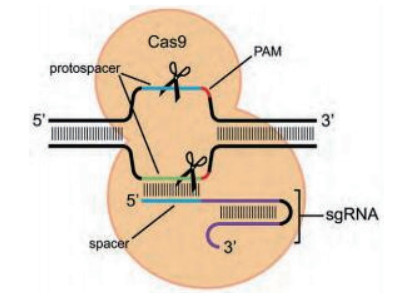
3 CRISPR/Cas作用机制 3.1 TypeⅡ CRISPR/Cas系统基本原理一个简易的TypeⅡ CRISPR/Cas系统必定要含有Cas9和gRNA,Cas9核酸酶具有改造crRNA和破坏双链DNA的双重功能[13],位于gRNA 5'端的20个左右的碱基决定CRISPR/Cas9系统在应用时Cas9核酸酶特异性切割的部位,主要负责决定CRISPR/Cas9系统的特异性。
基本原理(图 2):首先,Cas9蛋白与gRNA形成复合体;其次,上述复合体切割与gRNA的spacer互补的基因组DNA序列;随后造成双链DNA的损伤;最后通过体内的NHEJ或HR修复机制将引入基因突变[14]。
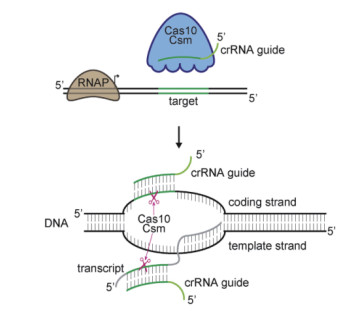
3.2 TypeⅢ CRISPR/Cas系统基本原理TypeⅢ CRISPR/Cas系统包含的两种类型中以A型研究较为突出,这里以type Ⅲ-A为例。A型干扰的mRNA首先转录形成DNA,Cas10-Csm核糖核蛋白体,在crRNA的引导下对与crRNA互补的DNA双链或单链进行切割[16],从而造成DNA的损伤(图 3)。
4 CRISPR/Cas系统之间的比较TypeⅡ CRISPR/Cas系统在最近几年研究火热,其突出的优点在于结构简单,系统容易设计,仅需要较短的gRNA和Cas9核酸酶就可以完成对特定靶基因的人工编辑,但是该系统存在较严重的脱靶把效应,使其广泛应用受到限制。
TypeⅢ CRISPR/Cas系统与其他两种类型相比存在自己独有的亮点:(1)TypeⅢ CRISPR/Cas系统只有在靶DNA转录的情况下才发挥功能。(2)Cas10-Csm复合物不仅具有执行引导RNA切割DNA的能力,也具有切割RNA的能力。这些突出的优点揭示了一种全能型的CRISPR免疫系统[16]。
5 CRISPR/Cas系统最新突破性研究随着生物学研究的迅猛发展,CRISPR/Cas系统也出现令人难以置信的新突破,为基因组编辑技术的广泛应用开辟了更为广阔的道路。
5.1 技术创新真核细胞具有较为复杂的转录调控系统,基因组中不同的位点采用不同的调控方式,利用这一原则,通过延长引导RNA使其包含效应蛋白的结合位点,从而开发了一种CRISPR支架RNA(scRNA),利用scRNA能够开发多基因转录程序,在控制一部分基因激活的同时又抑制了另一部分基因[17]。这一突破为基因表达程序的构建提供了强有力的技术支撑,也为细胞重组和研究代谢途径等开辟新的道路。
在TypeⅡ CRISPR/Cas系统中Cas9具有重要的作用,初始的Cas9大多都是来源于酿脓链球菌即SpCas9,但是在金黄色酿脓葡萄球菌中发现了一种新的Cas9即SaCas9,在相关实验发现体内调节基因组编辑效率方面SaCas9要比SpCas9高出很多[18]。一种高效的Cas9核酸酶的发现攻克了基因组编辑面临的一大挑战,在东京大学相关研究设计了一种光激活的Cas9,能够光控CRISPR/Cas9基因组编辑,在空间和时间上更好地控制RNA引导的核酸酶的活性[19];Sato实验室也相继推出了光活化系统来切割目标DNA序列,进行光活化的基因组修饰,他们采用每个Cas9片段与一个二聚化的结构域相融合,创建了光活化的Cas9工具;Alexander Deiters的实验室利用遗传编码的光照技术,在Cas9蛋白中插入硝基苯笼罩的赖氨酸,最终发现这种新型的Cas9系统能够让内源的基因表达沉默。这些技术的革新使人们能够更好的了解复杂的基因网络,也能很好的应用于医学方面。
5.2 疾病研究基因组编辑技术在医学方面的应用也越来越多,如进行基因治疗和构建疾病模型等。
5.2.1 构建疾病模型骨髓瘤是一种进行性肿瘤性疾病,目前对它的致病机理还不是很清楚,因此构建该疾病的模型,对于研究其致病机理和药物筛选有重要的作用。
小鼠的骨髓瘤疾病模型已被构建,研究小组将gRNA和表达Cas9蛋白的慢病毒表达载体构建出简易的TypeⅡ CRISPR/Cas系统,然后将表达5种微小gRNA慢性病毒载体组合在一起,导入小鼠的造血干细胞中,从而形成恶性肿瘤的单克隆;最后,将细胞注射到小鼠体内,结果表明CRISPR/Cas系统在多基因作用的模型建立上是有效的,能够较好的反应人类的疾病[20]。
最近,Broad研究所和麻省理工学院David H.Koch综合癌症研究所的诸多科学家们利用Cas9核酸酶技术在构建的癌症动物模型中系统地“敲除”(关闭)了整个基因组的所有基因,从而揭示出与肿瘤进化和转移紧密相关的部分基因,同时也表明Cas9核酸酶筛选技术也成为在体内系统分析肿瘤进化过程中基因表型的可靠技术[21]。为医学研究和临床治疗肿瘤,提供了新的思路和技术。
5.2.2 基因治疗目前对于HIV-1型艾滋病的治疗尚不存在显著的治疗手段。Ebina等[22]尝试利用CRISPR/Cas9技术对HIV-1感染的T细胞的LTR区进行打靶,结果显示它能有效降低细胞内前病毒的水平;韩英伦等[23]利用CRISPR/Cas9技术对哺乳动物的造血干细胞进行改造,成功遏制了HIV-1病毒的感染作用,与此同时发现,它还能作用HIV-1原病毒的LTR区彻底清除原病毒,对CRISPR/Cas9技术进行改造实验发现它可以识别并激活HIV-1原病毒,如果结合HAART药物,结果能够彻底清除体内的HIV-1原病毒。
EB病毒感染能够引起肿瘤和非肿瘤性疾病,严重危害人类健康,Wang等[24]利用CRISPR/Cas9技术体处理EB病毒潜伏感染的人类淋巴瘤细胞,结果发现有25%感染的细胞被清除,50%的细胞中,EB病毒负荷明显减少。由此可以预测,从人类感染的细胞中完全除去EB病毒,用于治疗相关疾病指日可待。
CRISPR/Cas9技术除了上述在病毒感染疾病基因治疗中取得显著成效以外,对一些遗传性疾病,例如:白内障、地中海贫血、囊性纤维病等基因突变引起的相关疾病研究中也已取得显著成果。
5.3 植物学研究从基因组的角度出发对植物基因进行人工改造从而培育优良品种具有非常重要的意义,由于外源DNA与基因组靶基因发生同源重组效率很低,因此对植物基因组进行人工改造就显得异常困难。受序列的限制以往的CRISPR载体往往是两个载体,也即表达gRNA和Cas9元件位于不同的载体,共转化效率较低,为了克服这一困难,我们可以试想利用同源重组技术和改进后的定点突变技术将基因特异的靶序列构建到通用载体中得到表达特异基因gRNA的入门载体,将其与目标载体同源重组从而构造特异的基因表达载体。
CRISPR/Cas系统在植物学方向的研究很多,但大多局限于研究去修饰和改造一个或多个靶位点。最近,华南农业大学研究人员利用PCR技术扩增形成多重gRNA表达带,再将其组装到CRISPR/Cas9表达载体上,从而发现一种适合于单子叶和双子叶植物,高效且方便的多重基因组编辑的强大的CRISPR/Cas9系统[25],为后续进行植物基因组改造、整合和培育新的品种提供技术支撑。
5.4 昆虫学研究在昆虫方向的研究大多局限于采用CRISPR/Cas9基因编辑技术,目前已经在果蝇、家蚕、埃及伊蚊体内利用CRISPR/Cas9基因编辑技术进行基因敲除和插入研究,为进一步研究基因功能提供新的思路和技术手段。
2014年,Liu等[26]在家蚕细胞中利用CRISPR/Cas9系统进行6个基因的敲除,在敲除Bm702基因的同时实现外源基因在该位点的成功插入;2015年,辛虎虎[27]在家蚕丝腺相关基因Bmsage的研究中采用CRISPR/Cas9系统进行基因的敲除和功能分析取得良好效果;2015年,Kistler等[28]在埃及伊蚊产卵后的3 h内注射gRNA与Cas9,敲除Wtrw基因并插入包含stop位点的ssODN供体片段,采用Cas9核酸酶系统对目的DNA进行切割和HR修复,在埃及伊蚊内插入ECFP阅读框,子代可在荧光显微镜下显示特殊的蓝色,便于形态学观察。利用CRISPR/Cas系统对昆虫基因功能的研究和注释,为人类了解和探究大自然产生深远的影响。
6 结语目前,CRISPR/Cas系统相关的基因组编辑技术已在多个研究领域广泛应用,尽管其中潜在着许多待解决的问题,如TypeⅡ CRISPR/Cas系统中DNA解旋是本身具有解旋能力还是由于DNA复制或转录的过程引发DNA解旋[11],如何确保临床应用中的安全性和有效性等;作为RNA编辑的TypeⅢ CRISPR/Cas系统目前仍有分子机制尚不明确。但作为第三代基因组编辑技术工具无论是从方法上还是实验成本上都是传统基因修饰和第二代基因组编辑技术无法取代的。
基因组编辑技术普遍存在细胞毒性和脱靶效应两大问题,由于CRISPR/Cas9系统自身存在脱靶效应,也使其在推广研究应用中受到或多或少的限制,对于降低TypeⅡ CRISPR/Cas系统的脱靶效应,可以考虑以下几个方面:(1)适当减少Cas9蛋白和gRNA的递送量。(2)对打靶位点进行严谨的筛选和过滤。(3)Cas9n和gRNA配对使用,形成5'端突出的末端。(4)精确设计和控制gRNA的长度以此降低gRNA和非目标序列的结合率。
CRISPR/Cas系统目前多数还处于基础研究,有些深层次的技术还有待于开发和应用,例如TypeⅠCRISPR/Cas系统还未见报道,2015年TypeⅢ CRISPR/Cas系统在Cell上发布有突破性进展,但随着基础学科的不断深入,CRISPR/Cas系统也会得到更为广阔的发展和应用前景。CRISPR/Cas系统的不断优化和提升,对未来医学领域:医学研究与医学临床治疗、药物开发、耐受药物细胞模型的建立与筛选、人源化器官的克隆移植;分子标记领域:药物靶向追踪、活细胞中特定结构基因构象分析;基因组学领域:功能基因研究、高通量功能基因鉴定与筛选、基因组改造与整合,CRISPR/Cas系统都已表现出巨大的应用潜力和研究开发价值。
| [1] | 谢科, 饶力群. 基因组编辑技术在植物中的研究进展与应用前景. 中国生物工程杂志, 2013, 33(6): 99–104. |
| [2] | Gaj T, Gersbach CA, Barbas CF 3rd. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends Biotechnol, 2013, 31 (7): 397–405. DOI:10.1016/j.tibtech.2013.04.004 |
| [3] | Godde JS, Bickerton A. The repetitive DNA elements called CRISPRs and their associated genes: evidence of horizontal transfer among prokaryotes. Mol Evol, 2006, 62 : 718–729. DOI:10.1007/s00239-005-0223-z |
| [4] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8 : 172. DOI:10.1186/1471-2105-8-172 |
| [5] | Mojica FJ, Diez-Villasenor C, Soria E, Juez G. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. Mol Microbiol, 2000, 36 : 244–246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [6] | Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats. Genome Biol, 2007, 8 : R61. DOI:10.1186/gb-2007-8-4-r61 |
| [7] | 常振义, 严维, 刘东风, 等. CRISPR/Cas技术研究进展. 农业生物技术学报, 2015, 23(9): 1196–1206. |
| [8] | 张瑞. 对CRISPR-Cas系统的简单介绍. 农业与技术, 2015, 35(2): 16–17. |
| [9] | Richter H, Randau L, Plagen A. Exploiting CRISPR/Cas: Interference mechanisms and applications. Int J Mol Sci, 2013, 14 : 14518–14531. DOI:10.3390/ijms140714518 |
| [10] | 方锐, 畅飞, 孙照霖, 等. CRISPR/Cas9介导的基因组定点编辑技术. 生物化学与生物物理进展, 2013, 40(8): 691–702. |
| [11] | 刘志国. CRISPR/Cas9系统介导基因组编辑的研究进展. 畜牧兽医学报, 2014, 45(10): 1567–1583. |
| [12] | 郑小梅, 张晓立, 于建东, 等. CRISPR-Cas9介导的基因组编辑技术的研究进展. 生物技术进展, 2015, 5(1): 1–9. |
| [13] | Fu Y, Foden JA, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013, 31 (9): 822–826. DOI:10.1038/nbt.2623 |
| [14] | Wieden heft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea. Nature, 2012, 482 (7385): 331–338. DOI:10.1038/nature10886 |
| [15] | 翟礼嘉, 郭东姝, 张金喆, 等. CRISPR/Cas系统在植物基因组编辑中的应用. 生命科学, 2015, 27(1): 64–70. |
| [16] | Samai P, Pyenson N, Jiang WY, et al. Co-transcriptional DNA and RNA Cleavage during TypeⅢ CRISPR-Cas Immunity. Cell, 2015, 161 : 1164–1174. DOI:10.1016/j.cell.2015.04.027 |
| [17] | Zalatan JG, Lee ME, Almeida R, et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell, 2015, 160 : 339–350. DOI:10.1016/j.cell.2014.11.052 |
| [18] | Ann Ran F, Cong L, Zhang F, et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature, 2015, 520 (7546): 186–191. DOI:10.1038/nature14299 |
| [19] | Nihongak Y, Kawano F, Nakajima T, et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing. Nature Biotechnology, 2015 : 1–7. |
| [20] | Heckl D, Kowalczyk MS, Yudovich D, et al. Generation of mouse models of myeloid malignancy with combinatorial genetic lesions using CRISPR-Cas9 genome editing. Nat Biotechnol, 2014, 32 (9): 941–946. DOI:10.1038/nbt.2951 |
| [21] | Chen SD, Sanjana NE, Zheng KJ, et al. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell, 2015, 160 : 1246–1260. DOI:10.1016/j.cell.2015.02.038 |
| [22] | Ebina H, Misawa N, Kanemura Y, et al. Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus. Sci Rep, 2013, 3 (2510): 1–7. |
| [23] | 韩英伦, 李庆伟. CRISPR/Cas9基因组编辑技术在HIV-1感染治疗中的应用进展. 遗传, 2016, 38(1): 9–16. |
| [24] | Wang J, Quake S R. RNA-guided endonuclease provides a therapeutic strategy to cure latent herpesviridae infection. Proc Natl Acad Sci USA, 2014, 111 (36): 13157–13162. DOI:10.1073/pnas.1410785111 |
| [25] | Ma XL, Zhang QY, Zhu QL, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Mol Plant, 2015, 8 (8): 1274–1284. DOI:10.1016/j.molp.2015.04.007 |
| [26] | Liu Y, Ma S, Wang X, et al. Highiy efficient muitiplex targeted mutagenesis and genomic stucture variation in Bombyx mori cells using CRISPR/Cas9. Insect Biochem Mol Biol, 2014, 49 : 35–42. DOI:10.1016/j.ibmb.2014.03.010 |
| [27] | 辛虎虎. CRISPR/Cas9基因敲除技术在家蚕基因功能研究中的应用[D].杭州:浙江大学, 2015. |
| [28] | Kistler KE, Vosshall LB, Matthews BJ. Genomeengineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep, 2015, 11 (1): 51–60. DOI:10.1016/j.celrep.2015.03.009 |