α-芋螺毒素是烟碱型乙酰胆碱受体(nAChRs)的拮抗剂,而乙酰胆碱结合蛋白(AChBPs)与nAChRs的胞外配体结合结构域同源性较高,AChBPs与nAChRs的药理特性和离子通道激活机制相似,因此AChBPs通常作为研究nAChRs的结构模板[1, 2]。多个AChBPs的x-ray结构已被解析,通过其晶体结构,我们更清楚了解nAChRs的结构。AChBPs有3种,分别是Lymnaea stagnalis、Aplysia californica、Bulinus truncatus,简称为Ls-AChBP、Ac-AChBP、Bt-AChBP,它们结构相类似[3, 4],α-芋螺毒素主要与Ac-AChBP结合。α-芋螺毒素与Ac-AChBP共结晶结构不仅帮助我们剖析nAChRs的生理和病理功能,而且有助于发现药效更好的α-芋螺毒素[4, 5]。
目前已有5个α-芋螺毒素与AChBPs共结晶结构被解析,它们分别是PnIA(A10L D14K)(PDB:2BR8)[5]、ImI(PDB:2C9T,2BYP)[6, 7]、TxIA(A10L)(PDB:2UZ6)[8]、BuIA(PDB:4EZ1)和GIC(PDB:5CO5)[9]与Ac-AChBP共结晶的结构。其中GIC与Ac-AChBP的复合物结构是我们报道的分辨率较高的共结晶结构。α-芋螺毒素GIC是从杀手芋螺(Conus geographus)中发现的作用于α3β2 nAChRs的毒素。α3β2 nAChRs是位于脑部的神经受体,与记忆、认识相关。α-芋螺毒素GIC作用于α3β2 nAChRs的IC50最低浓度为1.1 nmol/L,是目前发现最强作用于α3β2 nAChRs的毒素,这一点使它的结构与功能倍受关注,另外,它与Ac-AChBP结合的IC50值也较低(29 nmol/L),它与Ac-AChBP的共结晶的方法可为其他新型α-芋螺毒素与Ac-AChBP或nAChRs共结晶提供借鉴,为开发治疗疼痛、成瘾、帕金森症、阿尔茨海默氏症和癫痫等与nAChRs相关疾病的药物提供理论依据[9]。
蛋白结构的解析关键是要获得高分辨率的蛋白晶体。要获得高质量的蛋白晶体,必须对蛋白结晶条件进行筛选与优化[10-12]。蛋白结晶过程通常是把沉淀剂(池液)加到蛋白质溶液表面,沉淀剂从蛋白质溶液中吸走水分而引起蛋白结晶。结晶条件是指不同的沉淀剂及其摩尔浓度、pH值等因素。这些条件(因素)有上万种,因此需要利用机器人进行对结晶条件的大量筛选和优化,才能得到分辨率高的晶体。本研究采用正交实验,并利用机器人及HAMPTON RESEARCH试剂盒对α-芋螺毒素GIC与Ac-AChBP共结晶条件进行筛选与优化,以期获得高分辨率的晶体。
1 材料与方法 1.1 材料Ac-AChBP(AChBP from Aplysia californica)基因[13]根据NCBI提供的序列(NM_001204599)设计并由上海生工公司合成,在两端分别引入BamH Ⅰ、Xho Ⅰ酶切位点。大肠杆菌宿主菌DH10Bac,昆虫表达载体pFastBac1,昆虫细胞Sf9(Spodoptera frugiperda),HAMPTON RESEARCH结晶试剂盒PEG/Ion1,PEG/Ion2,Index,Crystal Screen,SaltRX等由本实验室保存。
1.2 方法 1.2.1 蛋白表达与纯化在昆虫细胞中分泌表达Ac-AChBP,分泌表达的培养基上清液经镍柱纯化后,用凝胶色谱进一步纯化。本实验所用凝胶色谱柱是superdex200(GE Healthcare),利用AKTA(GE Healthcare)系统分离纯化,流动相HBS溶液(10 mmol/L Hepes,150 mmol/L NaCl,pH7.2)流速为1 mL/min,将蛋白样品浓缩到1 mL后上样,利用AKTA程序过柱,自动收集样品。将收集样品利用SDS-PAGE检测,并用液氮速冻后置于-80℃保存[13-16]。
1.2.2 α-芋螺毒素GIC的合成与纯化α-芋螺毒素GIC由上海吉尔生化公司合成,并用C18柱(Vydac,Hesperia,CA,USA)经高效液相色谱纯化,收集纯化的样品(与标准品比较达到99.8%)冷冻干燥备用[9]。
1.2.3 Ac-AChBP蛋白结晶将纯化的Ac-AChBP浓缩至约20 mg/mL。使用液-液扩散法在291 k长晶体。具体如下:在96孔板中,用HAMPTON RESEARCH试剂PEG/Ion1,PEG/Ion2,Index,Cystal Screen,Sa-ltRX当池液,用结晶机器人CRYSTAL GRYPHON(Art robbins instrusmets)点样,将浓缩蛋白液与池液按1:1点坐滴,点样量为0.15 μL,在显微镜下观察晶体生长[9]。
1.2.4 α-芋螺毒素与Ac-AChBP共结晶α-芋螺毒素与Ac-AChBP共结晶前,先把它们混合,然后把混合好的溶液去点晶体。已知1 mol Ac-AChBP亚基与1 mol α-芋螺毒素结合(虽然理论上它们按摩尔比1:1结合,但为了使α-芋螺毒素能充分结合到Ac-AchBP中,把α-半螺毒素的量增加了10倍)。利用ExPASy Protparam tool分析Ac-AChBP吸光指数为1.5,其分子量为27 kD,取出Ac-AChBP蛋白储备液(浓度为1.0 mg/mL)1 mL。按以下公式计算Ac-AChBP蛋白亚基储备液的mol数为:

α-芋螺毒素GIC的C端已酰胺化,其分子量为1 610.4 kD,用纯化的α-芋螺毒素GIC重量除以其分子量可得到GIC的摩尔数。把α-芋螺毒素GIC按摩尔比为10:1与Ac-AChBP亚基共混。把混和液在室温放置2 h使其结合,然后13 000 r/min离心15 min,再用凝胶色谱纯化。收集Ac-AChBP与α-芋螺毒素结合的凝胶纯化色谱峰片段,将其浓缩至约20 mg/mL。使用坐滴蒸气扩散法在291 k长晶体,点完晶体后,在显微镜下观察到晶体生长[13-16]。
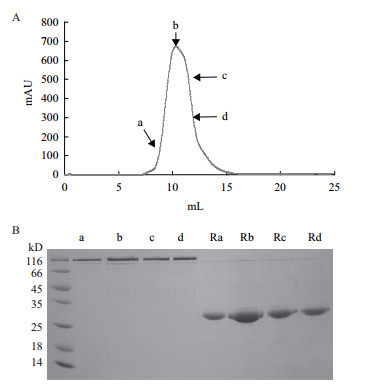
2 结果 2.1 Ac-AChBP的制备在昆虫细胞中分泌表达Ac-AChBP,并用凝胶色谱纯化。Ac-AChBP的凝胶纯化色谱(图 1-A)显示,其出峰位置在12.5 mL,根据Superdex200柱的说明,峰位置为12.5 mL表示约为135 kD的相对分子量。从不同的峰位置a-d收集组分作进一步的SDS-PAGE胶分析(图 1-B)。图 1-B中a-d是Ac-AChBP蛋白非变性的SDS-PAGE胶图,即上样时,没有加还原剂和煮沸样品。Ra-Rd所示是变性的SDS-PAGE胶图,即上样时,加还原剂DDT和煮沸样品5 min。a-d条带在SDS-PAGE胶顶部(图 1-B),显示它们非变性状态是五聚体,因为其是球状蛋白,电泳图只显示分子量为116 kD,比实际线型分子量(135 kD)要小。R a-d所示是变性的Ac-AChBP,其由五聚体变为单体,分子量约为27 kD;正好是表达出来的重组蛋白五聚体分子量的1/5,这一点也说明变性的Ac-AChBP状态为单体。活性状态为五聚体。
|
| 图 1 凝胶色谱(A)和SDS-PAGE(B)分析Ac-AChBP蛋白 A:Ac-AChBP的凝胶色谱图,a-d指不同的出峰位置;B:SDS-PAGE分析图A中不同峰位置的片段a-d |
在研究α-芋螺毒素GIC与Ac-AChBP共结晶条件之前,先研究Ac-AChBP的结晶条件。原因是共结晶中Ac-AChBP是个五聚体的大蛋白,而α-芋螺毒素GIC只结合在Ac-AChBP上的小多肽。因此Ac-AChBP结晶的条件是研究共结晶的前提。Ac-AChBP结晶条件可以借鉴在共结晶中。
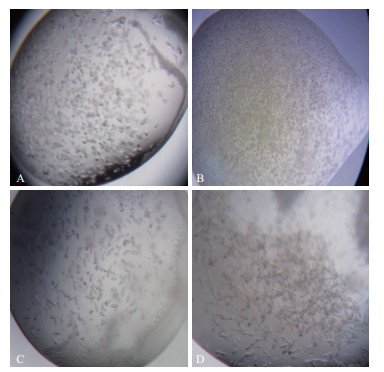
利用机器人初步筛选Ac-AChBP的结晶,选用结晶试剂盒PEG/Ion1,PEG/Ion2,Index,Crystal Screen,Wizard 1,SaltRX均能得到晶体,把得到的晶体送往上海同步辐射光源收集数据,SaltRX的4个条件(表 1和图 2)得到的晶体分辨率较好。在这4个条件中,15号(图 2-A)晶体条件得到的晶体数据最好,分辨率为3 Å。
|
| 图 2 SaltRX试剂盒初步筛选得到晶体 A:SaltRX试剂盒15号条件筛选得到晶体;B:SaltRX试剂盒49号条件筛选得到晶体;C:SaltRX试剂盒67号条件筛选得到晶体;D:SaltRX试剂盒73号条件筛选得到晶体 |
接着对15号晶体条件进行优化,优化实验设计如表 2所示,对36个结晶条件进行实验(两因素,六水平),并把得到的晶体送去收集数据,其中最好的晶体数据分辨率为2.4 Å,其生长条件为:0.8 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.6。
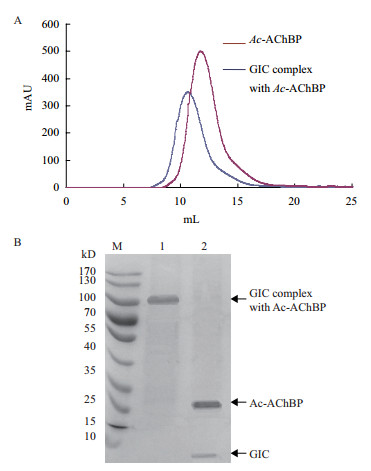
得到2.4 Å的Ac-AChBP晶体后,进一步研究其与α-芋螺毒素共结晶,共结晶前,先把它们共结合,结合的方法按1.2.4所示,α-芋螺毒素与Ac-AChBP结合后,用凝胶色谱纯化。图 3-A蓝线显示Ac-AChBP与α-芋螺毒素GIC结合的凝胶纯化色谱,红线是结合前的Ac-AChBP。图中蓝线比红线分子量大,说明Ac-AChBP已与α-芋螺毒素GIC结合。图 3-B是收集图 3-A蓝线峰组分进行SDS-PAGE胶分析,条带1是活性五聚体Ac-AChBP结合α-芋螺毒素GIC,其分子量显示为100-130 kD之间。而条带2是变性的蓝线组分,变性后,五聚体Ac-AChBP变为单体,分子量为27 kD。变性后,α-芋螺毒素GIC也解离出来,显示在SDS-PAGE胶底部。
|
| 图 3 凝胶色谱(A)、SDS-PAGE胶(B)分析α-芋螺毒素和Ac-AChBP结合 A:α-芋螺毒素结合Ac-AchBP前后凝胶色谱比较;红线是结合前的Ac-AChBP凝胶色谱,蓝线是结合后的Ac-AChBP凝胶色谱。B:SDS-PAGE胶分析α-芋螺毒素和Ac-AChBP结合,1:活性状态的α-芋螺毒素和Ac-AChBP结合条带;2:变性后条带;M:蛋白Marker |
收集以上共结合的凝胶色谱片段(蓝线峰部分),用机器人去点样长晶体,结晶池液采用优化Ac-AChBP晶体所得的的条件:0.8 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.6,也得到α-芋螺毒素GIC与Ac-AChBP共结晶的晶体,但由于受α-芋螺毒素GIC的影响,其晶体长得不如Ac-AChBP好,还需进一步优化。
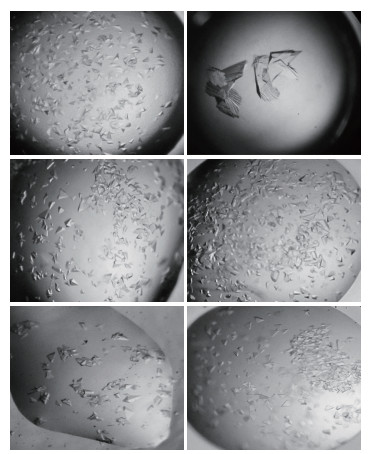
优化采用以上优化Ac-AChBP的方法(表 2),即改变不同沉淀剂的浓度和pH值,以期获得高质量晶体。表 2优化条件下长出的共结晶晶体如图 4所示(遴选),把优化条件下长出来的晶体送往上海同步辐射光源收集数据,其中最好的共结晶数据分辨率为2.1 Å,晶体如图 5所示,其生长条件为0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8。收集一套完整的数据,经HKL2000处理后数据其他指标也较好。
|
| 图 4 共结晶条件优化得到的晶体(遴选) |
|
| 图 5 优化的α-芋螺毒素GIC与Ac-AChBP共结晶2.1 Å晶体 |
目前已发表的α-芋螺毒素与AChBPs共结晶结构采用的结晶条件都有所不同,PnIA(A10L D14K)采用的结晶条件是:17%-18%(W/V)PEG 3350,170-200 mmol/L Na2SO4,100 mmol/L Bis-Tris propane,pH7.5[5];TxIA(A10L)采用的结晶条件是:20% polyethyleneglycol 3350 and bistrispropane at pH8.5[8];ImI在两个条件都得到晶体,它们分别是:11%-14% PEG-4000,0.1 mol/L Tris,pH7.5[6]和100 mmol/L sodium acetate(pH5.5)and polyethylene glycol 5000 monomethylether[7];我们也在两个条件下得到GIC的共结晶,分别是:1.5 mol/L lithium sulfate monohydrate,0.1 mol/L Tris,pH8.5[9]和0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8。以上看出,不同的α-芋螺毒素与AChBPs共结晶其结晶条件不同,同一α-芋螺毒素得到高分辨率的结晶条件也可以是多个。结晶条件千变万化,这可能与结晶的方法及蛋白的性质、纯度、缓冲液等因素有关。
本实验采用的结晶方法是液-液扩散法(Liquid-liquid diffusion),其原理如图 6所示,当把沉淀剂滴加到蛋白质溶液表面,它通过与蛋白质溶液接触面扩散到蛋白质溶液中,沉淀剂从蛋白质溶液中吸走水分,从而使蛋白质溶液浓度不断增大。因此在两溶液接触面形成浓度梯度,随着沉淀剂扩散进入的量越多,蛋白质溶液浓度就越大。蛋白质由不饱和溶液变成饱和溶液,再变成过饱和溶液,从而开始结晶[10]。

|
| 图 6 液-液扩散法结晶原理 |
我们利用LtIA、TxIB、LvIA、TxID、RegIA、GIC等多种α-芋螺毒素与Ac-AChBP共结晶[9, 17-20],在以上条件下有LtIA、LvIA和GIC得到共结晶晶体。优化后GIC分辨率较高,LtIA和LvIA共结晶晶体的优化条件待进一步探讨。不同的α-芋螺毒素对Ac-AChBP结晶影响,需要不同的结晶条件才能得到分辨率高的晶体。另外,每种α-芋螺毒素的溶解度不同,它们的溶解度对共结晶也有影响。
共结晶前采用浓度较稀的Ac-AChBP蛋白液与浓度较稀的α-芋螺毒素溶液先共混好,这种方法可以使α-芋螺毒素更好溶解,因为它们先混合在较稀的溶液中,结合后还要经过凝胶色谱纯化,所以这种方法不仅使Ac-AchBP和α-芋螺毒素充分溶解和结合,还可以除去α-芋螺毒素冻干粉中的杂质对结晶的影响。
我们试用另外一种共结晶方法,即把得到的Ac-AChBP晶体浸泡在α-芋螺毒素溶液中,浸泡时间为两周,观察α-芋螺毒素能否结合到晶体内部。但晶体送往上海同步光源收集数据后,分析结果显示α-芋螺毒素没有结合上Ac-AChBP,这说明α-芋螺毒素与Ac-AChBP共结晶的方法最好还是Ac-AChBP与α-芋螺毒素溶液先混合好再结晶,这样它们才容易结合并共结晶。
我们先研究Ac-AChBP蛋白的结晶条件,然后在此基础上再研究共结晶条件,可以避免浪费α-芋螺毒素,而且还可以分析每种α-芋螺毒素对共结晶条件的影响。
4 结论本研究表达与纯化海兔乙酰胆碱结合蛋白(Ac-AChBP),并把它与α-芋螺毒素GIC共结晶,采用正交实验,并利用结晶机器人及HAMPTON RESEARCH试剂盒对其结晶条件进行筛选与优化,在0.6 mol/L Ammonium citrate dibasic,0.1 mol/L Sodium acetate trihydrate pH4.8的条件下生长出较好质量的晶体,其分辨率达到2.1 Å。
| [1] | Van Dijk WJ, Klaassen RV, Schuurmans M, et al. Crystal structure of an ACh-binding protein reveals the ligand-binding domain of nicotinic receptors. Nature, 2001, 411 (6835): 2692–2676. |
| [2] | Smit AB, Syed NI, Schaap D, et al. A glia-derived acetylcholine-binding protein that modulates synaptic transmission. Nature, 2001, 411 (6835): 261–268. DOI:10.1038/35077000 |
| [3] | Hansen SB, Talley TT, Radic Z, et al. Structural and ligand recognition characteristics of an acetylcholine-binding protein from Aplysia californica. The Journal of Biological Chemistry, 2004, 279 (23): 24197–24202. DOI:10.1074/jbc.M402452200 |
| [4] | Celie PH, Klaassen RV, van Rossum-Fikkert SE, et al. Crystal structure of acetylcholine-binding protein from Bulinus truncatus reveals the conserved structural scaffold and sites of variation in nicotinic acetylcholine receptors. The Journal of Biological Chemistry, 2005, 280 (28): 26457–26466. DOI:10.1074/jbc.M414476200 |
| [5] | Celie PH, Kasheverov IE, Mordvintsev DY, et al. Crystal structure of nicotinic acetylcholine receptor homolog AChBP in complex with an α-conotoxin PnIA variant. Nature Structural & Molecular Biology, 2005, 12 (7): 582–588. |
| [6] | Hansen SB, Sulzenbacher G, Huxford T, et al. Structures of Aplysia AChBP complexes with nicotinic agonists and antagonists reveal distinctive binding interfaces and conformations. The EMBO Journal, 2005, 24 (20): 3635–3646. DOI:10.1038/sj.emboj.7600828 |
| [7] | Ulens C, Hogg RC, Celie PH, et al. Structural determinants of selective alpha-conotoxin binding to a nicotinic acetylcholine receptor homolog AChBP. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103 (10): 3615–3620. DOI:10.1073/pnas.0507889103 |
| [8] | Dutertre S, Ulens C, Büttner R, et al. AChBP-targeted α-conotoxin correlates distinct binding orientations with nAChR subtype selectivity. The EMBO journal, 2007, 26 (16): 3858–3867. DOI:10.1038/sj.emboj.7601785 |
| [9] | Lin B, Xu M, Zhu X, et al. From crystal structure of alpha-conotoxin GIC in complex with Ac-AChBP to molecular determinants of its high selectivity for alpha3beta2 nAChR. Scientific Reports, 2016, 6 : 22349. DOI:10.1038/srep22349 |
| [10] | Gorrec F. Protein crystallization screens developed at the MRC Laboratory of Molecular Biology. Drug Discovery Today, 2016, 21 (5): 819–825. DOI:10.1016/j.drudis.2016.03.008 |
| [11] | Liu J, Yin DC, Guo YZ, et al. Selecting temperature for protein crystallization screens using the temperature dependence of the second virial coefficient. PLoS One, 2011, 6 (3): e17950. DOI:10.1371/journal.pone.0017950 |
| [12] | Juarez-Martinez G, Steinmann P, Roszak AW, et al. High-throughput screens for postgenomics:studies of protein crystallization using microsystems technology. Analytical Chemistry, 2002, 74 (14): 3505–3510. DOI:10.1021/ac0112519 |
| [13] | Lin B, Meng H, Bing H, et al. Efficient expression of Acetylcholine-binding protein from Aplysia californica in Bac-to-Bac System. Biomed Res Int, 2014, 2014 : 691480. |
| [14] | Wang N, Shi X, Jiang L, et al. Structure of MERS-CoV spike receptor-binding domain complexed with human receptor DPP4. Cell Research, 2013, 23 (8): 986–993. DOI:10.1038/cr.2013.92 |
| [15] | 林波, 孟海玲, 吴勇, 等. 转染乙酰胆碱结合蛋白基因到昆虫细胞的研究. 生命科学研究, 2014, 18(1): 1–5. |
| [16] | 林波, 孟海玲, 吴勇, 等. 静水椎螺乙酰胆碱结合蛋白在Bac-to-Bac系统中的表达、纯化与结晶. 生物技术通报, 2014(8): 126–131. |
| [17] | Luo S, Akondi KB, Zhangsun D, et al. Atypical alpha-conotoxin LtIA from Conus litteratus targets a novel microsite of the alpha3beta2 nicotinic receptor. The Journal of Biological Chemistry, 2010, 285 (16): 12355–12366. DOI:10.1074/jbc.M109.079012 |
| [18] | Luo S, Zhangsun D, Zhu X, et al. Characterization of a novel alpha-conotoxin TxID from Conus textile that potently blocks rat alpha3beta4 nicotinic acetylcholine receptors. Journal of Medicinal Chemistry, 2013, 56 (23): 9655–9663. DOI:10.1021/jm401254c |
| [19] | Luo S, Zhangsun D, Wu Y, et al. Characterization of a novel alpha-conotoxin from conus textile that selectively targets alpha6/alpha3beta2beta3 nicotinic acetylcholine receptors. The Journal of Biological Chemistry, 2013, 288 (2): 894–902. DOI:10.1074/jbc.M112.427898 |
| [20] | Kompella SN, Cuny H, Hung A, et al. Molecular basis for differential sensitivity of alpha-Conotoxin RegⅡA at rat and human neuronal nicotinic acetylcholine receptors. Molecular Pharmacology, 2015, 88 (6): 993–1001. DOI:10.1124/mol.115.100503 |






