2. 汕头大学广东省海洋生物技术重点实验室,汕头 515063
2. Guangdong Provincial Key Laboratory of Marine Biotechnology, Shantou University, Shantou 515063
多功能酶(multifunctional enzymes,MFEs)通常是指具有多种生理功能的酶[1]。按照酶的作用机理,一般可以将多功能酶分为:条件多功能酶,底物多功能酶和催化多功能酶[1]。条件多功能酶是指在不同反应条件下具有不同催化活性的酶,底物多功能酶强调能作用于不同底物,催化多功能酶则指用相同的作用位点催化不同的生物转化反应。一般认为多功能酶可以在不加大基因组的前提下扩大生物体的生物功能[2],此外多功能酶可以在生化或信号途径中转换其功能使生物体更好适应环境[3]。
α淀粉酶是得到研究和应用最广泛的淀粉水解酶[4]。按照CAZy数据库根据催化活性保守区的划分[5],当前报道的大多数α淀粉酶(EC 3.2.1.1)属于糖苷水解酶13家族(GH13),其次归属于GH57家族,还有少数属于GH119和GH126[4]。糖苷水解酶分子中一般除了催化区还有底物结合模块[6, 7]。目前报道的淀粉酶的淀粉结合模块主要包括10个糖结合模块家族(Carbohydrate-Binding Module,CBM),分别是:CBM20、CBM21、CBM25、CBM26、CBM34、CBM41、CBM45、CBM48、CBM53和CBM58[4],此外还有新近建立的CBM69[8]和CBM74[9]。琼胶酶(Agarase)是可以降解琼脂糖的酶,分别归属于糖苷水解酶GH16、GH50、GH86、GH96和GH118家族[10]。目前科学界已经在各种生物体中发现了一些多功能淀粉酶[11-14],Liu等[15]最近就报道具有琼胶酶和卡拉胶酶活性的α淀粉酶Amy63。但目前报道的拥有琼胶酶活性的淀粉酶还非常少。
产琼胶酶海洋细菌ZC1是2012年建立的细菌新种Aquimarina agarilytica的典型菌株[16],其基因组已经得到测序[17],在其基因组中发现了两个淀粉酶编码基因。考虑到菌株ZC1可以在含淀粉而不含琼胶的培养基中产生琼胶酶,推测该菌株可能会产生同时具有淀粉酶和琼胶酶活性的酶。本研究拟通过克隆和重组表达菌株ZC1的一个淀粉酶编码基因amy440以验证其重组蛋白可能具有的淀粉酶和琼胶酶活性。
1 材料与方法 1.1 材料 1.1.1 试剂质粒提取试剂盒、DNA凝胶回收试剂盒、细菌基因组提取试剂盒购自广州东盛生物科技有限公司;蛋白质分子量标准购于江苏碧云天研究所(Beyotime);DNA分子量标准、DNA聚合酶(Primer STAR HS DNA polymerase)、dNTP Mixture、限制性内切酶、T4 DNA连接酶、克隆质粒载体pMD19-T购自大连宝生物有限公司;琼脂糖(Agarose LE)系由台湾MD生物公司生产,其他试剂均为国产分析纯。
1.1.2 材料在产琼胶酶海洋细菌ZC1的基因组中,发现了两个推测的淀粉酶编码基因,其中一个命名为amy440。
1.2 方法 1.2.1 淀粉酶基因amy440的生物信息学分析运用Blast程序分析蛋白序列相似性(http://blast.ncbi.nlm.nih.gov/),在线工具motif分析保守区(http://www.genome.jp/tools/motif/),用软件BioEdit进行序列比对,用软件Mega 6.0[18]构建系统进化树(Neighbor-Joining)。
1.2.2 淀粉酶基因amy440的克隆和重组表达载体构建使用Primer Premier 5.0软件设计特异性引物,所设计的引物如表 1所示。
使用细菌基因组提取试剂盒提取菌株ZC1的基因组DNA,使用表 1中引物扩增淀粉酶基因,扩增体系如下。
PCR反应体系:2×Primer STAR Buffer 25 μL,Primer STAR HS DNA polymerase 2.5 U,dNTP Mixture各200 μmol/L,正反引物各50 pmol,DNA模板0.1 μg,用水补足至50 μL。
PCR反应条件:95℃预变性4 min;94℃变性30 s,55℃退火30 s,72℃延伸2.5 min,30个循环数;72℃延伸15 min。
使用质粒小提试剂盒提取表达载体pET-32a(+)表达载体。使用限制性内切酶Sac I与Sal I同步双酶切基因amy440的PCR扩增产物和表达载体。将连接好的重组载体转化大肠杆菌E. coli DH5α感受态细胞,置于37℃培养。通过T7引物PCR扩增来筛选重组子,目的克隆送测序(华大基因)。将经过验证的重组载体转化大肠杆菌E. coli Rosetta(DE3)pLyS,同样用T7引物进行转化验证。同时设立空载体对照:将提取的pET-32a(+)质粒直接转化E. coli Rosetta(DE3)pLyS感受态以作为诱导表达时的空载体对照。
1.2.3 目的基因的重组表达将重组菌培养于含氨苄青霉素(50 μg/mL)的LB液体培养基中,当细菌生长至对数生长期时,加入终浓度为0.1 mmol/L的IPTG诱导目的基因的表达(16℃、150 r/min诱导20 h)后,取100 mL菌液收集菌体,向菌体中加入20 mL Tris-HCl缓冲液(20 mmol/L,pH8.0)重悬菌体,在冰浴中超声破碎菌体(超声破碎条件:功率300 W,工作3 s,间歇3 s,全程15 min)。分别取胞外发酵液、菌体总蛋白、菌体破碎上清和破碎沉淀等组分进行SDS-PAGE检测诱导表达效果。同时设置未诱导对照和空载体对照。
1.2.4 包涵体蛋白的制备和复性对诱导表达的重组菌进行超声破碎,离心收集包涵体沉淀(12 000 r/min,15 min)。用适量的缓冲液(20 mmol/L Tris-HCl,pH8.0,0.5 mol/L NaCl,2 mol/L尿素,2%Triton)洗涤包涵体沉淀,最后用适量的包涵体溶解缓冲液(20 mmol/L Tris-HCl,pH8.0,0.5 mol/L NaCl,8 mol/L尿素,100 mmol/L β-巯基乙醇,2%Triton)重悬,室温搅拌过夜。12 000 r/min,4℃离心15 min,收集上清液,用SDS-PAGE检测包涵体溶解效果。用透析缓冲液稀释溶解的包涵体蛋白浓度至约100 μg/mL,依次用分别含6、4、2、1和0.5 mol/L尿素的透析液进行透析,在4℃透析,每4-6 h更换新鲜透析液一次,高速离心去除沉淀,最后用磷酸缓冲液(pH7.4)透析过夜,高速离心去除沉淀,上清进行超滤浓缩。
1.2.5 活性电泳测定淀粉酶和琼胶酶活性在12%的分离胶(不含SDS)中分别加入1%的淀粉、1%的低熔点琼脂糖作为酶的反应底物,以复性的包涵体蛋白为电泳样品。电泳结束后,将分离胶置于20 mmol/L Tris-HCl(pH8.0)缓冲液中,37℃孵育30 min,取出分离胶用碘液对其染色,分别观察淀粉酶和琼胶酶活性。
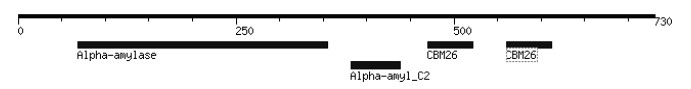
2 结果 2.1 淀粉酶amy440基因的生物信息学分析淀粉酶amy440的编码基因长2 190 bp,编码730个氨基酸(GenBank accession number=WP_010181598)。Blast分析表明,淀粉酶amy440与GenBank数据库中其他淀粉酶序列最高只有45%的序列相似性,在线工具motif的分析(图 1)表明,淀粉酶amy440的N端存在一个α淀粉酶活性催化区,在C端一侧有两个属于CBM26家族的底物结合模块。因此,amy440应属于α淀粉酶。在与目前已经得到研究的属于GH13、GH57、GH119和GH126等家族的α淀粉酶构建的蛋白序列系统进化树(图 2)表明,amy440与GH13家族的α淀粉酶聚在同一分支,因此amy440应属于GH13家族。
|
| 图 1 淀粉酶amy440的序列分析 |

|
| 图 2 淀粉酶amy440的系统进化树 |
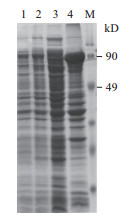
淀粉酶amy440的理论蛋白分子量约为81 kD,加上载体中的融合蛋白,理论上重组蛋白分子量约为95 kD。经SDS-PAGE检测(图 3),对比未诱导的样品,经诱导表达的重组菌菌体破碎液的沉淀中发现有过量表达的目的条带,说明表达的目的蛋白主要以包涵体的形式存在,需要进行包涵体蛋白的制备和复性。
|
| 图 3 重组菌表达的检测 1:未诱导表达的重组菌菌体蛋白;2:诱导表达的重组菌菌体蛋白;3:诱导表达的重组菌菌体破碎后的上清液;4:诱导表达的重组菌菌体破碎后的沉淀;M:蛋白分子量标准 |
提取的包涵体蛋白经复性后进行SDS-PAGE分析,结果(图 4)显示,在电泳凝胶上显示比较单一的条带,且分子量大小与预期相符,说明纯化效果较好,可以用于下一步的活性检测。

|
| 图 4 SDS-PAGE检测复性后的包涵体蛋白 M:蛋白分子量标准;1:复性后的包涵体蛋白 |
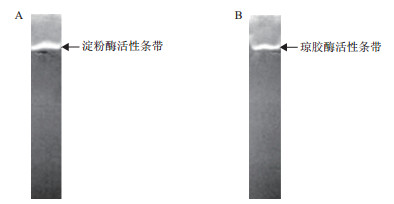
电泳结束后,蛋白凝胶上的淀粉酶条带水解附近的淀粉,电泳凝胶上其他部位淀粉不受影响,经碘液染色,淀粉酶所在部位的淀粉已被水解而不着色,显示无色透明条带,其他部位仍含有淀粉而着色(图 5-A),说明包涵体蛋白具有淀粉酶活性,也即证实编码蛋白amy440具有淀粉酶活性。
|
| 图 5 非变性凝胶电泳检测酶活性 |
电泳结束后,蛋白凝胶上的琼胶酶条带水解附近的琼脂糖,电泳凝胶上其他部位琼脂糖不受影响,经碘液染色,琼胶酶所在部位的琼脂糖已被水解而不着色,其他部位仍含有琼脂糖而着色(图 5-B),说明包涵体蛋白具有琼胶酶活性,也即证实编码蛋白amy440具有琼胶酶活性。
3 讨论目前发现的双功能糖苷水解酶中有的具有两个催化区[19],一个催化区负责一种酶活性;有的只有一个催化区[20-22],同一个催化区赋予两种酶活性。本研究中的淀粉酶amy440含有α淀粉酶的活性催化区,而并没有当前已知的琼胶酶活性催化保守区,推测只有一个催化区,属于催化多功能,因此amy440的主要活性应该在于淀粉酶,琼胶酶活性是次要的活性。这与目前已报道的唯一同时具有淀粉酶和琼胶酶活性的多功能淀粉酶Amy63相似[15]。虽然均具有淀粉酶和琼胶酶活性,Amy63与amy440的蛋白序列相似性只有28%左右,这可能与它们的物种亲缘关系较远有关:Amy63来自变形菌门弧菌属,而amy440来自拟杆菌门。并且Amy63拥有的催化区属于GH70家族,而amy440催化区属于GH13家族。这也说明同时赋予淀粉酶和琼胶酶活性的催化区不只来自一个GH家族。
一般认为CBM能起到帮助酶催化区附着底物的作用,已有研究表明CBM对酶的催化活性也有重要影响,可以提升酶的催化效率[23, 24]。按照CAZy数据库的记录,目前有80种CBM。当前与淀粉酶有联系的CBM有10多种。CBM26是其中一种常见的底物结合模块,通常主要存在GH13家族α淀粉酶的C端或催化区之后[4, 25]。用在线工具motif对Amy63进行分析后,发现Amy63没有目前已知的CBM保守区,而在amy440的C端部分存在两个CBM26保守区。含有CBM26也从侧面说明amy440应属于GH13家族α淀粉酶,与系统进化分析一致。目前已有研究显示,CBM26的数量可能与淀粉酶活性有关[26, 27],CBM26数量越多,淀粉酶活性也越高。但相关研究还很少,需要更多研究来探讨CBM与淀粉酶活性的关系。目前已得到研究的琼胶酶中含有的CBM主要是CBM6[28, 29]和CBM13[30, 31],拥有这些CBM的琼胶酶主要归属于GH16家族。按照CAZy数据库的记录,当前未有琼胶酶附带CBM26模块。
有关多功能酶的多功能催化活性的分子机制是今后的研究重点。对于淀粉酶amy440,有待进一步研究其催化结构域的结构及其多功能催化活性的关键氨基酸。
4 结论基因amy440是产琼胶酶海洋新种细菌ZC1的淀粉酶编码基因,长2 190 bp,编码730个氨基酸,序列分析表明,其属于糖苷水解酶13家族的α淀粉酶。通过对淀粉酶基因amy440进行克隆和重组原核表达,发现amy440的重组蛋白同时具有淀粉酶和琼胶酶活性。
| [1] | Cheng XY, Huang WJ, Hu SC, et al. A global characterization and identification of multifunctional enzymes. PLoS ONE, 2012, 7 (6): e38979. DOI:10.1371/journal.pone.0038979 |
| [2] | Jeffery CJ. Moonlighting proteins. Trends in Biochemical Sciences, 1999, 24 : 8–11. DOI:10.1016/S0968-0004(98)01335-8 |
| [3] | Jeffery CJ. Molecular mechanisms for multitasking: recent crystal structures of moonlighting proteins. Current Opinion in Structural Biology, 2004, 14 : 663–668. DOI:10.1016/j.sbi.2004.10.001 |
| [4] | Janeček Š, Svensson B, MacGregor EA. α-Amylase: an enzyme specificity found in various families of glycoside hydrolases. Cellular and Molecular Life Sciences, 2014, 71 (7): 1149–1170. DOI:10.1007/s00018-013-1388-z |
| [5] | Cantarel BL, Coutinho PM, Rancurel C, et al. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics. Nucleic Acids Research, 2009, 37 : D233–238. DOI:10.1093/nar/gkn663 |
| [6] | Cuskin F, Flint J, Gloster T, et al. How nature can exploit nonspecific catalytic and carbohydrate binding modules to create enzymatic specificity. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109 (51): 20889–20894. DOI:10.1073/pnas.1212034109 |
| [7] | Abbott D, van Bueren A. Using structure to inform carbohydrate binding module function. Current Opinion in Structural Biology, 2014 : 32–40. |
| [8] | Peng H, Zheng Y, Chen M, et al. A starch-binding domain identified in α-amylase (AmyP) represents a new family of carbohydrate-binding modules that contribute to enzymatic hydrolysis of soluble starch. FEBS Letters, 2014, 588 (7): 1161–1167. DOI:10.1016/j.febslet.2014.02.050 |
| [9] | Valk V, Lammol/Lerts van Bueren A, van der Kaaij RM, et al. Carbohydrate Binding Module 74 is a novel starch binding domain associated with large and multi-domain α-amylase enzymes. FEBS Journal, 2016, 283 (12): 2354–2368. DOI:10.1111/febs.13745 |
| [10] | Xie W, Lin BK, Zhou Z, et al. Characterization of a novel β-agarase from an agar-degrading bacterium Catenovulum sp. X3. Applied Microbiology and Biotechnology, 2013, 97 (11): 4907–4915. DOI:10.1007/s00253-012-4385-5 |
| [11] | Han P, Zhou P, Hu S, et al. A novel multifunctional α-amylase from the thermophilic fungus Malbranchea cinnamomea: biochemical characterization and three-dimensional structure. Applied Biochemistry and Biotechnology, 2013, 170 (2): 420–435. DOI:10.1007/s12010-013-0198-y |
| [12] | Cao H, Gao G, Gu Y, et al. Trp358 is a key residue for the multiple catalytic activities of multifunctional amylase OPMA-N from Bacillus sp. ZW2531-1. Applied Microbiology and Biotechnology, 2014, 98 (5): 2101–2111. DOI:10.1007/s00253-013-5085-5 |
| [13] | Han X, Lin B, Ru G, et al. Gene cloning and characterization of an α-amylase from Alteromonas macleodii B7 for Enteromorpha polysaccharide degradation. Journal of Microbiology and Biotechnology, 2014, 24 (2): 254–263. DOI:10.4014/jmb.1304.04036 |
| [14] | Xu Q, Cao Y, Li X, et al. Purification and characterization of a novel intracellular α-amylase with a wide variety of substrates hydrolysis and transglycosylation activity from Paenibacillus sp. SSG-1. Protein Expression and Purification, 2016 . DOI:10.1016/j.pep.2016.04.007 |
| [15] | Liu G, Wu S, Jin W, et al. Amy63, a novel type of marine bacterial multifunctional enzyme possessing amylase, agarase and carrageenase activities. Scientific Reports, 2016, 6 : 18726. DOI:10.1038/srep18726 |
| [16] | Lin BK, Lu GY, Zheng YD, et al. Aquimarina agarilytica sp. nov., a novel agarolytic species isolated from red alga. International Journal of Systematic and Evolutionary Microbiology, 2012, 62 : 869–873. DOI:10.1099/ijs.0.027136-0 |
| [17] | Lin BK, Lu GY, Li SK, et al. Draft genome sequence of the Novel agarolytic bacterium Aquimarina agarilytica ZC1. Journal of Bacteriology, 2012, 194 (10): 2769. DOI:10.1128/JB.00311-12 |
| [18] | Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis Version 6.0. Molecular Biology and Evolution, 2013, 30 : 2725–2729. DOI:10.1093/molbev/mst197 |
| [19] | Ye L, Su X, Schmitz G, et al. Molecular and biochemical analyses of the GH44 module of CbMan5B/Cel44A, a bifunctional enzyme from the hyperthermophilic bacterium Caldicellulosiruptor bescii. Applied and Environmental Microbiology, 2012, 78 (19): 7048–7059. DOI:10.1128/AEM.02009-12 |
| [20] | Huy ND, Thayumanavan P, Kwon T, et al. Characterization of a recombinant bifunctional xylosidase/arabinofuranosidase from Phanerochaete chrysosporium. Journal of Bioscience and Bioengineering, 2013, 116 (2): 152–159. DOI:10.1016/j.jbiosc.2013.02.004 |
| [21] | Xue X, Wang R, Tu T, et al. The N-terminal GH10 domain of a multimodular protein from Caldicellulosiruptor bescii is a versatile Xylanase/β-Glucanase that can degrade crystalline cellulose. Appl Environ Microbiol, 2015, 81 (11): 3823–3833. DOI:10.1128/AEM.00432-15 |
| [22] | Yang W, Bai Y, Yang P, et al. A novel bifunctional GH51 exo-α-l -arabinofuranosidase/ endo-xylanase from Alicyclobacillus sp. A4 with significant biomass-degrading capacity. Biotechnology for Biofuels, 2015, 8 (1): 1–11. DOI:10.1186/s13068-014-0179-6 |
| [23] | Hoffmam ZB, Zanphorlin LM, Cota J, et al. Xylan-specific carbohydrate-binding module belonging to family 6 enhances the catalytic performance of a GH11 endo-xylanase. New Biotechnology, 2016, 33 (4): 467–472. DOI:10.1016/j.nbt.2016.02.006 |
| [24] | Li S, Yang X, Bao M, et al. Family 13 carbohydrate-binding module of alginate lyase from Agarivorans sp. L11 enhances its catalytic efficiency and thermostability, and alters its substrate preference and product distribution. Fems Microbiology Letters, 2015 . DOI:10.1093/femsle/fnv054 |
| [25] | Majzlova K, Janecek S. Two structurally related starch-binding domain families CBM25 and CBM26. Biologia, 2014, 69 (9): 1087–1096. |
| [26] | Rodriguez-Sanoja R, Ruiz B, Guyot JP, et al. Starch-binding domain affects catalysis in two Lactobacillus α-amylases. Applied and Environmental Microbiology, 2005, 71 : 297–302. DOI:10.1128/AEM.71.1.297-302.2005 |
| [27] | Guillen D, Santiago ML, Linares LK, et al. Alpha-amylase starch binding domains: cooperative effects of binding to starch granules of multiple tandemly arranged domains. Applied and Environmental Microbiology, 2007, 73 (12): 3833–3837. DOI:10.1128/AEM.02628-06 |
| [28] | Hsu P, Wei C, Lu W, et al. Extracellular production of a novel endo-β-agarase AgaA from Pseudomonas vesicularis MA103 that cleaves agarose into neoagarotetraose and neoagarohexaose. International Journal of Molecular Sciences, 2015, 16 (3): 5590–5603. DOI:10.3390/ijms16035590 |
| [29] | Michel G, Barbeyron T, Kloareg B, et al. The family 6 carbohydrate-binding modules have coevolved with their appended catalytic modules toward similar substrate specificity. Glycobiology, 2009, 19 (6): 615–623. DOI:10.1093/glycob/cwp028 |
| [30] | Lu X, Chu Y, Wu Q, et al. Cloning, expression and characterization of a new agarase-encoding gene from marine Pseudoalteromonas sp.. Biotechnology Letters, 2009, 31 (10): 1565–1570. DOI:10.1007/s10529-009-0042-1 |
| [31] | Shi Y, Lu X, Yu W, et al. A new β-agarase from marine bacterium Janthinobacterium sp. SY12. World Journal of Microbiology & Biotechnology, 2008, 24 (11): 2659–2664. |





