肥胖的发病率在我国日益上升,2013年针对我国大于20岁的成年人进行统计,肥胖发病率为8.8%,超重人群则达到55.7%[1]。肥胖的发生是脂肪异常堆积的结果,对脂肪细胞分化机制的深入研究有益于为肥胖的治疗提供有效策略[2]。脂肪细胞的分化过程是多条信号通路以及大量转录因子协同级联调控的结果[3]。近期研究发现kruppel样锌指转录因子家族(Kruppel-like factor,KLF)成员广泛参与脂肪细胞分化[4]。在人前脂肪细胞和小鼠3T3-L1细胞中过表达KLF7可以显著抑制脂滴产生,并显著抑制脂肪细胞特异性基因PPARγ、C/EBPα、aP2和adipsin的表达[5]。此外,有研究显示长链非编码RNA(Long non-coding RNA,lncRNA)也参与脂肪细胞分化过程的调控[6]。lncRNA是一类片段长度大于200 bp的内源性RNA分子,人类基因组中只有大约1%具有蛋白编码潜能,然而在特定的发育节点却有大约70%的基因组转录,其中多数转录本属于lncRNA,它们通常由RNA聚合酶Ⅱ转录,经RNA剪接过程成熟,具有谱系特异性并行使多种生物学功能。例如,胞核lncRNA参与形成核糖核蛋白复合体从而修饰染色体结构影响基因转录活性,胞浆lncRNA可以直接或通过吸附microRNA调控mRNA的稳定[7-9]。Sun等[10]通过比对原代棕色和白色脂肪细胞、前体脂肪细胞以及成熟脂肪细胞的转录组,发现脂肪细胞分化过程中,175个lncRNAs被特异调控,而且C/EBPα和PPARγ可以结合于大量lncRNA的启动子区域,通过RNAi抑制其中10个lncRNAs可以显著抑制脂滴形成。
本研究拟通过分析lncRNA AK043773的基因定位,鉴定其在脂肪细胞分化过程中的表达和功能,旨在为进一步探索lncRNA AK043773在脂肪细胞分化中的分子作用机制奠定研究基础。
1 材料与方法 1.1 材料小鼠骨髓基质细胞ST2、前脂肪细胞3T3-L1和质粒pCDNA3.1(+)由本实验室保存;SPF级雄性6周龄C57BL/6小鼠购自北京华阜康生物科技股份有限公司;Trans5α感受态细胞、TransStart FastPfu DNA Polymerase和T4 DNA连接酶购自北京全式金生物技术有限公司;BamH I和EcoRⅤ内切酶购自美国Thermo Fisher;质粒提取试剂盒和凝胶回收试剂盒购自北京天根生化科技公司;Lipofectamine 2000购自美国Invitrogen;α-MEM和DMEM/H培养基购自美国Hyclone;胎牛血清FBS购自德国SeraPro;RNA提取试剂E.Z.N.A. Total RNA Kit购自美国Omega Bio-tek;Reverse Transcription Kit购自美国Thermo Fisher;SGExcel Fast SYBR Mixture kits及特异性引物购自生工生物工程有限公司;诱导试剂胰岛素(Insulin)、地塞米松(Dex)、3-异丁基-1-甲基黄嘌呤(IBMX)和油红O购自美国Sigma;吲哚美辛(Indo)购自美国Cyagen Biosciences Inc。
1.2 方法 1.2.1 细胞的培养与成脂分化诱导小鼠骨髓基质细胞ST2用含10% FBS的α-MEM培养基,前脂肪细胞3T3-L1用含10% FBS的DMEM/H培养基,置于37℃、5% CO2培养箱内培养,待细胞长至80%以上时传代。将细胞接种于12孔板,细胞密度达90%以上时进行成脂分化诱导。成脂诱导分化利用经典的激素鸡尾酒法,诱导试剂包含0.05 mmol/L 3-异丁基-1-甲基黄嘌呤、5 mg/L胰岛素、0.05 mmol/L吲哚美辛和0.05 μmol/L地塞米松。
1.2.2 pCDNA3.1-AK043773载体的构建设计合成扩增AK043773片段的特异性引物,以C57BL/6小鼠肝脏DNA为模板,经FastPfu DNA聚合酶扩增获得AK043773片段全长,将克隆片段与pCDNA3.1(+)载体分别进行BamH I和EcoRⅤ内切酶双酶切,胶回收PCR片段及载体大片段后用T4 DNA连接酶连接,连接产物转化至大肠杆菌Trans5α感受态细胞,提取单克隆后双酶切鉴定,鉴定正确克隆送交Invitrogen测序,测序正确载体命名为pCDNA3.1-AK043773载体。载体引物上游:5'-GCG GAT CCG AGA GGG CTG GAC TTC AG-3',下游5'-CCG GAT ATC CTT TTT TTA TAG AGC CTT GCA TTG G-3'。下划线代表酶切位点,其中GGATCC为BamH I,GATATC为EcoRⅤ。
1.2.3 pCDNA3.1及pCDNA3.1-AK043773载体转染转染前1 d,将ST2细胞接种于12孔板,转染当天细胞融合度达70%-90%时进行转染。转染试剂采用Lipofectamine 2000,转染6 h后细胞换液,具体实验步骤依说明书为准。转染24 h后可进行成脂细胞分化诱导;转染48 h后可检测转染效率。
1.2.4 RT-qPCR检测KLF7和lncRNA AK043773的表达水平ST2和3T3-L1细胞诱导成脂分化3 d后收取细胞,pCDNA3.1及pCDNA3.1-AK043773载体转染ST2细胞48 h后收取细胞,按照E.Z.N.A. Total RNA Kit说明书分别提取各组细胞总RNA,利用Reverse Transcription Kit将0.5 μg总RNA逆转录为cDNA。逆转录的条件:25℃ 10 min;42℃ 60 min;70℃ 10 min。
逆转录合成的cDNA作为模板,qPCR检测KLF7和lncRNA AK043773的表达。根据SGExcel Fast SYBR Mixture kits试剂盒说明书加入qPCR所需的2×Mix、cDNA和特异性引物反应。反应体系为20 μL,内参为β-actin。反应条件为:95℃预变性2 min;95℃变性30 s、57℃退火15 s、72℃延伸15 s,40个循环。PCR产物采用溶解曲线法分析,以确定样品扩增的特异性。采用2-ΔΔCt法计算目的基因实验组与对照组的表达差异,Ct值指反应管中荧光信号达到一定阈值时所经历的循环数。所有实验重复3次。KLF7 qPCR引物上游5'-ACT GAC AAA CAA ACA GAC CCA G-3',下游5'-GGT AGC GTT CCA ACT CAA GG-3';lncRNA AK043773 qPCR引物上游5'-ACG CCA GGG ATG GCA TTA-3',下游5'-GAG CCT TGC ATT GGT CAG T-3';β-actin qPCR引物上游5'-AAG ACC TCT ATG CCA ACA CAG-3',下游5'-GGA GGA GCA ATG ATC TTG ATC-3'。
1.2.5 油红O染色及OD值检测pCDNA3.1及pCDNA3.1-AK043773载体转染ST2细胞24 h后,进行成脂细胞分化诱导,待脂滴成熟后弃细胞培养基,1×PBS洗2次;4%多聚甲醛固定15 min后弃液,1×PBS洗1次;加入60%异丙醇,室温放置1-2 min后弃液;加入60%油红O染液,染色5 min,蒸馏水漂洗2次,于倒置显微镜下观察并拍照。拍照后弃水,将细胞培养板倒置晾干后,每孔加400 μL异丙醇萃取油红O,常温放置20 min,移至96孔板检测吸光度,波长设置520 nm。
1.2.6 统计学方法采用SPSS17.0软件进行统计处理。实验数据以均数±标准差(x±s)表示,两组间比较采用独立样本t检验。P < 0.05为差异有统计学意义。
2 结果 2.1 lncRNA AK043773的基因位点分析经UCSC数据库(http://genome.ucsc.edu/)检索,lncRNA AK043773基因全长2 720 bp,位于KLF7基因4号外显子的下游(图 1)。

|
| 图 1 lncRNA AK043773的基因位点分析 |
为确定AK043773的编码能力,经蛋白编码能力预测数据库CPAT:Coding-Potential Assessment Tool(http://lilab.research.bcm.edu/cpat/index.php)分析,AK043773的蛋白编码能力得分为0.13,作为对照,KLF7基因(NCBI序列参考编号为NM_033563.2)的蛋白编码能力得分为0.98。由此可见,AK043773编码蛋白的潜能极低,属于非编码RNA(图 2)。
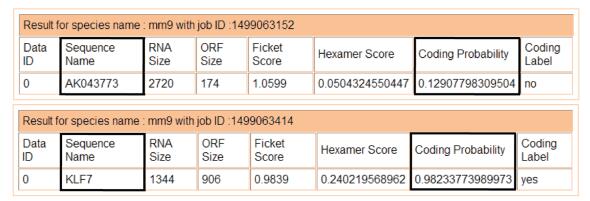
|
| 图 2 lncRNA AK043773的蛋白编码能力分析 |
ST2和3T3-L1细胞通过经典的激素鸡尾酒法成脂诱导分化3 d后,与未成脂分化细胞分别提取总RNA,逆转录后进行RT-qPCR检测。结果(图 3)显示,与未成脂分化的细胞相比,在成脂分化的3T3-L1和ST2细胞中,KLF7分别显著下降至0.80和0.47,lncRNA AK043773分别显著下降至0.74和0.49。
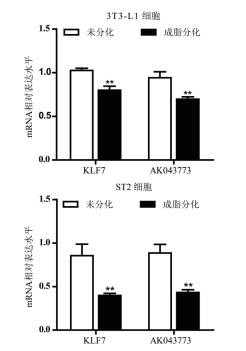
|
| 图 3 KLF7和lncRNA AK043773在3T3-L1和ST2细胞成脂前后的表达水平 **P < 0.01(成脂分化组VS.未分化组) |
以雄性C57BL/6小鼠肝脏DNA为模板,通过PCR特异性扩增2 720 bp的AK043773基因全长,通过BamH I和EcoR Ⅴ双酶切位点克隆至pCDNA3.1(+)载体,命名为pCDNA3.1-AK043773。测序结果正确,PCR特异性扩增片段与pCDNA3.1-AK043773载体双酶切后进行琼脂糖凝胶电泳,结果均显示载体构建成功,扩增片段及酶切结果见图 4。
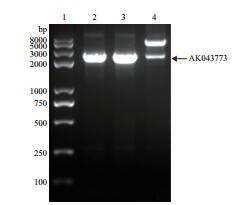
|
| 图 4 AK043773基因PCR特异性扩增、pCDNA3.1-AK043773载体PCR特异性扩增及其双酶切鉴定 1:DNA Marker Trans 2K plus Ⅱ;2:以雄性C57BL/6小鼠肝脏DNA为模板,PCR特异性扩增的AK043773基因片段;3:以构建的pCDNA3.1-AK043773载体为模板,PCR特异性扩增的AK043773基因片段;4:用BamH I和EcoR Ⅴ双酶切pCDNA3.1-AK043773载体结果 |
pCDNA3.1及pCDNA3.1-AK043773载体分别转染ST2细胞48 h后收取细胞,分别提取总RNA,逆转录后进行RT-qPCR检测。结果(图 5)显示,与pCDNA3.1载体转染组相比,转染pCDNA-3.1-AK043773载体可以显著提高ST2细胞中lncRNA AK043773的表达水平至13.06倍。

|
| 图 5 pCDNA3.1及pCDNA3.1-AK043773载体转染ST2细胞后lncRNA AK043773的表达 ***P < 0.001(pCDNA3.1-AK043773载体转染组VS. pCDNA3.1载体转染组) |
pCDNA3.1及pCDNA3.1-AK043773载体分别转染ST2细胞后诱导成脂分化,待脂滴成熟后进行油红O染色,pCDNA3.1-AK043773载体转染组较pCDNA3.1载体转染组细胞脂滴明显减少;异丙醇萃取油红O后经OD520nm波长吸光值检测,可见pCDNA3.1-AK043773载体转染组较pCDNA3.1载体转染组下降30%(图 6)。
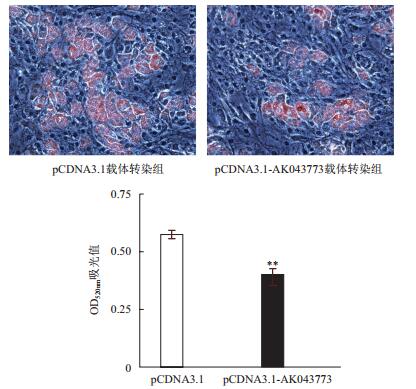
|
| 图 6 lncRNA AK043773对脂肪细胞分化的影响 油红O染色放大倍数为200×;**P < 0.01(pCDNA3.1-AK043773载体转染组VS. pCDNA3.1载体转染组) |
肥胖被定义为体脂超标,肥胖患病率的持续上升使得脂肪组织及脂肪细胞生成被广泛关注[11]。脂肪组织是非常活跃重要的内分泌器官,对能量及代谢稳态的维持至关重要[12]。脂肪细胞是脂肪组织的主要组成部分,脂肪细胞主要由成纤维样前脂肪细胞分化发育而来,脂肪细胞的分化是多种转录因子级联反应及各种调控因子调节的结果[13-15]。近期有研究显示lncRNA广泛参与生命活动的发育和分化进程[16-18]。2013年,Sun等[10]首次针对体外培养的小鼠的前白色、棕色脂肪细胞以及分化成熟的白色、棕色脂肪细胞,筛选得到polyA尾转录谱然后进行RNA测序,鉴定了175个差异表达的lncRNA,其中很多脂肪细胞富集的lncRNA启动子区有PPARγ和C/EBPα的结合位点,用RNAi抑制这些lncRNA的表达可以有效抑制成脂。2014年,Cooper等[19]在小鼠3T3-L1细胞中发现lncRNA NEAT1可以与SRp40通过调控PPARγ2的剪接影响成脂。2016年,Huang等[20]发现lncRNA H19可以通过组蛋白去乙酰化酶的表观修饰抑制骨髓来源间充质干细胞的成脂分化。这些研究结果提示lncRNA也参与了脂肪细胞的分化生成阶段。
在研究前期,对原代培养的小鼠骨髓基质细胞进行脂肪细胞诱导分化,72 h后采用芯片技术获得脂肪细胞分化前后的lncRNA及mRNA表达谱。发现在成脂分化的细胞中KLF7表达显著下调,该结果与文献报道一致[5],而位于KLF7基因4号外显子下游的lncRNA AK043773协同下调。经蛋白编码能力预测数据库Coding Potential Calculator(http://cpc.cbi.pku.edu.cn/programs/run_cpc.jsp)分析,lncRNA AK043773不具备编码蛋白的潜能。通过RT-qPCR验证了芯片结果,从而提示lncRNA AK043773可能参与脂肪细胞分化的调控,而且其可能发挥负向调控的功能。通过构建lncRNA AK043773的过表达载体并转染ST2细胞,经成脂诱导后证明了我们的猜想,即过表达lncRNA AK043773可以抑制脂肪细胞分化。
后续研究将验证lncRNA AK043773的蛋白编码潜能,通过5'和3'cDNA末端快速克隆(RACE)实验确定其全长,通过分离脂肪分化前后细胞的胞核和胞浆部分,采用RT-qPCR技术确定lncRNA AK043773的亚细胞定位,通过设计合成lncRNA AK043773的siRNA验证其在脂肪细胞分化中的功能。本研究发现lncRNA AK043773位于KLF7基因的下游,后续研究还将探讨其是否通过顺式作用影响KLF7的表达发挥其抑制脂肪细胞分化的功能,从而进一步深入的研究lncRNA AK043773发挥调控功能的分子机制,为阐释脂肪细胞分化进程的调控提供新理论。
4 结论本研究发现了一个新的lncRNA AK043773,其位于KLF7基因4号外显子的下游,在成脂分化的ST2和3T3-L1细胞中表达下调,在ST2细胞中过表达lncRNA AK043773可以抑制脂肪细胞分化。
| [1] |
Ng M, Fleming T, Robinson M, et al. Global, regional, and national prevalence of overweight and obesity in children and adults during 1980-2013: a systematic analysis for the Global Burden of Disease Study 2013[J]. The Lancet, 2014, 384(9945): 766-781. DOI:10.1016/S0140-6736(14)60460-8 |
| [2] |
Heinonen S, Saarinen L, Naukkarinen J, et al. Adipocyte morphology and implications for metabolic derangements in acquired obesity[J]. International Journal of Obesity, 2014, 38(11): 1423-1431. DOI:10.1038/ijo.2014.31 |
| [3] |
Guo L, Li X, Tang QQ. Transcriptional regulation of adipocyte differentiation: a central role for CCAAT/enhancer-binding protein(C/EBP)β[J]. J Biol Chem, 2015, 290(2): 755-761. DOI:10.1074/jbc.R114.619957 |
| [4] |
Wu Z, Wang S. Role of kruppel-like transcription factors in adipogenesis[J]. Developmental Biology, 2013, 373(2): 235-243. DOI:10.1016/j.ydbio.2012.10.031 |
| [5] |
Kawamura Y, Tanaka Y, Kawamori R, et al. Overexpression of Kruppel-like factor 7 regulates adipocytokine gene expressions in human adipocytes and inhibits glucose-induced insulin secretion in pancreatic β-cell line[J]. Molecular Endocrinology, 2006, 20(4): 844-856. DOI:10.1210/me.2005-0138 |
| [6] |
Knoll M, Lodish HF, Sun L. Long non-coding RNAs as regulators of the endocrine system[J]. Nature Reviews Endocrinology, 2015, 11(3): 151-160. DOI:10.1038/nrendo.2014.229 |
| [7] |
Djebali S, Davis CA, Merkel A, et al. Landscape of transcription in human cells[J]. Nature, 2012, 489(7414): 101-108. DOI:10.1038/nature11233 |
| [8] |
Boon RA, Jaé N, Holdt L, et al. Long Noncoding RNAs[J]. Journal of the American College of Cardiology, 2016, 67(10): 1214-1226. DOI:10.1016/j.jacc.2015.12.051 |
| [9] |
Sigova AA, Mullen AC, Molinie B, et al. Divergent transcription of long noncoding RNA/mRNA gene pairs in embryonic stem cells[J]. Proceedings of the National Academy of Sciences, 2013, 110(8): 2876-2881. DOI:10.1073/pnas.1221904110 |
| [10] |
Sun L, Goff LA, Trapnell C, et al. Long noncoding RNAs regulate adipogenesis[J]. Proceedings of the National Academy of Sciences, 2013, 110(9): 3387-3392. DOI:10.1073/pnas.1222643110 |
| [11] |
Smith U. Abdominal obesity: a marker of ectopic fat accumulation[J]. The Journal of Clinical Investigation, 2015, 125(5): 1790-1792. DOI:10.1172/JCI81507 |
| [12] |
Coelho M, Oliveira T, Fernandes R. Biochemistry of adipose tissue: an endocrine organ[J]. Archives of Medical Science, 2013, 9(2): 191-200. |
| [13] |
Musri MM, Párrizas M. Epigenetic regulation of adipogenesis[J]. Current Opinion in Clinical Nutrition & Metabolic Care, 2012, 15(4): 342-349. |
| [14] |
Cristancho AG, Lazar MA. Forming functional fat: a growing understanding of adipocyte differentiation[J]. Nature Reviews Molecular Cell Biology, 2011, 12(11): 722-734. DOI:10.1038/nrm3198 |
| [15] |
Chen Q, Shou P, Zheng C, et al. Fate decision of mesenchymal stem cells: adipocytes or osteoblasts?[J]. Cell Death & Differentiation, 2016, 23(7): 1128-1139. |
| [16] |
Fatica A, Bozzoni I. Long non-coding RNAs: new players in cell differentiation and development[J]. Nature Reviews Genetics, 2014, 15(1): 7-21. |
| [17] |
Dey BK, Mueller AC, Dutta A. Long non-coding RNAs as emerging regulators of differentiation, development, and disease[J]. Transcription, 2014, 5(4): e944014-1-e944014-9. |
| [18] |
Luo S, Lu JY, Liu L, et al. Divergent lncRNAs regulate gene expression and lineage differentiation in pluripotent cells[J]. Cell Stem Cell, 2016, 18(5): 637-652. DOI:10.1016/j.stem.2016.01.024 |
| [19] |
Cooper DR, Carter G, Li P, et al. Long non-coding RNA NEAT1 associates with SRp40 to temporally regulate PPARγ2 splicing during adipogenesis in 3T3-L1 Cells[J]. Genes, 2014, 5(4): 1050-1063. DOI:10.3390/genes5041050 |
| [20] |
Huang Y, Zheng Y, Jin C, et al. Long non-coding RNA H19 inhibits adipocyte differentiation of bone marrow mesenchymal stem cells through epigenetic modulation of histone deacetylases[J]. Sci Rep, 2016, 6(28897): 1-13. |




