核酸适配体(Aptamer)通常是指在体外利用指数富集的配基系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)筛选得到的核酸(DNA或RNA)片段,它的结构多样,可形成发卡、假结、凸环、G-四联体等复杂的空间结构,与靶标发生类似抗原-抗体反应的构象识别。它因类似抗体的功能,被称作化学抗体,且在较高亲和力和特异性的基础上,具有靶标范围广、筛选周期短、免疫原性低、不依赖动物、成本低、易重复、稳定性好、便于合成和修饰等优于抗体的特点[1]。核酸适配体发展潜力巨大,它能否取代抗体一直以来是人们关注的热点。SELEX技术的目的是在巨大的初始随机寡核苷酸文库中富集到特定靶标的亲和力高、特异性强的核酸适配体,怎样快速高效的获得性能优异的核酸适配体是制约其应用发展的关键问题之一,本文结合近些年核酸适配体的研究进展,阐述了SELEX技术不同环节涉及到的优化改良策略,有助于突破SELEX发展面临的筛选效率低的瓶颈,使得越来越多的核酸适配体应用到实际应用中去。
1 SELEX技术的改进策略 1.1 初始随机寡核苷酸文库初始随机寡核苷酸文库是SELEX筛选的起点,对初始随机寡核苷酸文库进行一定的设计与修饰可提高核酸适配体的筛选成功率。
1.1.1 初始随机寡核苷酸文库的设计初始随机文库的多样性是影响SELEX筛选的关键因素之一,而单链寡核苷酸的随机序列则是文库多样性的主要来源[2]。随机序列的设计常见下列几种情况:第一种类型最为常见,它的构成是A、G、C、T/U四种碱基的平均分布(图 1-A),这样不仅使得文库具有最大的多样性,而且还维持了序列空间与核酸二级及三级结构之间的平衡[3]。第二种类型是富含G或G+C的随机序列(图 1-B),由于它更易形成G-四联体,因而比普通的文库具有更强的稳定性,Choi等[4]利用该类序列筛选到的核酸适配体具有核酸酶抗性及更好的蛋白结合能力,能被细胞更好的吸收,并具有选择性的抗肿瘤增殖活性。第三种类型是部分随机化序列(图 1-C),它在随机区域掺入部分已知的对某靶标具有一定结合能力的序列片段来进行再次筛选,Santosh等[5]认为这样可以更快的去发现新的功能分子;Ahmad等[6]将两段凝血酶的核酸适配体序列在随机区域组建成的二价适体,它的亲和力相较与单体适体提高了200倍左右,这也为多价适体或者说多功能适配体的研究提供了方向。第四种类型是特殊设计的随机序列(图 1-D),它可根据研究者的特殊研究目的进行设计,Hao等[7]在随机序列中加入一段固定区域,利用核酸序列与靶标作用前后的结构转换达到检验金属离子Hg2+和Cu2+的目的。

|
| 图 1 初始随机寡核苷酸文库的设计类型 |
初始核酸文库的改良除了可以根据不同的研究需求进行特定的设计,还可通过对核酸文库进行特定的修饰来提高筛选的效率,即通过对核酸序列中核苷酸的碱基或者骨架进行修饰,不仅可以降低核酸酶的易感性,而且能增加核酸的结构多样性。Yu等[8]筛选到了人凝血酶的TNA核酸适配体,Kd值为200 nmol/Lol/L,其中TNA就是核酸骨架经过修饰的异源核酸XNAs中的一种。Kimoto等[9]在初始随机文库中掺入了多达3个的Ds核苷酸,其中Ds为携带疏水基7-(2-噻吩基)咪唑并[4,5-b]吡啶的非天然核苷酸,利用该特殊修饰的核酸文库筛选到了血管内皮细胞生长因子VGF-165和干扰素-λ的核酸适配体,两者Kd值分别为0.65 pmol/L和0.038 nmol/Lol/L,亲和力比天然核苷酸组成的核酸适配体提高了100多倍。Sefah等[10]以乳腺癌细胞(MDA-MB-231)为靶标,筛选得到核酸适配体ZAP-2012,其中ZAP-2012是由A、T、G、C四种天然核苷酸及Z、P两种非标准核苷酸组合而成,如果将Z、P用天然核苷酸替换,ZAP-2012的亲和力会大幅下降。
1.2 筛选方法为了提高核酸适配体的筛选效率,筛选方法出现了诸多新的尝试和改良,这表现在筛选过程的多个方面。首先,从靶标的固定介质而言,它的演变就衍生出多种SELEX筛选方法,目前最常用是以磁珠作为靶标固定介质的磁珠-SELEX。此外,还可以固定核酸文库或者采用非固定的模式,毛细管电泳-SELEX就很好的回避了固定方式的问题;细胞-SELEX甚至可以全细胞作为靶标,很大程度的保留了靶物质的天然构像。其次,对核酸文库的优化也是诸多改良中的一种,加尾-SELEX等回避了筛选后需要面对的截短优化等环节。随着筛选效率的不断提高,微流体-SELEX将基于芯片技术的筛选平台引入到筛选,微阵列-SELEX则可以多项筛选同时进行,筛选自动化成为一种新的诉求和发展趋势。最后,相较传统筛选技术的盲目重复,结合高通量的SELEX对文库的富集情况提供了强大的数据支持,在降低了盲目性的同时也指导了筛选的进行,提高了效率。本文将结合上述尝试和改良所涉及到的方法做具体介绍。
1.2.1 毛细管电泳-SELEX毛细管电泳-SELEX(Capillary Electrophoresis-SELEX,CE-SELEX)技术[11]可直接在溶液中借助毛细管电泳的高效分离能力将迁移效率不同的物质进行分离,反应体系中的游离核酸与核酸-靶标复合物的迁移能力不同,借助毛细管电泳-SELEX技术可实现快速分离,从而提高了SELEX的筛选效率。Mendonsa等[12]利用CE-SELEX只需4轮筛选,就得到了IgE的核酸适配体,解离常数Kd值为40 nmol/Lol/L;Yang等[13]利用CE-SELEX经过3轮筛选得到了小分子靶标卟啉NMM的核酸适配体。相较于传统的筛选方法,它们都大大缩短了实验周期,而且毛细管电泳装置进样体积小,节约了样本。同时,基于CE-SELEX技术,还有平衡混合物的非平衡毛细管电泳(NECEEM)、Non-SELEX、平衡混合物的平衡毛细管电泳(ECEEM)、扫集毛细管电泳(SweepCE)、微流控自由流电泳(μFFE)等多种筛选模式[11],进一步促进了CE-SELEX技术在核酸适配体筛选领域的发展应用。
1.2.2 细胞-SELEX核酸适配体的筛选大多针对特定的靶标展开,细胞-SELEX(cell-SELEX)是个例外,它以全细胞作为靶物质,不需要在筛选开始前获取靶标的详细情况,这不仅在一定程度上拓宽了SELEX筛选的靶标范围,同时也挖掘了核酸适配体作为一个分子工具在疾病相关的生物标记物的发现、细胞分子的识别、诊断与治疗等方面的巨大潜力[14],使得核酸适配体的研究具有更多实用价值。同时,cell-SELEX以其特有的优势避免了靶标的纯化过程,保留了细胞表面分子的天然构象,它涉及到的靶细胞衍生自各种组织或器官,如心、脑、肺、肝、肾、胰腺、乳腺、卵巢和结肠等[15],这对核酸适配体在相关疾病的诊断与治疗方面意义重大。体内SELEX(in vivo SELEX)相较细胞-SELEX面对更加复杂的筛选环境,它可在活体中筛选核酸适配体,Jing等[16]利用构建的肝内结直肠癌转移小鼠模型,通过静脉注射将2′-氟嘧啶修饰的RNA序列文库注入小鼠体内进行筛选,得到了RNA解旋酶的核酸适配体。
1.2.3 磁珠-SELEX将靶标固定在固体支持物上是一种比较常见的SELEX筛选方法,随着SELEX筛选的不断发展,固体支持物演变出了诸多种类,如微孔板、亲和层析柱、硝酸纤维素膜、表面等离子共振芯片、微流体芯片、琼脂糖珠、磁珠等;磁珠-SELEX(Mag-SELEX)的优势在于具有球形的展示面便于靶标的充分展示以及较为便捷的磁性分离特性,同时,各种亲和标签与相应的亲和树脂(如:组氨酸标签6xHis-与镍柱琼脂糖树脂、谷胱甘肽巯基转移酶标签GST-与谷胱甘肽树脂、麦芽糖结合蛋白标签MBP-与直链淀粉树脂)和偶联化学(如氨基、羧基和巯基等)使得该类方法具有广泛的应用范围[17]。Ye等[18]利用Mag-SELEX筛选到了氯霉素的核酸适配体。Xu等[19]将荧光标记和Mag-SELEX结合,即利用荧光磁珠(FluMag-SELEX)筛选到了多氯联苯的核酸适配体,并利用筛选到的核酸适配体构建金纳米探针用于检测多氯联苯,检出限为0.1-100 ng/mL。对于不易固定的小分子靶标,还可采用固定文库的方式,即捕获-SELEX(capture-SELEX),它可将核酸文库固定到磁珠上用于捕获易于结合的靶物质;Paniel等[20]设计初始文库时在单链核酸的随机区域引入一个对接序列,通过该序列与固定在磁珠上的捕获核酸以互补配对的方式将初始文库固定在磁珠上,通过capture-SELEX的方法筛选到了小分子靶标青霉素G的核酸适配体。基于Mag-SELEX及固定文库的筛选方法,Wang等[21]利用颗粒展示技术(Particle display),即通过乳化PCR将溶液中的单链核酸以105个拷贝的方式固定展示到每个磁珠上,然后与荧光标记的靶标孵育,通过流式细胞仪逐轮分选出结合能力强的单链核酸。
1.2.4 加尾SELEX初始文库中的核酸片段通常是由中间的随机区域,加上两边20 nt左右的固定区组成,加尾SELEX(Tailored SELEX)筛选所用的核酸序列则不含两边的固定序列。这一方面降低了固定区域由于参与随机区域的退火而对筛选造成的影响;另一方面则是回避了其他SELEX筛选方法需要面对的筛选后的截短处理等问题。加尾SELEX的具体做法是在随机文库的两端加上较短的固定序列,它与引物序列通过一段接头序列连接,经过PCR扩增之后,经碱裂处理掉两端序列后即可进入下一轮筛选[22]。类似的,基因组SELEX中用于PCR扩增的引物序列可能会与中间的基因组序列配对,干扰靶标与基因组序列的结合,Wen等结合加尾SELEX的原理,发展出了无引物基因组SELEX(primer-free genomic SELEX)[23];两种方法可将生物体基因组制成寡核苷酸文库,从中筛选生物活性分子的天然识别序列,为研究基因调控及代谢调节提供了新的技术平台[23]。
1.2.5 微流控SELEX微流控SELEX(Microfluidic SELEX)是将微流控芯片技术结合到了核酸适配体的筛选过程中的一种高效、快速的自动化筛选方法;它主要包括溶胶-凝胶微流控SELEX(Sol-gel microfluidic SELEX)和结合磁珠筛选的微流控SELEX(Magnetic bead-based microfluidic SELEX)。其中,溶胶-凝胶微流控SELEX是将靶标包裹在具有纳米多孔结构的硅酸盐玻璃制成的溶胶凝胶中,核酸经纳米多孔结构扩散到达靶标,基于磁珠的微流控SELEX则是将靶标固定在磁珠微球上,然后再在微流体芯片上完成富集工作。Bae等[24]利用溶胶-凝胶微流控SELEX经过7轮筛选得到了不需经化学修饰的小分子靶标黄嘌呤的核酸适配体,Kd值为4.2 µmol/L。基于磁珠的微流控SELEX技术先后出现了CMACS芯片系统、MMS芯片系统、其中MMS芯片引入了可被外部磁体磁化的铁磁材料制作的微通道,提高了分离效率,回避了CMACS芯片系统可能会面临的芯片通道内部微气泡引起的流体扭曲及磁珠在微通道的堆集等问题。另外,基于MMS系统也出现了很多改良,Wang等[25]在MMS芯片系统上增加了正筛选和负筛选单元,经7轮筛选后得到了肌红蛋白Myo的核酸适配体,Kd值在4.93 nmol/L和6.38 nmol/L之间;Huang等[26]在MMS芯片基础上加入了竞争性测定芯片用于检测核酸和靶标间的亲和力和特异性,并运用该系统成功筛选到了甲胎蛋白的核酸适配体,Kd值为2.7 nmol/L。Huang等[27]将筛选涉及的结合、分离和扩增3个步骤整合到了一个芯片上,每轮筛选仅需1 h左右,大大提高了筛选的效率。当然,微流控SELEX依赖于芯片的集成,而芯片的制作对专业性要求较高,这也在一定程度上限制了它的运用发展。
1.2.6 微阵列SELEX微阵列平台可被用于研究基因表达及蛋白-核酸互作,因此也有研究者将其用于SELEX筛选中来,即微阵列SELEX(Microarray SELEX),它的优势在于可以同时进行多项筛选,大大提高了筛选的效率。Liu等[28]利用蛋白质微阵列和微流控芯片结合的方法,通过将乳铁蛋白固定在蛋白质微阵列芯片上,然后与核酸文库相互作用,并利用牛血清白蛋白,α-乳清蛋白,β-乳球蛋白,酪蛋白设置了反筛,通过微阵列扫描仪实时监测富集过程,经7轮筛即得到了乳铁蛋白的核酸适配体,Kd值为1.04 ± 0.50 nmol/L。与大多数其他SELEX方法(~1016序列)中使用的随机库的大小相比,由于微阵列( < 105序列)的低容量,使用微阵列来进行SELEX筛选便受到一定程度的限制。Cho等[29]将微流控技术、高通量测序和原位核酸适配体阵列合成技术三者结合,构建了在筛选的同时定量测定核酸适配体亲和性和特异性的系统(QPASS),并且利用该系统筛选到了血管生成素Ang2的核酸适配体,Kd值最低在20.5 ± 7.3 nmol/L。
1.2.7 高通量SELEX传统的核酸适配体的筛选过程就像一个黑盒,它是通过重复的若干轮筛选后,挑取50-100个单克隆进行序列分析,这在一方面会导致某些高亲和力序列的丢失,另一方面,每一轮次级文库制备主要依赖于PCR扩增,但PCR往往存在偏好性扩增的现象,这会导致初始库中的低频高亲和力序列随着筛选的进行而不断丢失,而最后高频出现的序列也不一定就具有高的亲和能力。利用高通量测序技术对每一轮筛选进行高通量的序列测定和分析,可达到对整个富集过程的监测效果,不仅能及时的调整筛选压力以提高筛选效率,减少筛选的盲目性,而且可以更深层次的解析核酸适配体结构和功能间的关系。Cho等[30]利用微流控SELEX技术在靶向血小板衍生生长因子(PDGF-BB)的筛选过程中引入高通量测序技术,经3轮筛选得到Kd值小于3 nmol/L的核酸适配体,亲和力比传统SELEX方法提高了3-8倍,特异性提高了2-4倍。Dausse等[31]利用微流体芯片和表面等离子共振相结合的方式进行靶向X盒结合蛋白1(XBP1)的SELEX筛选,通过表面等离子共振装置对每一轮进行亲和力进行评估,在第4轮之后观测到了表面等离子信号的明显变化,最终经过六轮筛选得到了XBP1的核酸适配体,Kd值达8 nmol/L,通过对第2轮和第4轮进行高通量测序分析发现5′-GUUGCGG-3′基序的频率由4.5%上升至43%,也即在两轮筛选后,在没有观察到表面等离子信号的变化情况下也能看到富集的趋势,加快了筛选的进程。
1.3 次级文库的制备单链次级文库作为每一轮SELEX筛选的起点,如何保证每一轮次及文库中单链核酸的质量和产量是核酸适配体筛选成功与否的关键性问题之一。本文主要介绍如下几种常见的单链制备方法:不对称PCR法、酶解法、磁珠法、特殊引物结构法及其他一些综合性方法,并结合每种方法的自身特点,阐述利弊。
1.3.1 不对称PCR制备ssDNA利用不对称PCR制备ssDNA是核酸适配体筛选过程中次级文库制备最为常见的方法如图 2所示,它通过控制非限制引物与限制性引物的比例,一般为(50-100):1,达到制备ssDNA的目的。不对称PCR反应扩增初期的产物主要是dsDNA,当限制性引物用尽之后,非限制性引物引导下的线性扩增产物主要为ssDNA,整个过程中引物的比例至关重要,需要进行相关的优化实验[32]。不对称PCR制备单链简易又经济,但也存如下问题:其一,需要经过PAGE胶或者琼脂糖等手段将ssDNA和dsDNA进行分离来保证ssDNA的纯度;其二,PCR扩增过程会产生不同类型、不同长度的副产物[33],随着筛选的进行,需要及时的优化调整PCR条件才能确保ssDNA的质量符合要求。
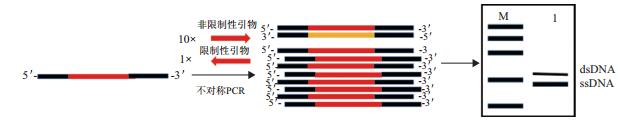
|
| 图 2 不对称PCR制备ssDNA的原理图 |
λ核酸外切酶的最佳作用底物是5′磷酸化的dsDNA,5′磷酸化的dsDNA经酶解作用之后就剩下了非磷酸化的单链核酸[34];λ核酸外切酶法制备ssDNA的过程如下(图 3):首先,利用带磷酸标记的下游引物经PCR扩增产生5′磷酸化的dsDNA;其次,借助λ核酸外切酶催化5′磷酸化的dsDNA,使其反义链从5′-磷酸末端逐步放出5′-单核苷酸而被降解掉;最后利用乙醇沉淀等方式将正义链回收即为所需的ssDNA。利用λ核酸外切酶法制备ssDNA操作简便,节约时间成本,但是伴随的酶解不完全等问题也会影响ssDNA的纯度。

|
| 图 3 酶切法制备ssDNA的原理图 |
磁珠法制备ssDNA综合利用了生物素与链霉亲和素之间较强的非共价相互作用(解离常数约为4×10-14 M)及磁性微球在磁性环境下易于操纵的优点,磁珠法制备ssDNA的过程如下(图 4):首先,利用生物素标记的引物经PCR扩增获得生物素化的dsDNA;其次,生物素化的dsDNA与链霉亲和素包被的磁珠在适当条件下孵育,形成dsDNA-生物素-链霉亲和素-磁珠系统;最后在NaOH的作用下,使dsDNA解链,不带生物素的核酸链经乙醇沉淀回收即为所需的ssDNA。借助磁珠这一介质本身的磁性特点,使ssDNA制备过程简单易行,目前被很多实验室广泛采用。但也有报道称NaOH可能会破坏生物素和链霉亲和素之间的疏水性,范德华力,氢键等相互作用,导致生物素标记的核酸链也解离到溶液中与互补链结合,从而影响单链核酸特定三级结构的形成。为了尽量减少该类情况,需要对NaOH的用量进行适当的探究[35]。

|
| 图 4 磁珠法制备ssDNA的原理图 |
不等长PCR法制备ssDNA的实质就是通过特殊设计的引物结构来进行PCR扩增,造成PCR产物长度上的差异,然后经琼脂糖或者PAGE胶进行分离后,切胶回收获得所需的单链核酸,原理如图 5。特殊引物结构的设计常见有如下两种:一种是下游引物在与模板的互补序列后面加上六乙二醇的间臂,阻止正义链的延伸,同时在间臂后面加上20个碱基左右的polyA来增加反义链与正义链的长度差异[36];另外一种的下游引物在其模板互补序列的后面加上一段富含GC的区域,富含GC的区域具有较高的Tm值,使其在PCR的延伸阶段保持茎环结构,从而阻止正义链的延伸,使得正义链和反义链间形成长度差异[37]。
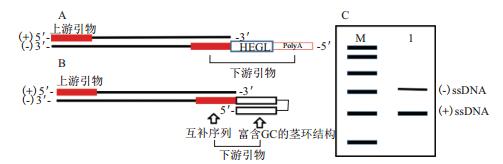
|
| 图 5 利用特殊引物结构制备ssDNA的原理图 |
除了上述几种常见的制备ssDNA的方法,还有一些综合性的方法被报道,如利用不对称PCR和磁珠法结合来制备单链。该方法流程如图 6。首先,利用对称PCR扩增得到生物素化的PCR产物;其次,将该生物素化的PCR产物再通过生物素标记的引物经不对称PCR扩增,将得到的PCR产物与链霉亲和素包被的磁珠作用;最后,经乙醇沉淀回收即可得到目的ssDNA。基于PCR的单链制备过程会面临副产物的问题,不同长度的单链作为副产物影响实验结果但是又很难定量和清除,磁珠法制备单链则面临生物素化单链解离等问题。若把这两种方法结合起来,能够很好的回避这两个难题[38]。
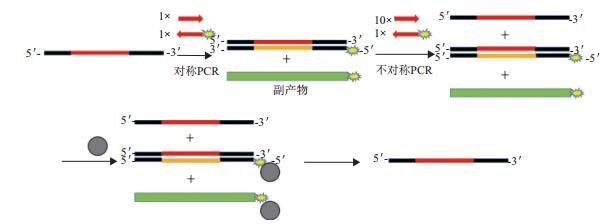
|
| 图 6 利用综合性方法制备ssDNA的原理图 |
随着核酸适配体研究的深入,SELEX筛选之后的Post-SELEX阶段也越来越受到人们关注。如果说目前的SELEX筛选过程是一个“黑盒”,那么Post-SELEX阶段就是一个试错的过程。Post-SELEX主要是对筛选到的核酸适配体进行相关的修饰改造,以期获得性能更佳的核酸适配体。Macugen可治疗年龄相关性黄斑变性,也是首个基于核酸适配体的药物,它的做法是在鉴定和截短初始适体序列后,对其尾端做了2’-O-Me,2’-NH2和2’-F等序列修饰,使得最终核酸适配体的亲和力、IC50和生物半衰期均提高了数量级,进而进入测试并被批准上市[39]。常见的Post-SELEX修饰方法有截短、化学修饰、重组、定点突变等。截短主要是将一些与靶标相互作用不重要的部分移除。Gao等[40]通过对获得的漆沟藻毒素的核酸适配体进行截短,获得的核心区域具有17.7 nmol/L的较低的Kd值。化学修饰常见于核苷酸的糖环、碱基及磷酸骨架部位[41]。Lanford等[42]发现锁核酸(Locked Nucleic acid)通过与miR-122互补可对丙型肝炎病毒有效持久的抑制,其中锁核酸是一种双环状的核苷酸衍生物。Zhang等[43]构建多价核酸适配体的给药系统,获得了更强的靶标结合力及细胞内化能力。Zheng等[44]通过定点突变和优化截短将石房蛤毒素核酸适配体的亲和力提高了30多倍。
3 展望核酸适配体作为一种新型分子识别工具,其在分析检测、临床诊断、疾病靶向治疗、新药研发、生物标志物发现、生物成像等领域发展潜力巨大,但SELEX筛选的不同环节往往会遇到不同的问题,这使得目前为止并没有一个标准的程序可以适用于各种不同靶标,筛选和表征仍是现阶段制约核酸适配体应用发展的关键问题。本文针对SELEX筛选的不同环节阐述了相应的改良与尝试,相信随着微流体、微阵列、纳米技术、新型测序方法及计算工具等新技术新方法与SELEX技术的不断融合和发展,核酸适配体的筛选与表征将会更加的方便快捷,并逐渐走向标准化、自动化。另外,针对目前存在的不足,核酸适配体的研究可以加强以下几个方面的研究:(1)发展和完善核酸适配体相关的数据库信息,将不同领域筛选到的适配体序列信息包括选择条件、结构特征、亲和力、特异性、解离常数等相关参数收集起来,综合数据库的发展可以为筛选提供一定的参考和指导;(2)加强核酸适配体与靶标互作关系的研究,找出结合位点作用机制,或者结合不同靶标自身结构特点,设计模拟出可能的核酸适配体序列,增大筛选的成功率。
| [1] | Radom F, Jurek PM, Mazurek MP, et al. Aptamers:molecules of great potential[J]. Biotechnology Advances, 2013, 31 (8): 1260–1274. DOI:10.1016/j.biotechadv.2013.04.007 |
| [2] | Luo X, Mckeague M, Pitre S, et al. Computational approaches toward the design of pools for the in vitro selection of complex aptamers[J]. RNA, 2010, 16 (11): 2252–2262. DOI:10.1261/rna.2102210 |
| [3] | Blind M, Blank M. Aptamer Selection Technology and Recent Advances[J]. Molecular Therapy-Nucleic Acids, 2015, 4 (1): e223. |
| [4] | Choi EW, Nayak LV, Bates PJ. Cancer-selective antiproliferative activity is a general property of some G-rich oligodeoxynucleotides[J]. Nucleic Acids Research, 2010, 38 (5): 1623–1635. DOI:10.1093/nar/gkp1088 |
| [5] | Santosh B, Yadava PK. Nucleic acid aptamers:research tools in disease diagnostics and therapeutics[J]. Biomed Research International, 2014, 2014 (4): 540451–540463. |
| [6] | Ahmad KM, Xiao Y, Soh HT. Selection is more intelligent than design:improving the affinity of a bivalent ligand through directed evolution[J]. Nucleic Acids Research, 2012, 40 (22): 11777–11783. DOI:10.1093/nar/gks899 |
| [7] | Hao Q, Csordas AT, Wang J, et al. Rapid and label-free strategy to isolate aptamers for metal ions[J]. Acs Nano, 2016, 10 (8): 7558–7565. DOI:10.1021/acsnano.6b02558 |
| [8] | Yu H, Zhang S, Chaput JC. Darwinian evolution of an alternative genetic system provides support for TNA as an RNA progenitor[J]. Nature Chemistry, 2012, 4 (3): 183–187. DOI:10.1038/nchem.1241 |
| [9] | Kimoto M, Yamashige R, Matsunaga K, et al. Generation of high-affinity DNA aptamers using an expanded genetic alphabet[J]. Nature Biotechnology, 2013, 31 (5): 453–457. DOI:10.1038/nbt.2556 |
| [10] | Sefah K, Yang Z, Bradley KM, et al. In vitro selection with artificial expanded genetic information systems[J]. Proc Natl Acad Sci U S A, 2013, 111 (4): 1449–54. |
| [11] | 杨歌, 魏强, 赵新颖, 等. 蛋白质的核酸适配体筛选的研究进展[J]. 色谱, 2016, 34(4): 370–381. |
| [12] | Mendonsa SD, Bowser MT. In vitro evolution of functional DNA using capillary electrophoresis[J]. Journal of the American Chemical Society, 2004, 126 (1): 20–21. DOI:10.1021/ja037832s |
| [13] | Yang J, Bowser MT. CE-SELEX selection of catalytic DNA aptamers for a small molecule porphyrin target[J]. Analytical Chemistry, 2013, 85 (3): 1525–1530. DOI:10.1021/ac302721j |
| [14] | Chen M, Yu Y, Jiang F, et al. Development of Cell-SELEX technology and its application in cancer diagnosis and therapy[J]. International Journal of Molecular Sciences, 2016, 17 (12): 2079. DOI:10.3390/ijms17122079 |
| [15] | Zhao L, Tan W, Fang X. Introduction to aptamer and cell-SELEX[M]// Tan W, Fang X. Aptamers Selected by Cell-SELEX for Theranostics. Springer-Verlag Benlon Heidelberg, 2015:1-11. |
| [16] | Jing M, Ray P, Liu J, et al. In vivo selection against human colorectal cancer xenografts identifies an aptamer that targets RNA helicase Protein DHX9[J]. Molecular Therapy-Nucleic Acids, 2016, 5 (4): e315. |
| [17] | Ozer A, Pagano JM, Lis JT. New technologies provide quantum changes in the scale, speed, and success of SELEX methods and aptamer characterization[J]. Molecular Therapy Nucleic Acids, 2014, 3 (2): e183. |
| [18] | Ye D, Gao Z, Wang L, et al. Selection and identification of chloramphenicol-specific DNA aptamers by Mag-SELEX[J]. applied Biochemistry & Biotechnology, 2016, 180 (8): 1–13. |
| [19] | Xu S, Yuan H, Chen S, et al. Selection of DNA aptamers against polychlorinated biphenyls as potential biorecognition elements for environmental analysis[J]. Analytical Biochemistry, 2012, 423 (423): 195–201. |
| [20] | Paniel N, Istamboulié G, Triki A, et al. Selection of DNA aptamers against penicillin G using Capture-SELEX for the development of an impedimetric sensor[J]. Talanta, 2017, 162 : 232–240. DOI:10.1016/j.talanta.2016.09.058 |
| [21] | Wang J, Gong Q, Maheshwari N, et al. Particle display:a quantitative screening method for generating high-affinity aptamers[J]. Angewandte Chemie, 2014, 53 (19): 4796–4801. DOI:10.1002/anie.201309334 |
| [22] | Vater A, Jarosch F, Buchner K, et al. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach:tailored-SELEX[J]. Nucleic Acids Research, 2003, 31 (31): e130. |
| [23] | 邵宁生, 李少华, 黄燕苹. SELEX技术及Aptamer研究的新进展[J]. 生物化学与生物物理进展, 2006, 33(4): 329–335. |
| [24] | Bae H, Ren S, Kang J, et al. Sol-gel SELEX circumventing chemical conjugation of low molecular weight metabolites discovers aptamers selective to xanthine[J]. Nucleic Acid Therapeutics, 2013, 23 (6): 443–449. DOI:10.1089/nat.2013.0437 |
| [25] | Wang Q, Liu W, Xing Y, et al. Screening of DNA aptamers against myoglobin using a positive and negative selection units integrated microfluidic chip and its biosensing application[J]. Analytical Chemistry, 2014, 86 (13): 6572–6579. DOI:10.1021/ac501088q |
| [26] | Huang CJ, Lin HI, Shiesh SC, et al. An integrated microfluidic system for rapid screening of alpha-fetoprotein-specific aptamers[J]. Biosensors & Bioelectronics, 2012, 35 (1): 50–55. |
| [27] | Huang CJ, Lin HI, Shiesh SC, et al. Integrated microfluidic system for rapid screening of CRP aptamers utilizing systematic evolution of ligands by exponential enrichment(SELEX)[J]. Biosensors & Bioelectronics, 2010, 25 (7): 1761–1766. |
| [28] | Liu X, Li H, Jia W, et al. Selection of aptamers based on a protein microarray integrated with a microfluidic chip[J]. Lab on A Chip, 2016, 17 (1): 178–185. |
| [29] | Cho M, Soo OS, Nie J, et al. Quantitative selection and parallel characterization of aptamers[J]. Proceedings of the National Academy of Sciences of the United states of America, 2013, 110 (46): 18460–18465. DOI:10.1073/pnas.1315866110 |
| [30] | Cho M, Xiao Y, Nie J, et al. Quantitative selection of DNA aptamers through microfluidic selection and high-throughput sequencing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107 (35): 15373–15378. DOI:10.1073/pnas.1009331107 |
| [31] | Dausse E, Barré A, Aimé A, et al. Aptamer selection by direct microfluidic recovery and surface plasmon resonance evaluation[J]. Biosensors & Bioelectronics, 2016, 80 (8): 418–425. |
| [32] | Citartan M, Tang TH, Tan SC, et al. Asymmetric PCR for good quality ssDNA generation towards DNA aptamer production[J]. Songklanakarin Journal of Science & Technology, 2012, 34 (2): 125–131. |
| [33] | Tolle F, Wilke J, Wengel J, et al. By-product formation in repetitive PCR amplification of DNA libraries during SELEX[J]. PLoS One, 1932, 9 (12). |
| [34] | Citartan M, Tang TH, Tan SC, et al. Conditions optimized for the preparation of single-stranded DNA(ssDNA)employing lambda exonuclease digestion in generating DNA aptamer[J]. World Journal of Microbiology and Biotechnology, 2011, 27 (5): 1167–1173. DOI:10.1007/s11274-010-0563-8 |
| [35] | Wakimoto Y, Jiang J, Wakimoto H. Isolation of single-stranded DNA[J]. Current Protocols in Molecular Biology, 2014, 107 : 2. |
| [36] | Stoltenburg R, Reinemann C, Strehlitz B. SELEX-a(r)evolutionary method to generate high-affinity nucleic acid ligands[J]. Biomolecular Engineering, 2007, 24 (4): 381–403. DOI:10.1016/j.bioeng.2007.06.001 |
| [37] | Cao X, Li S, Chen L, et al. Combining use of a panel of ssDNA aptamers in the detection of Staphylococcus aureus[J]. Nucleic Acids Research, 2009, 37 (14): 4621–4628. DOI:10.1093/nar/gkp489 |
| [38] | Guo WM, Kong KW, Brown CJ, et al. Identification and characterization of an eIF4e DNA aptamer that inhibits proliferation with high throughput sequencing[J]. Molecular Therapy Nucleic Acids, 2014, 3 (12): e217. |
| [39] | Ruckman J, Green LS, Beeson J, et al. 2′-Fluoropyrimidine RNA-based aptamers to the 165-amino acid form of vascular endothelial growth factor(VEGF165). Inhibition of receptor binding and VEGF-induced vascular permeability through interactions requiring the exon 7-encoded domain[J]. Journal of Biological Chemistry, 1998, 273 (32): 20556–20567. DOI:10.1074/jbc.273.32.20556 |
| [40] | Gao S, Hu B, Zheng X, et al. Gonyautoxin 1/4 aptamers with high-affinity and high-specificity:From efficient selection to aptasensor application[J]. Biosensors & Bioelectronics, 2016, 79 : 938–944. |
| [41] | Wang RE, Wu H, Niu Y, et al. Improving the stability of aptamers by chemical modification[J]. Current Medicinal Chemistry, 2011, 18 (27): 4126–4138. DOI:10.2174/092986711797189565 |
| [42] | Lanford RE, Hildebrandteriksen ES, Petri A, et al. Therapeutic silencing of microRNA-122 in primates with chronic hepatitis C virus infection[J]. Science, 2010, 327 (5962): 198–201. DOI:10.1126/science.1178178 |
| [43] | Zhang Z, Ali MM, Eckert MA, et al. A polyvalent aptamer system for targeted drug delivery[J]. Biomaterials, 2013, 34 (37): 9728–35. DOI:10.1016/j.biomaterials.2013.08.079 |
| [44] | Zheng X, Hu B, Gao SX, et al. A saxitoxin-binding aptamer with higher affinity and inhibitory activity optimized by rational site-directed mutagenesis and truncation[J]. Toxicon Official Journal of the International Society on Toxinology, 2015, 7 : 41–47. |




