红豆杉(Taxus chinensis R.)别名紫杉,系红豆杉科红豆杉属常绿乔木,根据其生长地域性和生物学特征型,在全世界可分为至少14个品种[1-2]。对红豆杉的研究热点起源于1971年美国化学家Wani等[3]从短枝红豆杉的树皮中分离出具有强抗癌活性的紫杉烷二萜类化合物紫杉醇。随后,1979年Horwitz和Schiff等[4]发现紫杉醇具有强效的抗肿瘤作用,其作用机制独特,是通过与微管蛋白质结合,使细胞有丝分裂受阻,从而对抑制肿瘤细胞的生长。1992年,美国FDA批准紫杉醇上市。目前,紫杉醇是治疗乳腺癌和转移性卵巢癌的最好药物之一,同时紫杉醇对肺癌、食道癌也有显著疗效,对肾炎及细小病毒炎症也有显著的治疗效果[5]。除紫杉醇外,红豆杉中还有很多其他的生物活性成分[6]。由此可见,红豆杉作为世界上濒临灭绝的天然珍稀植物,不但有极高的经济价值,更具有重要的的药用价值。如今红豆杉主要应用于紫杉醇提取,但由于红豆杉树中紫杉醇的低含量,导致红豆杉在被大量需求的同时也造成大量浪费,影响红豆杉的开发应用。
近年来,随着高通量测序技术等分子生物学手段的迅速发展,包括miRNA在内的红豆杉转录组学研究也取得了一定的成就[7-11]。miRNA是一类内源性非编码的小分子单链RNA,长度在19-24个核苷酸(nt)之间,在动植物发育过程中发挥着复杂且重要的作用。红豆杉中存在着大量的miRNA,并对红豆杉植株本身生长发育以及紫杉醇生物合成具有重要作用[12],但由于红豆杉基因组信息的缺乏,阻碍了红豆杉miRNA的研究,而获取高质量总RNA是进行miRNA研究的重要前提。
植物组织中RNA提取比较困难,由于植物中多酚、多糖等对RNA提取的干扰和RNA酶对RNA的降解,导致难以有效地分离纯化植物组织中的RNA。到目前为止,提取植物总RNA的方法已有不少[13, 14],其中最常见的是Trizol法、CATB法及试剂盒。Trizol法快速、高效,用时较短,但得到的RNA往往杂质较多,需要进一步纯化。CATB法获得的RNA纯度高、完整性较好,但操作较复杂、提取时间较长,缺乏有效的RNA酶抑制剂,容易被RNA酶降解。使用试剂盒提取植物总RNA速度快、用时短,并含有有效的抑制RNase的活性成分,但一般价格昂贵,一次提取量很少。此外,现有的提取方法均不能实现植物RNA的大量提取,无法满足后续试验需要。因此在现有的方法上进行改良,进而得到大量高质量的RNA是非常有必要的。考虑成本问题,本研究在已有的植物总RNA提取方法Trizol法和CATB法[15]的基础上进行优化,初步实现了高质量RNA的大量制备,通过凝胶柱层析法显著提高红豆杉总RNA的纯度并能得到了富含miRNA的小分子RNA。
1 材料与方法 1.1 材料 1.1.1 原料与试剂红豆杉叶,采自湖南长沙麓溪峪休闲度假山庄,取新鲜叶液氮保存;拟南芥miR156对照品,购自广州市锐博生物科技有限公司,纯度为98%;尿嘧啶核苷酸对照品,购自上海楷洋生物技术有限公司,纯度≥98%;RNase A,购自上海碧云天生物技术有限公司;实验用水为超纯水;其他的试剂均为分析纯。
1.1.2 仪器与设备恒温加热水浴锅,郑州长城科工贸有限公司;电子天平,奥豪斯国际贸易(上海)有限公司;闪式提取器,河南智晶生物科技股份有限公司;低温高速离心机,长沙英泰仪器有限公司;高效液相色谱仪,LC-2010A,Shimadzu corporation Kyoto Japan;BioSpec-nano紫外分光光度计,Shimadzu corporation Kyoto Japan;数控超声清洗器,昆山市超声仪器有限公司;Pre-HPLC北京创新通恒科技有限公司。
1.2 方法 1.2.1 CATB法 1.2.1.1 焦碳酸二乙酯(Diethy pyrocarbonate,DEPC)水溶液的配制焦碳酸二乙酯2mL,加入1 000 mL蒸馏水,室温搅拌4 h后静置12 h以上,使用前121℃高压灭菌30 min。
1.2.1.2 CTAB提取液的配制CTAB 10 g,PVP 10 g,NaCl 93.6 g,EDTA 7.306 g,加入已灭菌的DEPC水溶液400 mL,置于65℃水浴锅水浴1 h,趁热使用。
1.2.1.3 总RNA提取(1)称取新鲜红豆杉叶片100 g,加入400 mL CTAB提取液,β-巯基乙醇10 mL,闪式提取器高速涡旋15 min后,置于65℃水浴锅水浴30 min。(2)涡旋液转入500 mL离心瓶中,3 500 r/min离心10 min。(3)上清液中加入等体积氯仿:异戊醇(24:1,V/V),在4℃,10 000 r/min离心10 min。(4)将上清移至新离心管中,加入1/4体积10 mol/L LiCl,混匀,4℃沉淀过夜。(5)4℃,10 000 r/min离心10 min,沉淀用75%乙醇洗涤两次,4℃,10 000 r/min离心5 min,获取沉淀,-20℃保存备用。(6)沉淀用N2吹干至无乙醇味,加入2 mL已灭菌的DEPC水溶液至沉淀恰好溶解。取少量样品进行HPLC分析和紫外分光光度测定。
1.2.2 Trizol法 1.2.2.1 Trizol提取液的配制异硫氰酸胍125 g,8-羟基喹啉0.1 g,加入灭菌后DEPC水溶液149 mL,0.75 mol/L柠檬酸钠溶液8.8 mL,10%十二烷基肌氨酸钠溶液13.2 mL,2 mol/L NaAc溶液25 mL,重蒸酚250 mL,混合均匀,4℃储存备用。
1.2.2.2 总RNA提取(1)称取新鲜红豆杉叶片24 g,加入240 mL Trizol提取液,闪式提取器高速涡旋2 min。(2)涡旋液室温放置30 min,使样品充分裂解后,转入500 mL离心瓶中,3 500 r/min离心5 min。(3)上清液中加入1/5体积氯仿,振摇混匀后于4℃,10 000 r/min离心15 min。(4)上清液加入等体积异丙醇,室温放置20 min,于4℃,10 000 r/min离心10 min,去上清,获取沉淀。(5)沉淀用适量75%乙醇洗涤2-3次,4℃,10 000 r/min离心5 min,取沉淀,-20℃储存备用。(6)沉淀用N2吹干至无乙醇味,加入2 mL已灭菌的DEPC水溶液使沉淀恰好溶解。取少量样品进行HPLC分析和紫外分光光度测定。
1.2.3 Trizol法的条件优化 1.2.3.1 Trizol提取液作用体积的优化以1.2.2.2的RNA提取方法为基本条件,称取新鲜红豆杉叶6 g,按叶液比(m/V)3:20、3:30、3:40、3:50、3:60,分别加入Trizol提取液,其余操作相同,得到用同样体积的DEPC水溶液溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品的浓度与纯度,以确定最佳的Trizol提取液作用体积。
1.2.3.2 裂解时间的优化以1.2.2.2的RNA提取方法为基本条件,将涡旋液分别在室温中放置5 min、10 min、15 min、20 min、25 min、30 min,其余操作相同,得到用同样体积的DEPC水溶液溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品的浓度与纯度,以确定最佳的样品裂解时间。
1.2.3.3 氯仿作用量的优化以1.2.2.2的RNA提取方法为基本条件,按上清液与氯仿比(V/V)1:1/5、1:1/4、1:1/3、1:1/1、1:1,加入氯仿,其余操作相同,得到用同样体积的DEPC水溶液溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品的浓度与纯度,以确定最佳的氯仿作用量。
1.2.4 红豆杉RNA检测 1.2.4.1 高效液相色谱法分析色谱条件:Shodex OHpak SB-804 HQ凝胶柱(8.0 mm×300 mm;10 μm),流动相:0.02 mol/L磷酸盐缓冲液—0.1‰叠氮钠;流速:0.5 mL/min;检测波长:260 nm;进样量:10 μL。
拟南芥miR156对照品溶液的配制:取拟南芥miR156双链对照品5 nmol(约100 μg),置于1.5 mL离心管内,加入500 μL灭菌后DEPC水溶液溶解,摇匀即得,过0.45 μm滤膜,置于4℃冰箱冷藏,备用。
尿嘧啶核苷酸对照品溶液的配制:精密称取尿嘧啶核苷酸对照品5.03 mg,置于1.5 mL离心管内,加入1 mL灭菌后DEPC水溶液溶解,摇匀即得,过0.45 μm滤膜,置于4℃冰箱冷藏,备用。
红豆杉总RNA供试品溶液的配制:经不同方法提取的红豆杉总RNA沉淀,按红豆杉叶:DEPC水溶液(m/V = 25:2)的比例加入已灭菌的DEPC水溶液使沉淀溶解,涡旋混匀,过0.45 μm滤膜,即得。
红豆杉RNA酶解溶液的配制:经Trizol法提取的红豆杉RNA沉淀,加入灭菌后DEPC水溶液溶解至浓度为100 μg/mL,经检测无RNA降解后,按每9 μg样品量加入10 μL RNase A的比例加入RNase A,于37℃中保温60 min,即得。
1.2.4.2 紫外分光光度测定经不同方法提取的红豆杉总RNA沉淀,加入已灭菌的DEPC水溶液至沉淀恰好溶解,涡旋混匀,吸取2 μL,检测样品在260 nm下的吸光度与浓度,测定样品的OD260/280与OD260/230的比值。
1.2.5 红豆杉总RNA的纯化 1.2.5.1 PVP法以1.2.2.2的RNA提取方法为基本条件,按物料比(m/m)1:0.1在Trizol提取液中加入PVP,其余操作相同,得到DEPC水溶液恰好溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品的浓度与纯度。
1.2.5.2 β-巯基乙醇法以1.2.2.2的RNA提取方法为基本条件,按物料比(m/V)2:1在Trizol提取液中加入β-巯基乙醇,其余操作相同,得到DEPC水溶液恰好溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品的浓度与纯度。
1.2.5.3 高盐沉淀法以1.2.2.2的RNA提取方法为基本条件,上清液加入1/2体积异丙醇和1/2体积高盐溶液(1.2 mol/L NaCl)共同沉淀,室温放置20 min,于4℃,10 000 r/min离心10 min,弃去上清液,获取沉淀,其余操作相同,沉淀用DEPC水溶液恰好溶解,HPLC和紫外分光光度测定,计算样品的浓度与纯度。
1.2.5.4 低浓度乙醇沉淀法以1.2.2.2的RNA提取方法为基本条件,氯仿抽提后,上清液加入1/15体积3 mol/L NaAc(pH 5.2)和1/5体积的无水乙醇,振荡混匀,冰浴静置1 h,促进多糖沉淀,于4℃,10 000 r/min离心10 min,取上清液,加入等体积异丙醇,其余操作相同,得到DEPC水溶液恰好溶解的红豆杉总RNA样品,HPLC和紫外分光光度测定,计算样品浓度与纯度。
1.2.5.5 凝胶柱层析法称取新鲜红豆杉叶25 g,以1.2.2.2的RNA提取方法为基本条件提取红豆杉总RNA,获得沉淀用4 mL灭菌后DEPC水溶液溶解,Sephadex G-100型凝胶装柱(15 mm×60 cm),湿法上样,流速约为0.35 mL/min,灭菌后DEPC水溶液洗脱约一个柱体积后接取流分,合并,冻干后加入500 μL灭菌后DEPC水溶液溶解,HPLC分析和紫外分光光度测定,计算样品浓度与纯度。
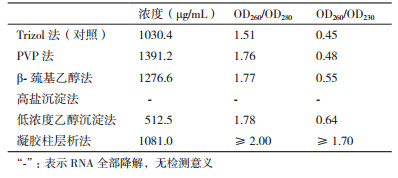
2 结果 2.1 红豆杉RNA的HPLC分析图 1是红豆杉RNA的HPLC分析图谱,16 min至17 min出峰的主要是富含miRNA的小分子RNA成分,而18 min至22 min出峰的则是红豆杉RNA降解产生的碎片和核苷酸类成分。
|
| 图 1 红豆杉RNA的HPLC分析图谱 |
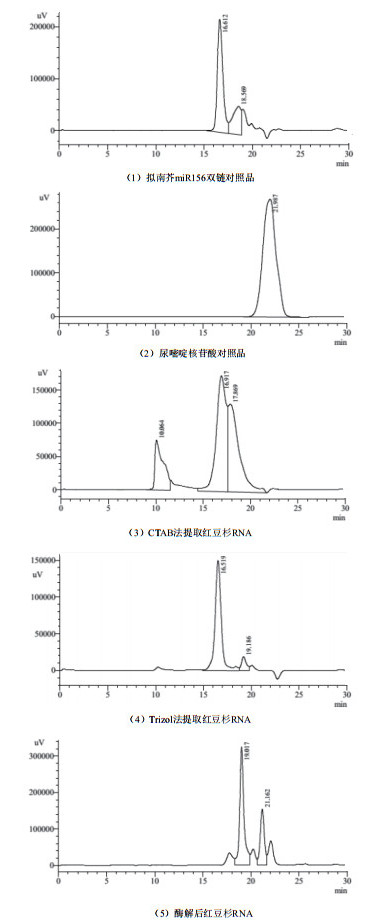
HPLC结果显示,不同量Trizol提取液作用下,红豆杉总RNA没有出现明显的降解现象,可以用于进一步考察分析。由图 2可知,随着Trizol提取液作用体积的增加,红豆杉总RNA的OD260/OD230比值和OD260/OD280比值没有显著差别,但浓度逐渐增大,达到3:40时转向平稳。因为Trizol提取液能迅速破碎细胞,使核蛋白复合体中的蛋白质变性沉淀并释放出RNA,作用体积过小,RNA释放不完全,作用体积过大,会增加反应成本,并导致提取时间延长,增大RNA的降解可能。因此综合考虑,红豆杉叶与Trizol提取液的作用体积比可以控制为3:40。
|
| 图 2 Trizol提取液作用体积对红豆杉总RNA的影响 |
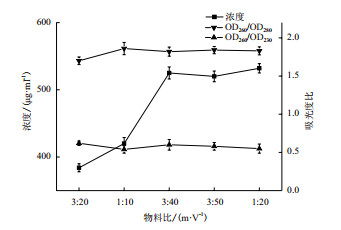
HPLC结果显示,在不同裂解时间的作用下,红豆杉总RNA没有出现明显的降解现象,可以用于进一步考察分析。由图 3可知,随着裂解时间的增加,红豆杉总RNA的OD260/OD230比值、OD260/OD280比值均没有显著差别,而浓度在20 min时有小幅度的下降,当延长至30 min时又回升,但影响没有统计学差异,这是由于闪式提取器的高速涡旋作用和Trizol提取液的强裂解性,能迅速破碎植物细胞并释放出RNA,因此裂解时间的延长对红豆杉RNA的提取量基本没有影响。考虑时间过长可能会增加红豆杉RNA降解的可能性,可以将裂解时间控制为5 min。
|
| 图 3 裂解时间对红豆杉RNA的影响 |
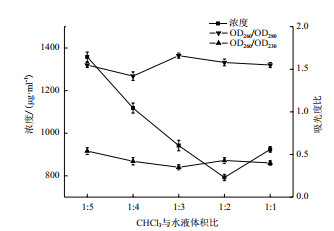
HPLC结果显示,在不同氯仿量的作用下,红豆杉总RNA没有出现明显的降解现象,可以用于进一步考察分析。由图 4可知,随着氯仿作用体积的增加,红豆杉总RNA的OD260/OD230比值和OD260/OD280比值没有显著差别,但浓度逐渐降低,达到1:1时有小幅度回升。产生浓度降低的原因可能是氯仿作用体积过高,导致蛋白质发生大量迅速的变性沉淀,同时裹携RNA共同沉淀,致使红豆杉RNA的提取量减少。但氯仿作用量过小,可能造成蛋白质沉降不完全,影响RNA的纯度。因此,综合考虑,确定氯仿与水液的作用体积比为1:5。
|
| 图 4 氯仿作用量对红豆杉总RNA的影响 |
通过考察Trizol提取液作用体积、裂解时间及氯仿作用量对红豆杉总RNA的影响,得到红豆杉叶中总RNA的较优提取条件为:红豆杉叶与Trizol提取液的作用体积比为3:40;裂解时间为5 min;氯仿与水液的体积比为1:5。经该条件提取后,可得到浓度约为1 100 μg/mL,OD260/OD280在1.6左右及OD260/OD230在0.5左右的红豆杉总RNA样品。
2.3 红豆杉总RNA的纯化结果结果如表 1所示,在同样的提取条件下,PVP法和β-巯基乙醇法虽然能提高红豆杉总RNA的提取浓度,并没有表现出明显的纯化效果,酚类物质没有很好的去除。低浓度乙醇沉淀法有一定的纯化效果,但在去除多糖的同时裹携部分RNA一同沉降,大幅度降低了总RNA的提取浓度。凝胶柱层析法能显著的提高红豆杉总RNA的纯度,除去其中的酚类、多糖、蛋白质等杂质。通过对不同的红豆杉总RNA的纯化方法的考察,确定使用凝胶柱层析法纯化总RNA,最终可以得到浓度约为1 000 μg/mL OD260/OD280在2.00-2.20之间及OD260/OD230大于1.70的高质量RNA样品。
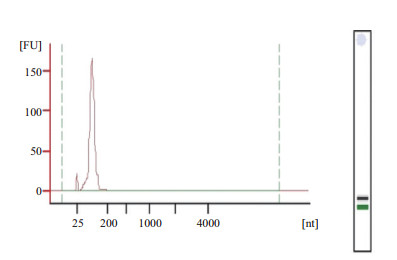
取经过Sephadex G-100型凝胶柱纯化后的冻干粉,加入500 μL灭菌后DEPC水溶解。应用Agilent 2100 Bioanalyzer测定分离得到的小分子RNA的纯度及完整性,由图 5可知,所得的小分子RNA分离干净,纯度较好,无显著的降解现象(OD260/OD280 = 2.15,OD260/OD230 = 1.81)。达到sRNA文库构建的要求,后续可以进行HiSeq测序及miRNA的鉴定。
|
| 图 5 红豆杉miRNA的分离结果 |
由于植物中富含多酚、多糖类次级代谢物质,严重干扰植物总RNA的提取,并会影响逆转录,基因扩增,文库构建等下续操作。CTAB法和Trizol法均能提取得到红豆杉总RNA,但前者在提取过程中更容易产生较大程度的降解,形成大量的RNA碎片与核苷酸。防止RNA大量降解是RNA提取过程中首先要注意的问题,植物组织破碎瞬间释放的内源性RNA酶及提取过程中所用器材等携带的外源性RNA酶都有可能将RNA降解,因此抑制RNA酶的活性至关重要。Trizol法中使用的异硫氰酸胍是有效的RNA酶抑制剂,在裂解组织的同时能对RNA酶产生强烈的变性作用,此外8-羟基喹啉及重蒸酚也可以造成一定的抑制作用,减少红豆杉RNA降解,得到比较完全的红豆杉RNA。而CTAB法中所用的提取试剂缺乏强有力的RNA酶抑制剂,使得RNA在提取过程中容易被RNA酶降解,若额外再加入RNA酶抑制剂,则导致提取成本提高,不利于红豆杉RNA的大量制备。而市场上研制的一些专门用于提取植物总RNA的试剂盒,虽然提取总RNA速度快,用时短,但价格昂贵,一次提取量很少,不能满足后续的需求。因此,综合考虑,选择使用Trizol法提取红豆杉总RNA。PVP能与酚类化合物形成螯合物,使之不能成为多酚氧化酶的底物而抑制酚类的氧化[16];β-巯基乙醇法具有强还原性,能抑制酚类物质氧化成醌类[17];低浓度乙醇和高盐能沉淀多糖,分离多糖与RNA[18-20]。但PVP法、β-巯基乙醇、低浓度乙醇和高盐沉淀法不能得到明显的纯化效果。而凝胶柱层析法能显著的提高红豆杉总RNA的纯度,除去其中的酚类、多糖、蛋白质等杂质,这为以后纯化高质量的RNA提供一个有效快速的方法。
本研究方法与现有的提取方法相比虽然转移率略有降低,但大大降低了成本,并首次实现了植物RNA的大量提取,为其后续试验研究提供充足的物质基础,便于进一步植物miRNA的研究。整个提取过程,具有简便、快速的特点,可为其他植物总RNA的提取提供参考。
4 结论本实验以湖南长沙红豆杉叶为材料,建立以Trizol法提取RNA,用闪式提取器快速大量提取植物总RNA的方法。从红豆杉叶总RNA的提取到纯化,考虑降解因素、提取效率、样品纯度等因素,确定了较优提取条件:红豆杉叶与Trizol提取液的作用体积确定为3:40;裂解时间确定为5 min;氯仿与水液的作用体积确定为1:5;在总RNA的研究基础上使用凝胶柱层析法对红豆杉miRNA进行分离纯化,得到富含miRNA的小分子RNA,且凝胶柱层析法能显著的提高红豆杉总RNA的纯度,除去其中的酚类、多糖、蛋白质等杂质,确定了高质量红豆杉总RNA提取纯化方案。最终平均每25 g红豆杉叶中可提取得到总RNA样品2 200 μg左右,转移率约十万分之八,首次实现了植物总RNA的大量提取。
| [1] | Hao DC, Xiao PG, Yang L. Phylogenetic relationships of the genus Taxus inferred from chloroplast intergenic spacer and nuclear coding DNA[J]. Biol Pharm Bull, 2008, 31 (2): 260–265. DOI:10.1248/bpb.31.260 |
| [2] | Hao DC, Xiao PG, Huang BL, et al. Interspecific relationships and origins of Taxaceae and Cephalotaxaceae revealed by partitioned Bayesian analyses of chloroplast and nuclear DNA sequences[J]. Plant Syst Evol, 2008, 276 (1-2): 89–104. DOI:10.1007/s00606-008-0069-0 |
| [3] | Wani MC, Taylor HL, Harold L, et al. Plant antitumor agents. Ⅵ. Isolation and structure of taxol, a novel antileukemic and antitumor agent from Taxus brevifolia[J]. J Amer Chem Soc, 1971, 93 (9): 2325–2327. DOI:10.1021/ja00738a045 |
| [4] | Schiff PB, Fant J, Horwitz SB, et al. Promotion of microtubule assembly in vitro by taxol[J]. Nature(London), 1979, 277 (5698): 665–667. DOI:10.1038/277665a0 |
| [5] | 张静. 植物红豆杉的抗癌药用价值研究[J]. 中国药业, 2014(1): 1–3. |
| [6] | 李良松, 冯仲科, 刘德庆. 红豆杉名实与功用通考[J]. 中国中药杂志, 2011(12): 1682–1685. |
| [7] | Martin RC, Liu PP, Nonogaki H. MicroRNAs in seeds:modified detection techniques and potential applications[J]. Can J Bot, 2006, 84 (2): 189–198. DOI:10.1139/b05-141 |
| [8] | Liu PP, Montgomery TA, Fahlgren N, et al. Repression of AUXIN RESPONSE FACTOR10 by microRNA160 is critical for seed germination and post-germination stages[J]. Plant J, 2007, 52 (1): 133–46. DOI:10.1111/j.1365-313X.2007.03218.x |
| [9] | Nodine MD, Bartel DP. MicroRNAs prevent precocious gene expr-ession and enable pattern formation during plant embryogenesis[J]. Genes Dev, 2010, 24 (23): 2678–2692. DOI:10.1101/gad.1986710 |
| [10] | Kang MM, Zhao Q, Zhu DY, et al. Characterization of microRNAs expression during maize seed development[J]. BMC Genomics, 2012 : 13, 360. |
| [11] | Abdin MZ, Israr M, Rehman RU, et al. A novel antimalarial drug:Biochemical and molecular approaches for enhanced production[J]. Planta Med, 2003, 69 (4): 289–299. DOI:10.1055/s-2003-38871 |
| [12] | Hao D, Yang L, Liu M, et al. Identification of Taxus microRNAs and their targets with high-throughput sequencing and degradome analysis[J]. Physiol Plant, 2012, 146 : 388–403. DOI:10.1111/ppl.2012.146.issue-4 |
| [13] | 赵双宜, 吴耀荣, 夏光敏. 介绍一种简单高效的植物总RNA提取方法[J]. 遗传, 2002, 34(3): 337–338. |
| [14] | 李宏, 王新力. 植物组织RNA提取的难点及对策[J]. 生物技术通报, 1999(1): 36–39. |
| [15] | 杨成君, 王军, 周莉, 等. 红果人参叶片RNA的提取[J]. 生物技术通报, 2008(1): 136–139. |
| [16] | Couch JA, Fritz PJ. Isolation of DNA from plants high in polyphenolics[J]. Plant Mol Biol Rep, 1990, 8 (1): 8–12. DOI:10.1007/BF02668875 |
| [17] | Bahloul M, Burkard G. An improved method for the isolation of total RNA from spruce tissues[J]. Plant Mol Biol Rep, 1993, 11 (3): 212–215. DOI:10.1007/BF02669847 |
| [18] | Fang G, Hammar S, Grumet R. A quick and inexpensive method for removing polysaccharides from plant genomic DNA[J]. BioTechniques, 1992, 13 (1): 52–54, 56. |
| [19] | Chang SJ, Puryear J, Cairney J. A simple and efficient method for isolating RNA from pine trees[J]. Plant Mol Biol Rep, 1993, 11 (2): 113–116. DOI:10.1007/BF02670468 |
| [20] | Manning K. Isolation of nucleic acids from plants by differential solvent precipitation[J]. Anal Biochem, 1991, 195 (1): 45–50. DOI:10.1016/0003-2697(91)90292-2 |