CRISPR(Clustered regularly interspaced short palindromic repeats)系统最早发现于细菌中,据降解外源遗传物质方式的不同,该系统被分为了3种类型[1]。Ⅰ型、Ⅲ型系统中参与的蛋白众多,皆涉及多种蛋白复合物[2]。Ⅱ型CRISPR/Cas9系统构成与作用相对简单,因而被广泛应用[3]。该系统中的crRNA与tracrRNA分别具有靶定核酸位点与结合Cas9蛋白的能力,可招募Cas9蛋白剪切降解外源核酸物质[4]。应用时,结合crRNA和tracrRNA形成的sgRNA(single-guide RNA)与优化后的Cas9蛋白相互作用,可在真核生物中介导特定的DNA双链断裂(DSB)[5]。再经修复途径NHEJ和HDR,可分别诱导目的序列的突变和插入[6]。相比于ZFN及TALEN,CRISPR/Cas9系统有更高频率出现的核酸靶向位点N18NGG,则具有更大的作用范围,可更精细的编辑与加工[7]。CRISPR/Cas9也有脱靶现象[8]。其靶向序列的错配会导致脱靶,产生错误剪切[9]。对此,改造Cas9蛋白成为Cas9 nickase(Cas9n)功能缺陷型蛋白,只造成DNA单链断裂;设计靶定正、负双链的多对sgRNA,只有当造成的正、负链缺口足够接近时,才会形成DSB,如此可大大降低脱靶效应造成的DNA损伤及其影响[10]。
目前CRISPR/Cas9广泛应用于编码基因,而在长链非编码RNA(lncRNA)上的报道却不多见[11]。LncRNA是一类长度超过200 nt、不编码蛋白质的RNA分子[12]。功能上,lncRNA或是作为转录活性分子的分子信号;或结合蛋白或核酸并影响其功效;或引导蛋白定位于目的位点;或作为蛋白与核酸组装的媒介与平台,在表观遗传、转录及转录后等水平调控基因的表达[13]。LncRNA HOTAIR(HOX transcript antisense RNA),其5' 端和3' 端可分别与PCR2和LSD1相结合,参与表观遗传学调控[14]。它与多种肿瘤的发生与发展有关,在乳腺癌,肺癌,胃癌,子宫内膜癌等多种癌组织中表达水平均高于其正常组织[15]。然而,其作用的具体机制研究尚不完全[16]。
利用CRISPR/Cas9系统编辑HOTAIR基因,对cas9技术应用的完善以及全面探究HOTAIR功能具有重要意义。然而,单一sgRNA介导的DNA单位点断裂已不能高效改变HOTAIR的功能表型[17]。再添加一个sgRNA,形成pgRNA(paired-guide RNA),分别靶向HOTAIR基因的上、下游,产成两个DSB,以达到切割HOTAIR基因片段的目的。
DLC1(Deleted in Liver Cancer 1)基因为原发性人肝细胞癌中常见的基因缺失片段,也在多种人类癌症中缺失或沉默[18]。DLC1可使RhoA、Cdc42活性抑制,可诱导细胞凋亡[19]。p53对可修复的DNA损伤,可使细胞停留在G1期,细胞生长抑制;当DNA损伤不可修复时,p53则诱导细胞程序性死亡[20]。而过表达p53抑制癌细胞的活性的同时也会引起细胞的过早衰老[21]。p21可有效结合并抑制细胞周期蛋白激酶CDK2/4等的活性,引起细胞周期停滞[22]。另一方面,可下调Caspase-3和ROS的生成,抑制细胞的凋亡,但其不会诱导细胞凋亡[23]。pRB(Rb蛋白)在多种不同的肿瘤细胞中都有缺失或者突变[24]。pRB的去磷酸化抑制E2F的活性,下调细胞周期蛋白以及细胞周期蛋白激酶表达,从而达到抑制肿瘤的作用[25]。
本研究拟利用pgRNA/Cas9系统在HeLa细胞中试图敲除长链非编码RNA HOTAIR,并以此研究HOTAIR变化对细胞功能及常见肿瘤抑制因子的影响,验证HOTAIR变化对子宫颈癌细胞的重要意义,同时为HOTAIR的作用途径提出新的方向。
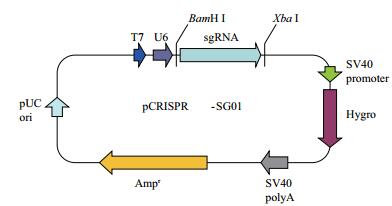
1 材料与方法 1.1 材料 1.1.1 细胞与质粒所选用的人类宫颈癌细胞HeLa购买于美国ATCC公司;Cas9表达质粒CP-C9NU-01和sgRNA表达质粒pCRISPR-SG01皆购自于广州复能基因有限公司。CP-C9NU-01带有mCherry标签和G418(neo)抗性基因,pCRISPR-SG01带有潮霉素B(Hygromycin B)抗性基因。
1.1.2 培养基高糖DMEM细胞培养基:使用标准高糖DMEM干粉(GIBCO)配制,加入800 mL超纯水,加入碳酸氢钠3.7 g,HEPES 2.383 g,调节pH至7.4,并定容至1 L。0.22 μm滤膜过滤除菌。LB液体培养基:氯化钠1 g,酵母提取物0.5 g,蛋白胨1 g,100 mL超纯水,20 min,12℃高压灭菌。细胞完全培养基:配制好的高糖DMEM培养基中加入10%的胎牛血清(FBS,Mini),100 U/mL青霉素和100 mg/mL链霉素。
1.1.3 主要试剂氨苄青霉素(Amp,100 mg/mL);噻唑兰试剂(MTT,5 mg/mL);G418抗生素(100 mg/mL);潮霉素B抗生素(100 mg/mL);焦碳酸二乙酯试剂(DEPC);二甲基亚砜(DMSO)。
1.1.4 酶和引物限制性内切酶BamH I和Xba I购自NEB公司;蛋白酶K,RNA水解酶A,T4连接酶和实时定量PCR SYBR Green购自于Thermo公司;RNA反转录试剂盒购自于TaKaRa公司。各引物序列见表 1。
根据HOTAIR基因序列,在其上、下游寻找的N18NGG序列,通过与人类基因组库(Dec.2013-GRCh8/hg38)匹配,在其基因上、下游各选取3个靶向目的位点设计sgRNA(命名为U1、U2、U3、D1、D2、D3)。PCR扩增得到目的条带,通过限制性酶BamH I和Xba I酶切,将目的片段克隆到载体pCRISPR-SG01上,并进行测序验证(武汉擎科)。得到的6个sgRNA表达质粒,根据其上、下游位置两两配对,得到9组pgRNA(分别为D1U1、D1U2、D1U3、D2U1、D2U2、D2U3、D3U1、D3U2、D3U3),形成pgRNA文库。
1.2.2 细胞培养及转染从液氮罐中取出冻存的细胞,置于37℃水浴中,使其迅速融化。3 000 r/min低速离心5 min,弃去上清,加入含有10% FBS,100 U/mL青霉素和100 mg/mL链霉素的DMEM高糖培养基,吹打混匀,放入5% CO2,37℃的无菌恒温箱中培养。转染前一天,取对数生长期的细胞消化,接种至6孔板中,每孔接种1×106细胞。转染时,细胞密度达到70%-80%。取2 μg Cas9蛋白表达质粒CP-C9NU-01,按照转染试剂与质粒DNA(3:1)的比例,加入转染试剂(PEI)以及适量的无血清培养基,混匀,室温孵育15 min。将混匀后的脂质体-DNA复合物加入已接种细胞的细胞培养板中,轻轻混匀,置于细胞培养箱中继续培养。24 h后更换新鲜培养基,并加入抗生素G418进行筛选。按照以上的转染条件,通过转染试剂PEI将pgRNA导入细胞中(每组pgRNA转染总质量2 μg,其中上、下游sgRNA各1 μg)。转染24 h后更换新鲜培养基,并加入抗生素G418和潮霉素B进行筛选。
1.2.3 用药最适浓度选择及细胞筛选取对数生长期的细胞消化,接种至6孔板中,每孔接种1×106细胞。按0、2、4、6、8和10 μg/mL设置G418的用药终浓度。持续用药14 d后,细胞全部致死的最小浓度作为最适用药浓度;相同地,潮霉素B以0、1、2、3和4 μg/mL设置用药终浓度,14 d后以细胞全部致死的最小浓度作为最适用药浓度。对转染后的HeLa细胞以最适浓度的抗生素进行筛选,构建稳定表达的细胞株。
1.2.4 基因组DNA提取及PCR检测收集细胞,加入1 mL组织裂解液,65℃水浴1 h,裂解细胞;加入2 μL蛋白酶K(20 U/μL)和4 μL RNA酶A(20 mg/mL)置于50℃水浴消化;利用饱和酚、氯仿抽提DNA,加入等体积的异丙醇沉降,用1 mL 75%乙醇洗涤,经干燥后加入无菌水溶解,定量。针对HOTAIR基因上特异性序列G1、G2,以基因组DNA为模板,通过PCR扩增,确定CRISPR/Cas9对HOTAIR基因的编辑情况。
1.2.5 RNA提取及qRT-PCR检测收集细胞,加入1 mL Trizol Reagent,反复吹打,使其充分裂解;加入200 μL氯仿,剧烈震荡30 s,冰上放置15 min;4℃,12 000 r/min离心15 min;将上层液体转移至RNase-free的EP管中,并加入0.5 mL异丙醇,混匀;4℃,12 000 r/min离心,弃掉上清,并加入70%乙醇,洗涤RNA;7 500 r/min离心10 min,弃掉上清,室温干燥。加入适量DEPC水溶解并定量。使用gDNA Eraser反转录试剂盒(TaKaRa)按照操作手册将RNA反转录成单链cDNA。以cDNA为模板,扩增目的基因,通过荧光实时定量PCR对目的基因HOTAIR以及相关肿瘤抑制因子p21、p53、pRB和DLC1的RNA水平进行分析。
1.2.6 MTT增殖检测取对数生长期的细胞消化,接种至96孔板中,每孔接种1×103细胞,每组设置6个复孔。培养24 h后,每孔中加入10 μL MTT试剂(5 mg/mL),置于培养箱中孵育4 h;弃上清加入100 μL的DMSO,室温下避光振荡10 min,待结晶完全溶解后,通过美联免疫检测仪OD570处测量隔空的吸光值。每24 h检测一次,连续测量8 d,绘制细胞增值曲线。
1.2.7 细胞划痕实验细胞伤口愈合的快慢可以直接反应出细胞迁移能力的大小。取对数生长期的细胞消化,接种至6孔板中,每孔接种1×106细胞。培养24 h后,使用无菌枪头在细胞培养板中划痕,枪头要垂直,确保划痕宽度一致;PBS缓冲液清洗细胞2-3次,并加入新鲜培养基。置于细胞培养箱中继续培养,分别在划痕后0、24和48 h对划痕同一位置处观察拍照。
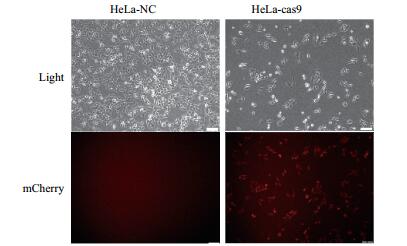
2 结果 2.1 在HeLa细胞中稳定转染Cas9核酸酶为了构建CRISPR-Cas9敲除HOTAIR的稳定遗传细胞株,首先将Cas9蛋白表达质粒CP-C9NU-01(图 1),通过化学转染试剂PEI导入HeLa细胞。以终浓度为800 μg/mL的G418药物连续筛选。质粒上携带有mCherry的荧光基团,在激发光的作用下,细胞发出红光。通过激光共聚焦显微镜观察,结果(图 2)表明,转染Cas9蛋白表达质粒CP-C9NU-01的细胞在激发光作用下,能看到细胞发出红光;未转染的对照组则未观察到红光。以上结果表明,Cas9蛋白已在HeLa细胞中稳定表达。

|
| 图 1 核酸酶Cas9表达质粒图谱 |
|
| 图 2 细胞荧光分析 Light:自然光;mCherry:红色荧光标签;HeLa-NC:未转染Cas9核酸酶的HeLa细胞;HeLa-cas9:转染Cas9核酸酶的HeLa细胞 |
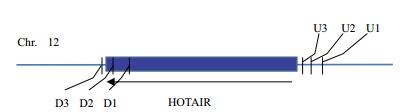
在HOTAIR基因两端选取合适的靶向序列N18NGG,通过与人类基因组库(Dec.2013-GRCh8/hg38)匹配。在HOTAIR基因上、下游挑选与靶位点匹配的序列,并设计引物,分别命名为U1、U2、U3、D1、D2和D3(图 3)。PCR扩增得到目的片段,经限制性酶BamH I和Xba I酶切,将其克隆至表达载体pCRISPR-SG01上(图 4)。按照sgRNA上、下游位置,两两配对形成pgRNA文库(分别命名为D1U1、D1U2、D1U3、D2U1、D2U2、D2U3、D3U1、D3U2和D3U3)。
|
| 图 3 HOTAIR sgRNA靶向位点示意图 D:downstream,HOTAIR下游;U:upstream,HOTAIR上游 |
|
| 图 4 sgRNA表达质粒图谱 |
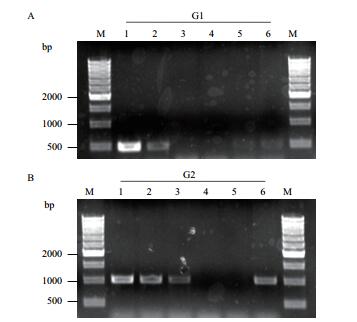
通过脂质体转染试剂PEI将pgRNA导入稳定表达Cas9核酸酶的HeLa细胞中,并通过抗生素G418(终浓度800 μg/mL)和潮霉素B(终浓度300 μg/mL)进行筛选。将筛选后得到细胞扩大培养,提取基因组DNA。通过对HOTAIR基因序列分析,选取两段特异性的序列G1、G2,以基因组DNA为模板,分别用鉴定引物G1 PF、G1 PR和G2 PF、G2 PR进行扩增,确定在pgRNA的引导下cas9核酸酶是否对HOTAIR基因片段进行切除。转染cas9核酸酶的稳定细胞株HeLa-cas9作为对照。以筛选得到的细胞株D1U3及D3U1基因组DNA为模板,用引物G1 PF、G1 PR进行扩增,没有得到与预期大小相符的特异性的条带(图 5-A);以筛选得到的细胞株D3U1及D3U3基因组DNA为模板,用引物G2 PF、G2 PR进行扩增,没有得到特异性的条带(图 5-B)。结果表明,筛选得到的D3U1细胞株为在pgRNA的引导下,Cas9核酸酶切除了D3U1细胞株中HOTAIR基因片段。
|
| 图 5 HOTAIR基因组PCR检测 A:G1位点PCR产物琼脂糖凝胶电泳图。B:G2位点PCR产物琼脂糖凝胶电泳图。G1:495 bp;G2:1024 bp;M:1 kb marker;1:NC,HeLa-cas9细胞;2:D1U1;3:D1U3;4:D3U1;5:D3U3;6:D2U2。2-6:分别为转染pg-RNA的HeLa-cas9细胞 |
对D3U1细胞株中HOTAIR的RNA水平进行qRT-PCR检测,结果(图 6)表明,与正常的对照组HeLa细胞相比其HOTAIR水平下调60%左右。

|
| 图 6 D3U1细胞株qRT-PCR检测 D3U1细胞株中HOTAIR表达下调60%。HeLa-cas9:稳定转染Cas9蛋白的细胞株;D3U1:转染pgRNA D3及U1的HeLa-cas9细胞。**:P < 0.01 |
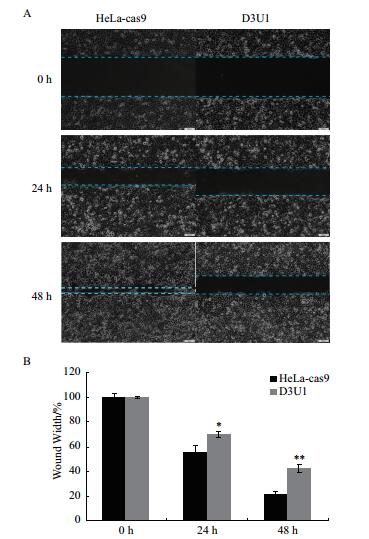
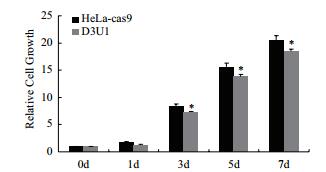
为了确定cas9对HOTAIR的敲除效果,针对筛选得到的稳定遗传细胞株D3U1的细胞迁移和增殖能力进行检测。相比对照组HeLa-cas9细胞株,D3U1细胞株的细胞迁移能力要明显降低(图 7);同时细胞的增殖能力也有了显著性的减弱(图 8)。
|
| 图 7 划痕实验检测细胞迁移能力 A:划痕实验检测细胞迁移能力的变化;B:统计结果表明:相比对照组,D3U1细胞的迁移能力明显降低。*:P < 0.05;**:P < 0.01,下同 |
|
| 图 8 MTT实验检测细胞增殖能力变化情况 相比对照组,D3U1细胞增殖能力明显减弱。HeLa-cas9:稳定转染Cas9蛋白的细胞株;D3U1:转染pgRNA D3及U1的HeLa-cas9细胞 |
为了深入了解HOTAIR功能,对集中重要的肿瘤因子DLC1、p21、p53和pRB的RNA水平进行分析。结果(图 9)显示,相比对照组,D3U1细胞株中p21和pRB的转录水平显著性增加,而DLC1和p53的转录水平则明显降低。由此可见,HOTAIR与肿瘤因子之间存在着紧密联系。

|
| 图 9 HOTAIR下调细胞株D3U1中肿瘤抑制因子RNA水平检测 HeLa-cas9:稳定转染Cas9蛋白的细胞株;D3U1:转染pgRNA D3及U1的HeLa-cas9细胞 |
CRISPR/Cas9系统敲除技术因其自身易操作,高效率,高通量的优点,在使用范围和作用效率上优于ZFN和TALEN[26];而针对长链非编码RNA编辑应用方面,因后者无法编码蛋白,其功能主要通过自身RNA二级结构[27]与核酸[28],蛋白的相互作用而实现[29]。因此仅依赖一条sgRNA介导序列突变的传统方法已无法改变部分长链非编码RNA功能。因此,本研究在该系统基础上中再添加一个sgRNA,形成pgRNA,介导长链非编码RNA基因的片段切割,已达到敲除目的。该方法已被Zhu等[30]用于长链非编码RNA AAVS1与LINC01087的敲除,并取得了不错的效果。
长链非编码RNA HOTAIR与肿瘤的转移与侵袭,发生和发展有着密切关系,但其具体作用机制尚未完全明了[31]。为了更好地探究HOTAIR的功能及分子机制,利用CRISPR/Cas9技术对lncRNA HOTAIR进行基因编辑。该实验首先构建了稳定遗传表达Cas9核酸酶的HeLa细胞株,因其可通过导入特定的sgRNA对多种目的基因进行编辑,则具有重要的应用价值;之后在该细胞株中导入靶向HOTAIR基因上、下游的pgRNA,介导HOTAIR基因片段的切割。实验结果显示,利用该方法筛选得到的HeLa细胞中HOTAIR表达水平下调约60%,并未完全敲除。对此,我们推测HOTAIR基因的完全敲除可能会导致细胞致死。Goyal等[32]在其研究中表明CRISPR/Cas9系统敲除HOTAIR基因具有局限性。由于HOTAIR是一类基因间长链非编码RNA,位于基因HOXC12与HOXC13之间,其敲除会影响周边基因的表达。因此,在后续相关实验中,我们应选取转录相对独立,而且敲除后不会对邻近基因产生影响的的长链非编码RNA进行研究。
筛选得到HOTAIR下调的HeLa细胞株,对其细胞迁移和增殖能力检测,验证pgRNA/Cas9介导的HOTAIR敲低是能有效地引起细胞功能的改变。该结果与其它文献报道一致,HOTAIR表达水平的降低能抑制细胞的迁移与增殖[33-35]。在该细胞株中检测相关肿瘤抑制因子随HOTAIR下调的影响变化,以此建立HOTAIR与肿瘤抑制因子之间的联系,为阐释HOTAIR功能探究新的作用机制。DLC1作为一个肿瘤抑制因子,其启动子高甲基化时,自身突变或缺失,会促进相关肿瘤细胞的增殖,迁移与侵袭[36-37]。HOTAIR的5' 端与PRC2结合,可引起基因启动子区域DNA甲基化的改变[37]。该机制可能作用在DLC1启动子上,下调HOTAIR可能是通过改变DLC1启动子区域甲基化程度,从而导致其转录水平受到影响。其具体机制有待实验进行验证。
P53作为p21的上游基因,可促进p21的转录表达。而检测结果表明下调HOTAIR抑制HeLa细胞迁移与增殖,促进p21转录的同时,p53转录水平下降,暗示着HOTAIR对p21以及p53的调控可能通过不同的途径来完成。Zhai等[38]指出HOTAIR可促进p53启动子的甲基化水平,从而抑制p53表达;Zheng等[39]发现抑制HOTAIR表达同样可以显著性增加p53表达。由于基因的RNA与蛋白水平并没有直接的相关性,RNA水平的变化情况并不能完全正确的反应蛋白水平的变化情况,因此还需通过Western Blot检测HOTAIR与p53蛋白水平之间的关系。另一方面,下调HOTAIR明显增加p21和pRB RNA水平,由于p21和pRB与细胞周期存在紧密联系,都可直接影响细胞周期,使细胞生长停滞,故其转录水平的上升很有可能是细胞迁移与增殖阻滞的主要原因。
本研究利用CRISPR/Cas9编辑lncRNA HOTA-IR,并检测该系统是否能有效影响其功能表型及相关肿瘤抑制因子转录,因而对后者如何具体受到HOTAIR作用和影响上并未进一步探究。尽管如此,通过在HOTAIR与肿瘤抑制因子之间建立联系,也为HOTAIR作用机制的研究提出了新的方向。
4 结论本实验通过设计靶定HOTAIR基因的pgRNA,在HeLa细胞中利用CRISPR/Cas9系统可有效下调长链非编码RNA HOTAIR的表达水平。细胞划痕和MTT增殖实验表明使用该方法下调HOTAIR可有效抑制细胞的迁移与增殖,改变细胞的表型。同时,利用该方法得到的HOTAIR下调细胞株可用于对HOTAIR在HeLa细胞内功能的探究,为揭示HOTAIR的作用机制提供了的新方向。
| [1] | Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation[J]. Annual Review of Genetics, 2011, 45 : 273–297. DOI:10.1146/annurev-genet-110410-132430 |
| [2] | 殷朝敏, 范秀芝, 史徳芳, 等. CRISPR/Cas基因编辑技术及其在真菌中的应用[J]. 生物技术通报, 2017, 33(3): 58–65. |
| [3] | Zheng PX, Chan YC, Chiou CS, et al. Clustered regularly interspaced short palindromic repeats Are emm type-specific in highlyprevalent group a Streptococci[J]. PLoS One, 2015, 10 : 145–223. |
| [4] | 张凯丽, 李瑞, 胡桐桐, 等. CRISPR/Cas9技术的发展及在基因组编辑中的应用[J]. 生物技术通报, 2016(32): 47–60. |
| [5] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339 : 823–826. DOI:10.1126/science.1232033 |
| [6] | Bortesi L, Fischer R. The CRISPR/Cas9 system for plant genome editing and beyond[J]. Biotechnology Advances, 2015, 33 : 41–52. DOI:10.1016/j.biotechadv.2014.12.006 |
| [7] | Yasue A, Mitsui SN, Watanabe T, et al. Highly efficient targeted mutagenesis in one-cell mouse embryos mediated by the TALEN and CRISPR/Cas systems[J]. Scientific Reports, 2014, 4 : 5705. |
| [8] | Gao Y, Wu H, Wang Y, et al. Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects[J]. Genome Biology, 2017, 18 : 13. DOI:10.1186/s13059-016-1144-4 |
| [9] | Koo T, Lee J, Kim JS. Measuring and reducing off-target activities of programmable nucleases including CRISPR-Cas9[J]. Molecules and Cells, 2015, 38 : 475–481. DOI:10.14348/molcells.2015.0103 |
| [10] | McCaffrey J, Sibert J, Zhang B, et al. CRISPR-CAS9 D10A nickase target-specific fluorescent labeling of double strand DNA for whole genome mapping and structural variation analysis[J]. Nucleic Acids Research, 2016, 44 : e11. DOI:10.1093/nar/gkv878 |
| [11] | Ho TT, Zhou N, Huang J, et al. Targeting non-coding RNAs with the CRISPR/Cas9 system in human cell lines[J]. Nucleic Acids Research, 2015, 43 : e17. DOI:10.1093/nar/gku1198 |
| [12] | Martens-Uzunova ES, Bottcher R, Croce CM, et al. Long noncoding RNA in prostate, bladder, and kidney cancer[J]. European Urology, 2014, 65 : 1140–1151. DOI:10.1016/j.eururo.2013.12.003 |
| [13] | Hayes EL, Lewis-Wambi JS. Mechanisms of endocrine resistance in breast cancer: an overview of the proposed roles of noncoding RNA[J]. Breast Cancer Research: BCR, 2015, 17 : 40. DOI:10.1186/s13058-015-0542-y |
| [14] | 李雨薇, 王裕民, 张雪莹, 等. 长链非编码RNA HOTAIR在恶性肿瘤中的研究进展[J]. 生物化学与生物物理进展, 2015, 42(3): 228–235. |
| [15] | Miao Z, Ding J, Chen B, et al. HOTAIR overexpression correlated with worse survival in patients with solid tumors[J]. Minerva Medica, 2016, 107 : 392–400. |
| [16] | Zhou X, Chen J, Tang W. The molecular mechanism of HOTAIR in tumorigenesis, metastasis, and drug resistance[J]. Acta Biochimica et Biophysica Sinica, 2014, 46 : 1011–1015. DOI:10.1093/abbs/gmu104 |
| [17] | Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343 : 84–87. DOI:10.1126/science.1247005 |
| [18] | Zimonjic DB, Popescu NC. Role of DLC1 tumor suppressor gene and MYC oncogene in pathogenesis of human hepatocellular carcinoma: potential prospects for combined targeted therapeutics[J]. Int J Oncol, 2012, 41 : 393–406. DOI:10.3892/ijo.2012.1474 |
| [19] | Yang C, Wu D, Jia J, et al. DLC1 as a regulator of proliferation, invasion, cell cycle, and apoptosis in cutaneous squamous cell carcinoma[J]. Tumour Biology, 2013, 34 : 2633–2643. DOI:10.1007/s13277-013-0813-0 |
| [20] | Chen J. The Cell-Cycle Arrest and apoptotic functions of p53 in tumor initiation and progression[J]. Cold Spring Harbor Perspectives in Medicine, 2016, 6 : a026104. DOI:10.1101/cshperspect.a026104 |
| [21] | Nicolai S, Rossi A, Di Daniele N, et al. DNA repair and aging: the impact of the p53 family[J]. Aging, 2015, 7 : 1050–1065. DOI:10.18632/aging.v7i12 |
| [22] | Cazzalini O, Scovassi AI, Savio M, et al. Multiple roles of the cell cycle inhibitor p21(CDKN1A) in the DNA damage response[J]. Mutation Research, 2010, 704 : 12–20. DOI:10.1016/j.mrrev.2010.01.009 |
| [23] | He H, Baldwin GS. Rho GTPases and p21-activated kinase in the regulation of proliferation and apoptosis by gastrins[J]. Int J Biochem Cell Biol, 2008, 40 : 2018–2022. DOI:10.1016/j.biocel.2008.05.002 |
| [24] | Hutcheson J, Witkiewicz AK, Knudsen ES. The RB tumor suppressor at the intersection of proliferation and immunity: relevance to disease immune evasion and immunotherapy[J]. Cell Cycle, 2015, 14 : 3812–3819. DOI:10.1080/15384101.2015.1010922 |
| [25] | Schaal C, Pillai S, Chellappan SP. The Rb-E2F transcriptional regulatory pathway in tumor angiogenesis and metastasis[J]. Advances in Cancer Research, 2014, 121 : 147–182. DOI:10.1016/B978-0-12-800249-0.00004-4 |
| [26] | Gaj T, Gersbach CA, Barbas CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31 : 397–405. DOI:10.1016/j.tibtech.2013.04.004 |
| [27] | Johnsson P, Lipovich L, Grander D, et al. Evolutionary conservation of long non-coding RNAs; sequence, structure, function[J]. Bba-Gen Subjects, 2014, 1840 : 1063–1071. DOI:10.1016/j.bbagen.2013.10.035 |
| [28] | He S, Zhang H, Liu HH, et al. LongTarget: a tool to predict lncRNA DNA-binding motifs and binding sites via Hoogsteen base-pairing analysis[J]. Bioinformatics, 2015, 31 : 178–186. DOI:10.1093/bioinformatics/btu643 |
| [29] | Zhu JJ, Fu HJ, Wu YG, et al. Function of lncRNAs and approaches to lncRNA-protein interactions[J]. Sci China Life Sci, 2013, 56 : 876–885. DOI:10.1007/s11427-013-4553-6 |
| [30] | Zhu S, Li W, Liu J, et al. Genome-scale deletion screening of human long non-coding RNAs using a paired-guide RNA CRISPR-Cas9 library[J]. Nature Biotechnology, 2016, 34 : 1279–1286. DOI:10.1038/nbt.3715 |
| [31] | Bhan A, Mandal SS. LncRNA HOTAIR: A master regulator of chromatin dynamics and cancer[J]. Biochimica et Biophysica Acta, 2015, 1856 : 151–164. |
| [32] | Goyal A, Myacheva K, Gross M, et al. Challenges of CRISPR/Cas9 applications for long non-coding RNA genes[J]. Nucleic Acids Research, 2016, 45 (3): e12. |
| [33] | Dong L, Hui L. HOTAIR promotes proliferation, migration, and invasion of ovarian cancer SKOV3 cells through regulating PIK3R3[J]. Med Sci Monit, 2016, 22 : 325–331. DOI:10.12659/MSM.894913 |
| [34] | Yiwei T, Hua H, Hui G, et al. HOTAIR interacting with MAPK1 regulates ovarian cancer skov3 cell proliferation, migration, and invasion[J]. Medical Science Monitor, 2015, 21 : 1856–1863. DOI:10.12659/MSM.893528 |
| [35] | Zhou C, Ye L, Jiang C, et al. Long noncoding RNA HOTAIR, a hypoxia-inducible factor-1alpha activated driver of malignancy, enhances hypoxic cancer cell proliferation, migration, and invasion in non-small cell lung cancer[J]. Tumour Biology, 2015, 36 : 9179–9188. DOI:10.1007/s13277-015-3453-8 |
| [36] | Kang Z, Xu F, Zhang QA, et al. Correlation of DLC1 gene methylation with oncogenic PIK3CA mutations in extramammary Paget's disease[J]. Modern Pathology, 2012, 25 : 1160–1168. DOI:10.1038/modpathol.2012.65 |
| [37] | Bujko M, Kober P, Rusetska N, et al. Aberrant DNA methylation of alternative promoter of DLC1 isoform 1 in meningiomas[J]. Journal of Neuro-oncology, 2016, 130 : 473–484. DOI:10.1007/s11060-016-2261-3 |
| [38] | Zhai N, Xia Y, Yin R, et al. A negative regulation loop of long noncoding RNA HOTAIR and p53 in non-small-cell lung cancer[J]. Onco Targets and Therapy, 2016, 9 : 5713–5720. DOI:10.2147/OTT |
| [39] | Zheng H, Min J. Role of long noncoding RNA HOTAIR in the growth and apoptosis of osteosarcoma cell MG-63[J]. BioMed Research International, 2016, 57 : 57–64. |





