2. 上海交通大学药学院,上海 200240
2. School of Pharmacy, Shanghai Jiao Tong University, Shanghai 200240
细辛(Asari Radix et Rhizoma)是常用解表类中药,主治风寒表证、痰饮咳嗽,兼具镇痛消炎作用,被广泛应用于中医临床,来源于马兜铃科细辛属(Asarum)植物北细辛A. heterotropoides Fr. Schmidt var. mandshuricum(Maxim.) Kitag.、汉城细辛A. sieboldii Miq. var. seoulense Nakai.、华细辛A. sieboldii Miq.干燥的根和根茎[1]。除药用外,细辛在其它方面也具有很大的应用价值。近几年的研究表明,细辛全草含有多糖、叶酸、多种微量元素和氨基酸,畜禽长期合理使用可提高机体代谢能力、促进对营养物质的消化吸收,是一种潜在的新型优良饲料添加剂[2];在植物源农药、香精香料、日用化工等方面也颇具开发价值[3]。然而,有关细辛生物学特性和资源学方面的基础研究却十分薄弱,对细辛次生代谢产物积累的动态变化规律及其内在机制缺乏了解,人工栽培仍停留在粗放的状态,因而难以准确掌控细辛药材的质量。植物代谢组的特征,包括化学成分的组成和比例,是植物个体和种群在各种内外因素的综合作用下,生物机体复杂的生理生化活动过程的最终体现,具体而言,是次生代谢物在其生物合成过程中受到各项影响因素的作用,相关基因表达的结果。因此,阐明次生代谢途径,分离相关基因,比较基因在不同发育阶段、不同组织器官中的表达差异,对于理解药用植物次生代谢物含量变化的规律是必不可少的。
实时荧光定量PCR(qRT-PCR)是基因表达分析的重要工具,该技术具有灵敏性高、特异性强、快速准确等特点,有效解决了常规PCR假阳性率高等缺点[4-6]。但在数据处理过程中,qRT-PCR的结果容易受到不同因素的影响而缺少一致性,因此,选用稳定表达的基因作为内参基因,实现实验结果的标准化,对提高实验结果分析的准确性非常关键。理想的内参基因应在所有组织和细胞类型中均能表达,且不受任何因素的影响,然而,任何基因的恒定表达都只是在一定类型的细胞或实验因素下有范围的恒定,并非绝对地稳定表达[7, 8]。事实上,同一基因在不同种属、同一植物不同组织和不同时期的表达丰度都存在变化[9]。因此,在对目的基因进行差异表达分析时,首先需要筛选到可以作为内参基因使用的合适的基因。
根据已有的研究报道,植物基因表达分析中作为内参基因的有很多,本研究选取常用的SAND-1、ACT、18S rRNA、CYP-2、GAPDH、TUB作为候选内参基因,设计特异性引物,以华细辛在营养生长期、花期和花后期等不同生长时期的根、根茎、叶片、叶柄、花等不同组织部位的材料为样品进行相对定量PCR检测,并用geNorm、NormFinder、BestKeeper、Delta CT等软件进行统计学分析,力图筛选出华细辛中表达稳定性较强的内参基因,为研究华细辛基因差异表达奠定基础。
1 材料与方法 1.1 材料 1.1.1 研究材料实验所用研究材料为华细辛。分别取华细辛营养生长期的根(Root A)、根茎(Rhizoma A)、叶(Leaf A)、叶柄(Petiole A),花期的根(Root B)、根茎(Rhizoma B)、叶(Leaf B)、叶柄(Petiole B),花(Flower),花后期的根(Root C)、根茎(Rhizoma C)、叶(Leaf C)、叶柄(Petiole C),将它们清洗干净并在液氮中研磨成粉,置于-80℃冰箱中保存备用。
1.1.2 主要试剂总RNA提取试剂盒RNAprep Pure Plant Kit(Tiangen公司)、逆转录试剂盒FastQuant RT Kit(With gDNase)(Tiangen公司)、荧光定量PCR试剂盒SuperReal PreMix Plus(SYBR Green)(Tiangen公司)。其余试剂若未说明均为国产分析纯。引物由上海英骏公司合成。
1.2 方法 1.2.1 总RNA的提取及逆转录根据Tiangen RNA-prep Pure Plant Kit说明书进行总RNA的提取,用1.2%琼脂糖凝胶电泳检测,确保各样品28S与18S电泳条带亮度之比约为2:1,且无基因组DNA残留。根据Tiangen FastQuantRT Kit(With gDNase)说明书进行逆转录反应,合成的cDNA作为荧光定量PCR反应的模板。
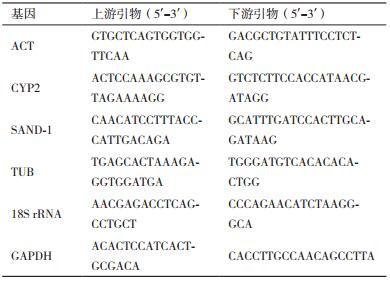
1.2.2 候选内参基因的选择及引物设计本研究共选取6个候选内参基因,分别为SAND-1、ACT、18SrRNA、CYP-2、GAPDH和TUB,引物序列见表 1。在qRT-PCR中,可通过熔解曲线判断引物的特异性。
本实验在Applied Biosy-stems StepOne PCR仪上进行。反应体系(20 µL)如下:2×SuperReal PreMix Plus 10 µL,50×ROX Reference Dye 2 µL,cDNA模板(100 ng)0.1 µL,上游引物(10 µmol/L)0.6 µL,下游引物(10 µmol/L)0.6 µL,RNase-free ddH2O 6.7 µL。
反应程序为:预变性95℃ 15 min;95℃ 10 s,60℃ 20 s,72℃ 30 s,在延伸阶段检测荧光强度,收集信号,40个循环;72℃ 10 s。每个处理设置3个重复,并以ddH2O代替模板设置阴性对照。
1.2.4 数据分析在对内参基因的表达稳定性进行评价之前,须构建标准曲线,即将cDNA模板进行5倍的梯度稀释,根据1.2.3的程序进行荧光定量PCR反应,将所得结果(Ct值)运用Excel计算内参基因的相关系数(R2),然后根据公式lg(1+E)=R2计算出扩增效率(E)[10]。目前认为只有扩增效率在一定的范围(90%-105%)时,才能进行下一步数据分析。
根据公式Q=EΔCT,其中,ΔCT= Ctmin-Ct样品,E为基因扩增效率(当扩增效率接近100%时,E默认为2),利用Excel计算出每个扩增样品的表达量Q[11]。在上述基础上,进一步利用geNorm[12]、NormFinder[7]、BestKeeper[13]、Delta CT软件[14]对各内参基因的稳定性进行统计学分析。
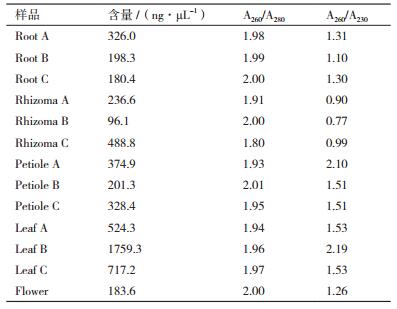
2 结果 2.1 总RNA提取琼脂糖凝胶电泳结果(图 1)显示,总RNA中28S和18S两条主带清晰,且28S条带大约为18S条带亮度的两倍,说明总RNA有良好的完整性。紫外分光光度法测得的A260/A280为1.8-2.0,表明总RNA纯度高,浓度适宜(表 2)。所提总RNA符合下一步实验的要求。
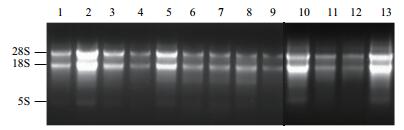
|
| 图 1 华细辛不同生长时期不同组织总RNA电泳图 1:花;2:叶C;3:叶A;4:叶柄C;5:叶柄A;6:根茎C;7:根茎A;8:根C;9:根A;10:根B;11:根茎B;12:叶B;13:叶柄B |
通过梯度稀释cDNA样本作为模板进行qPCR扩增后,绘制每个内参基因的标准曲线,得到引物相关参数(表 3)。从表中可以看到6个内参基因的斜率均在-3.6--3.2内,相关系数R2均高于定量PCR反应要求的0.98,说明其线性关系很好,扩增效率为92%-102%,且熔解曲线均只有单一峰,不存在非特异性扩增。上述结果表明各内参基因均符合实时荧光定量PCR对扩增效率的要求,可进行后续实验。
通过对目的基因在mRNA水平上的表达量进行定量测定,计算每个基因Ct值。Ct值大小反映了对应基因的表达丰度高低,Ct值越小,表达丰度越高。图 2可以看出,这6个基因的Ct值为21-35,其中18S rRNA丰度最高(Ct < 25)。

|
| 图 2 六个内参基因的Ct值分析 |
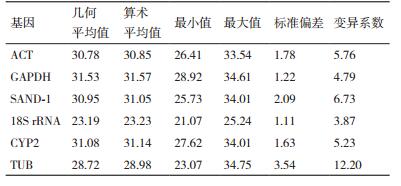
利用geNorm程序对各候选内参基因的表达稳定性进行分析。通过计算基因的表达稳定值(M)并对其进行排序,M越小,基因稳定性越高。分析结果(图 3)显示,不同样品间各候选内参基因表达稳定值M大小顺序为18S rRNA < GAPDH < ACT < CYP-2 < SAND-1 < TUB,18S rRNA的M最小,表达最稳定,TUB稳定性最差。
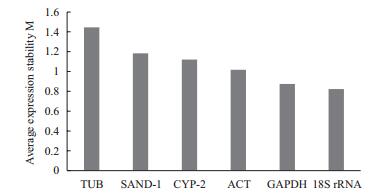
|
| 图 3 候选基因在华细辛不同样品中表达稳定性的geNorm分析 |
BestKeeper程序主要通过比较Ct值的标准偏差和变异系数来选择表达最稳定的基因,标准偏差和变异系数越小,稳定性越好;反之,稳定性越差。分析结果(表 4)显示,在所有样品中,ACT、GAPDH、SAND-1、18SrRNA、CYP2、TUB的标准偏差分别为1.78、1.22、2.09、1.11、1.63、3.54,18S rRNA最小,为1.11,表达最稳定,TUB表达稳定性最差。变异系数的大小顺序为18S rRNA < GAPDH < CYP-2 < ACT < SAND-1 < TUB,18S rRNA表达最稳定,TUB表达稳定性最差,与标准偏差结果一致。综上,18S rRNA在各个样品中表达最稳定。
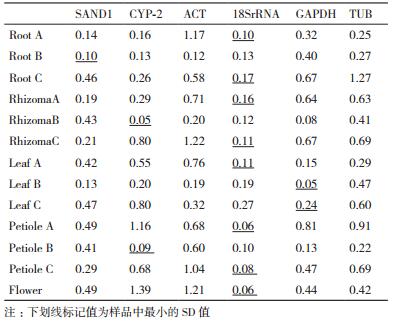
ΔCt法是通过比较候选内参基因之间的ΔCt值来筛选表达最稳定的内参基因的分析方法,即通过计算各基因的平均标准偏差(SD值),对基因的表达稳定性进行排序,平均标准偏差越小,基因稳定性越高。
结果(表 5)表明,在Root A、Root C、Rhizoma A、Rhizoma C、Leaf A、Petiole A、Petiole C、Flower共8个样品中,18S rRNA均具有最小的SD值(在表格中已用下划线标出),表明在此样品中稳定性最强;在样品Rhizoma B与Petiole B中,CYP-2表达稳定性最强;而在样品Leaf B与Leaf C中,GAPDH具有最强的表达稳定性;Root B中,SD值为GAPDH>TUB>CYP-2=18S rRNA>ACT>SAND-1,即SAND-1表达稳定性最强。而图 4也直观地表明候选内参基因18S rRNA在华细辛大多数样品中具有较小的SD值,且各样品间SD值相对其它候选内参基因差距较小,是相对表达最稳定的内参基因。虽说有5个样品中表达最稳定的并不是18S rRNA,但是筛选内参基因主要筛选在大多数样品中都具有较强稳定性的内参基因,综合来看,在华细辛中适合作为基因差异表达的内参基因为18S rRNA。
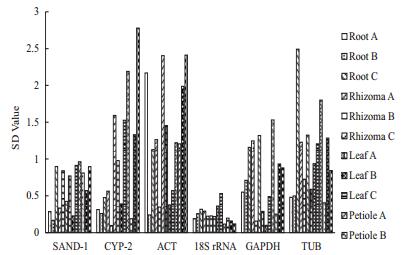
|
| 图 4 ΔCt法分析候选基因在华细辛不同样品中表达稳定性 |
利用NormFinder程序分析6个候选内参基因的表达稳定性,稳定值越小,基因表达越稳定。分析结果(图 5)显示,各样品中候选内参基因表达稳定性依次为18S rRNA>GAPDH>ACT>SAND-1>CYP-2>TUB,18S rRNA表达最稳定。这与geNorm、BestKeeper、Delta CT法分析的结果一致。
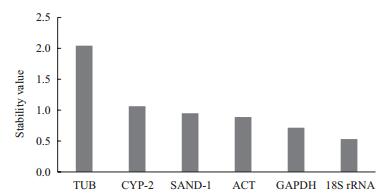
|
| 图 5 NormFinder分析候选内参基因在华细辛不同样品中的表达 |
荧光定量PCR是定量检测基因表达水平最重要的方法,为了获得准确、可靠的实时荧光定量PCR结果,实验方案设计合理,引物设计合适,RNA提取质量高、聚合酶扩增效率高、cDNA的合成以及PCR准备过程中的准确操作和防止污染等实验过程的每一步操作都很重要[12]。此外,选择合适的内参基因也是准确分析目标基因表达变化的重要条件。
在qRT-PCR分析中,内参基因的使用被认为是最适当的标准化方法,使用不理想的内参基因将对数据的分析造成偏差[15, 16]。在植物中,常用的内参基因有肌动蛋白基因(ACT)、18S核糖体RNA(18S rRNA)、3-磷酸甘油醛脱氢酶基因(GAPDH)、转录延伸因子基因(EF-1α)、多聚泛素酶基因(UBQ)、α微管和β微管蛋白基因(TUA、TUB)以及亲环蛋白基因(CYP)等[17]。但是内参基因常常表现为在某些实验条件下表达较稳定,而在另一些条件下欠稳定。因此,研究目标基因的表达情况时,研究者需要结合各自的实验条件和样品类型,根据物种、基因家族等的不同,选择合适而稳定的内参基因进行校正和标准化,才能有助于得到可靠的实验结果。到目前为止,很多植物在不同条件下的合适的内参基因已经被筛选出来,如孙美莲等[18]指出,比较茶树不同组织中的基因表达差异时,β-ACT作为校正内参基因最合适。Iskandar等[19]研究表明,在β-ACT、β-TUB、GAPDH与25S rRNA四个候选基因中,25S rRNA在甘蔗各组织中表达始终一致,是理想的内参基因;而GAPDH在成熟茎节间表达也较稳定,亦可作为研究甘蔗表达量的合适内参基因。Mascia等[20]分析了番茄的8个内参基因在5种不同植物病毒侵染条件下的表达稳定性,结果表明,UBI和GAPDH在所有样品中表达都相对稳定,而ACT在侵染样品中表达稳定性最好。因此,选择合适的内参基因是实验结果准确的前提条件。
4 结论本研究选取SAND-1、ACT、18S rRNA、CYP2、GAPDH、TUB为候选内参基因,分析其在华细辛不同样品中的表达稳定性。结果表明,在geNorm、BestKeeper、Delta CT、NormFinder四种不同的软件分析中,表达最稳定的内参基因均趋向18S rRNA,而4个软件是基于不完全相同的算法来对内参基因稳定性进行衡量的。可见,在研究华细辛不同时期不同组织中相关基因的表达时,18S rRNA作为内参基因是较为合适可靠的。
| [1] | 国家药典委员会. 《中华人民共和国药典》(2015版一部)[M]. 北京: 中国医药科技出版社, 2015. |
| [2] | 王晓丽, 金礼吉, 续繁星, 等. 中草药细辛研究进展[J]. 亚太传统医药, 2013(97): 68–71. |
| [3] | 韩俊艳, 孙川力, 纪明山. 中药细辛的研究进展[J]. 中国农学通报, 2011, 27(9): 46–50. |
| [4] | Valasek MA, Repa JJ. The power of real-time PCR[J]. Advances in Physiology Education, 2005, 29 (3): 151–159. DOI:10.1152/advan.00019.2005 |
| [5] | Mackay IM. Real-time PCR in the microbiology laboratory[J]. Clinical Microbiology and Infection, 2004, 10 (3): 190–212. DOI:10.1111/j.1198-743X.2004.00722.x |
| [6] | 赵焕英, 包金凤. 实时荧光定量PCR技术的原理及其应用研究进展[J]. 中国组织化学和细胞化学杂志, 2007, 16(4): 492–497. |
| [7] | Andersen CL, Jensen JL, Ørntoft TF. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64 (15): 5245–5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [8] | 张艳君, 朱志峰, 陆融. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展, 2007, 34(5): 546–550. |
| [9] | Guénin S, Mauriat M, Pelloux J, et al. Normalization of qRT-PCR data: the necessity of adopting a systematic, experimental conditions-specific, validation of references[J]. Journal of Experimental Botany, 2009, 60 (2): 487–493. DOI:10.1093/jxb/ern305 |
| [10] | 周兰, 张利义, 张彩霞, 等. 苹果实时荧光定量PCR分析中内参基因的筛选[J]. 果树学报, 2012(6): 965–970. |
| [11] | 吴文凯, 刘成前, 周志刚, 等. 用于莱茵衣藻荧光定量PCR分析的内参基因选择[J]. 植物生理学报, 2009, 45(7): 667–672. |
| [12] | Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3 (7): research0034.1. |
| [13] | Pfaffl MW, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26 (6): 509–515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [14] | Silver N, Best S, Jiang J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Molecular Biology, 2006, 7 (1): 33. DOI:10.1186/1471-2199-7-33 |
| [15] | Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR[J]. Nature Protocols, 2006, 1 (3): 1559–1582. DOI:10.1038/nprot.2006.236 |
| [16] | Dheda K, Huggett JF, Chang JS, et al. The implications of using an inappropriate reference gene for real-time reverse transcription PCR data normalization[J]. Analytical Biochemistry, 2005, 344 (1): 141–143. DOI:10.1016/j.ab.2005.05.022 |
| [17] | Dheda K, Huggett JF, Bustin SA, et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR[J]. Biotechniques, 2004, 37 : 112–119. |
| [18] | 孙美莲, 王云生, 杨冬青, 等. 茶树实时荧光定量PCR分析中内参基因的选择[J]. 植物学报, 2010, 45: 579–587. DOI:10.3969/j.issn.1674-3466.2010.05.007 |
| [19] | Iskandar HM, Simpson RS, Casu RE, et al. Comparison of reference genes for quantitative real-time polymerase chain reaction analysis of gene expression in sugarcane[J]. Plant Molecular Biology Reporter, 2004, 22 (4): 325–337. DOI:10.1007/BF02772676 |
| [20] | Mascia T, Santovito E, Gallitelli D, et al. Evaluation of reference genes for quantitative reverse-transcription polymerase chain reaction normalization in infected tomato plants[J]. Molecular Plant Pathology, 2010, 11 (6): 805–816. |