2. 中国科学院天津工业生物技术研究所,天津 300308
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Science; Tianjin 300308
天然产物的糖基化在生物体中发挥着重要作用[1-4],不仅使代谢产物变得丰富多样,而且可改变化合物的理化性质,如增加溶解度、增强稳定性、促进在活细胞中的储存和积累以及调节其化学性质和生物活性。酚类化合物广泛存在于自然界中,从简单的酚酸到复杂的黄酮类和花青素等[5]。据报道,酚类化合物具有抗衰老、抗氧化、抗恶性细胞增殖等功能[6]。酚类化合物的糖基化在植物和微生物中有着重要的意义,如改变花的颜色,增加苷元的可溶性和降低外源物质的毒性等[7-8]。此外,酚类化合物糖基化可提高其生物活性,如柚皮苷、熊果苷和水杨苷等[7, 9]。有研究发现,将水飞蓟素A进行糖基化得到水飞蓟素A-7-O-β-吡喃葡萄糖苷后,其生物利用率显著提高[10]。糖基化修饰可以使外源化合物的理化性质与生物活性发生明显变化,利用生物技术改变天然产物的糖基化类型从而获得更多的天然产物及其衍生物成为该领域的研究热点[11-17]。
Ma等[18]利用RACE技术从红景天植株中分离得到糖基转移酶UGT73B6,并证明该酶可以将酪醇转化为红景天苷。UGT73B6是首个报道的参与红景天苷合成的糖基转移酶。本实验室利用UGT73B6实现了红景天苷和天麻素在大肠杆菌中异源合成,并且产物中同时合成了酪醇、对羟基苯甲酸和对羟基苯甲醛葡萄糖基化副产物[19]。因此,UGT73B6对小分子酚类化合物具有一定的底物宽泛性。
本研究为进一步探索糖基转移酶UGT73B6潜在底物,利用大肠杆菌全细胞生物转化的方法,在UGT73B6重组菌中饲喂7种不同种类的酚类化合物,以期实现其相应的糖苷化合物的合成,旨为酚苷类化合物的生物合成提供案例参考。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒大肠杆菌DH5α、BL21(DE3)由本实验室保存。质粒pET28a购于Novagen公司。质粒pGEX-4T-1-ugt73b6由上海捷瑞公司合成。
1.1.2 试剂Phusion超保真DNA聚合酶购自New England Biolabs公司;限制性内切酶Nde I、Bam HI购自Fermentas公司;T4连接酶购自TaKaRa Biotechnology公司;3,5-二羟基甲苯、4-甲氧基苯酚、间甲基苯酚、对羟基苯丙酸、白藜芦醇、愈创木酚、香豆酸标准品购自百灵威公司;异丙基-β-D-硫代半乳糖苷(IPTG)购自北京索莱宝生物科技有限公司。
1.1.3 培养基LB培养基:酵母粉5 g/L,胰蛋白胨10 g/L,氯化钠10 g/L;M9培养基:磷酸氢二钠12.8 g/L,磷酸氢二钾3 g/L,氯化钠0.5 g/L,氯化铵1 g/L,葡萄糖2 g/L,硫酸镁0.24 g/L,氯化钙11.1 g/L。
1.1.4 实验底物本研究选择了7种小分子的酚类化合物作为UGT73B6的潜在底物进行全细胞转化实验。所用底物及其结构见表 1。
ugt73b6(GenBank accession number:AY547304)来自库页红景天(Rhodiola sachalinensis),由上海捷瑞公司合成,为了在大肠杆菌中使用进行了密码子优化,用ugt73b6syn来表示。合成的ugt73b6syn基因片段被克隆到pGEX-4T-1载体,由捷瑞公司构建成pGEX-4T-1-ugt73b6。以限制性内切酶Nde I和BamH I对pET28a质粒和pGEX-4T-1-ugt73b6分别进行酶切;通过凝胶电泳回收载体及基因片段;以载体与基因片段摩尔数比为1:3进行连接,获得质粒pET28a-ugt73b6。质粒pET28a-ugt73b6转化至BL21(DE3),获得重组菌株BL21(DE3)/pET28a-ugt73b6。质粒pET28a转入到BL21(DE3)中,获得菌株BL21(DE3)/pET28a。
1.2.2 SDS-PAGE检测蛋白表达分别挑取BL21(DE3)/pET28a与BL21(DE3)/pET28a-ugt73b6菌株单克隆于3 mL液体LB中,37℃培养12 h;按体积比1:100转接至50 mL液体LB培养基,37℃培养至OD600为0.6-0.8,加入终浓度0.1 mmol/L的IPTG于16℃进行蛋白诱导24 h,收集发酵液2 mL,12 000 r/min离心10 min;加入200 μL的5 × loading buffer,沸水浴10 min,12 000 r/min离心10 min,上样量5 μL。
1.2.3 全细胞转化挑取重组菌BL21(DE3)/pET28a-ugt73b6与BL21(DE3)/pET28a单克隆,分别接种至3 mL液体LB中,37℃培养12 h;按体积比1:100转接至50 mL液体LB培养基,37℃培养至OD600为0.6-0.8,加入终浓度0.1 mmol/L的IPTG于16℃进行蛋白诱导,蛋白诱导时间为24 h;4 000 r/min离心10 min,收集菌体,加入50 mL M9Y发酵培养基(含0.025% Yeast Extract)进行重悬;培养基中添加终浓度为2 mmol/L的催化底物,于30℃继续培养,48 h后取样进行产物检测。
1.2.4 发酵产物HPLC和LC-MS分析检测取发酵液1 mL,12 000 r/min离心10 min,收集上清。HPLC检测系统是岛津液相色谱仪;检测条件为:HPLC流动相A =水(含0.1%甲酸),B =甲醇;流速= 1 mL/min,溶液配比为二元浓度梯度,洗脱条件:0-5 min 5% B,6-45 min 5% B到100% B;进样量20 μL;液相色谱柱为Agela Innoval C18柱(4.6 × 250 mm);UV检测波长254 nm。
LC-MS检测:配有紫外检测器的安捷伦1260系统和配有ESI离子源探针的bruker microQ-TOF Ⅱ质谱仪,检测条件包括:Agela Innoval C18柱(4.6 × 250mm);UV检测波长254 nm;流动相A =水(含0.1%甲酸),B =甲醇;流速= 1mL/min,溶液配比为二元浓度梯度,洗脱条件:0-5 min 5% B,6-45 min 5% B到100% B;进样量20 μL;ESI正离子源,分子量扫描范围50-800。
1.2.5 糖苷化合物的分离制备按照1.2.3所述方法进行发酵液的制备,发酵总量为1 L。发酵结束后,将发酵液4 000 r/min离心15 min,取上清。将预处理好的大孔树脂SP-825(树脂用量为发酵液体积的50%)装入层析柱,加入发酵液离心后的上清,流速可控制在2 mL/min,使树脂充分吸附发酵产物,待发酵液流尽后,用两倍柱体积的水洗脱残留在树脂柱中的杂质,之后用80%乙醇洗脱两个柱体积,收集洗脱液,分离得到的溶液用旋转蒸发仪减压浓缩。最后,用1-2 mL甲醇将分离得到的各组分从旋蒸瓶中转移出来。HPLC分离条件为:岛津高效液相色谱半制备仪,测定条件包括:检测器SPD-20A,检测波长254 nm;流动相A =水(含0.1%体积甲酸)B =甲醇;流速= 4 mL/min;洗脱条件:65%A和35%B;进样量100 μL。
1.2.6 糖苷化合物的NMR检测样品经干燥后溶于500 μL为氘代甲醇转入直径2.5 mm核磁管中,用Bruker Avance Ⅲ 400 MHz核磁共振仪测定1H NMR。
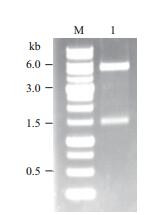
2 结果 2.1 载体pET28a-ugt73b6的验证重组载体pET28a-ugt73b6用Nde I和BamH I酶切验证,得到大小为1.5 kb和5.3 kb两条带(图 1),与理论大小一致,并送金唯智测序公司进行了DNA测序验证。
|
| 图 1 载体pET28a-ugt73b6构建 M:DNA Marker;1:载体pET28a-ugt73b6用Nde I/Bam H I双酶切 |
BL21(DE3)/pET28a与BL21(DE3)/pET28a-ugt73b6菌株按照1.2.2的方法发酵并诱导蛋白,制备样品,进行蛋白胶电泳,染色,脱色后SDS-PAGE如图 2所示,UGT73B6蛋白条带大小为53.9 kD,说明目的蛋白成功表达。

|
| 图 2 BL21(DE3)/pET28a与BL21(DE3)/pET28a-ugt73b6SDS-PAGE检测图 M:蛋白marker;1:BL21(DE3)/pET28a总蛋白;2:BL21(DE3)/pET28a-ugt73b6总蛋白 |
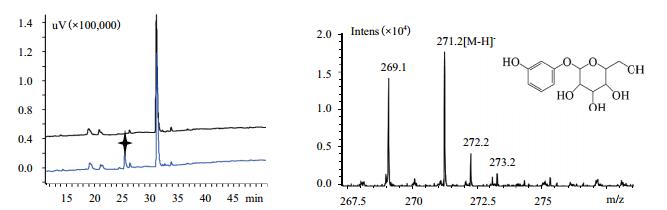
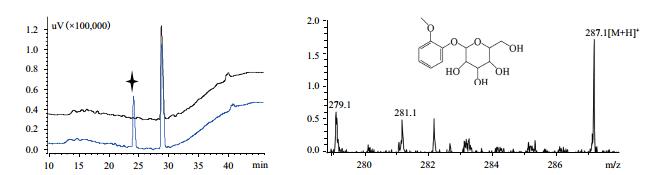
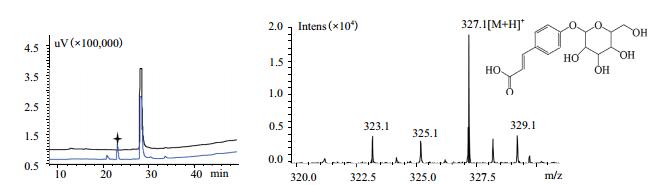
按方法1.2.3进行全细胞转化实验,对菌株BL21(DE3)/pET28a和菌株BL21(DE3)/pET28a-ugt73b6进行发酵培养,取48 h的发酵液进行HPLC检测,结果如图 2-9所示,其中菌株BL21(DE3)/pET28a均无新化合物产生,菌株BL21(DE3)/pET28a-ugt73b6中7种底物都有新化合物的合成(四角星所对应的峰)。对新化合物进行LC-MS检测,结果表明7种底物都发生葡萄糖基化,因此推断糖基转移酶UGT73B6对所选取的7种小分子的酚类化合物都有催化活性,生成了糖苷化合物。
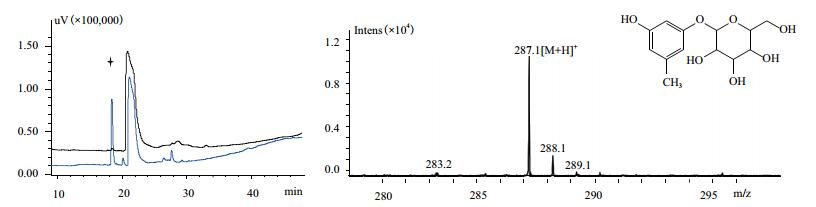
|
| 图 3 菌株BL21(DE3)/pET28a-ugt73b6添加底物3,5-二羟基甲苯HPLC(A)及LC-MS(B)检测结果 |
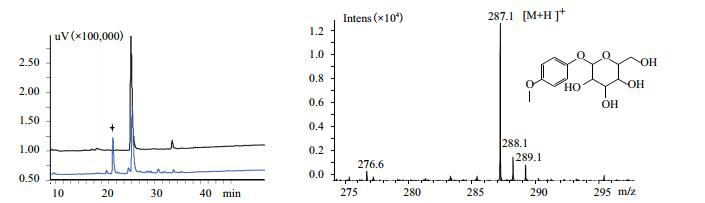
|
| 图 4 菌株BL21(DE3)/pET28a-ugt73b6添加底物4-甲氧基苯酚HPLC(A)及LC-MS(B)检测结果 |
|
| 图 5 菌株BL21(DE3)/pET28a-ugt73b6添加底物间甲基苯酚HPLC(A)及LC-MS(B)检测结果 |
|
| 图 6 菌株BL21(DE3)/pET28a-ugt73b6添加底物愈创木酚HPLC(A)及LC-MS(B)检测结果 |
|
| 图 7 菌株BL21(DE3)/pET28a-ugt73b6添加底物香豆酸HPLC(A)及LC-MS(B)检测结果 |

|
| 图 8 菌株BL21(DE3)/pET28a-ugt73b6添加对羟基苯丙酸HPLC(A)及LC-MS(B)检测结果 |
|
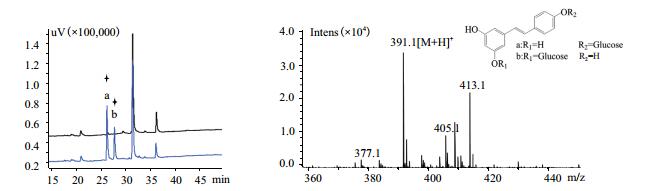
| 图 9 菌株BL21(DE3)/pET28a-ugt73b6添加白藜芦醇HPLC(A)及LC-MS(B)检测结果 |
为进一步确定图 9中a与b两种白藜芦醇糖苷化合物的结构,对这两种化合物进行1H NMR鉴定。化合物a的1H NMR显示,该化合物结构中含有一个对位取代的苯环[δH = 7.38(d,J = 8.6 Hz,2 H),6.77(d,J = 8.6 Hz,2 H)],一个间位三取代的苯环[δH = 6.95(d,J = 2.1 Hz,1 H),6.77(d,J = 8.6 Hz,2 H)],一个双键[7.08(d,J = 16.3 Hz,1 H),6.89(d,J = 16.3 Hz,1 H)],这与苷元白藜芦醇结构一致,另外化合物含有典型的葡糖糖苷分子信号[4.94(d,J = 7.5 Hz,1 H),3.94(dd,J = 12.0,2.2 Hz,1H),3.70(dd,J = 12.0,6.3 Hz,1H),3.56-3.33(m,4 H)],结合桥头碳上氢的耦合常数为7.5 H,可确定为β构型糖苷键,参照相关文献[20-21]可以最终确定保留时间为25.5 min的化合物a为白藜芦醇-4' -O-β-D-葡萄糖苷;另一保留时间为27.5 min的化合物b的1H NMR显示,该化合物同样具有白藜芦醇苷元结构[δ = 7.45(d,J = 8.8 Hz,2 H),7.08(d,J = 8.8 Hz,2 H),7.00(d,J = 16.3 Hz,1 H),6.88(d,J = 16.3 Hz,1 H),6.47(d,J = 2.2 Hz,2 H),6.18(t,J = 2.1 Hz,1 H)],高场区信号显示化合物为葡萄糖苷产物,根据桥头碳原子上氢的耦合常数为7.5 H,可确定为β构型糖苷键,结合文献报道[20-21]化合物b鉴定为白藜芦醇-3-O-β-D-葡萄糖苷。
3 讨论本研究通过微生物转化实验,利用糖基转移酶UGT73B6,饲喂7种不同的酚类化合物,首次实现利用糖基转移酶UGT73B6获得相应的8种糖苷化合物。
基于3,5-二羟基甲苯、4-甲氧基苯酚、间甲基苯酚、对羟基苯丙酸、愈创木酚和香豆酸结构特点,可根据HPLC及LC-MS检测结果确定所对应糖苷化合物的结构。底物白藜芦醇因具有多个羟基,为了确定具体的催化位置,对其进行放大发酵实验,提取分离获得催化产物,经NMR检测,确定了糖基转移酶可以分别催化白藜芦醇3和4' 位的羟基形成单糖苷化产物。这表明糖基转移酶UGT73B6能够催化白藜芦醇合成不同结构类型的糖苷化合物,为其多羟基底物合成结构多样的糖苷化合物提供了一种有效的方法。
基因ugt73b6来源于植物红景天,本研究中仅对其DNA序列进行了密码子优化,为了使蛋白在大肠杆菌中更好的表达,接下来可以对诱导剂IPTG浓度和蛋白诱导温度进行优化,以确定更合适的蛋白诱导条件,未来工作中还可以通过提高蛋白的可溶性等方面来提高蛋白的表达及活性[22],从而进一步提高糖苷化合物的产量。另外,可以对ugt73b6基因进行易错PCR筛选以及定点饱和突变,从而得到催化效率更高的突变体,以提高产物的产量;并且可以进一步探索糖基转移酶ugt73b6的底物宽泛性,研究该酶对底物的催化规律。
生物转化法修饰天然产物,不仅仅应用于酚类化合物,还可以应用于萜类、甾体类、生物碱以及黄酮类等多种结构类型的化合物,在增加天然化合物的结构多样性方面具有极大的潜力[23]。我国天然产物资源丰富,在天然化合物领域拥有良好的研究基础,目前已经分离鉴定了大量的不同结构类型的天然产物,可进一步将生物转化技术引入到天然产物的研究中,包括资源开发、药物设计和新的活性先导化合物的发现与筛选等各个环节,从而开发出拥有自主知识产权、并具有中国特色的创新药物[24]。
糖基化可赋予多酚类化合物新的特性及生物学活性,是新药筛选的一个重要手段,逐渐成为国内外天然药物开发利用研究的热点。本研究中得到的8种糖苷类化合物都具有重要的实用价值及研究价值,研究选取的7种酚类化合物底物,其中,白藜芦醇是天然的抗氧化剂,能够降低血液黏稠度、保持血液畅通等;间甲基苯酚主要用作农药中间体,生产杀虫剂;4-甲氧基苯酚可用作乙烯基型塑料单体的阻聚剂、紫外线抑制剂和化妆品的抗氧化剂等;愈创木酚和3,5-二羟基甲苯多用于香料、染料的合成;香豆酸和对羟基苯丙酸是重要的医药和材料中间体。糖基化也许能够赋予它们新的生物活性,为进一步研究其药用功能奠定基础。
4 结论本研究在大肠杆菌BL(DE3)中,利用糖基转移酶UGT73B6,饲喂7种不同底物,成功获得相对应的糖苷化合物。不仅展示了糖基转移酶UGT73B6对酚类化合物的底物宽泛性,并且为天然化合物的分子结构修饰提供了一种新的途径。
| [1] | Modolo LV, Li L, Pan H, et al. Crystal structures of glycosyltransferase UGT78G1 reveal the molecular basis for glycosylation and deglycosylation of (iso) flavonoids[J]. Journal of Molecular Biology, 2009, 392 (5): 1292–1302. DOI:10.1016/j.jmb.2009.08.017 |
| [2] | Gachon CM, Langlois-Meurinne M, Saindrenan P. Plant secondary metabolism glycosyltransferases: the emerging functional analysis[J]. Trends in Plant Science, 2005, 10 (11): 542–549. DOI:10.1016/j.tplants.2005.09.007 |
| [3] | Jones P, Vogt T. Glycosyltransferases in secondary plant metabolism: tranquilizers and stimulant controllers[J]. Planta, 2001, 213 (2): 164–174. DOI:10.1007/s004250000492 |
| [4] | Bowles D, Isayenkova J, Lim EK, et al. Glycosyltransferases: managers of small molecules[J]. Current Opinion in Plant Biology, 2005, 8 (3): 254–263. DOI:10.1016/j.pbi.2005.03.007 |
| [5] | Cheynier V. Phenolic compounds: from plants to foods[J]. Phytochemistry Reviews, 2012, 11 (2-3): 153–177. DOI:10.1007/s11101-012-9242-8 |
| [6] | Lin D, Xiao M, Zhao J, et al. An Overview of plant phenolic compounds and their importance in human nutrition and management of Type 2 diabetes[J]. Molecules, 2016, 21, 1374 : 1–19. |
| [7] | Woo HJ, Kang HK, Nguyen TT, et al. Synthesis and characterization of ampelopsin glucosides using dextransucrase from Leuconostoc mesenteroides B-1299CB4: glucosylation enhancing physicoche-mical properties[J]. Enzyme and Microbial Technology, 2012, 51 (6-7): 311–318. DOI:10.1016/j.enzmictec.2012.07.014 |
| [8] | Raab T, Barron D, Vera FA, et al. Catechin glucosides: occurrence, synthesis, and stability[J]. Journal of Agricultural and Food Chemistry, 2010, 58 (4): 2138–2149. DOI:10.1021/jf9034095 |
| [9] | Blanchard S, Thorson JS. Enzymatic tools for engineering natural product glycosylation[J]. Current Opinion in Chemical Biology, 2006, 10 (3): 263–271. DOI:10.1016/j.cbpa.2006.04.001 |
| [10] | Li D, Roh SA, Shim JH, et al. Glycosylation of genistin into soluble inclusion complex form of cyclic glucans by enzymatic modification[J]. Journal of Agricultural and Food Chemistry, 2005, 53 (16): 6516–6524. DOI:10.1021/jf050732g |
| [11] | Leadlay PF. Combinatorial approaches to polyketide biosynthesis[J]. Current Opinion in Chemical Biology, 1997, 1 (2): 162–168. DOI:10.1016/S1367-5931(97)80005-1 |
| [12] | Floss HG. Antibiotic biosynthesis: from natural to unnatural compounds[J]. Journal of Industrial Microbiology & Biotechnology, 2001, 27 (3): 183–194. |
| [13] | Mendez C, Salas JA. Altering the glycosylation pattern of bioactive compounds[J]. Trends in Biotechnology, 2001, 19 (11): 449–456. DOI:10.1016/S0167-7799(01)01765-6 |
| [14] | Fu X, Albermann C, Jiang J, et al. Antibiotic optimization via in vitro glycorandomization[J]. Nature Biotechnology, 2003, 21 (12): 1467–1469. DOI:10.1038/nbt909 |
| [15] | Yang J, Hoffmeister D, Liu L, et al. Natural product glycorandomization[J]. Bioorganic & Medicinal Chemistry, 2004, 12 (7): 1577–1584. |
| [16] | Rupprath C, Schumacher T, Elling L. Nucleotide deoxysugars: essential tools for the glycosylation engineering of novel bioactive compounds[J]. Current Medicinal Chemistry, 2005, 12 (14): 1637–1675. DOI:10.2174/0929867054367167 |
| [17] | Melancon CE 3 rd, Thibodeaux CJ, et al. Glyco-stripping and glyco-swapping[J]. ACS Chemical Biology, 2006, 1 (8): 499–504. DOI:10.1021/cb600365q |
| [18] | Ma LQ, Liu BY, Gao DY, et al. Molecular cloning and overexpression of a novel UDP-glucosyltransferase elevating salidroside levels in Rhodiola sachalinensis[J]. Plant Cell Reports, 2007, 26 (7): 989–999. DOI:10.1007/s00299-007-0317-8 |
| [19] | Bai Y, Bi H, Zhuang Y, et al. Production of salidroside in metabolically engineered Escherichia coli[J]. Scientific Reports, 2014, 4 : 6640. |
| [20] | Zhang Z, Yu B, Schmidt R. Synthesis of mono- and di-O-β-d-glucopyranoside conjugates of (E)-resveratrol[J]. Synthesis, 2006, 2006 (8): 1301–1306. DOI:10.1055/s-2006-926394 |
| [21] | Zhou M, Hamza A, Zhan CG, et al. Assessing the regioselectivity of OleD-catalyzed glycosylation with a diverse set of acceptors[J]. Journal of Natural Products, 2013, 76 (2): 279–286. DOI:10.1021/np300890h |
| [22] | 田宁, 咸莫, 胡仰栋, 等. 产香叶醇重组大肠杆菌发酵培养基的优化[J]. 林产化学与工业, 2015, 35(4): 131–137. |
| [23] | Gantt RW, Goff RD, Williams GJ, et al. Probing the aglycon promiscuity of an engineered glycosyltransferase[J]. Angewandte Chemie International Edition, 2008, 47 : 8889–8892. DOI:10.1002/anie.v47:46 |
| [24] | 刘庆鑫, 李慧梁, 柳润辉, 等. 微生物转化在天然产物研究中的应用[J]. 药学实践杂志, 2012, 30(5): 321–325. |