盐胁迫、高温、干旱、水淹、机械损伤、空气污染和化学污染等非生物胁迫会引起植物内源胁迫乙烯分泌增多,而过量乙烯导致植物生长发育受阻或死亡[1-3]。1-氨基环丙烷-1-羧酸(1-aminocyclopropane-1-carboxylate,ACC)是乙烯合成的前体物质,而ACC脱氨酶可将ACC分解为氨和α-酮丁酸,降低乙烯浓度,缓解乙烯对植物产生的不良反应,从而促进植物生长发育,提高其耐盐性[4-8]。在土壤、植物根际和根内生环境中产ACC脱氨酶的细菌是普遍存在的[9-10],研究表明,植物根际促生菌(Plant growth promoting rhizobacteria,PGPR)、内生细菌(Endophytic bacteria)及木霉菌(Trichoderma sp.)等内生真菌(Endophytic fungi)均可产生ACC脱氨酶[11-12],尤其是对PGPR而言,利用ACC脱氨酶降低植物体内的乙烯水平,缓解盐胁迫对植物造成的损伤是其促进植物生长和抗逆的主要机制[5, 13-19]。和其他耗时较长,工作量大而效率低的传统ACC脱氨酶细菌的分离方法相比,通过PCR手段对编码ACC脱氨酶的acdS基因片段进行扩增的方法,高效快速且灵敏度高,具有良好的应用推广价值。快速筛选具ACC脱氨酶活性的菌株对于进一步挖掘多功能PGPR菌种资源,制备微生物肥料,促进农林业可持续发展具有着重要的生产应用价值[20-21]。本研究以期从多种盐生植物以及杨树根际、根内生部位分离培养所得大量细菌中,通过选择合适的引物,建立PCR反应体系,快速筛选产ACC脱氨酶菌株。
1 材料与方法 1.1 材料 1.1.1 根系及根际土样品采集采样地点为山东省青岛市黄岛区滨海大道风河大桥风河入海口和浙江省杭州市富阳区新登镇杨树良种基地,共采集10种盐生植物:盐地碱蓬(Suaeda salsa)、砂引草(Messerschmidia sibirica)、肾叶打碗花(Calystegia soldanella)、西伯利亚蓼(Polygonum sibiricum)、糙叶苔草(Carex scabrifolia)、筛草(Carex kobomugi)、滨藜(Atriplex patens)、无翅猪毛菜(Salsola komarovii)、芦苇(Phragmites australis)以及地肤(Kochia scoparia)和美洲黑杨(Populus deltoids)的根部及其根际土。将植物样品和土壤样品迅速装入塑封袋中。于冰盒中暂时存放,48 h内运输至实验室进行细菌分离培养。
1.1.2 主要试剂细菌基因组DNA提取试剂盒(离心柱形),Axygen生物公司,中国;1-Aminocyc-lopropane-1-carboxylic Acid(Merck公司);16S rRNA序列扩增引物由上海生物工程技术服务有限公司合成。
1.1.3 培养基1/10 TSA培养基、PIA培养基、LB培养基、PAF培养基、DF培养基(以硫酸铵或ACC为唯一氮源)和ADF培养基[15]。
1.2 方法 1.2.1 植物根系表面消毒和研磨用水将植物根系样品表面的杂物冲洗干净,再将植物根系样品修剪成段。在超净台中对植物样品进行表面消毒处理:先用75%的乙醇溶液(按体积比用无菌水稀释)浸泡样品1 min,接着用次氯酸钠溶液(0.5%的活性氯)浸泡样品5-10 min(具体时间视植物根系的幼嫩程度而定),再将样品放入装有90%的乙醇溶液的烧杯中漂洗次氯酸钠,最后用无菌水冲洗样品5次。随后,将已消毒的根系组织剪碎,置于无菌的50 mL离心管中,向管内倒入适量1×PBS缓冲液(以刚没过样品为准)。将研磨枪枪头置于酒精灯外焰上灼烧后于无菌水中冷却。将上述50 mL离心管插入碎冰中冰浴,将灭菌处理过的研磨枪枪头伸入管内调至最低档研磨样品。另取无菌的50 mL离心管,剪取一块无菌的纱布封在管口,将所得植物根系样品匀浆倒入无菌纱布封口的离心管内,得到根系研磨液,5℃冰箱内保存。
1.2.2 根际土的收集取植物根系样品,抖落根外土,将带有根际土的植物根系样品修剪成段置于装有1×PBS缓冲液的50 mL离心管中,在离心管外标注好对应的样品编号。将离心管放置在涡旋振荡器(Vortex-Genie 2,MOBIO公司)上充分振荡5 min,使根际土悬浮于缓冲液中。取出根系样品,将根际土悬浮液10℃下6 000×g离心15 min(德国Sigma 3K15台式高速冷冻离心机),倒掉上清液,收集根际土,放置于5℃冰箱内保存。
1.2.3 内生细菌和根际细菌的分离纯化本实验采用传统连续稀释法分离植物内生细菌和根际细菌[20]。取1 mL根系研磨液或1 g根际土,加至99 mL 1×PBS缓冲液,重新悬浮并充分混匀,随后按照1:10的比例依次进行系列稀释,分别得到10-3、10-4和10-5 3个浓度的土壤悬浮液。选取其中10-4和10-5两个浓度梯度的土壤悬液,充分混匀后各取100 µL,涂布于1/10 TSA、PIA培养基表面。每种培养基不同样品各浓度重复3次,作好标记。22℃避光培养3-4 d,及时观察细菌菌落。挑取少量菌体用“连续划线法”和“三线法”在LB培养基上进行纯化,直至出现单菌落。
1.2.4 产ACC脱氨酶细菌的分离和保存采用富集定向筛选[15]和连续稀释[22]的方法分离产ACC脱氨酶的细菌。取1 mL根系研磨液或根际土悬浮液,分别经PAF培养基、以硫酸铵或ACC为唯一氮源的DF培养基和ADF培养基进行筛选和培养,最后挑取不同形态的单菌落在LB培养基上划线纯化。
对分离纯化后所得细菌进行低温冷冻保藏,具体方法为:在超净台中以镊子夹取灭菌处理后的牙签从LB培养基上挑取细菌单菌落,放入50 mL液体LB培养液中,在28℃旋转摇床200 r/min条件下液体发酵12 h后,收集1 mL菌液以5 000×g转速离心10 min,弃去上清液,将菌体溶于灭菌处理过的50%甘油,利用移液枪吹打混匀后,将所得悬浊液加入冷冻管中于超低温冰箱(-70℃)中保藏。
1.2.5 细菌基因组的提取和PCR扩增挑取细菌单菌落至LB培养基中,28℃振荡培养至对数期取适量菌液,以5 000×g转速离心收集菌体,使用细菌基因组提取试剂盒,依照其步骤提取基因组。使用Q5000超微量紫外分光光度计(美国Quawell公司)测定DNA浓度,并根据OD260/280比值检测样品DNA纯度,-20℃保存样品。
以细菌基因组DNA为模板,对16S rRNA片段基因进行PCR扩增。引物为细菌16S rRNA通用引物356F和1064R,扩增区域为V3-V5可变区,扩增体系共50 μL,其中模板3 μL(约50 ng),正向反向引物各0.5 μL(浓度50 μmol/L),25 μL 2×Taq MasterMix(不含染料)(北京康为世纪生物科技有限公司),最后用无菌去离子水补足至50 μL。PCR扩增条件为90℃预变性4 min;94℃变性40 s,58℃退火50 s,72℃延伸1 min,30个循环;72℃延伸10 min,4℃保存。PCR产物用1.0%的琼脂糖凝胶分离,再用DNA纯化试剂盒(Axygen生物公司,中国)对目的片段进行胶回收并送至杭州擎科生物有限公司测序。
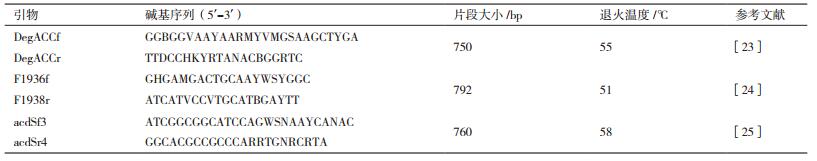
1.2.6 利用PCR技术筛选产ACC脱氨酶的细菌提取富集筛选得到的细菌菌株的DNA溶液作为PCR反应的模板,选择合适的引物来PCR扩增acdS片段从而筛选具ACC脱氨酶活性的细菌。本研究参考相关文献选用DegACCf/DegACCr、F1936f/ F1938r和acdSf3/acdSr4三对引物[23-25],引物信息详情见表 1。
(1)DegACCf和DegACCr使用DegACCf和DegACCr这对引物进行PCR扩增,采用50 μL体系:上下游引物DegACCf和DegACCr各加0.5 μL,DNA模板3 μL,2×Taq-MasterMix 25 μL和21 μL去离子水。PCR扩增程序设置为:90℃预变性4 min;95℃变性40s,55℃退火1 min,72℃延伸1 min,35次循环;72℃延伸5 min;4℃保存。PCR扩增所得产物直接进行1%琼脂糖凝胶(m/V)电泳检测。(2)F1936f和F1938r使用F1936f和F1938r这对引物进行PCR扩增,采用50 μL体系:上下游引物F1936f和F1938r各加0.5 μL,DNA模板3 μL,2×Taq-MasterMix 25 μL及21 μL去离子水。PCR程序设置为:90℃预变性4 min;95℃变性40 s,51℃退火1 min,72℃延伸1 min,35次循环;72℃延伸5min;4℃保存。PCR扩增所得产物直接进行1%琼脂糖凝胶电泳检测。(3)acdSf3和acdSr4使用acdSf3和acdSr4这对引物,采用50 μL PCR扩增体系:上下游引物acdSf3和acdSr4各加0.5 μL,DNA模板2 μL,2×Taq-MasterMix 25 μL及22 μL去离子水。采取递减PCR(touchdown-PCR)的方法:94℃预变性10 min;94℃变性40 s,65℃退火50 s,72℃延伸1 min,每个循环退火温度降低0.4℃,15个循环后退火降至58℃,循环20次;72℃延伸10 min,4℃保存。PCR扩增所得产物直接进行1%琼脂糖凝胶电泳检测。(4)acdS基因克隆测序将电泳后所得与目的片段大小一致的条带切胶,送往生工生物工程(上海)股份有限公司进行TA克隆测序。
1.2.7 核苷酸序列比对和系统发育分析16S rRNA和acdS基因测序结果用BioEdit v7.0.9和Genedoc v2.7.0等序列比对软件进行编辑,去除两端不准确序列后,在GenBank数据库中用BLAST功能(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行相似性搜索和同源性比对。调取匹配度较高的已登记菌株相应序列,通过ClustX1.81软件进行多序列比对,利用MEGA v5.1软件通过Neighbor-Joining法分别构建基于16S rRNA和acdS基因的系统发育进化树。其中,构建16S rRNA系统发育树时选择Asticcacaulis solisilvae、A. benevestitus、A. exxentricus和Brevundimonas sp.作为外群,构建acdS基因系统发育树时选择Rhizobium leguminosarum、R. gallicum、Sinorhizaobium meliloti和Pseudomonas sp.作为外群。生成outree文件后,使用FigTree v1.4.2软件(http://tree.bio.ed.ac.uk/software/figtree/)对系统发育树进行编辑和注释。
1.2.8 acdS基因编码的氨基酸序列的比对与系统发育分析acdS基因原始序列进行编辑,与GenBank数据库中已描述菌株对比,去除核苷酸序列内含子,得到编码ACC脱氨酶氨基酸的基因序列。去除内含子的基因序列进行blastx和Protein BLAST搜索,调取匹配度高的已知菌株acdS基因序列及氨基酸序列,通过ClustX1.81软件进行多序列比对,利用MEGA v5.1软件选用Poisson Correction模型构建基于细菌acdS基因核苷酸和氨基酸序列的系统发育进化树。
2 结果 2.1 盐生植物和杨树根际根内生细菌分离情况经分离纯化、富集培养和16S rRNA分子鉴定(图 1-A),从10种盐生植物和杨树样品中所得根际细菌154株,根内生细菌93株,共计247株。基于其16S rRNA片段在NCBI数据库进行BLAST比对,对其分类地位进行初步鉴定,结果显示其中243个菌株属于变形菌门(Proteobacteria),分属于γ变形菌纲(Gammaproteobacteria)、β变形菌纲(Betaproteobacteria)、α变形菌纲(Alphaproteobacteria);剩余4个菌株属于厚壁菌门(Firmicutes)芽胞杆菌纲(Bacilli)。各个分类单元中,内生细菌和根际细菌所占比例相当,无明显差异。内生细菌和根际细菌宿主和类别分布无明显规律,说明其宿主特异性较低。
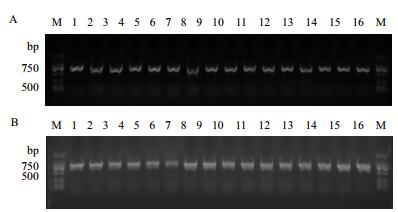
|
| 图 1 基于16S rRNA和acdS基因对16个代表性菌株PCR扩增产物电泳图谱 A:基于16S rRNA的电泳检测结果,目的条带片段大小约650 bp;B:基于acdS基因的电泳检测结果,目的条带片段大小约750 bp;M:DNA marker(康为世纪公司)扩增条带;1-16为代表性16个菌株的电泳条带 |
PCR扩增产物电泳结果显示,使用引物对DegACCf/DegACCr扩增出现较多非特异性条带且各片段大小不一,提高退火温度后,非特异性条带较多,且具有假阳性现象。使用引物对F1936f/F1938r的PCR扩增产物电泳结果中特异性条带较多,其中大小在790 bp左右的条带较多,但克隆测序结果表明扩增出的片段不是目的基因acdS基因。acdSf3和acdSr4引物的PCR扩增产物电泳结果中,条带清晰且特异性较好,片段大小在750 bp左右,与目的片段相符,扩增成功率高(图 1-B)。因此,本实验最终确定采用的是acdSf3/acdSr4引物对及其PCR反应体系。
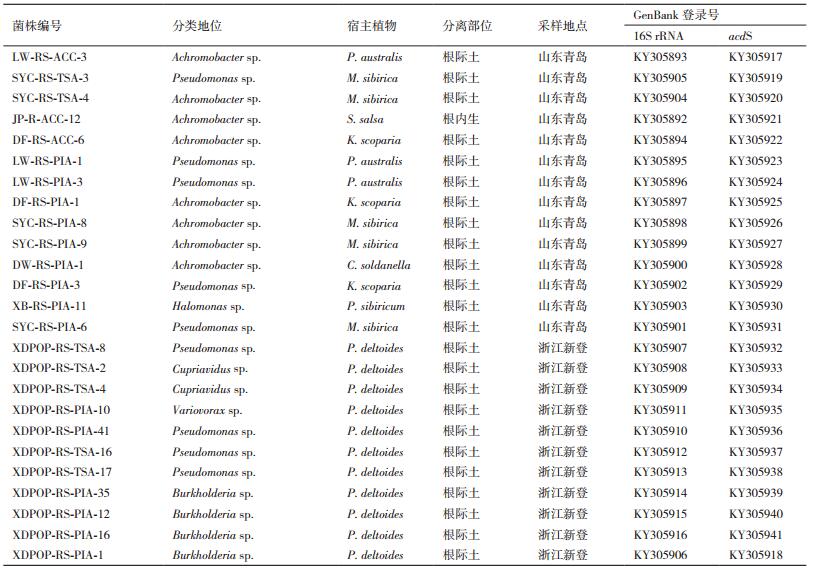
2.3 筛选得到25个产ACC脱氨酶的菌株采用acdSf3/acdSr4引物对及其PCR反应体系,对编码ACC脱氨酶的acdS序列基因片段进行扩增和克隆测序,结果显示,其中25株细菌的基因组中可扩增出acdS基因。该25株菌株相关信息及16S rRNA、acdS基因的GenBank登录号,见表 2。
基于16S rRNA构建的系统发育树中可以看出(图 2),具ACC脱氨酶活性的24株根际细菌和1株内生细菌分布于β变形菌纲(Betaproteobacteria)的伯克氏菌属(Burkholderia sp.)、贪铜菌属(Curiavidus sp.)、多嗜菌属(Variovorax sp.)和无色杆菌属(Achromobacter sp.),以及γ变形菌纲(Gamaproteobacteria)的假单胞杆菌属(Pseudomonas sp.)和盐单胞菌属(Halomonas sp.)内,共计2个纲6个属。在归类于β变形菌纲的15株细菌中,宿主植物为盐生植物根际或根系的8个细菌全部分布于无色杆菌属,且亲缘关系较近;而伯克氏菌属、贪铜菌属和多嗜菌属内则均为杨树根际细菌。γ变形菌纲10个细菌中,9个分离自盐生植物或杨树根际的细菌属于假单胞菌属,且属内多样性较丰富。系统发育树中步移值大于等于50的在分支节点标出,分支长度与同源进化呈正比关系。
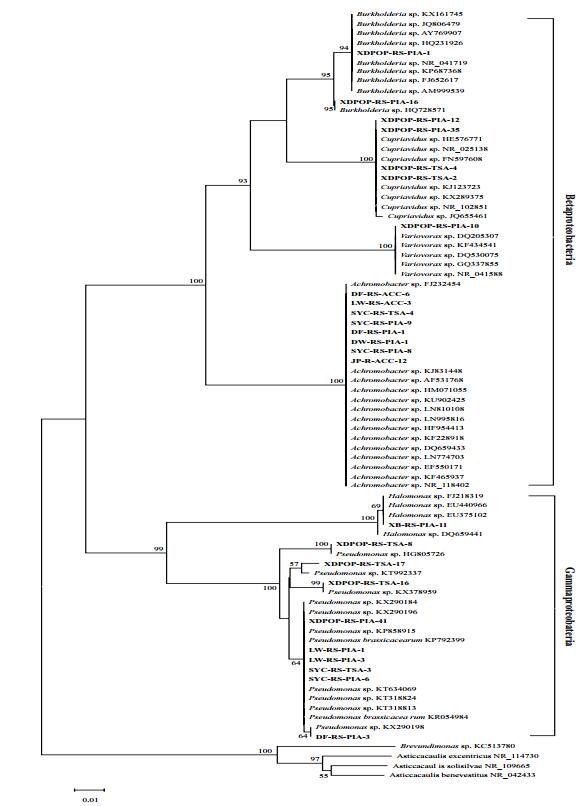
|
| 图 2 基于16S rRNA构建系统发育进化树 图中加粗字体部分表示本研究中25种产ACC脱氨酶的细菌菌株,下同 |
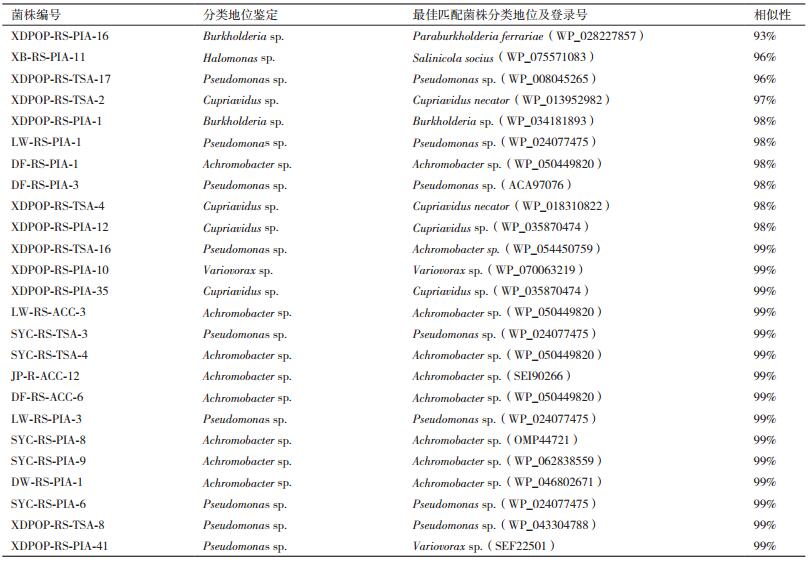
使用acdSf3和acdSr4这对引物对acdS基因扩增,获得相应基因片段,去掉内含子并翻译为相应氨基酸序列后在NCBI数据库内与已报道菌株进行BLAST比对,结果显示同源性为93%-99%(表 3),基于acdS核苷酸和氨基酸序列构建的系统发育树分别如图 3和图 4所示。
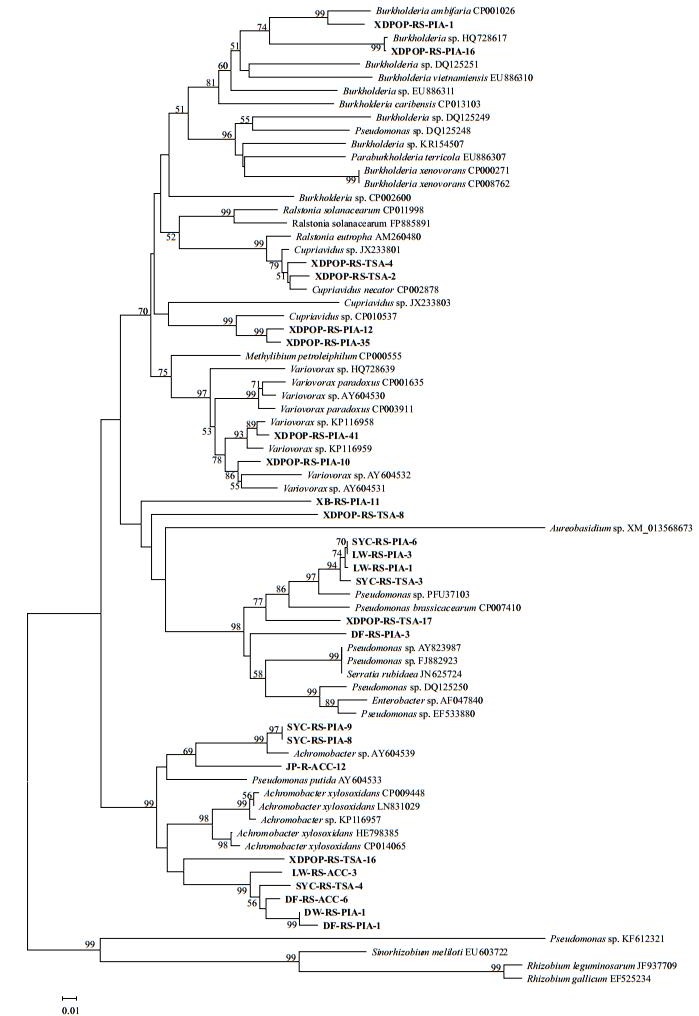
|
| 图 3 基于acdS基因构建系统发育进化树 |
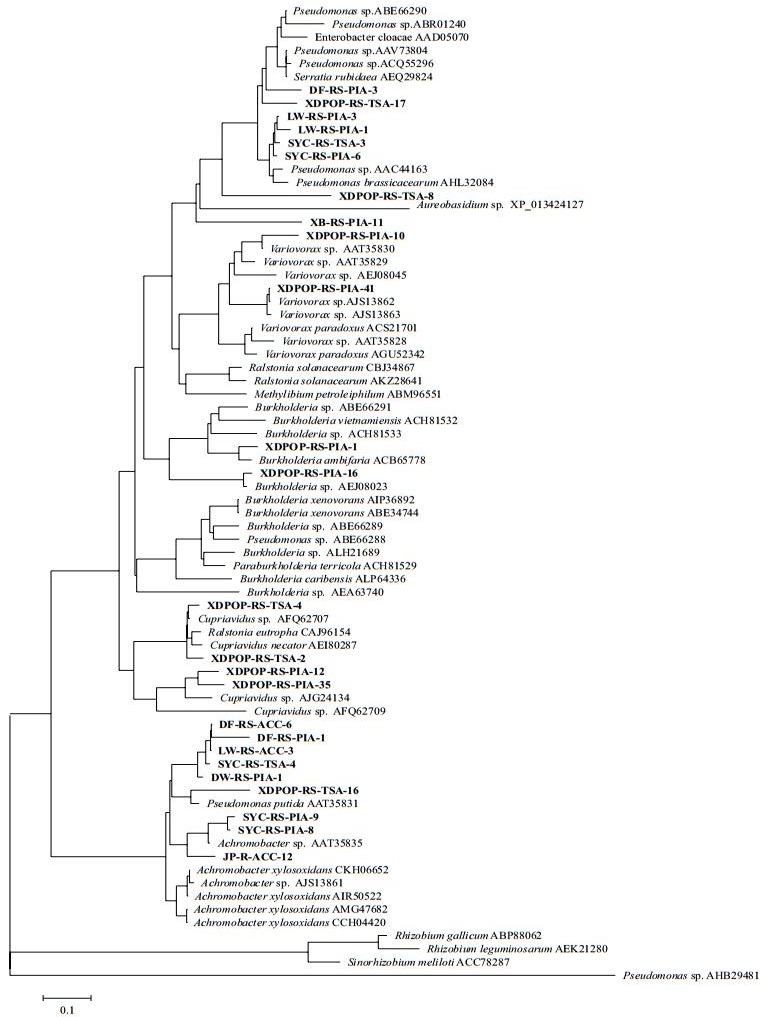
|
| 图 4 基于acdS基因序列编码的氨基酸序列构建系统发育进化树 |
通过基于acdS基因核苷酸序列构建的系统发育树(图 3)和基于acdS基因编码的氨基酸序列构建的系统发育树(图 4)可看出,25株具ACC脱氨酶活性的细菌主要可归类于两大基因簇。系统发育树中上部分基因簇中16个菌株分属4个亚群,其中LW-RS-PIA-1、LW-RS-PIA-3、SYC-RS-TSA-3和SYC-RS-PIA-6等4个假单胞菌属(Pseudomonas sp.)细菌同源性较高,功能最为相近,DF-RS-PIA-3及XDPOP-RS-TSA-17两个假单胞菌(Pseudomonas sp.)与LW-RS-PIA-1等4个假单胞菌的同源性较为接近;而XDPOP-RS-TSA-8(Pseudomonas sp.)、XB-RS-PIA-11(Halomonas sp.)则各形成一个相对独立的分支。XDPOP-RS-PIA-16和XDPOP-RS-PIA-1(Burkholderia sp.)属于一个亚群,从图 3中可看出该亚群与其它3个亚群的同源性较近,但是从图 4中可看出该亚群形成较独立分支,功能较为特殊。中下部分基因簇中包含9个菌株,由图 3所示,DW-RS-PIA-1、DF-RS-PIA-1、DF-RS-ACC-6、LW-RS-ACC-3、SYC-RS-TSA-4等5个无色杆菌(Achromobacter sp.)及XDPOP-RS-TSA-16(Pseudomonas sp.)同源性较高,为一个亚群,由图 4所示,XDPOP-RS-TSA-16(Pseudomonas sp.)在功能上形成一个独立的分支;另外,由图 3、图 4中可看出,SYC-RS-PIA-8和SYC-RS-PIA-9两个无色杆菌(Achromobacter sp.)与JP-R-ACC-12(Pseudo-monas sp.)为另外一个亚群,功能相近同源性较高。
3 讨论本研究对10种不同盐碱地指示性盐生植物以及杨树根际、根系内部进行分离培养和分子鉴定,共获得247株细菌,归类于4大纲10个目,多样性较丰富。并通过筛选引物、优化PCR反应条件等方法建立了一套从中稳定高效的快速筛选具ACC脱氨酶活性细菌的方法。在前人研究基础上,本实验选择了DegACCf/DegACCr、F1936f/F1938r以及acdSf3/acdSr4等3对编码细菌ACC脱氨酶的acdS基因片段的引物进行扩增[23-25]。最终只有acdSf3/acdSr4引物对可以成功扩增出acdS基因;虽然其余两对引物在一些报道中被认为行之有效[23-24],如基于宏基因组方法检测环境样品acdS基因的遗传多样性,但在本实验中对纯菌株的扩增效果反而不佳,说明在利用PCR技术扩增目标片段时,需要根据实际的实验情况来选择合适的引物。基于acdS基因编码的氨基酸序列进行系统发育分析,可将这些菌株进一步分为两大类群,某些菌株如XDPOP-RS-PIA-16(Burkholderia sp.)、XB-RS-PIA-11(Halomonas sp.)同源性最低,氨基酸结构的新颖性可能决定了在酶活和功能方面的特殊性,有待于今后进一步探索。
本研究通过PCR检测手段,发现ACC脱氨酶细菌在根际和根内生环境遗传多样性丰富,是一类优质植物促生抗逆微生物资源。在建立具ACC脱氨酶活性的菌株的资源库的基础上,今后的研究工作将围绕定量测试菌株ACC脱氨酶活性展开,旨在筛选出高酶活ACC脱氨酶产生菌,这对增强林木耐盐性和沿海造林工程等研究都具有重要的科学意义,对进一步开发促生、增强抗逆性的菌剂应用于林木高质量育苗也有良好的应用前景。另外,与耗时长、工作量大而效率低的传统ACC脱氨酶产生菌分离方法相比,本研究为快速筛选具ACC脱氨酶活性的细菌建立了PCR体系,对同类研究具有较重要的参考价值和指导意义。
4 结论本研究利用PCR技术,以编码ACC脱氨酶的acdS基因为标记,分别使用acdSf3/acdSr4、DegACCf/DegACCr和F1936f/F1938r三对引物对多种盐生植物和杨树根际、根内生细菌进行检测,实验结果表明使用acdSf3/acdSr4引物可有效扩增出细菌acdS基因。利用该PCR技术筛选具有ACC脱氨酶活性的菌株是高效便捷的方法。
致谢 感谢青岛农业大学辛华老师在样品采集过程中提供的帮助。| [1] | Hyodo H. Stress/wound ethylene[M]// Mattoo AK, Suttle JC. The Plant Hormone Ethylene. Boca Raton:CRC Press, 1991:65-80. |
| [2] | Hasegawa PM, Bressan RA, Zhu JK, et al. Plant cellular and molecular responses to high salinity[J]. Annual Review of Plant Biology, 2000, 51 (1): 463–499. DOI:10.1146/annurev.arplant.51.1.463 |
| [3] | Mahajan S, Tuteja N. Cold, salinity and drought stresses:an overview[J]. Archives of Biochemistry and Biophysics, 2005, 444 (2): 139–158. DOI:10.1016/j.abb.2005.10.018 |
| [4] | Morgan PW, Drew MC. Ethylene and plant responses to stress[J]. Physiologia Plantarum, 1997, 100 (3): 620–630. DOI:10.1111/ppl.1997.100.issue-3 |
| [5] | Glick BR. Modulation of plant ethylene levels by the bacterial enzyme ACC deaminase[J]. FEMS Microbiology Letters, 2005, 251 (1): 1–7. DOI:10.1016/j.femsle.2005.07.030 |
| [6] | Nadeem SM, Zahair ZA, Naveed M, et al. Rhizobacteria capable of producing ACC-deaminase may mitigate salt stress in wheat[J]. Soil Science Society of America Journal, 2010, 74 : 533–542. DOI:10.2136/sssaj2008.0240 |
| [7] | Deepti B, Nidhi B, Deepamala M, et al. ACC deaminase containing Arthrobacter protophormiae induces NaCl stress tolerance through reduced ACC oxidase activity and ethylene production resulting in improved nodulation and mycorrhization in Pisum sativum[J]. Journal of Plant Physiology, 2014, 171 : 884–894. DOI:10.1016/j.jplph.2014.03.007 |
| [8] | Singh RP, Shelke GM, Kumar A, et al. Biochemistry and genetics of ACC deaminase:a weapon to "stress ethylene" produced in plants[J]. Frontiers in Microbiology, 2015, 6 : 937. |
| [9] | Duan J, Müller KM, Charles TC, et al. 1-aminocyclopropane-1-carboxylate(ACC)deaminase genes in rhizobia from southern Saskatchewan[J]. Microbial Ecology, 2006, 57 (3): 423–436. |
| [10] | Glick BR, Cheng Z, Czarny J, et al. Promotion of plant growth by ACC deaminase-producing soil bacteria[J]. European Journal of Plant Pathology, 2007, 119 (3): 329–339. DOI:10.1007/s10658-007-9162-4 |
| [11] | Brotman Y, Landau U, Cuadros-Inostroza á, et al. Trichoderma-plant root colonization:escaping early plant defense responses and activation of the antioxidant machinery for saline stress tolerance[J]. PLoS Pathogens, 2013, 9 : e1003221. DOI:10.1371/journal.ppat.1003221 |
| [12] | Nascimento FX, Rossi MJ, Soares CR, et al. New insights into 1-aminocyclopropane-1-carboxylate(ACC)deaminase phylogeny, evolution and ecological significance[J]. PLoS One, 2014, 9 : e99168. DOI:10.1371/journal.pone.0099168 |
| [13] | Glick BR, Cheng Z, Czarny J, et al. Promotion of plant growth by ACC deaminase-producing soil bacteria[J]. European Journal of Plant Pathology, 2007, 119 (3): 329–339. DOI:10.1007/s10658-007-9162-4 |
| [14] | Glick BR, Penrose DM, Li J. A model for the lowering of plant ethylene concentrations by plant growth-promoting bacteria[J]. Journal of Theoretical Biology, 1998, 190 (1): 63–68. DOI:10.1006/jtbi.1997.0532 |
| [15] | Penrose DM, Glick BR. Methods for isolating and characterizing ACC deaminase-containing plant growth promoting rhizobacteria[J]. Physiologia Plantarum, 2003, 118 (1): 10–15. DOI:10.1034/j.1399-3054.2003.00086.x |
| [16] | 刘琛, 赵宇华, 傅庆林, 等. ACC脱氨酶活性菌株的筛选, 鉴定及其对茄子耐盐性的影响[J]. 浙江大学学报:农业与生命科学版, 2008, 34(2): 143–148. |
| [17] | 田磊, 姜云, 陈长卿, 等. 一株人参内生1-氨基环丙烷-1-1羧酸(ACC)脱氨酶活性细菌的筛选, 鉴定及其对宿主生长的影响[J]. 微生物学报, 2014, 54(7): 760–769. |
| [18] | 方芳, 刘佳莉, 史煦涵, 等. 石油污染土壤中植物根际促生菌的筛选及特性分析[J]. 生物技术通报, 2012, 6: 154–158. |
| [19] | 窦雅静, 康丽华, 陆俊锟, 等. 黑木相思根瘤菌ACC脱氨酶活性的研究[J]. 中南林业科技大学学报, 2014, 34(11): 77–83. DOI:10.3969/j.issn.1673-923X.2014.11.016 |
| [20] | 谢明杰, 程爱华. 我国微生物肥料的研究进展及发展趋势[J]. 微生物学杂志, 2010, 20(4): 42–45. |
| [21] | 姚军朋, 姚拓, 王小利. ACC脱氨酶的应用研究进展与评述[J]. 生物技术, 2010, 20(2): 87–91. |
| [22] | Jogaiah S, Abdelrahman M, Tran LS, et al. Characterization of rhizosphere fungi that mediate resistance in tomato against bacterial wilt disease[J]. Journal of Experimental Botany, 2013, 64 : 3829–3842. DOI:10.1093/jxb/ert212 |
| [23] | Blaha D, Prigent-Combaret C, Mirza MS, et al. Phylogeny of the 1-aminocyclopropane-1-carboxylic acid deaminase-encoding gene acdS in phytobeneficial and pathogenic Proteobacteria and relation with strain biogeography[J]. FEMS Microbiology Ecology, 2006, 56 (3): 455–470. DOI:10.1111/fem.2006.56.issue-3 |
| [24] | Nikolic B, Schwab H, Sessitsch A. Metagenomic analysis of the 1-aminocyclopropane-1-carboxylate deaminase gene(acdS)operon of an uncultured bacterial endophyte colonizing Solanum tuberosum L[J]. Archives of Microbiology, 2011, 193 (9): 665–676. DOI:10.1007/s00203-011-0703-z |
| [25] | Li Z, Chang S, Ye S, et al. Differentiation of 1-aminocyclopropane-1-carboxylate(ACC)deaminase from its homologs is the key for identifying bacteria containing ACC deaminase[J]. FEMS Microbiology Ecology, 2015, 91 (10): fiv112. DOI:10.1093/femsec/fiv112 |