2. 中国科学院大学,北京 100049;
3. 中国科学院遗传与发育生物学研究所农业资源研究中心 中国科学院农业水资源重点实验室,石家庄 050021
2. University of Chinese Academy of Sciences, Beijing 100049;
3. Key Laboratory of Agricultural Water Resources, Center for Agricultural Resources Research, Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Shijiazhuang 050021
病毒是目前已知的最简单的生命单位,通常是由DNA或RNA组成的基因组和外壳蛋白(衣壳)两部分构成。病毒在自然界分布广泛,是地球上最丰富的生物体[1-2]。噬菌体是侵染细菌的病毒统称,细菌是地球上最多的活生物体,仅在海洋中细菌数量就高达1.2×1029[3]。噬菌体数量远大于细菌数量,保守估计海洋中的噬菌体总数高达1031[4-6]。在海洋环境中噬菌体每秒与寄主发生高达1023次感染几率,以维持噬菌体在环境中的数量[7]。
通常学术界认为噬菌体的研究史可以划分为3个时期:细菌病毒最早于1915年和1917年被Twort和D’Herelle发现,当时科学家期望利用噬菌体进行医疗方面的开发研究,主要应用于治疗细菌感染。然而到20世纪40年代随着抗生素的发现,噬菌体便迅速退出了治疗细菌感染的领域,这一目标逐渐被放弃,这被认为是噬菌体研究的第一个时期。需要说明的是,近年来伴随着细菌抗耐药性的增强和超级细菌的出现,噬菌体疗法的研究又开始复苏。噬菌体研究的第二个时期是从20世纪70年代开始的,在这一时期,科学家发现噬菌体是现代分子生物学研究的一个很好的工具,对推动分子生物学发展起到重要的作用,故此有“高等生物的分子生物学并不是站在巨人的肩膀上,而是站在矮小的噬菌体上”这样的说法[8]。噬菌体研究的第三个阶段是从21世纪初开始的,科学家(主要是海洋生物学家)发现噬菌体通过感染寄主,导致细胞裂解,细胞中营养元素释放到环境中,间接地促进了环境中有机和无机营养元素的循环,是元素生物地球化学循环的重要驱动者之一;噬菌体能侵染多个宿主,是物种间基因水平转移的重要媒介,它直接影响到微生物群落结构和物种进化,是决定微生物多样性的关键影响因子[9];同时也发现噬菌体基因结构组成非常复杂,大量未知的遗传基因被认为是基因库中的“暗物质”,对这些暗物质基因的研究,有助于揭开生命的起源、演替和更迭规律,有助于确定噬菌体在生态系统中的地位和作用,从这一时期开始,科学家们开启了研究病毒生态学的热潮[10]。
尽管噬菌体广泛存在于各种生境中,但学术界对其数量和遗传基因多样性的研究还存在许多瓶颈问题需要克服。首先是人们对自然界中噬菌体认识的传统的方法主要是依靠寄主细胞诱捕和电子显微镜观察。然而自然环境中决大部分噬菌体的宿主目前尚处于不可培养状态[11],从而限制了噬菌体的获取,其次是目前尚缺乏通用的标记基因用于病毒或者噬菌体基因多样性研究,即没有发现诸如像原核生物16S rDNA和真核生物18S rDNA中存在共有的保守序列,用于基因多样性研究[12]。但近年来,随着噬菌体基因组学的发展,发现噬菌体某些家族的结构或功能蛋白氨基酸片段序列高度保守,利用这些保守片段氨基酸序列设计引物,可以直接采用PCR扩增技术,从环境中获得噬菌体特定家族基因序列,用于噬菌体遗传多样性分析[13]。这些基因包括编码噬蓝藻体壳组装蛋白的g20基因[14-16]、编码T4型噬菌体主要壳蛋白的g23基因[17-18]、T7型噬菌体的DNA合成酶pol基因[19-20]、编码蓝藻噬菌体光系统Ⅱ(PSⅡ)反应中心蛋白D1和D2蛋白的psbA和psbD基因[21-22],磷酸盐调节基因phoH基因[23-26]等。前文对自然环境中编码g23基因和g20基因多样性研究进展已进行了综述[27-28],本文以最新报道的phoH基因为目标,综述近年来海洋和稻田环境中噬菌体该基因多样性的研究进展,目的向研究者推荐一种用于噬菌体生态学研究新的标记基因,从而促进该基因在噬菌体基因多样性研究中的应用,促进病毒生态学研究进展。
1 噬菌体辅助代谢基因——phoH基因噬菌体辅助代谢基因(Auxiliary metabolic genes,AMGs)是指与其寄主具有同源关系的基因。噬菌体AMG是从寄主那里获得的,但其基因序列又与寄主的代谢基因有所区别。通常我们认为噬菌体的生存完全依赖于寄主细胞的生命活动,不会携带冗余的代谢基因,但最近科学家针对部分海洋噬菌体全基因组测序和海洋病毒宏基因组学分析发现,一些噬菌体携带与寄主同源的代谢基因,这些基因包括蓝藻噬菌体参与光合作用的基因psbA和psbD[29],噬菌体焦磷核苷酸水解酶基因mazG[30]及磷酸盐调节基因phoH[23-26]等。蓝藻噬菌体携带辅助代谢基因psbA和psbD的功能被科学家解读为在寄主细胞光合系统受到光损失时,噬菌体启动自身携带的光合基因以弥补修复寄主的光合作用系统,从而使噬菌体更好地繁殖[21, 31-32]。但关于噬菌体phoH基因是否和怎样参与寄主的磷营养吸收还没有明确的研究报道。尽管对噬菌体phoH基因的代谢功能尚不明确,但研究者发现该基因可以作为研究噬菌体基因多样性一个好的分子标记基因。
Goldsmith等[25]通过使用来自大肠杆菌(Escherichia coli)菌株K-12亚型MG1655,原绿球藻(Prochlorococcus)菌株NATL1A和NATL2A的基因组的磷酸盐调节基因与GenBank数据库中的病毒子集进行BLAST相似性检索。在检测的35个磷酸盐调节基因中[33],只有5个基因(phoH,pstS,phoA,phoE和ugpQ)在噬菌体基因组中被发现。比对结果表明,在GenBank数据库中完全测序的602个噬菌体基因组中有42个携带phoH基因,而携带pstS、phoA、phoE和ugpQ基因噬菌体的基因分布只出现在9、2、1和3个噬菌体基因组中。为此,Goldsmith等认为phoH基因可以作为一种新的分子标记基因用于研究海洋噬菌体多样性。与目前普遍使用的研究噬菌体多样性其他分子标记基因,如g23、g20、DNA pol和psbA等不同,phoH基因不局限于某一特定形态的病毒家族[34],同时该基因在一些侵染自养和异养细菌的噬菌体,甚至在感染自养真核微生物病毒中都有可能存在,故此该基因的使用范围更宽泛。
基于此,美国南佛罗里达大学的Goldsmith等[25]通过比较聚球藻(Synechococcus)噬菌体S-PM2,原绿球藻噬菌体P-SSM2和P-SSM4,以及弧菌(Vibrio)噬菌体KVP40的phoH基因的全长序列,设计出了简并性引物vPhoHf(5’-TGC RGG WAC AGG TAA RAC AT-3’)和vPhoHr(5’-TCR CCR CAG AAA AYM ATT TT-3’),他们首次采用该引物PCR扩增海水病毒DNA样品,评估了海水中噬菌体phoH基因的多样性。
2 海洋环境中噬菌体phoH基因多样性对噬菌体phoH基因多样性的研究主要集中在海洋水体环境。Goldsmith等[25]采用其设计的简并性引物vPhoHf和vPhoHr率先对大西洋马尾藻海深度分别为0米、200米、500米和1 000米海水中病毒的DNA进行PCR扩增,通过氨基酸比对构建的系统发育树表明,来自该海域海水噬菌体phoH序列形成5个类群(Group 1-5)。其中,Group 1和Group 5分布广,在所有深度剖面的样品中均检测得到;Group 2含有大部分的可培养的蓝藻噬菌体的phoH序列和环境样品克隆,但在深度500 m的样品中未检出;Group 3中的克隆序列在光照区域较为丰富,并随着深度增加而减小,在深度1 000米的样品中未检出,Group 3进一步被划分为3个亚群,分别为Group 3a、3b和3c;Group 4包含的克隆均来自于1 000 m深度的样品中。在5个类群中,只有Group 2含有可培养的蓝藻噬菌体phoH序列,其他4个类群全部由未知的环境噬菌体克隆组成。该研究表明,在马尾藻海域环境中存在着独特的噬菌体类群,且这些噬菌体中phoH序列分布随采样深度不同存在显著差异。
为了探究海洋中噬菌体phoH基因分布是否存在地域性,Goldsmith等[25]进一步对6个海域(墨西哥湾、北冰洋、不列颠哥伦比亚沿海水域、地中海、马尾藻海、挪威海岸水域)0-100米深度水体病毒浓缩物的DNA样品进行PCR扩增。构建的系统发育树表明,在前面发现的5个噬菌体类群基础上,还发现1个新的噬菌体phoH类群(Group 6)。其中,Group 1中80%以上的序列来自挪威海岸水域(Raunefjorden);而Group 2仅在马尾藻海和墨西哥湾这两个研究地点被发现;Group 3主要存在于采自不列颠哥伦比亚海湾的样品中;Group 4包含的克隆主要来自北冰洋王湾(Kongsfjorden);Group 5中的噬菌体也主要存在于马尾藻海的海水中;而Group 6则主要来自Raunefjorden和墨西哥湾的海水样品中。在6个类群中,除Group 2含有分离得到的蓝藻噬菌体外,其他5个类群组成均为环境克隆。该结果进一步表明,目前学术界获得的可培养噬菌体只能表征海洋噬菌体多样性的极小部分,海洋中存在大量的未知的噬菌体phoH基因资源。
在上述发现基础上,2015年Goldsmith等[35]对大西洋马尾藻百慕大海域研究站夏季(9月)和冬季(3月)采集的海水样品进行3年(2008、2010和2011年)的追踪研究。他们采用454高通量测序技术共获得了3 600个phoH基因操作分类单元(OTU),发现夏季和冬季噬菌体phoH基因分布显著不同,且噬菌体季节性差异明显大于年际间的差异。虽然采用高通量测序技术捕获到非常丰富的phoH基因序列,但大多数phoH序列属于几个主要的OTU,其中OTU1-OTU5平均占所有序列的52.4%,相对丰度最高的前18个OUT占所测得序列的82%。同样,从该海域获得的phoH基因划分到Group 1-Group 5中,没有发现新的类群,这与他们团队2011年报道的结果相似。不同年份研究获得相似的结果充分表明利用phoH基因表征的噬菌体基因多样性具有较强的可靠性。
3 稻田中噬菌体phoH基因多样性对GenBank上噬菌体基因组信息分析发现,有大约40%来自海洋环境的噬菌体携带phoH基因,与此相对应只有4%来自非海洋环境噬菌体携带phoH基因[25]。这一发现似乎限制了phoH基因在陆地生态系统噬菌体多样性研究中的应用。但需要指出的是,GenBank中噬菌体全基因组信息来源于可培养噬菌体,由于环境中可培养的细菌只占很小部分,这无疑限制了噬菌体的获得,进而导致自然环境中许多可能携带phoH基因的病毒不能被发现。另外,如果磷营养缺乏是诱发噬菌体携带辅助代谢phoH基因的前提条件,那么与海洋水体环境相比,非海洋的陆地环境可能不缺磷,由此提出是否非海洋生态系统噬菌体也携带phoH基因。
为了解开这一谜团,2016年Wang等[36]首次采用引物vPhoHf/vPhoHr对采集到我国东北4个稻田(大安、牡丹江、阎家岗和绥化)水体样品DNA进行PCR扩增。然后将PCR扩增产物采用胶纯化、克隆和Sanger测序方法,分析研究phoH基因多样性。从4个稻田水体中获得了424个噬菌体phoH基因序列,其中约90%的序列分布在含有自养细菌病毒分离株的phoH基因群中,据此判断这些序列可能来源于蓝藻病毒;其余10%的序列则分布在含有异养细菌病毒分离株的phoH基因群中,推断其来源于异养细菌病毒。将获得的phoH基因序列在NCBI数据库中进行BLASTp比对后发现,大约70%的基因序列与登录的序列的最高相似度均小于70%,表明稻田生态系统中存在大量未知的噬菌体phoH基因。该研究首次揭示出稻田生态系统中噬菌体也携带phoH基因,表明此基因也可以作为研究海洋以外其他陆地环境中噬菌体多样性的有效标记基因。
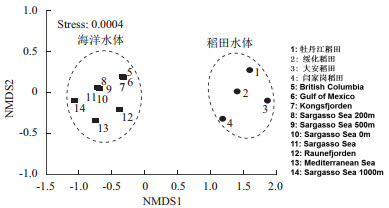
将来自稻田水体和海洋水体中噬菌体phoH基因一起构建系统进化树发现,在原有的6个海洋水体噬菌体phoH基因类群(Group 1-6) 的基础上[25],又将从稻田水体获得的大部分phoH基因序列建立了4个独特的类群和7个新的亚群,其中Groups 2、3、4、6为海洋水体和我国东北稻田水体共有类群;Group 1和Group 5在本次我国东北稻田水体中未检出,而类群Group α、Group β、Group γ和Group δ和亚群Group 2b、Group 3d、Group 3e、Group 6a、Group 6b、Group 6c和Group 6d为我国东北稻田特有类群,来自海洋环境中的克隆未被划分到这些类群中。研究还发现,不同稻田水体的噬菌体phoH基因序列分布到不同类群中的比例存在差异,说明稻田噬菌体群落结构组成具有明显的地理分布特征。采用非度量多维尺度分析(NMDS)方法对采自世界不同海洋水体和中国稻田水体中的噬菌体phoH基因集群分析发现,稻田与海洋环境中的噬菌体phoH基因群集差异非常明显,不同环境单独聚在一起(图 1)。该发现证明以phoH基因表征的噬菌体群落结构组成在稻田和海洋环境中完全不同,该结果进一步印证前期我们针对噬菌体g23和g20的研究发现[27, 37]。
4 沙漠环境中phoH基因多样性最近噬菌体在沙漠中的分布情况也得到了研究者的关注。2015年Adriaenssens等[38]调查纳米比亚沙漠石下生物宏病毒组中发现存在辅助代谢基因phoH。通过MetaVir鉴定出18条完整的phoH序列和23条部分phoH序列,构建的PhyML系统发生树显示大部分沙漠石下病毒phoH氨基酸序列与来自NCBI数据库中的已知噬菌体的phoH基因关系远,且海洋和沙漠石下病毒phoH基因分布在完全不同的进化枝上,该研究说明在陆地生态环境中存在独特的病毒或噬菌体的phoH基因序列。
5 展望噬菌体的发现己经有100多年的历史[39],一直受到研究者的高度关注。在过去数年里,一些用于研究环境噬菌体基因多样性的引物被设计出来,推动了噬菌体基因多样性的研究工作[18, 21, 40-41]。本文介绍的phoH基因是一种新的噬菌体分子标记基因,但到目前为止,针对该基因研究仅限于海洋[25]、稻田生态系统及沙漠生物中[36],是否在其他生态环境中适用尚未见报道。现有的研究结果表明,只有Group 2类群包含可分离培养的蓝藻噬菌体phoH基因序列,其他类群都不含有可培养的噬菌体,因此定性这些扩增得到的未知噬菌体phoH基因来源还有待明晰。另外,phoH基因是磷酸盐调节基因,其表达与磷酸盐胁迫相关,但为什么噬菌体也携带该基因;该基因在噬菌体中的作用是什么;不同磷营养水平是否能够影响噬菌体该基因的丰度和多样性,以及多大范围内噬菌体携带该基因等问题还不明确。对上述问题的研究将会在一定程度上丰富和推动环境病毒生态学研究的进展。
| [1] | Fuhrman JA. Marine viruses and their biogeochemical and ecological effects[J]. Nature, 1999, 399 (6736): 541. DOI:10.1038/21119 |
| [2] | Sabehi G, Shaulov L, Silver DH, et al. A novel lineage of myoviruses infecting cyanobacteria is widespread in the oceans[J]. Proc Natl Academy of Sciences, 2012, 109 (6): 2037–2042. DOI:10.1073/pnas.1115467109 |
| [3] | Whitman WB, Coleman DC, Wiebe WJ. Prokaryotes:the unseen majority[J]. Proc Natl Academy of Sciences, 1998, 95 (12): 6578–6583. DOI:10.1073/pnas.95.12.6578 |
| [4] | Breitbart M, Rohwer F. Here a virus, there a virus, everywhere the same virus?[J]. Trends in Microbiology, 2005, 13 (6): 278–284. DOI:10.1016/j.tim.2005.04.003 |
| [5] | Suttle CA. Viruses in the sea[J]. Nature, 2005, 437 (7057): 356. DOI:10.1038/nature04160 |
| [6] | Weinbauer MG. Ecology of prokaryotic viruses[J]. FEMS MicroBiol Rev, 2004, 28 (2): 127–181. DOI:10.1016/j.femsre.2003.08.001 |
| [7] | Suttle CA. Marine viruses-major players in the global ecosystem[J]. Nature Reviews. Microbiology, 2007, 5 (10): 801. DOI:10.1038/nrmicro1750 |
| [8] | Brüssow H. Bacteriophage-host interaction:from splendid isolation into a messy reality[J]. Current Opinion in Microbiology, 2013, 16 (4): 500–506. DOI:10.1016/j.mib.2013.04.007 |
| [9] | Hambly E, Suttle CA. The viriosphere, diversity, and genetic exchange within phage communities[J]. Current Opinion in Microbiology, 2005, 8 (4): 444–450. DOI:10.1016/j.mib.2005.06.005 |
| [10] | Mann NH. The third age of phage[J]. PLoS Biology, 2005, 3 (5): e182. DOI:10.1371/journal.pbio.0030182 |
| [11] | Ward DM, Weller R, Bateson MM. 16S rRNA sequences reveal numerous uncultured microorganisms in a natural community[J]. Nature, 1990, 345 (6270): 63–65. DOI:10.1038/345063a0 |
| [12] | Edwards RA, Rohwer F. Opinion:viral metagenomics[J]. Nature Reviews. Microbiology, 2005, 3 (6): 504. DOI:10.1038/nrmicro1163 |
| [13] | Rohwer F, Edwards R. The Phage Proteomic Tree:a genome-based taxonomy for phage[J]. Journal of Bacteriology, 2002, 184 (16): 4529–4535. DOI:10.1128/JB.184.16.4529-4535.2002 |
| [14] | Fuller NJ, Wilson WH, Joint IR, et al. Occurrence of a sequence in marine cyanophages similar to that of T4 g20 and its application to PCR-based detection and quantification techniques[J]. Appl Environ Microbiol, 1998, 64 (6): 2051–2060. |
| [15] | Jameson E, Mann NH, Joint I, et al. The diversity of cyanomyovirus populations along a North-South Atlantic Ocean transect[J]. The ISME Journal, 2011, 5 (11): 1713. DOI:10.1038/ismej.2011.54 |
| [16] | Sullivan MB, Coleman ML, Quinlivan V, et al. Portal protein diversity and phage ecology[J]. Environ Microbiol, 2008, 10 (10): 2810–2823. DOI:10.1111/j.1462-2920.2008.01702.x |
| [17] | Filée J, Tétart F, Suttle CA, et al. Marine T4-type bacteriophages, a ubiquitous component of the dark matter of the biosphere[J]. Proc Natl Acad Sci USA, 2005, 102 (35): 12471–12476. DOI:10.1073/pnas.0503404102 |
| [18] | Jia Z, Ishihara R, Nakajima Y, et al. Molecular characterization of T4-type bacteriophages in a rice field[J]. Environ Microbiol, 2007, 9 (4): 1091–1096. DOI:10.1111/emi.2007.9.issue-4 |
| [19] | Breitbart M, Miyake JH, Rohwer F. Global distribution of nearly identical phage-encoded DNA sequences[J]. FEMS Microbiology Letters, 2004, 236 (2): 249–256. DOI:10.1111/fml.2004.236.issue-2 |
| [20] | Chen F, Wang K, Huang S, et al. Diverse and dynamic populations of cyanobacterial podoviruses in the Chesapeake Bay unveiled through DNA polymerase gene sequences[J]. Environ Microbiol, 2009, 11 (11): 2884–2892. DOI:10.1111/emi.2009.11.issue-11 |
| [21] | Mann NH, Cook A, Millard A, et al. Marine ecosystems:bacterial photosynthesis genes in a virus[J]. Nature, 2003, 424 (6950): 741–741. |
| [22] | Sullivan MB, Lindell D, Lee JA, et al. Prevalence and evolution of core photosystem Ⅱ genes in marine cyanobacterial viruses and their hosts[J]. PLoS Biology, 2006, 4 (8): e234. DOI:10.1371/journal.pbio.0040234 |
| [23] | Williamson SJ, Rusch DB, Yooseph S, et al. The Sorcerer Ⅱ Global Ocean Sampling Expedition:metagenomic characterization of viruses within aquatic microbial samples[J]. PLoS One, 2008, 3 (1): e1456. DOI:10.1371/journal.pone.0001456 |
| [24] | Thompson L R, Zeng Q, Kelly L, et al. Phage auxiliary metabolic genes and the redirection of cyanobacterial host carbon metabolism[J]. Proc Natl Academy of Sciences, 2011, 108 (39): E757–E764. DOI:10.1073/pnas.1102164108 |
| [25] | Goldsmith DB, Crosti G, Dwivedi B, et al. Development of phoH as a novel signature gene for assessing marine phage diversity[J]. Appl Environ Microbiol, 2011, 77 (21): 7730–7739. DOI:10.1128/AEM.05531-11 |
| [26] | Dwivedi B, Xue BJ, Lundin D, et al. A bioinformatic analysis of ribonucleotide reductase genes in phage genomes and metagenomes[J]. BMC Evolutionary Biology, 2013, 13 : 33. DOI:10.1186/1471-2148-13-33 |
| [27] | 王光华, 刘俊杰. 自然环境中T4型噬菌体g23基因多样性研究进展[J]. 微生物学报, 2011, 51(6): 732–739. |
| [28] | 荆瑞勇, 王光华. 不同自然环境下噬藻体g20基因多样性研究进展[J]. 微生物学报, 2013, 53(11): 1149–1157. |
| [29] | Millard A, Clokie MRJ, Shub DA, et al. Genetic organization of the psbAD region in phages infecting marine Synechococcus strains[J]. Proc Natl Acad Sci USA, 2004, 101 (30): 11007–11012. DOI:10.1073/pnas.0401478101 |
| [30] | Bryan MJ, Burroughs NJ, Spence EM, et al. Evidence for the intense exchange of MazG in marine cyanophages by horizontal gene transfer[J]. PLoS One, 2008, 3 (4): e2048. DOI:10.1371/journal.pone.0002048 |
| [31] | Lindell D, Jaffe JD, Johnson ZI, et al. Photosynthesis genes in marine viruses yield proteins during host infection[J]. Nature, 2005, 438 (7064): 86. DOI:10.1038/nature04111 |
| [32] | Lindell D, Sullivan MB, Johnson ZI, et al. Transfer of photosynthesis genes to and from Prochlorococcus viruses[J]. Proc Natl Acad Sci USA, 2004, 101 (30): 11013–11018. DOI:10.1073/pnas.0401526101 |
| [33] | Hsieh YJ, Wanner BL. Global regulation by the seven-component Pi signaling system[J]. Current Opinion in Microbiology, 2010, 13 (2): 198–203. DOI:10.1016/j.mib.2010.01.014 |
| [34] | Adriaenssens EM, Cowan DA. Using signature genes as tools to assess environmental viral ecology and diversity[J]. Appl Environ Microbiol, 2014, 80 (15): 4470–4480. DOI:10.1128/AEM.00878-14 |
| [35] | Goldsmith DB, Parsons RJ, Beyene D, et al. Deep sequencing of the viral phoH gene reveals temporal variation, depth-specific composition, and persistent dominance of the same viral phoH genes in the Sargasso Sea[J]. Peer J, 2015, 3 : e997. DOI:10.7717/peerj.997 |
| [36] | Wang X, Liu J, Yu Z, et al. Novel groups and unique distribution of phage phoH genes in paddy waters in northeast China[J]. Scientific Reports, 2016, 6 : 38428. DOI:10.1038/srep38428 |
| [37] | Jing R, Liu J, Yu Z, et al. Phylogenetic distribution of the capsid assembly protein gene (g20) of cyanophages in paddy floodwaters in Northeast China[J]. PLoS One, 2014, 9 (2): e88634. DOI:10.1371/journal.pone.0088634 |
| [38] | Adriaenssens EM, Van Zyl L, De Maayer P, et al. Metagenomic analysis of the viral community in Namib Desert hypoliths[J]. Environ Microbiol, 2015, 17 (2): 480–495. DOI:10.1111/1462-2920.12528 |
| [39] | Twort FW. An investigation on the nature of ultramicroscopic viruses[J]. The Lancet, 1915, 186 (4814): 1241–1243. DOI:10.1016/S0140-6736(01)20383-3 |
| [40] | Zhong Y, Chen F, Wilhelm SW, et al. Phylogenetic diversity of marine cyanophage isolates and natural virus communities as revealed by sequences of viral capsid assembly protein gene g20[J]. Appl Environ Microbiol, 2002, 68 (4): 1576–1584. DOI:10.1128/AEM.68.4.1576-1584.2002 |
| [41] | Zeidner G, Preston CM, Delong EF, et al. Molecular diversity among marine picophytoplankton as revealed by psbA analyses[J]. Environ Microbiol, 2003, 5 (3): 212–216. DOI:10.1046/j.1462-2920.2003.00403.x |