城市化建设过程中,人类活动会破坏湿地表层土壤结构,造成湿地土壤有机质含量等理化性质发生改变;另一方面,生产生活污废水未经处理排入湿地,带来湿地有机质含量增加[1]。城市湿地有机质的改变对湿地的结构和功能产生重要影响[2]。微生物是湿地生态系统中不可或缺的成员,这些微生物存在于湿地的土壤和植物的根系表面,通过参与营养物的循环和改善填料氧化还原条件,以实现处理污水的功能[3]。研究表明人工湿地中的微生物丰度及其与污染物去除有一定相关性[4-7],湿地微生物丰度与TN去除率显著正相关[8]。反硝化及厌氧氨氧化是最终去除污水中TN的途径,有机质及不同形态的氮是反硝化过程和厌氧氨氧化过程的电子供体或受体[9-10]。有机质缺乏往往是反硝化微生物脱氮的限制性步骤[11],研究表明高浓度有机质有利于反硝化微生物的生长[12]。国内外学者对湿地微生物的研究多集中于人工湿地微生物功能的研究,对城市湿地微生物基因丰度研究的报道相对较少[13],因此了解有机质浓度对城市湿地微生物基因丰度的影响,将有助于对城市湿地的深入研究。
城市湿地有机质的研究多分为两个方面:沉积物有机质和水体中溶解性有机质(DOM)。土壤/沉积物中的有机质和水中的有机质是湿地中有机质的联合体,二者相互转化,共同作用于湿地环境,影响湿地生态系统结构及功能。因此,有必要将土壤有机质和水体有机质共同考虑。湿地中土壤和水体界面是氮循环的重要场所,氮循环也受到各种理化环境的影响,因此通过探究表层土壤中氮循环相关功能基因对有机质的响应及分布特征,可以帮助我们了解氮元素在湿地中迁移转化的微生物学机制。
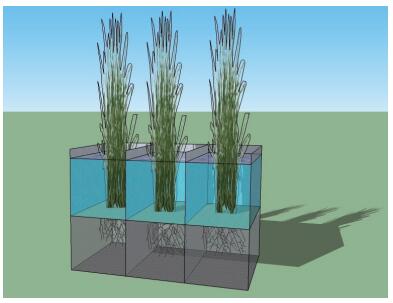
1 材料与方法 1.1 材料实验设计构建了小型模拟系统— —PVC板制成的顶部开口的水箱(长×宽×高=400 mm×400 mm×600 mm)。水箱底部填25 cm厚的湿地土壤,加25 cm深的上覆水,每个系统中种植10株挺水植物芦苇(Phragmites australis)。装置图见图 1。
|
| 图 1 模拟湿地装置示意图 |
实验设置6个处理组,每组3个平行,各实验处理组控制条件,见表 1。
A和B两种土壤,均采自上海市江湾湿地,土壤A是湿地内部河道的水岸交错带土壤,土壤B是在周边开发建设过程中受到破坏(表层土壤有被铲车清表)的湿地土壤。土壤A中总磷(TP)、总氮(TN)、有机质(OM)初始浓度分别为4.17±0.05、1.19±0.01、24.3±0.02 g/kg干重,土壤B中总磷(TP)、总氮(TN)、有机质(OM)初始浓度分别为1.13±0.02、0.57±0.01、13.7±0.04 g/kg干重。
系统中的上覆水取自流经同济大学校园景观河道,上覆水中TP、可溶性活性磷(SRP)、TN、氨氮(NH4+-N)、硝氮(NO3--N)、亚硝氮(NO2--N)、溶解性有机碳(DOC)初始浓度分别为0.055±0.001、0.035±0.001、1.11±0.11、0.54±0.01、0.36±0.06、0.069±0.001、7.09±0.51 mg/L,即为低浓度处理组。高浓度处理组即在采集的景观河道水中添加氯化铵、磷酸二氢钾及制备的樟树树叶浸出液控制上覆水中TN、TP、DOC浓度为10 mg/L、0.5 mg/L和30 mg/L左右。
芦苇采自江湾湿地,预培养4周后,选取大小一致的健壮植株种植在各实验装置中,每个实验装置种植10棵(密度62.5株/m2)。
1.2 方法 1.2.1 样品采集及保存实验末期上覆水放空后,按照梅花布点法采集5个点位的0-5 cm表层土壤,剔除植物根系后将每个装置的样品混合均匀后放于密封袋中,置于-25℃下冷冻保存待测。
1.2.2 土壤有机质的分析使用光度法[14]测定土壤有机质含量,分析仪器为UV-2800紫外分光光度计。
1.2.3 微生物的提取及分析采用OMEGA泥样基因组提取试剂盒(D5625-01 Soil DNA Kit)进行基因组DNA提取与纯化。通过1%琼脂凝胶电泳检测提取基因组DNA,并储存置于-25℃下冷冻保存。
后续引物设计及实时荧光定量PCR的工作交由上海美吉生物医药科技有限公司完成。本次检测所使用的目的基因引物信息见表 2,并稀释到10 μmol/L的浓度。对细菌的16S rRNA基因、古菌的16S rRNA基因、厌氧氨氧化菌的16S rRNA基因,和以下功能基因的靶片段氨单氧化酶基因(amoA)、亚硝酸盐氧化还原酶基因(nxrA)、与膜结合的硝酸还原酶基因(narG)、含铜亚硝酸盐还原酶基因(nirK)、含细胞色素cd1亚硝酸盐还原酶基因(nirS)、一氧化二氮还原酶基因(nosZ)进行定量分析[15]。
1.3 数据分析数据处理采用Excel 2013,数据取3组平行数据的平均值。各数据图的绘制采用Origin Pro 8.0,数据协方差分析采用SPSS 19.0,运用单因素方差分析,选取不同处理组为因素,考察6个实验处理组植物各项指标在0.05水平上是否存在显著性差异。
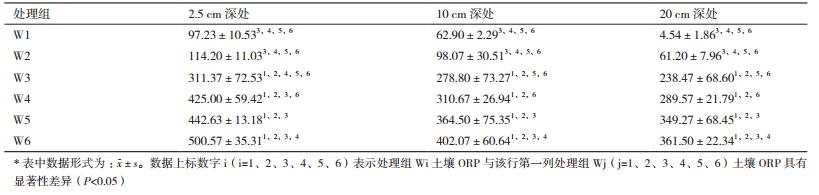
2 结果 2.1 湿地土壤理化参数表 3为实验末期不同实验组土壤ORP随深度变化的数据,各处理组土壤ORP值都随着深度增加而降低。土壤A(W1、W2) ORP显著低于土壤B(W3-W6)(P < 0.05)。W1、W2两个处理组在垂向上的差异则没有显著性(P > 0.05),W3-W6四个处理组土壤ORP垂向上的差异具有显著性(P < 0.05)。
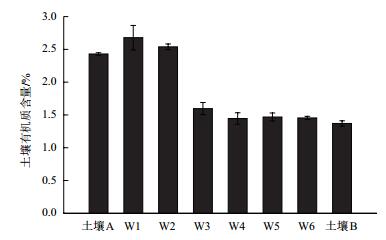
图 2是各处理组土壤有机质的含量,由图 2可知实验末期各处理组土壤表层有机质含量相较于原土壤都有所升高。土壤A处理组有机质含量显著高于土壤B处理组(P < 0.05);添加外源DOM的W1和W3两个处理组土壤表层有机质含量有显著的升高(P < 0.05)。
|
| 图 2 各处理组土壤表层有机质含量的变化 |
图 3是各处理组表层土壤中细菌和古菌16S rRNA基因的丰度。土壤A处理组(W1、W2) 细菌丰度显著高于土壤B处理组(W5、W6)(P < 0.05),添加外源氮、磷的W3、W4与W5、W6并未发现显著性的变化(P > 0.05),外源DOM的添加使得W1、W3、W5三个处理组细菌丰度要分别高于W2、W4、W6,上覆水中有机质的增加提高了表层土壤中供微生物生长的碳源。古菌的丰度要低于细菌,但同细菌类似,土壤A处理组中丰度更高。添加外源DOM的W3古菌丰度显著高于W4(P < 0.05)。
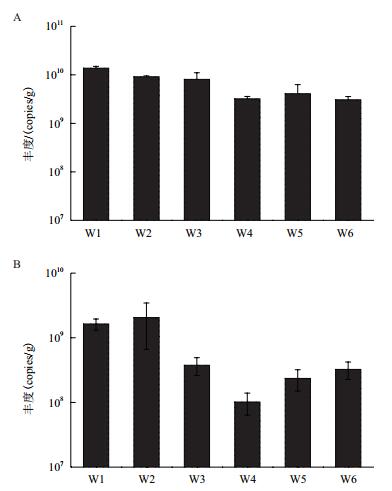
|
| 图 3 各处理组表层土壤细菌(A)、古菌(B)基因丰度 |
图 4所示为各处理组表层土壤中参与硝化过程的nxrA基因的丰度。nxrA基因是将NO2-N好氧转化为NO3--N的标志物[22]。W1、W2两个处理组基因丰度要显著高于其他4个处理组(P < 0.05),W1与W2之间以及W3、W4、W5、W6相互间没有显著性差异(P > 0.05),nxrA基因和反硝化相关的几个基因相比,丰度要低很多。amoA基因由于引物设计出现问题,在扩增过程中一直得不到稳定的结果。
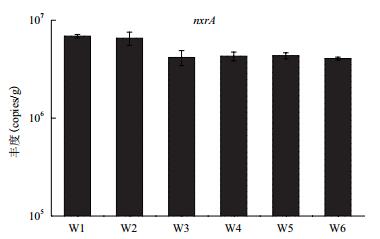
|
| 图 4 各系统表层土壤硝化过程相关基因绝对丰度 |
图 5是参与反硝化过程的4个重要基因narG、nirK、nirS和nosZ的丰度,对比各反硝化相关基因的丰度与细菌的丰度发现,反硝化细菌在总的细菌群落里所占的比例并不高,与Henry[23]的结果一致。
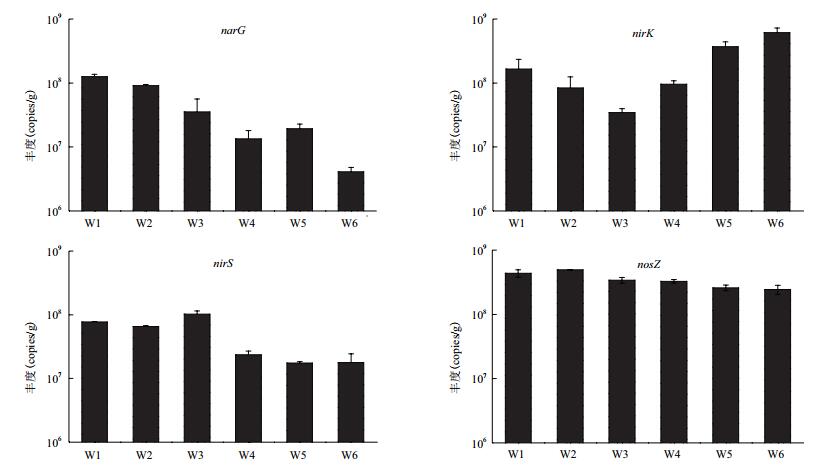
|
| 图 5 各处理组表层土壤反硝化过程相关基因丰度 |
narG基因是将NO3--N厌氧转化为NO2--N的标志物[24]。W1、W2中该细菌丰度要显著高于其他处理组(P < 0.05)。W1与W2之间也存在显著性差异(P < 0.05)。外源DOM的添加使得W1、W3、W5三个处理组narG基因丰度要分别高于W2、W4、W6。
nirK和nirS是亚硝酸还原酶的功能基因,参与NO2--N向N2O、NO的转化过程[25]。nirK基因的丰度要普遍高于nirS基因,这与García-Lledó[25]研究结果一致。两个细菌虽然在反硝化过程中作用一致,但这两个基因在湿地中的丰度却表现出不同的规律。首先,同一处理组中,nirK基因的丰度要普遍高于nirS基因;其次,这两种基因在各处理组之间的差异性也明显不同。nirK基因W1-W4系统中基因丰度要显著低于W5、W6(P < 0.05)。nirS基因的结果正相反,W5、W6系统中nirS基因丰度要低。
nosZ是编码N2O还原酶的基因,作用是完成N的彻底反硝化[26]。N2O还原为N2是反硝化作用的最后一步反应,受制于前反应,该基因在W1-W6处理组表层土壤中的丰度和narG基因、nirK基因一致。
2.2.4 厌氧氨氧化细菌基因丰度图 6是各处理组表层土壤厌氧氨氧化细菌的丰度,W1-W6表层土壤anammox丰度较高,实验中土壤一直处于淹水的状态,造成的厌氧的环境有利于anammox的增殖。W1、W2、W3三个处理组该细菌丰度要显著高于其他几个处理组(P < 0.05),与表层间隙水ORP结果相一致。
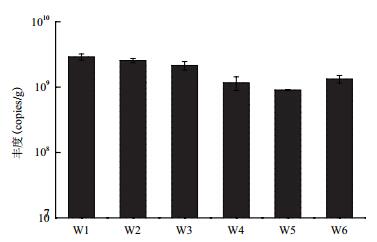
|
| 图 6 各处理组表层土壤厌氧氨氧化细菌丰度 |
上覆水中的外源DOM会通过直接沉降或被生物体吸收利用后再经过一系列的地球化学过程将其转移到土壤中[27],增加湿地表层土壤有机质含量[28]。本研究中,外源DOM添加条件下表层土壤有机质含量的增幅较小。外源溶解性的营养物质对土壤有机质的贡献需要通过生物的吸收、转化及死亡后的沉降等是一个长期的过程[27],表层土壤有机质主要来自湿地植物凋落物的生物降解[28-29]。本研究实验周期为5月21日到9月4日,是芦苇的生长期,凋落物较少,因此DOM增幅较小。基于以上,湿地表层土壤一旦被破坏,通过自然过程恢复其有机质含量很难,需要漫长的时间。
湿地表层土壤被破坏后可生物利用的有机质含量下降,造成微生物数量锐减[13, 30]。而可生物利用有机质的增加不仅增加微生物的多样性,还可增加微生物的基因丰度[31],湿地表层土壤中充足碳源也有助于古菌的生长[32]。本研究上覆水中DOM的添加提高了表层土壤中供微生物生长的碳提高微生物丰度,与Blanchet等[33]的研究一致。DOM的长期输入会使湿地土壤结构发生改变,从而使微生物群落发生变化[2]。
硝化过程相关基因丰度也受到土壤有机质含量的影响,有机质的增加可显著提高其基因丰度[34]。本研究中可能是土壤处于淹水状态,硝化作用受到抑制[35],硝化细菌nxrA基因的丰度在总的细菌群落里所占的比例并不高。
上覆水添加DOM增加了反硝化的碳源,并提供了厌氧的条件,提高湿地表层土壤narG基因丰度[12, 36]。Chen等[37]的研究也表明加入植物滤液的反硝化细菌丰度明显高于不加植物滤液的处理组,外加DOM有助于反硝化细菌的生长。本研究中narG基因丰度较高,能够减少NO3--N的积累[15]。在氮源、碳源以及氧化还原条件更有利于反硝化作用的W1、W2、W3处理组系统中nirS基因丰度显著要高于其他处理组(P < 0.05),nirK基因的结果正相反,也可能是因为相比于nirK基因,nirS基因受碳源的影响更大[38]。
对于nosZ基因来讲,N2O的还原受制于前反应,因此nosZ基因丰度变化规律表现出了和narG基因、nirK基因的一致性[15]。本研究中反硝化过程各个环节的narG、nirK、nosZ三个基因的丰度变化规律表现出了一致性:有机质较高的土壤A中各基因丰度显著高于土壤B(P < 0.05),因为反应底物高且厌氧环境更适合反硝化的进行;另外,上覆水添加氮和DOM的处理组中基因丰度显著高于无添加的W5、W6(P < 0.05),而只添加氮的W4则没有发现显著差异(P > 0.05)。表明碳源在反硝化过程中起到了重要的作用[15]。各处理组nirK和nirS的基因丰度显著高于nxrA基因的丰度,与Fu等[36]的研究一致,实验过程中可能存在短程硝化反硝化途径。
Anammox细菌基因丰度与表层间隙水ORP结果相一致,还原性的环境是影响厌氧氨氧化过程的关键因素。Trimmer等[39]研究指出土壤有机碳的含量和厌氧氨氧化的活性显著正相关,有机质有助于厌氧氨氧化的进行。Zhi等[15]研究结果表明当有机质含量高时,湿地完全没有亚硝氮和硝氮的积累,而厌氧氨氧化菌显著增加。anammox细菌为自养型细菌,以CO2为碳源,有机碳的存在可能会厌氧氨氧化细菌利用有机碳取代基质作为新的代谢途径,展现多样的代谢途径[40]。有研究表明低浓度有机物存在时可促进厌氧氨氧化菌的活性,但有机物浓度高时,厌氧氨氧化菌活性大大降低[41-43]。厌氧氨氧化细菌在总的细菌群落里所占的比例较高,厌氧氨氧化成为湿地氮循环另一种途径[15]。
4 结论上覆水添加外源有机质,表层土壤有机质含量增幅较小,没有显著性的变化(P > 0.05),土壤特性的改变需经历动植物残体的沉降累积、元素的地球化学循环等,湿地表层土壤被破坏后的自然恢复是一个漫长的过程。
土壤有机质含量显著影响细菌、古菌及氮循环各环节相关基因的丰度。表层土壤被破坏,土壤中有机质含量降低使得微生物及参与氮循环的基因丰度显著下降(P < 0.05)。同时提高上覆水中的外源氮和DOM含量会显著促进湿地微生物以及氮功能基因丰度的增加(P < 0.05)。
土壤有机质含量增加显著促进反硝化过程相关的narG、nirS、nosZ三个基因的丰度,且nirS基因受外加碳源的影响更大。
湿地土壤一直处于淹水状态,表层土壤中厌氧氨氧化基因丰度较高,具有较大厌氧氨氧化去除氮素的潜能。
| [1] | 李春晖, 郑小康, 牛少凤, 等. 城市湿地保护与修复研究进展[J]. 地理科学进展, 2009, 28(2): 271–279. DOI:10.11820/dlkxjz.2009.02.016 |
| [2] | 吴丰昌. 天然有机质及其与污染物的相互作用[M]. 北京: 科学出版社, 2010: 311. |
| [3] | USEPA. A Handbook of Constructed Wetlands[M]. New York: USEPA, 1994. |
| [4] | 杨旭. 微污染饮用水源人工湿地预处理效能与作用机理研究[D]. 哈尔滨: 哈尔滨工业大学, 2008. http://cdmd.cnki.com.cn/article/cdmd-10213-2009223981.htm |
| [5] | Ragusa SR, McNevin D, Qasem S, et al. Indicators of biofilm development and activity in constructed wetlands microcosms[J]. Water Research, 2004, 38 (12): 2865–2873. DOI:10.1016/j.watres.2004.03.039 |
| [6] | 王世和. 人工湿地污水处理理论与技术[M]. 北京: 科学出版社, 2007: 17-19. |
| [7] | 夏宏生, 蔡明, 向欣. 人工湿地净化作用与微生物相关性研究[J]. 广东水利水电, 2008(3): 4–8. |
| [8] | 杜刚, 黄磊, 高旭, 等. 人工湿地微生物数量与污染物去除的关系[J]. 湿地科学, 2013, 11(1): 13–20. |
| [9] | Angnes G, Nicoloso RS, Silva MLBD, et al. Correlating denitrifying catabolic genes with N2O and N2emissions from swine slurry composting[J]. Bioresource Technology, 2013, 140 (3): 368–375. |
| [10] | Faulwetter JL, Gagnon V, Sundberg C, et al. Microbial processes influencing performance of treatment wetlands:A review[J]. Ecological Engineering, 2009, 35 (6): 987–1004. DOI:10.1016/j.ecoleng.2008.12.030 |
| [11] | 代嫣然, 梁威, 吴振斌. 低碳高氮废水的人工湿地脱氮研究进展[J]. 农业环境科学学报, 2010, 29: 305–309. |
| [12] | Gao M, Liu J, Qiao Y, et al. Diversity and abundance of the denitrifying microbiota in the sediment of eastern china marginal seas and the impact of environmental factors[J]. Microbial Ecology, 2017, 73 (3): 602–615. DOI:10.1007/s00248-016-0906-6 |
| [13] | 裴希超, 许艳丽, 魏巍. 湿地生态系统土壤微生物研究进展[J]. 湿地科学, 2009, 7(2): 181–186. |
| [14] | 李婧. 土壤有机质测定方法综述[J]. 分析试验室, 2008, 27(S1): 154–156. |
| [15] | hi W, Ji G. Quantitative response relationships between nitrogen transformation rates and nitrogen functional genes in a tidal flow constructed wetland under C/N ratio constraints[J]. Water Research, 2014, 64 (7): 32–41. |
| [16] | Grosskopf R, Janssen PH, Liesack W. Diversity and structure of the methanogenic community in anoxic rice paddy soil microcosms as examined by cultivation and direct 16S rRNA gene sequence retrieval[J]. Appl Environ Microbiol, 1998, 64 (3): 960–969. |
| [17] | Scala DJ, Kerkhof LJ. Nitrous oxide reductase(nosZ)gene-specific PCR primers for detection of denitrifiers and three nosZ genes from marine sediments[J]. Fems Microbiology Letters, 1998, 162 (1): 61–68. DOI:10.1111/fml.1998.162.issue-1 |
| [18] | roback I. N, Enwall K, Jarvis A, Hallin S. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. Fems Microbiology Ecology, 2004, 49 (3): 401–417. DOI:10.1016/j.femsec.2004.04.011 |
| [19] | Tsushima I, Kindaichi T, Okabe S. Quantification of anaerobic ammonium-oxidizing bacteria in enrichment cultures by real-time PCR[J]. Water Research, 2007, 41 (4): 785–794. DOI:10.1016/j.watres.2006.11.024 |
| [20] | Yan TF, Fields MW, Wu LY, et al. Molecular diversity and characterization of nitrite reductase gene fragments(nirK and nirS)from nitrate-and uranium-contaminated groundwater[J]. Environ Microbiol, 2003, 5 (1): 13–24. DOI:10.1046/j.1462-2920.2003.00393.x |
| [21] | Poly F, Wertz S, Brothier E, Degrange V. First exploration of Nitrobacter diversity in soils by a PCR cloning-sequencing approach targeting functional gene nxrA[J]. FEMS Microbiology Ecology, 2008, 63 (1): 132–140. DOI:10.1111/fem.2008.63.issue-1 |
| [22] | Wang H, Ji G, Bai X, He C. Assessing nitrogen transformation processes in a trickling filter under hydraulic loading rate constraints using nitrogen functional gene abundances[J]. Bioresourouse Technology, 2015, 177 : 217–223. DOI:10.1016/j.biortech.2014.11.094 |
| [23] | Henry S, Bru D, Stres B, et al. Quantitative detection of the nosZ gene, encoding nitrous oxide reductase and comparison of the abundances of 16S rRNA, narG, nirK, and nosZ genes in soils[J]. Appl Environ Microbiol, 2006, 72 (8): 5181–5189. DOI:10.1128/AEM.00231-06 |
| [24] | López-Gutiérrez JC, Henry S, Hallet S, et al. Quantification of a novel group of nitrate-reducing bacteria in the environment by real-time PCR[J]. Journal of Microbiological Methods, 2004, 57 (3): 399–407. DOI:10.1016/j.mimet.2004.02.009 |
| [25] | García-Lledó A, Vilar-Sanz A, Trias R, et al. Genetic potential for N2O emissions from the sediment of a free water surface constructed wetland[J]. Water Research, 2011, 45 (17): 5621–5632. DOI:10.1016/j.watres.2011.08.025 |
| [26] | Stres B, Mahne I, Avgustin G, et al. Nitrous oxide reductase(nosZ)gene fragments differ between native and cultivated Michigan soils[J]. Applied & Environ Microbiol, 2004, 70 (1): 301–309. |
| [27] | 李绪录, 周毅频, 夏华永. 大鹏湾表层沉积物中碳、氮、磷的多年调查结果和有机质来源分析[J]. 环境科学学报, 2012, 32(5): 1113–1119. |
| [28] | 冯璐, 施肥对祁连山湿地植物群落结构及土壤有机质的影响[D]. 西宁: 青海师范大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10746-1015661619.htm |
| [29] | Xi M, et al. Assessment of the content, structure, and source of soil dissolved organic matter in the coastal wetlands of Jiaozhou Bay, China[J]. Physics and Chemistry of the Earth Parts Alblc.(2017), http://dx.doi.org/10.1016/j.pce. 2017. 03. 004. |
| [30] | 刘树, 梁漱玉. 芦苇湿地土壤有机质含量对芦苇产能的影响研究[J]. 现代农业科技, 2008(7): 232–234. |
| [31] | Landa M, Cottrel T, Kirchman D, et al. Changes in bacterial diversity in response to dissolved organic matter supply in a continuous culture experiment[J]. Aquatic Microbial Ecology, 2013, 69 (2): 157–168. DOI:10.3354/ame01632 |
| [32] | 李露, 陈宏春, 高大文. 七星河湿地厌氧氨氧化古细菌多样性及其与环境因子的相关性分析[J]. 环境科学学报, 2015, 35(4): 1097–1105. |
| [33] | Blanchet M, Femandez C, Joux F. Photoreactivity of riverine and phytoplanktonic dissolved organic matter and its effects on the dynamics of a bacterial community from the coastal Mediterranean Sea[J]. Prog Oceanogr, 2017 . DOI:10.1016/j.pocean.2017.03.003 |
| [34] | Riley KA, Stein OR, Hook PB. Ammonium removal in constructed wetland microcosms as influenced by presence and species of plants and organic carbon load[J]. J Environ Sci Health Part A, 2005, 40 (6-7): 1109–1121. DOI:10.1081/ESE-200055594 |
| [35] | 邱昭政, 罗专溪, 赵艳玲, 颜昌宇. 溶氧对富集培养的河口湿地表层沉积物氨氧化菌多样性及氨氧化速率的影响[J]. 2013, 34(2): 532-539. |
| [36] | Fu GP, Huang LK, Guo ZP, et al. Effect of plant-based carbon sources on denitrifying microorganisms in a vertical flow constructed wetland[J]. Bioresource Technology, 2017, 224 : 214–221. DOI:10.1016/j.biortech.2016.11.007 |
| [37] | Chen Y, Wen Y, Zhou Q, et al. Effects of plant biomass on denitrifying genes in subsurface-flow constructed wetlands[J]. Bioresource technology, 2014, 157 : 341–345. DOI:10.1016/j.biortech.2014.01.137 |
| [38] | Hallin S, Throb?ck IN, Dicksved J, et al. Metabolic profiles and genetic diversity of denitrifying communities in activated sludge after addition of methanol or ethanol[J]. Applied & Environ Microbiol, 2006, 72 (72): 5445–5452. |
| [39] | Trimmer M, Nicholls JC, Deflandre B. Anaerobic ammonium oxidation measured in sediments along the Thames estuary, United Kingdom[J]. Applied & Environ Microbiol, 2003, 69 (11): 6447–6454. |
| [40] | Lan CJ, Kumar M, Wang CC, et al. Development of simultaneous partial nitrification, anammox and denitrification(SNAD)process in a sequential batch reactor[J]. BioresourceTtechnology, 2011, 102 (9): 5514–5519. DOI:10.1016/j.biortech.2010.11.024 |
| [41] | Zhang SY, Wu P, Song YL, et al. Nitrogen removal using ANAMMOX and denitrification for treatment of municipal sewage[J]. Environmental Science, 2015, 36 (11): 4174–4179. |
| [42] | Yang PB, Li X, Huang Y, et al. Short or long term influence of phenol on nitrogen removal efficiency of ANAMMOX sludge[J]. Environmental Science, 2015, 36 (10): 3771–3777. |
| [43] | 管勇杰, 于德爽, 李津, 等. 有机碳源作用下厌氧氨氧化系统的脱氮效能[J]. 环境科学, 2017, 38(2): 654–664. |