随着全球工业化发展和现代化进程的加快,人们对石油及其制品的需求快速增加。为了满足人们日益增加的需求,石油的开采量和开采范围不断扩大,交易量逐年攀升,导致受污染的土壤和海域范围也在不断扩展。据估计每年造成环境污染泄露的石油在百万t以上。石油污染由于其高毒性和难降解性已对生物和生态环境造成了极大的危害,如何治理并恢复健康的生态环境已成为人类生存和社会经济可持续发展的瓶颈。微生物治理技术以其环境友好和费用低(仅为理化治理技术30%-50%)的优势,引起了相关领域的极大关注[1-2]。由于不同微生物的作用底物和作用机制不同,而石油组分相对较复杂,所处的环境条件也不尽相同,单一微生物很难达到理想的修复效果,因此高效石油降解菌的选择和协同作用很重要,掌握菌种资源分类特征和作用机制是面对微生物修复技术应用挑战和取得理想修复效果的关键[3]。
本文总结和梳理了石油污染与技术应用趋势、石油降解微生物聚类分析、石油微生物降解分子机制和石油污染的微生物修复技术应用等方面,梳理了石油污染微生物修复相关领域的研究进展。以期为微生物修复技术的高效和广泛应用提供支撑。
1 石油污染与技术应用趋势石油已经成为世界各国最主要的环境污染物之一[4-5],早在20世纪90年代,研究者就发现石油污染危害的持久性,石油污染不仅引起了水土环境质量的下降,而且导致了大批动植物死亡,甚至引发了物种基因的突变,对整个生态系统造成了难以弥补的危害[6]。据保守估计每年平均石油泄漏量在110万t左右,其中海洋石油泄漏有60-80万t之多[8-9]。以污染土壤的地下储油罐泄漏为例,20世纪90年代,美国经确认的地下渗漏储油罐达10万多个[10],至2001年短短10年间已上升至44万个[11]。与陆地石油污染相比,水体较土壤的流动性更强,污染扩散范围也更广。海洋作为第一大水体,石油污染已成为破坏海洋生态系统的第一大元凶。据统计,海洋石油污染发生的次数约占海洋总污染事件的80%[12]。自1969年发生第一次超级油船失事以来,世界上已有超过40处大的海洋泄漏。20世纪80年代初发生在英国北海的油田和油轮漏油事件,使斯堪的纳维亚半岛地区的海洋生物几乎绝迹。同时期80年代末,在美国阿拉斯加威廉王子海峡触礁的万t级“瓦德兹”号油轮,造成4万t原油倾泻入海,致使20多万海鸟和4千多头海獭死亡,至今经过近30年的生态恢复,威廉王子峡湾的海獭在掘食蛤蜊时,仍能刨到当年渗漏的石油。另一次令人震惊的海洋石油污染事件是21世纪90年代中东海湾战争导致的输油管大泄露,原油以每天数百万桶的速度从艾哈迈迪输油管中漏入大海,在波斯湾形成一片长56 km、宽16 km水面油膜,在短短月余时间内溢油总量达170万t,成为人类历史上最严重的原油泄漏事故。进入21世纪,随着世界经济的高速发展,世界各国对石油的需求也与日俱增,全球石油总产量已由20世纪的20亿t以上增加至2014年的42亿t[12]。海洋石油污染也越来越严重和频繁。据统计,新世纪前12年全世界共发生了77起大型海上溢油事件,以2010年发生13起、2011年10起最为频繁,其持续时间之长、溢油之多和危害之大令海洋生态环境遭受了巨大破坏[13]。2010年最严重的墨西哥湾油井爆炸事件,致使上百万加仑原油从墨西哥湾1 500 m海面下喷涌而出,所引发的海洋生态破坏程度堪比30多年前“瓦德兹”号事故。
我国的石油污染状况也不容乐观,我国生产的石油80%来自陆地,以油田基地周围的土壤污染最为严重。据初步统计,我国石化行业中,每年平均仅各种底泥的产量就有80万t[14]。我国华北油田周围的很多农田由于原油污染而无法耕种,部分石油化工区可高达10 g/kg土壤,远高于土壤污染临界值200 mg/kg[15]。每年继续新增污染土壤10万t[16]。严重的地方甚至污染到地下水资源,如山东淄博地区早在21世纪初地下水的最高含油就达到100 mg/L以上,超过国家标准(<0.1 mg/L﹚1 000倍以上[17]。我国地下储油罐泄漏情况虽然没有系统的统计资料,但仅就已经报道的加油站漏油事故已显示我国的储油罐问题不容忽视。石油污染也威胁到了我国的地表水资源,以黄河和辽河为例,黄河水系年平均含油最高可达4.82 mg/L,辽河水系年平均含油最高可达7.68 mg/L,远远超过了国家地面水环境质量标准确定的3类地表水标准(GB3838-2002,<0.1 mg/L)。我国海洋也是石油污染的重灾区。20世纪70年代末到80年代初,石油以10万t/年的速度进入我国近海海域,到80年代末该数值增至近20万t/年,之后呈逐年递增趋势[18]。如1983年巴拿马籍“东方大使”号油轮在青岛港出港行驶至中沙礁搁浅,导致3 343 t原油泄漏,污染海岸线230 km,同时对附近1000多hm2的水产养殖区造成严重污染。本世纪2004年末,巴拿马籍集装箱船“现代促进”轮与德国籍集装箱船“地中海伊伦娜”轮发生碰撞,导致1 200多t船舶燃料油溢出,在海上形成一条长约16.5 km的油带,造成珠江口海域严重污染。2011年,大连新港一艘利比里亚籍30万t油轮卸油,陆地输油管线爆炸引发大火和原油入海,溢油的总量达到了1 500 t,导致50 km2海面受污染,同年渤海蓬莱发生的19-3油田溢油事故,共造成劣四类海水面积800 km2之多,使整个渤海生态系统受到严重危害[11, 13, 19]。
石油污染对生物和生态环境造成的极大危害显而易见,未来的形势也更加严峻,对之进行治理也成为生态环境可持续发展中最迫切的事情。利用微生物降解石油污染物的成功案例最早见于20世纪80年代末美国治理“瓦德兹”油轮石油泄露项目,该项目利用微生物短时间内清除了石油污染,开创了生物修复治理石油海洋污染的先例。此后石油污染微生物修复相关领域的研究呈现了爆发式的蓬勃发展,在微生物菌种资源、降解机制、环境应用等方面都取得了一定成果。
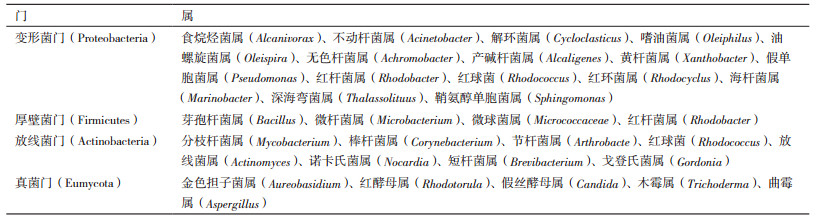
2 石油降解微生物归类分析油污降解功能微生物涉及了众多的细菌种属和部分真菌种属,分离及鉴定烷烃降解微生物是微生物治理技术应用的基础[20]。据研究报道统计,石油烃降解菌涉及到细菌7个门之多,近70个属,真菌13个属,放线菌10个属、蓝藻9个属、海藻14个属,共200多种微生物。根据系统发育树从门和亚门级对石油降解菌归类分析,结果显示细菌主要为变形菌门(proteobacteria)和厚壁菌门(Finnicutes)的种属,放线菌集中于放线菌门(Actinomycesbovis),真菌归属真菌门(Eumycota),又以子囊菌亚门(Ascomycetes)、半知菌亚门(Deuteromycotina)和担子菌亚门(Basidiomycotina)的种属为主[21]。结合国内外的研究报道,将研究比较深入的石油烃降解菌按照门级归类如表 1。从表 1可以看出石油降解菌归属有偏好,细菌以变形菌门和放线菌门种属占比较高,尤其以变形菌门凸显。
微生物利用石油烃底物的活性存在不同程度的差异,细菌是石油烃降解菌团中研究最早和最广泛的微生物种类[22-23]。仅以石油烃为唯一碳源的细菌就达到7个属之多,包括食烷烃菌属(Alcanivorax)、不动杆菌属(Acinetobacter)、解环菌属(Cycloclasticus)、嗜油菌属(Oleiphilus)、油螺旋菌属(Oleispira)、深海弯菌属(Thalassolituus)和海杆菌属(Marinobacter)。其中的食烷烃菌属、嗜油菌属、油螺旋菌属、深海弯菌属可以利用各种各样的直链和/或支链饱和烃。不动杆菌属(Acinetobacter)可以利用C10-40的大分子直链烷烃,而解环菌属则以多环芳烃为底物,其他属细菌可利用的底物具有多样性[24-28]。
油污土壤和油污海水中富集的微生物种群类别和数量也存在差异。石油污染土壤中主要累积的细菌有红杆菌(Rhodobacter)、红球菌(Rhodococcus)、节杆菌(Arthrobacter)和短杆菌(Brevibacterium),尤其是光合红细菌数量较未污染土壤高出很多[29-30]。有两类重要的光合细菌类荚膜红杆菌(Rhodobacter capsulata)和胶质红环菌属(Rhodocyclus gelatinosa)可代谢土壤石油污染物[31]。油污海洋中不仅会富集大量的石油降解细菌,也会富集大量的石油降解真菌。富集的石油降解细菌有:食烷烃菌(Alcanivorax)、海杆菌(Marinobacter)、无色杆菌(Achromobacter)、假单胞菌(Pseudomonas)、不动杆菌(Acinetobacter)、芽孢杆菌(Bacillus)、微球菌(Micrococcus)和产碱杆菌(Alcaligenes)等;富集的真菌包括金色担子菌(Aureobasidium)、红酵母菌(Rhodotorula)、假丝酵母菌(Candida)等[20, 32-33]。有些研究结果表明拟杆菌也是海洋石油降解菌群中的重要菌类,尤其在污染海洋的底泥样本中[34-36]。其中以对降解芳香烃的新鞘氨醇杆菌属(Novosphingobium)研究得最为深入[37-39]。
以上的研究报道说明不同种属的微生物对底物和环境条件具有一定的选择性和倾向性,这些属性是由微生物代谢石油烃的分子代谢机制决定的。
3 微生物降解石油的分子机制研究微生物石油降解机制是提升微生物治理效率的内在动力。微生物降解石油类化合物的难易程度不同,依次为直链烷烃>支链烷烃>小分子芳烃>环烷烃[2]。链烷烃主要通过好氧的单末端氧化、次末端氧化、双末端氧化和厌氧的直接脱氢方式降解[40-41]。各途径生成的脂肪酸再进行β氧化最终形成二碳脂肪酸和乙酰辅酶A,进入三羧酸循环放出CO2。相比于直/支链烷烃,环烷烃的结构决定了其更难于被微生物降解。环烷烃的好氧降解一般是在多功能氧化酶的催化下形成环醇,继而脱氢成环酮,再进一步氧化成环酯,也可开环成为脂肪二酸。芳香烃类因其结构中均含有苯环结构,所以其好氧降解的共同点是苯环结构在加氧酶催化下先加氧成苯醇,进而脱氢为苯酚、开环等一系列后续酶促反应,最后进入三羧酸循环[42-43]。对于好氧条件下难降解的芳香烃,在厌氧条件下降解的共同点是苯环结构在羧酸氧化酶的催化下生成苯甲酸,然后生成苯甲酰辅酶A[43-44]。
近年来,随着分子生物学和生物信息学技术的发展,石油微生物功能基因和蛋白的研究成为新的热点。早在2003年,Acinetobacter sp. ADPl和Pseudomonas aeruginosa PAOl两株石油降解菌的全基因组序列己经由耶鲁大学和华盛顿大学公布并进行功能基因组学分析,石油降解菌Alcanivorax borkumensis SK2的全基因组序列也由德国的GBF中心(GBF-German Research centre for Biotechnology)完成了破译[45]。随着基因测序技术的革命性发展,测序成本大幅度降低,众多的石油降解菌完成了基因组的测序工作,包括节杆菌、分支杆菌、芽孢杆菌、红球菌、鞘氨醇单胞菌和多个假单胞菌[46-49]。与此同时,对石油烃降解蛋白的研究也逐渐聚焦一些关键的烷烃降解酶,依据烷烃降解酶催化底物烷烃链的长度不同,可把烷烃降解酶分为3类:(1) 负责氧化短链烷烃的可溶性甲烷单加氧酶(sMMO)及与其类似酶[50-51];(2) 氧化中等链长烷烃的膜结合相关烷烃羟化酶Alk和细胞色素P450单氧酶[52-54];(3) 氧化长链烷烃的酶系统AlmA[41, 55-56, 58]。研究结果显示烷烃羟化酶是由跨膜羟化酶AlkB、胞质红素氧还蛋白AlkFG和红素氧还蛋白还原酶AlkT所组成,形成完整的3组分酶复合体,其中跨膜烷烃羟化酶AlkB以及细胞色素P450和黄素结合蛋白AlmA是目前研究较为广泛的石油降解酶,能够催化烷烃降解的起始反应。在所有已鉴定的烷烃羟化酶中都显示共有一个7个氨基酸组成的保守区NYXEHYG(L/M),该区域可被用来识别跨膜烷烃羟化酶[57]。不同降解菌中的P450蛋白依据基因差异可分别归属于267个不同家族[58]。对应于石油组分的复杂性,烷烃降解菌的酶系统也呈现出多样性,不同的烷烃降解酶可同时存在于同一菌种,同时这些降解酶系统的底物范围又可交叉或重叠[59-60]。
石油烃降解菌在长期适应环境中不仅发展出了以石油为底物的代谢系统,而且形成了有利于底物接近与获取的辅助系统。例如微生物细胞壁外的特殊吸收系统,该系统可产生表面活性剂使石油烃类化合物充分乳化,以利于细菌的吸收和利用[61]。据报道,有百种以上的微生物能产生表面活性剂。微生物产生的生物表面活性剂就其化学组成来分,主要分为糖脂类和脂肽类,这些表面活性剂通过增强烃类水溶性来提高微生物细胞的底物获取能力[62-63]。另外一些微生物则通过自身的趋化性运动与底物接触,如一些假单胞类污染物降解菌(Pseudorhonas pzctida G7、Pseudomonas pvtida RKdI和Psetidomonas sp. strain NCIB)对底物有一定的趋化行为[64-66]。
4 石油污染的微生物修复技术微生物修复技术的应用是微生物菌种资源获取和污染降解机制研究的最终目的。目前利用微生物对石油降解效果和影响因子的研究多集中在实验室条件下,野外和露天自然条件下的应用研究相对较少[67-71]。20世纪80年代末期,美国应用微生物技术成功清理了“瓦德兹”油轮石油泄漏造成的污染,开创了微生物修复技术治理石油污染的先例[72-74]。
随后的研究证明,石油微生物降解的程度和效率依赖于微生物的种类数量以及生物降解的环境条件[2, 75]。微生物作为石油污染修复主体,是微生物修复技术建立的核心。由于不同种属微生物作用的底物有差异,没有一种微生物能够单独降解所有的石油烃组分,必须通过多种微生物的联合氧化和偏利共生的协同作用才能取得满意效果[22]。因此依据环境中污染物的成分,利用不同微生物作用对象和作用机制的互利性,形成针对多组分底物的多样性微生物菌团,才能达到更好的微生物石油降解或环境修复效果。
微生物的降解效果不仅与自身代谢机制相关,同时也受到环境因子的影响。影响微生物降解效果的环境因子包括营养元素、氧气、温度和表面活性剂等。环境中的N、P营养元素是微生物成功降解石油的重要原料,只有满足一定的养分条件,微生物才能有效代谢石油污染物。有研究结果认为环境中C:N:P的最适比例为100:10:1[76]。为满足营养盐的浓度需求,不同修复环境所适宜的营养盐形态也不同。土壤环境施加水溶性的氮磷营养盐可有效促进烃类的生物降解,但海洋环境由于受到水流扩散等的影响,水溶性营养盐更容易稀释和流失,而水不溶或脂溶性营养盐于油相中能更好地保障微生物的利用[2, 77]。虽然营养元素的缺乏可限制微生物的生长,但过高的浓度也会对石油降解起抑制作用。表面活性剂的作用与营养盐相似,适量的表面活性剂会降低微生物利用石油的表面张力,促进石油烃降解菌的生长,但随着表面活性剂的持续增加菌量反而会减少[1, 78]。由于石油烃的好氧降解效率相对较高,氧作为电子受体可提高石油烃的降解效率[79-80]。厌氧条件下虽然部分微生物能降解石油,但降解速率相对较低[42],但在有氧条件下较难降解的姥鲛烷和植烷可在厌氧条件下发生降解[81]。温度也是影响石油降解效率的重要因子之一。虽然降解石油的微生物有嗜冷菌、嗜热菌和嗜中温菌,但大多数石油降解菌属嗜中温菌,最适温度在30℃左右,温度过高过低都会对降解效率产生抑制。另外,环境的酸碱条件也是影响微生物降解效果的关键因子,大部分的微生物最适PH在中性左右,酸性和碱性条件均不利于微生物的生长。
综上所述,微生物修复石油污染的效果,不仅与微生物自身的降解能力有关,而且与菌种之间的协同性、环境因子的适宜度等都有关系,所以要综合考虑各种因素,除了依据类别和降解机制选择合适的高效降解菌协作菌团外,还要为这些降解菌创造必要的生存、繁殖和降解条件,才能达到更好的环境污染修复效果。
| [1] | Das N, Chandran P. Microbial degradation of petroleum hydrocarbon contaminants:an overview[J]. Biotechnology Research Interna-tional, 2010, 2011 (1): 941810. |
| [2] | Leahy JG, Colwell RR. Microbial degradation of hydrocarbons in the environment[J]. Microbiological Reviews, 1990, 54 (3): 305–315. |
| [3] | Atlas RM, Cerniglia CE. Bioremediation of petroleum pollutants[J]. Bioscience, 1995, 35 (1-3): 317–327. |
| [4] | Holliger C, Gaspard S, Glod G, et al. Contaminated environments in the subsurface and bioremediation:organic contaminants[J]. Fems MicroBiol Rev, 2010, 20 (3-4): 517–523. |
| [5] | Kvenvolden KA, Cooper CK. Natural seepage of crude oil into the marine environment[J]. Geoo-Mar Lett, 2003, 23 (3-4): 140–146. DOI:10.1007/s00367-003-0135-0 |
| [6] | Alvarez PJ, Vogel TM. Substrate interactions of benzene, toluene, and para-xylene during microbial degradation by pure cultures and mixed culture aquifer slurries[J]. Applied & Environ Microbiol, 1991, 57 (10): 2981–2985. |
| [7] | Brooijmans RJ, Pastink MI, Siezen RJ. Hydrocarbon-degrading bacteria:the oil-spill clean-up crew[J]. Microbial Biotechnology, 2010, 2 (6): 587–594. |
| [8] | Kvenvolden KA, Cooper CK. Natural seepage of crude oil into the marine environment[J]. Geo-Marine Letters, 2003, 23 (3-4): 140–146. DOI:10.1007/s00367-003-0135-0 |
| [9] | Truax DD, Britto R, Sherrard JH. Bench-scale studies of reactor-based treatment of fuel-contaminated soils[J]. Waste Management, 1995, 15 (5): 351–357. |
| [10] | Agency EP. Cleaning up the nation's waste sites:markets and technology trends[M]//Cincinnatio, OH.US:Government Institutes, Inc. 2004:103-104. |
| [11] | Huang L, Xie J, Shi XF, et al. Research on the isolation, identifi-cation and degradation characteristics of a diesel oil degrading strain[J]. Advanced Materials Research, 2013, 642 (1): 206–210. |
| [12] | 2014年全球石油产量分国家统计情况, http://www.askci.com/news/chanye/2015/10/23/159481sgr.shtml |
| [13] | List of petroleum hydrocarbon spills https://en.wikipedia.org/wiki/List_of_petroleum hydrocarbon_spills. |
| [14] | 刘五星, 骆永明, 滕应, 等. 我国部分油田土壤及油泥的石油污染初步研究[J]. 土壤, 2007, 39(2): 247–251. |
| [15] | 何良菊, 魏德洲, 张维庆. 土壤微生物处理石油污染的研究[J]. 环境工程学报, 1999(3): 110–115. |
| [16] | 李广贺, 张旭, 卢晓霞. 土壤残油生物降解性与微生物活性[J]. 地球科学-中国地质大学学报, 2002, 27(2): 181–185. |
| [17] | 曹云者, 施烈焰, 李丽, 等. 石油烃污染场地环境风险评价与风险管理[J]. 生态毒理学报, 2007, 2(3): 265–272. |
| [18] | 蔡成翔, 焦淑菲, 尹艳镇. 我国近岸海域石油污染现状及其防治措施[J]. 化工技术与开发, 2012, 41(8): 14–17. |
| [19] | 重大海洋石油污染事件, http://www.ymhb.gov.cn/info/content.asp?info Id=2924. |
| [20] | Van Hamme JD, Singh A, Ward OP. Recent advances in petroleum microbiology[J]. Microbiology and Molecular Biol Rev:MMBR, 2003, 67 (4): 503–549. DOI:10.1128/MMBR.67.4.503-549.2003 |
| [21] | Ollivier B, Magot M. Petroleum microbiology[M]. American Society for Microbiology Press, 2005: 317-336. |
| [22] | Rahman KS, Rahman TJ, Kourkoutas Y, et al. Enhanced bioremediation of n-alkane in petroleum sludge using bacterial consortium amended with rhamnolipid and micronutrients[J]. Bioresource Technology, 2003, 90 (2): 159–168. DOI:10.1016/S0960-8524(03)00114-7 |
| [23] | Brooijmans RJW, Pastink MI, Siezen RJ, et al. Hydrocarbon-degrading bacteria:the oil-spill clean-up crew[J]. Microbial Biotechnology, 2010, 2 (6): 587–594. |
| [24] | Throneholst M, Wentzel A, Ellingsen TE, et al. Identification of novel genes involved in long-Chain n-Alkane degradation by Acinetobacter sp. Strain DSM 17874[J]. Applied & Environ Microbiol, 2007, 73 (10): 3327–3332. |
| [25] | Head IM, Swannell RPJ. Bioremediation of potroleum hydrocarbon contaminants in marine habitats[J]. Curr Opin Biotech, 1999, 10 : 234–239. DOI:10.1016/S0958-1669(99)80041-X |
| [26] | Yakimov MM, Timmis KN, Golyshin PN. Obligate oil-degrading marine bacteria[J]. Current Opinion in Biotechnology, 2007, 18 (3): 257–266. DOI:10.1016/j.copbio.2007.04.006 |
| [27] | Head IM, Jones DM, Röling WFM. Marine microorganisms make a meal of oil[J]. Nat Rev Microbiol, 2006, 4 (3): 173–182. DOI:10.1038/nrmicro1348 |
| [28] | Yakimov MM, Giuliano L, Gentile G, et al. Oleispira antarctica gen. nov. sp. nov. a novel hydrocarbonoclastic marine bacterium isolated from Antarctic coastal sea water[J]. International Journal of Systematic & Evolutionary Microbiology, 2003, 53 (3): 779–785. |
| [29] | Guzev VS, Khalimov EM, Volde MI, et al. Regulatory effect of glucose on hydrocarbon-oxidizing microorganisms in soil[J]. Microbiology, 1997 (2): 124–128. |
| [30] | Guzev VS, Volde MI, Kulichevskaya IS, et al. Effect of butyric acid on the physiological activity of hydrocarbon-oxidizing Rhodococci[J]. Microbiology, 2001, 70 (3): 263–269. DOI:10.1023/A:1010490924885 |
| [31] | Tkh M, Kirienko OA, Imranova EL. Effect of photosynthetic bacteria and compost on degradation of petroleum products in s petroleum hydrocarbon[J]. Applied Biochemistry & Microbiology, 2004, 40 (2): 181–185. |
| [32] | Hara A, Syutsubo K, Harayama S. Alcanivorax which prevails in oil-contaminated seawater exhibits broad substrate specificity for alkane degradation[J]. Environ Microbiol, 2003, 5 (9): 746–753. DOI:10.1046/j.1468-2920.2003.00468.x |
| [33] | 张士璀, 范晓, 马军英. 海洋生物技术和应用[M]. 第2版. 北京: 海洋生物技术出版社, 1997: 1-274. |
| [34] | Kasai Y, Kishira H, Syutsubo K, et al. Molecular detection of marine bacterial populations on beaches contaminated by the Nakhodka tanker oil-spill accident[J]. Environ Microbiol, 2001, 3 (4): 246–255. DOI:10.1046/j.1462-2920.2001.00185.x |
| [35] | Kostka JE, Prakash O, Overholt WA, et al. Hydrocarbon-degrading bacteria and the bacterial community response in gulf of Mexico beach sands impacted by the deepwater horizon oil spill[J]. Applied & Environ Microbiol, 2011, 77 (22): 7962–7974. |
| [36] | 高小玉, 明红霞, 陈佳莹, 等. 大连湾石油污染沉积物中细菌群落结构分析[J]. 海洋学报, 2014, 36(6): 58–66. |
| [37] | Wongwongsee W, Chareanpat P, Pinyakong O. Abilities and genes for PAH biodegradation of bacteria isolated from mangrove sediments from the central of Thailand[J]. Marine Pollution Bulletin, 2013, 74 (1): 95–104. DOI:10.1016/j.marpolbul.2013.07.025 |
| [38] | 崔志松, 邵宗泽. 一株海洋新鞘氨醇杆菌phe-8(Novosphingo-bium sp.)的PAHs降解基因和降解特性[J]. 厦门大学学报:自然科学版, 2006, 45(s1): 257–261. |
| [39] | Segura A, Hernández-Sánchez V, Marqués S, et al. Insights in the regulation of the degradation of PAHs in Novosphingobium sp. HR1a and utilization of this regulatory system as a tool for the detection of PAHs[J]. Science of the Total Environment, 2017 . |
| [40] | Adebusoye SA, Ilori MO, Amund OO, et al. Microbial degradation of petroleum hydrocarbons in a polluted tropical stream[J]. World Journal of Microbiology and Biotechnology, 2007, 23 (8): 1149–1159. DOI:10.1007/s11274-007-9345-3 |
| [41] | Wang W, Shao Z. Diversity of flavin-binding monooxygenase genes(almA)in marine bacteria capable of degradation long-chain alkanes[J]. FEMS Microbiol Ecol, 2012, 80 (3): 523–533. DOI:10.1111/fem.2012.80.issue-3 |
| [42] | Heider J, Schühle K. Anaerobic biodegradation of hydrocarbons including methane[M]// The Prokaryotes-Prokaryotic Physiology and Biochemistry. Springer-Verlag Berlin Heidelberg, 2013:605-634. |
| [43] | Hara E, Uchiyama H. Degradation of petroleum pollutant materials by fungi[OL/M]. Fungi as Bioremediators, 2013, 32:117-133. |
| [44] | Wang XB, Chi CQ, Nie Y. Degradation of petroleum hydrocarbons(C6-C40) and crude petroleum hydrocarbon by a novel Dietzia strain[J]. Bioresource Technology, 2011, 102 (17): 7755–7761. DOI:10.1016/j.biortech.2011.06.009 |
| [45] | Golyshin PN, Martins Dos Santos VA, Kaiser 0, et al. Genome sequence completed of Alcanivorax borkumensis, a hydrocarbon-degrading bacterium that plays a global role in petroleum hydrocarbon removal from marine systems[J]. J Biotechnol, 2003, 106 (2-3): 215–220. DOI:10.1016/j.jbiotec.2003.07.013 |
| [46] | Jin HM, Jeong H, Moon EJ, et al. Complete genome sequence of the polycyclic aromatic hydrocarbon-degrading bacterium Alteromonas sp. Strain SN2[J]. J Bacteriol, 2011, 193 (16): 4292–4293. DOI:10.1128/JB.05252-11 |
| [47] | Tang H, Yu H, Li Q, et al. Genome sequence of Pseudomonas putida strain B6-2, a superdegrader of polycyclic aromatic hydrocarbons and dioxin-like compounds[J]. Journal of Bacteriology, 2011, 193 (23): 6789–6790. DOI:10.1128/JB.06201-11 |
| [48] | Fondi M, Rizzi E, Emiliani G, et al. The genome sequence of the hydrocarbon-degrading Acinetobacter venetianus VE-C3[J]. Research in Microbiology, 2013, 164 (5): 439–449. DOI:10.1016/j.resmic.2013.03.003 |
| [49] | Hao K, Li H, Li F, et al. Complete genome sequence of Bacillus pumilus PDSLzg-1, a hydrocarbon-degrading bacterium isolated from oil-contaminated soil in China[J]. Genome Announc, 2016, 4 (5): pii:e010779–16. |
| [50] | Sullivan JP, Dickinson D, Chase HA. Methanotrophs, Methylosinus trichosporium OB3b, sMMO, and their application to bioremediation[J]. Crit Rev Microbiol, 1998, 24 (4): 335–373. DOI:10.1080/10408419891294217 |
| [51] | Siewert I, Limberg C. Low-molecular-weight analogues of the soluble methane monooxygenase(sMMO):from the structural mimicking of resting states and intermediates to functional models[J]. Chemistry, 2009, 15 (40): 10316–10328. DOI:10.1002/chem.v15:40 |
| [52] | Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases[J]. Enzyme & Microbial Technology, 2006, 39 (2): 235–251. |
| [53] | Belhaj A, Desnoues N, Elmerich C. Alkane biodegradation in Pseudomonas aeruginosa strains isolated from a polluted zone:identification of alkB and alkB-related genes[J]. Research in Microbiology, 2002, 153 (6): 339–344. DOI:10.1016/S0923-2508(02)01333-5 |
| [54] | Nebert DW, Roe AL, Dieter MZ, et al. Role of the aromatic hydrocarbon receptor and[Ah]gene battery in the oxidative stress response, cell cycle control, and apoptosis[J]. Biochemical Pharmacology, 2000, 59 (1): 65–85. DOI:10.1016/S0006-2952(99)00310-X |
| [55] | 王万鹏, 邵宗泽. 红灯食烷菌(Alcanivorax hongdengensis)黄素结合单加氧酶(AlmA)的基因克隆及其烷烃诱导表达[J]. 微生物学报, 2010, 50(8): 1051–1057. |
| [56] | 王丽萍. 大西洋表层海水烷烃降解菌及其烷烃羟化酶CYP153A基因的多样性分析[D]. 厦门: 国家海洋局第三海洋研究所, 2009. http://cdmd.cnki.com.cn/article/cdmd-85303-2009198276.htm |
| [57] | van Beilen JB, Neusenschwander M, Smits THM. Rubredoxins involved in alkane oxidation[J]. Bacteriol, 2002, 184 (6): 1722–1732. DOI:10.1128/JB.184.6.1722-1732.2002 |
| [58] | 付媛媛. 北部湾石油降解菌的多样性分析与咸水球形菌(Salinisphaera)的系统根进化及烷烃降解基因研究[D]. 厦门: 国家海洋局第三海洋研究所, 2011. http://cdmd.cnki.com.cn/Article/CDMD-85303-1011169121.htm |
| [59] | Haddock JD. Aerobic degradation of aromatic hydrocarbons:enzyme structures and catalytic mechanisms[M]. Springer Berlin Heidelberg, 2010. |
| [60] | 吴业辉. 海洋石油降解菌的分离鉴定及其降解酶基因的研究[D]. 厦门: 厦门大学, 2007. http://mobileir.xmu.edu.cn:8080/dspace/handle/2288/46299 |
| [61] | Wolfe MF, Olsen HE, Gasuad KA. Effect of initial petroleum hydrocarbon concentration and dispersant on crude petroleum hydrocarbon biodegradation in contaminated seawater[J]. Marine Environmental Research, 1999, 47 : 473–489. DOI:10.1016/S0141-1136(98)00132-9 |
| [62] | Chauhan A, Fazlurrahman, Qakeshott JG, Jain RK. Bacterial metabolism of polycyclic aromatic hydrocarbons:strategies for bioremediation[J]. Indian J Microbiol, 2008, 48 : 95–113. DOI:10.1007/s12088-008-0010-9 |
| [63] | Makkar RS, Rockne KJ. Comparison of synthetic surfactants and biosurfactants in enhancing biodegradation of polycyclic aromatic hydrocarbons[J]. Environmental Toxicology & Chemistry, 2003, 22 (10): 2280–2292. |
| [64] | Grimm A, Harwood C. NahY, a catabolic plasmid-encoded receptor required for chemotaxis of Pseudomonas putida to the aromatic hydrocarbon naphthalene[J]. Journal of Bacteriology, 1999, 181 (10): 3310–3316. |
| [65] | Samanta SK, Jain RK. Evidence for plasmid-mediated chemotaxis of Pseudomonas putidatowards naphthalene and salicylate[J]. Can J Microbiol, 2000, 46 : 1–6. DOI:10.1139/cjm-46-1-1 |
| [66] | Grimm AC, Harwood CS. Chemotaxis of Pseudomonas putirda to the polyaromatic hydrocarbon naphthalene[J]. Appl Environ Microbiol, 1997, 63 : 4111–4115. |
| [67] | Mearns AJ. Cleaning oiled shores:Putting bioremediation to]the test[J]. Spill Science & Technology Bulletin, 1997, 4 (4): 209–217. |
| [68] | Prince RC. Petroleum spill bioremediation in marine environments[J]. Crit Rev Microbiol, 1993, 19 (4): 217–242. DOI:10.3109/10408419309113530 |
| [69] | Swannell RP, Lee K, Mcdonagh M. Field evaluations of marine oil spill bioremediation[J]. Microbiol Rev, 1996, 60 (2): 342–365. |
| [70] | Mearns A, Venosa A, Lee K, et al. Field-Testigg Bioremediation Treating agents:Lessons From an Experime[J/OL]// International Oil Spill Conference, 2012:707-712. |
| [71] | Bao MT, Wang LN, Sun PY, et al. Biodegradation of crude oil using an efficient microbial consortium in a simulated marine environment[J/OL]. Marine Pollution Bulletin, 2012, 64(6):1177-1185. |
| [72] | Serpone N. Elizzetti EP[M]. New York: Wiley Publishing Inc, 1989: 10-11. |
| [73] | Lovley DR, Woodward JC, Chapelle FH. Stimulated anoxic biodegradation of aromatic hydrocarbons using Fe(Ⅲ)ligands[J]. Nature, 1994, 370 (6485): 128–131. DOI:10.1038/370128a0 |
| [74] | Sprintson A. Fundamentals and applications[J]. Synthesis, 2005, 65 (3): 119–120. |
| [75] | Varjani SJ. Microbial degradation of petroleum hydrocarbons[J]. Bioresource Technology, 2016, 223 : 277–286. |
| [76] | Nikolopoulou M, Kalogerakis N. Enhanced bioremediation of crude oil utilizing lipophilic fertilizers combined with biosurfactants and molasses[J]. Mar Pollut Bull, 2008, 56 (11): 1855–1861. DOI:10.1016/j.marpolbul.2008.07.021 |
| [77] | Delille D, Pelletier E. Effectiveness of bioremediation of crude oil contaminated subantarctic intertidal sediment:the microbial response[J]. Microbial Ecology, 2002, 44 (2): 118–126. DOI:10.1007/s00248-001-1047-z |
| [78] | Nikolopoulou M, Kalogerakis N, et al. Biostimulation strategies for fresh and chronically polluted marine environments with petroleum hydrocarbons[J]. Journal of Chemical Technology & Biotechnology, 2010, 84 (6): 802–807. |
| [79] | Berthe-Corti L, Höpner T. Geo-biological aspects of coastal petroleum hydrocarbon pollution[J]. Palaeogeography Palaeoclimatology Palaeoecology, 2005, 219 (1-2): 171–189. DOI:10.1016/j.palaeo.2004.10.020 |
| [80] | Margesin R, Hammerle M, Tscherko D. Microbial activity and community composition during bioremediation of diesel-oil-contaminated soil:effects of hydrocarbon concentration, fertilizers, and incubation time[J]. Microb Ecol, 2007, 53 (2): 259–269. DOI:10.1007/s00248-006-9136-7 |
| [81] | Caldwell SL, Laidler JR, Brewer EA, et al. Anaerobic oxidation of methane:mechanisms, bioenergetics, and the ecology of associated microorganisms[J]. Environ Sci Technol, 2008, 42 (18): 6791–6799. DOI:10.1021/es800120b |