2. 无锡市环境监测中心站, 无锡 214000
2. Wuxi Environmental Monitoring Centre, Wuxi 214000
蓝藻为一种原始而古老的藻类原核微生物,在适宜的水文、气象条件下易在富营养湖泊中大量繁殖而形成蓝藻水华,被认为是影响水质指标的重要因素[1]。许多蓝藻尤其是淡水水体中常见的铜绿微囊藻(Microcystis aeruginosa)会产生以微囊藻毒素为代表的有毒次生代谢产物,对人畜的肝脏及皮肤产生极大伤害,严重危害水体环境以及人类自身身体健康和生命安全[2-4],这也使得蓝藻水华问题成为国内外关注的重要环境问题之一。现今对蓝藻的鉴别方法主要有传统形态学和基于16S rRNA基因的分子生物技术,耗时耗力操作复杂且精度不够[5-7],因此,简便、快速、精确地鉴别蓝藻的方法是非常必要的。
基于质谱分析的蛋白质组学/生物信息学方法——基质辅助激光解吸/电离平衡飞行时间质谱(MALDI-TOF MS)可用来在属、种水平上鉴别微生物[8-10]。蛋白质因其大量丰富且在检测中较好的敏感性和特异性,常被用作生物标识物,尤其是核糖体蛋白质具有管家功能,稳定地高度表达于不同细胞和组织中[11-14],因此与MALDI-TOF MS结合可用于高通量常规分析和临床微生物学[15-17]。
笔者前期研究将此手段引入蓝藻鉴别领域,建立了以13个核糖体蛋白质(RP)为生物标识物的MALDI-TOF MS直接鉴别纯种蓝藻细胞的新方法。蓝藻中约52个核糖体蛋白质的氨基酸序列情报可以反映出不同藻株之间的差异,与目前主流的以16S rRNA基因序列作为生物标识物鉴定方法相比,无需遗传物质的提取扩增,快速、简便、样品用量少,可实现在株的水平上对蓝藻进行鉴定鉴别[18]。本研究在已建立的新方法基础上,以4种优势水华蓝藻按不同比例配置模拟湖泊中混合蓝藻样品,探讨所建立的方法对于从混合蓝藻样品中鉴别出M. aeruginosa的可行性,并分析M. aeruginosa在MALDI-TOF MS法下被检测出的敏感性,并将该方法应用于实际太湖水体中蓝藻的鉴别,旨在为今后蓝藻的日常程序化监测以及对淡水湖泊中的蓝藻爆发提前预警提供参考与依据。
1 材料与方法 1.1 材料 1.1.1 菌株来源本研究人工混合样品所用的四株水华优势蓝藻均来自日本国立环境研究所的藻种保存机构(MCC-NIES,Tsukuba,Japan),为铜绿微囊藻(Microcystis aeruginosa Lemmermann,NIES-843)即模式株、细小平裂藻(Merismopedia tenuissima Lemmermann,NIES-230)、爬行颤藻(Oscillatoria animalis Agardh ex Gomont,NIES-206)和近亲鱼腥藻(Anabaena affinis Lemmermann,NIES-1639);实际蓝藻样品为2016年水华易爆发期5月30日内太湖竺山湖和梅园水厂外采集的水样。
1.1.2 培养基及培养条件NIES-843的培养采用MA培养基,NIES-230为C培养基,NIES-206被培养于MDM琼脂培养基,NIES-1639为CB培养基,各培养基的配方均由MCC-NIES提供[19]。以光:暗比12 h:12 h为循环周期,光照密度15-25 μmol·m-2·s-1以及温度20℃为条件培养3-4周后用作实验。
1.1.3 主要仪器和试剂高速冷冻离心机(德国SIGMA 3-18K型和美国Beckman Allegra 64R型),均质研磨器(美国Biospec Mini Beadbeater-16型),飞行时间质谱仪(日本岛津Axima CFR Plus型)。TMA-1 buffer:Tris-HCl 10 mmol/L(pH 7.8)、NH4Cl 30 mmol/L、MgCl210 mmol/L、2-巯基乙醇6 mmol/L;混合液A:乙腈(ACN)500 μL、三氟乙酸(TFA)10 μL、去离子水500 μL;芥子酸基质溶液:芥子酸(SA)10 mg、500 μL混合液A。
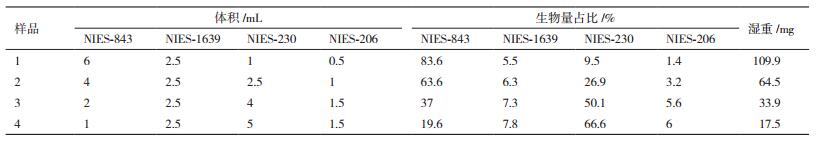
1.2 方法 1.2.1 藻株混合样品的准备4株实验用蓝藻的原始细胞密度见表 1所示,单位为cells/mL。从细胞形态学来说,4株蓝藻具有非常明显的区别,NIES-206和NIES-1639是长链状的,因而其生物量在生物学上经常用volume/mL表达,为了使这4株蓝藻的生物量具有可比性,通过测量单个细胞体积来使生物量转换成volume/mL的表达方式,因此本研究的4个混合样品的成分配比,见表 2。
所测样品只需离心和机械破碎的前处理即可获得70S核糖体亚基:样品做好标记进行超声波轻微除泡,移入15 mL离心管中入离心机离心10 min,操作条件:9 100×g,4℃;去除上清液,取出底部沉淀物转移至1.5 mL离心管内(离心管提前称量)离心10 min,操作条件:20 400×g,4℃;上一步操作后各样品加入160 μL的TMA-1缓冲液混匀,移入提前备好的含160 mg氧化锆-二氧化硅微粒的细胞破碎专用离心管,置入均质研磨器进行机械破碎,操作条件:3 000 r/min,破碎两次,每次45 s,间隔30 s;将机械破碎后的样品移入1.5 mL离心管离心25 min,操作条件:5 800×g,4℃;上述样品取上清液移入1.5 mL离心管高速离心4 h后去上清液,操作条件:64 000×g,4℃,获得核糖体亚单位沉淀物。
1.2.3 蛋白质原位结晶上述制备完成后,即进行点靶操作。在获得核糖体亚单位沉淀物中加入10 μL混合液A,充分混匀,先吸取2 μL该样品滴入专用样品板上,再滴入1.5 μL基质SA溶液覆盖,在空气中自然风干,使蛋白质原位结晶。
1.2.4 MALDI-TOF MS测定质荷比(m/z)设定在2-40 kD的范围内,通过一个配备N2的激光脉冲仪(λ= 337 nm,脉冲宽度为3 ns,频率为10 Hz)的Axima CFR加飞行时间质谱测定仪在正线性模式下得到质谱图。
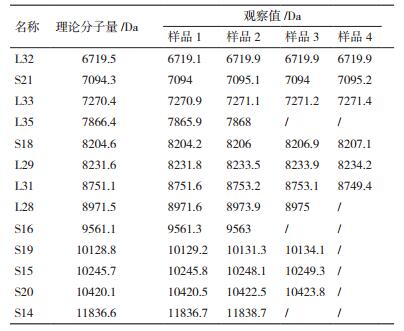
1.2.5 生物信息学分析利用国际数据库检索M. aeruginosa NIES-843的RP氨基酸全序列(http://www.ncbi.NLM.NIH.gov/),并通过蛋白分析专家系统中(http://www.expasy.org/)的序列等电点和分子质量分析软件,考虑“ N端规则”[20],计算出NIES-843的核糖体亚基蛋白的理论值。依据笔者之前的实验,选取其中13个RP作为生物标识物,将MALDI-TOF MS测定得出的质谱图与13个RP的理论值对比分析后鉴别蓝藻的种类。
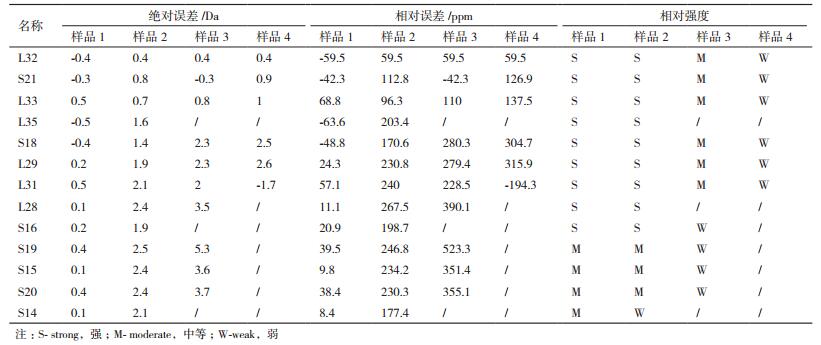
2 结果 2.1 人工混合蓝藻样品中RP的峰值的检测四种人工混合蓝藻样品经MALDI-TOF MS测定后质谱图见图 1。混合样品1和2中都能完全检测出这13个特征RP标识峰,样品3则包含10个特征RP标识峰,检出率为76.9%。而样品4仅能检测到6个特征RP标识峰,检出率为38.5%(表 3)。能从13个RP标识物中测得大于等于10个RP即检出率大于75%定义为能够鉴别该样品,上述结果表明建立的新方法可以在前3个混合蓝藻样品中鉴别出NIES-843,而不能从样品4中鉴别出NIES-843,对应的混合样品中能检测出的NIES-843所占的最低比例为37%。
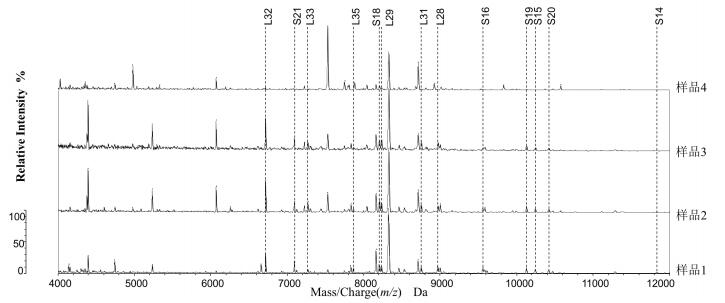
|
| 图 1 四种混合蓝藻样品的质谱图 13个核糖体亚基蛋白如图标注;L:大亚基;S:小亚基 |
随着混合样品中NIES-843配比的降低即从样品1逐步到样品4(表 2),RP标识物的观察值与理论值之间的误差随之相对增加而RP特征峰的相对强度则相应降低(表 4),如样品4只能检测出6个RP特征峰且强度都很弱。同时,从RPL32逐步到RPS14,同种样品的峰值强度也从S19之后有所降低,即当分子量大于10 kD(表 3)时,强度变弱,同时质谱图的信噪比也在随着RP特征峰强度的降低而反向增大(图 1)。上述结果表明随着所测目标蓝藻浓度的减少,测得的质谱图误差增大,稳定性降低,即鉴定效果减弱。
M. aeruginosa NIES-843可以在前3个混合蓝藻样品中被精确地鉴别出来,而不能在样品4中被成功识别,即样品3中NIES-843所含的生物量为新方法对其检测鉴别的最低检出限。最初所用的原始NIES-843的细胞密度为1.44×106 cells/mL,样品3中共使用NIES-843的体积为2 mL,因此对于在人工混合蓝藻样品中使用RP为生物标识物的MALDI-TOF MS法对M. aeruginosa NIES-843的最低检出限为2.88×106cells,相对应的混合样品中NIES-843所占的比例为37%。
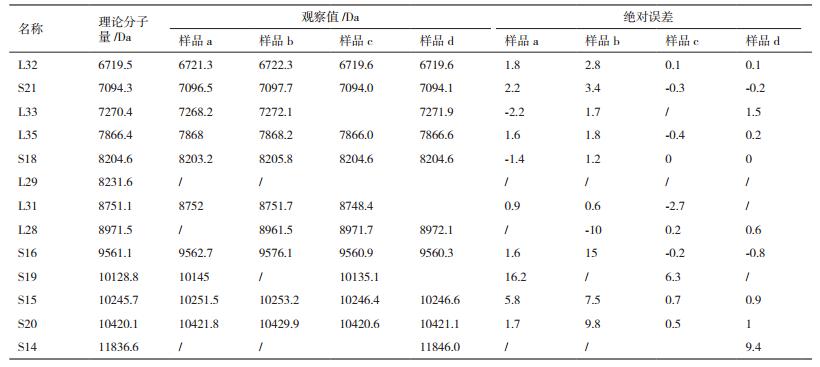
2.4 MALIDI-TOF MS法应用于实际太湖蓝藻实际太湖4个采样点的蓝藻样品a、b、c和d经MALDI-TOF MS测定后质谱图见图 2。四个实际样品中都能检测出这13个特征RP标识峰中的10个,检出率为76.9%。各个样品能检测出的特征峰并不完全相同,如只有样品d能够检测出RPS14(表 5)。依据实验室蓝藻样品能从13个RP标识物中测得大于等于10个RP即检出率大于75%定义为能够鉴别该样品,上述结果表明建立的新方法可以在这两个实际太湖蓝藻样品中鉴别出NIES-843,即新方法可以成功应用于实际湖泊中蓝藻的鉴别。同时太湖样品RP峰值的绝对误差与人工混合样品相比较,实际太湖样品a和b的误差值相对更大一点,而实际太湖样品c和d的误差值比人工混合样品2和样品3更低,甚至与人工混合样品1相当。
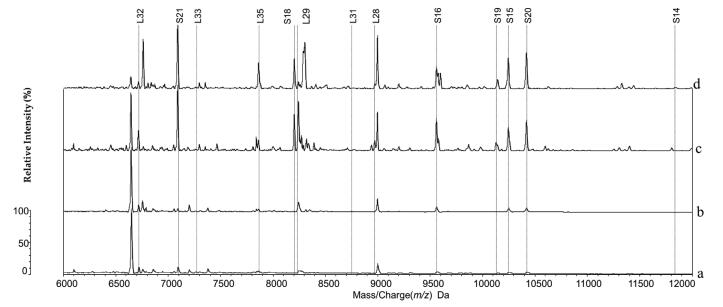
|
| 图 2 实际太湖蓝藻样品的质谱图 13个核糖体亚基蛋白如图标注;L:大亚基;S:小亚基 |
因日本诺贝尔得主田中耕一[21]的研究成果,新型分析化学方法被引入微生物鉴定领域:利用MALDI-TOF MS为手段,对微生物细胞直接测定,根据测定得到的特征波谱,以构成微生物细胞成分的多酯类或蛋白质作为生物标识物,对微生物进行鉴定[22]。但该法在测定微生物得到的波谱中,包括了多种成分的峰值,容易受到微生物的培养条件、测试条件及测试仪器等因素的影响,影响判断的准确性,因此,需要确立不易受上述各因素影响的生物标识物。Ochi和Ochiai[23-24]首先提出各物种都有其独特的RP氨基酸序列及其所决定的分子量大小,与在基因水平上决定着不同物种的16S rRNA基因指标一样,能反映出分子进化过程和亲缘关系,是物种分类的有力指标,可将RP作为鉴定微生物的标识蛋白质;Pineda等[25]也证实了RP作为生物标识物能成功用于细菌的鉴定。迄今为止,MALDI-TOF MS已广泛应用于临床及食品行业的各种微生物的快速、准确识别。
在前期研究中,笔者建立了利用MALDI-TOF MS法鉴别纯种蓝藻的新方法,因M. aeruginosa NIES-843的全基因及RP的氨基酸序列被测出[26],且其作为太湖中占主导地位的有毒蓝藻[27]被确立为模式株,也因此确立了13个特征RP为生物标识物。在此基础上为了分析新方法应用于实际湖泊中蓝藻日常监测鉴别的可行性,本研究首先将实验室中不同蓝藻菌株混合,为了模拟实际样品,藻株选择为实际湖泊中水华爆发时的优势种类;为了分析该方法的敏感性与实际采样体积的合理性,本研究中混合样品按模式株浓度递减设计,得出MALDI-TOF MS对M. aeruginosa的最低检出生物量,并依据其原始浓度计算出所需样品体积。滇池将1×106 cells/mL作为蓝藻水华的5级标准,同时也作为滇池蓝藻水华爆发的判断依据[28]。我国的太湖,目前主要以叶绿素a的浓度来判断蓝藻水华的发生以及发生的等级,通过叶绿色a的浓度与铜绿微囊藻细胞密度的转换,当密度为4.58×104 cells/mL到2.75×105 cells/mL之间常被认为发生蓝藻水华[29]。依据本研究中混合样品检测鉴别的结果与最低检测限,只需3 mL样品即可判别滇池蓝藻爆发,而15-50 mL水样即可判别太湖蓝藻的发生,这在环境监测中是非常合理的采样体积。当然,发生水华的水体中藻类不仅仅包含M. aeruginosa,所以在实际监测中水样的体积要高于计算出的检出体积,且通过离心或过滤的手段,水样的体积能够符合新建方法的预处理步骤。因此,本研究所确立的新方法不仅可以定性地鉴别混合蓝藻样品,而且它的检测灵敏度也能达到预测与预警的要求。在实验室试验成功且合理的情况下,本研究将所建立的新方法用于太湖实际蓝藻样品的鉴别,结果表明,该方法能成功应用于太湖水样中蓝藻的鉴别。
当然该方法还存在一些不足,目前还不能实现完全定量检测,且与实验室混合样品相比,实际蓝藻样品a和b中的误差值更大,原因可能是实际湖泊的水体成分复杂,存在各种干扰物质(如细菌、泥渣、各类杂质等)等,这还有待进一步研究。针对这一现象,首先可以在采样时采用浮游植物网进行采样,如样品c和样品d即采用该采样方法,过滤掉部分干扰物质且提高了蓝藻在水样中的浓度;其次在质谱测定时可以相应增加激光强度和激光击打次数来提高测量精确度和稳定性,因此太湖样品c和d的误差值则与实验室混合样品相当。同时混合样品中所选用的其他菌株的基因还未完全测序,因而将来随着蛋白质数据库的不断完善及发展,越来越多的蓝藻藻株将通过我们建立的MALDI-TOF MS法被检测鉴别出,这也将有助于弄清水华爆发时蓝藻的物种组成与形成机理,为蓝藻的预警预测提供有力依据。
4 结论本研究利用RP为生物标识物结合MALDI-TOF MS成功从实验室混合蓝藻样品中鉴别出目标M. aeruginosa,并得出对其最低生物量检测限及所占的比例,同时将建立的新方法成功应用于实际太湖蓝藻的鉴别,并验证了RP作为生物标识物鉴别蓝藻的可行性。
| [1] | 陈翔, 禹继华, 刘杰, 等. 洪泽湖蓝藻暴发的气象条件分析[J]. 安徽农业科学, 2010, 38(15): 8141–8142,8177. DOI:10.3969/j.issn.0517-6611.2010.15.159 |
| [2] | 甘南琴, 魏念, 宋立荣. 微囊藻毒素生物学功能研究进展[J]. 湖泊科学, 2017, 29(1): 1–8. |
| [3] | Backer LC, Landsberg JH, Miller M, et al. Canine cyanotoxin poisonings in the United States(1920s-2012):Review of suspected and confirmed cases from three data sources[J]. Toxins, 2013, 5 : 1597–1628. DOI:10.3390/toxins5091597 |
| [4] | Paerl, H W, Hall NS, Calandrino ES. Controlling harmful cyanobacterial blooms in a world experiencing anthropogenic and climatic-induced change[J]. Sci Total Environ, 2011, 409 (10): 1739–1745. DOI:10.1016/j.scitotenv.2011.02.001 |
| [5] | 朱冰川, 张军毅, 魏轲, 等. 浮游植物半定量活检法在蓝藻预警监测中的应用[J]. 环境监控与预警, 2011, 3(6): 8–11. |
| [6] | Srivastava A, Singh S, Ahn CY, et al. Monitoring approaches for a toxic cyanobacterial bloom[J]. Environmental Science & Technology, 2013, 47 (16): 8999–9013. |
| [7] | 刘洋, 胡佩茹, 马思三, 等. 实时荧光定量PCR方法检测南太湖入湖口产毒微囊藻[J]. 湖泊科学, 2016, 28(2): 246–252. DOI:10.18307/2016.0202 |
| [8] | Ciccimaro E, Blair IA. Stable-isotope dilution LC-MS for quantitative biomarker analysis[J]. Bioanalysis, 2010, 2 (2): 311–341. DOI:10.4155/bio.09.185 |
| [9] | Nakamura S, Sato H, Tanaka R, et al. Verification of ribosomal proteins of Aspergillus fumigatus for use as biomarkers in MALDI-TOF MS identification[J]. Mass Spectrometry, 2016, 5 (1): A0049. DOI:10.5702/massspectrometry.A0049 |
| [10] | Welker M. Proteomics for routine identification of microorganisms[J]. Proteomics, 2011, 11 (15): 3143–3153. DOI:10.1002/pmic.201100049 |
| [11] | Arnold RJ, Reilly JP. Observation of Escherichia coli ribosomal proteins and their posttranslational modifications by mass spectrometry[J]. Analytical Biochemistry, 1999, 269 (1): 105–112. DOI:10.1006/abio.1998.3077 |
| [12] | Christie-Oleza JA, Miotello G, Armengaud J. Proteogenomic definition of biomarkers for the large Roseobacter clade and application for a quick screening of new environmental isolates[J]. J Proteome Res, 2013, 12 (11): 5331–5339. DOI:10.1021/pr400554e |
| [13] | Sun LW, Kanae T, Hiroaki S, et al. Characterization of ribosomal proteins as biomarkers for matrix-assisted laser desorption/ioniza-tion mass spectral identification of Lactobacillus plantarum[J]. Rapid Communications in Mass Spectrometry, 2006, 20 (24): 3789–3798. DOI:10.1002/(ISSN)1097-0231 |
| [14] | 胡亚, 卢诚, 魏川川, 等. 家蝇核糖体蛋白S18基因的克隆及表达模式研究[J]. 生物技术通报, 2016, 32(6): 135–142. |
| [15] | Kanae T, Hiroaki S, Sun LW, et al. A simple intact protein analysis by MALDI-TOF-MS for characterization of ribosomal proteins of two genome-sequenced lactic acid bacteria and verification of their amino acid sequences[J]. J Proteome Res, 2007, 6 (10): 3899–3907. DOI:10.1021/pr070218l |
| [16] | Croxatto A, Prod'hom G, Greub G. Applications of MALDI-TOF mass spectrometry in clinical diagnostic microbiology[J]. FEMS Microbiology Reviews, 2012, 36 (2): 380–407. DOI:10.1111/j.1574-6976.2011.00298.x |
| [17] | Roda A, Mirasoli M, Roda B, et al. Recent developments in rapid multiplexed bioanalytical methods for foodborne pathogenic bacteria detection[J]. Microchimica Acta, 2012, 178 (1-2): 7–28. DOI:10.1007/s00604-012-0824-3 |
| [18] | Sun LW, Jiang WJ, Sato H, et al. Rapid Classification and Identification of Microcystis aeruginosa strains using MALDI-TOF MS and polygenetic analysis[J]. PLoS One, 2016, 11 (5): e0156275. DOI:10.1371/journal.pone.0156275 |
| [19] | Kasai F, Kawachi M, Erata M, et al. NIES-Collection, List of strains, microalgae and protozoa[M]. 7th ed. Tsukuba: National Institute for Environmental Studies, 2004. |
| [20] | Moerschell RP, Hosokawa Y, Tsunasawa S, et al. The specificities of yeast methionine aminopeptidase and acetylation of aminoterminal methionine in vivo. Processing of altered iso-1-cytochromes c created by oligonucleotide transformation[J]. Journal of Biological Chemistry, 1990, 265 (32): 19638–19643. |
| [21] | Yuuhiko T, Fumie K, Makoto M, et al. Multilocus sequence typing(MLST)reveals high genetic diversity and clonal population structure of the toxic cyanobacterium Microcystis aeruginosa[J]. Microbiology, 2007, 153 (11): 3695–3703. DOI:10.1099/mic.0.2007/010645-0 |
| [22] | Lay JO. MALDI-TOF mass spectrometry of bacteria[J]. Mass Spectrom Rev, 2001, 20 (4): 172–194. DOI:10.1002/(ISSN)1098-2787 |
| [23] | Ochi KJ. Heterogeneity of ribosomal-proteins among Streptomyces species and its application to identification[J]. Microbiology, 1989, 135 (10): 2635–2642. DOI:10.1099/00221287-135-10-2635 |
| [24] | Ochiai K, Uchida K, Kawamoto I. Similarity of ribosomal proteins studied by two-dimesional coetrophoresis for identification of gram-positive bacteria[J]. International Journal of Systematic Bacteriology, 1993, 43 (1): 69–76. DOI:10.1099/00207713-43-1-69 |
| [25] | Pineda FJ, Antoine MD, Demirev PA, et al. Microorganisms identification by matrix-assisted laser/desorption ionization mass spectrometry and model-derived ribosomal proteins biomarkers[J]. Analytical Chemistry, 2003, 15 : 3817–3822. |
| [26] | Takakazu K, Nobuyoshi N, Shinobu O, et al. Complete genomic structure of the bloom-forming toxic cyanobacterium Microcystis aeruginosa NIES-843[J]. DNA Research, 2007, 6 : 247–256. |
| [27] | 施丽梅, 蔡元锋, 孔繁翔, 等. 水华衰亡过程中微囊藻群体胶鞘结构及其元素组成的变化[J]. 湖泊科学, 2016, 28(5): 944–950. DOI:10.18307/2016.0503 |
| [28] | 李颖, 施择, 张榆霞, 等. 关于用藻密度对蓝藻水华程度进行分级评价的方法和运用[J]. 环境与可持续发展, 2014, 39(2): 67–68. |
| [29] | 于海燕, 周斌, 胡尊英, 等. 生物监测中叶绿素a浓度与藻类密度的关联性研究[J]. 中国环境监测, 2009(6): 40–43. |