2. 华东理工大学上海生物制造技术协同创新中心, 上海 200237
2. Shanghai Collaborative Innovation Center for Biomanufacturing, East China University of Science and Technology, Shanghai 200237
随着现代生物学DNA合成技术的飞速进步,大规模合成复杂代谢通路乃至微生物基因组成为现实,进而有力地推动了合成生物学的发展[1, 2]。利用人工设计构建的微生物作为细胞工厂进行化学品的绿色高效生产,有望为解决人类发展面临的资源、能源、健康、环境等若干重大问题提供解决方案。正因为如此,合成生物学在世界范围内得到了越来越广泛的关注,并成为生物技术的前沿学科。自从2006年加州大学伯克利分校Keasling教授研究组利用人工代谢途径在酵母体内合成青蒿酸以来[3],合成生物学已经在药物分子、生物能源、功能材料、保健食品等多种重要产品的合成方面取得重要进展。截至到2015年,全世界至少已有81家公司或研究机构的116种合成生物学产品得到开发应用,总产值高达52亿美元[4],预期2025年这一产值将增长到1 000亿美元[5]。
然而,由于人们对复杂生物系统的认识还非常有限,使得构建人工生物系统面临着巨大的困难:对生物元件的性能表征和机制解析不足,导致由多种蛋白质分子形成的装置和作用网络行为难以预测,而当这些装置和网络在细胞中发挥功能时,其行为又会受到细胞内多种环境因素的影响,使得人工生物体系往往无法按照预先的行为进行工作,严重限制了合成生物学愿景的实现。为解决这一问题,科学家们尝试通过定向进化技术来创造符合预期功能的微生物菌株。相比于常规的理性设计方法,定向进化不依赖于对生物系统的深入认识,而是模拟自然界达尔文进化原理,首先在实验室中构建大容量随机突变库,然后在施加人工选择压力的条件下,从库中筛选出极少数符合预期表型的个体,已经在多种酶基因、代谢途径、乃至微生物的改造中取得了成功[6-9],但定向进化成功的关键在于筛选方法的效率和准确性。常规筛选方法 (如摇瓶筛选、孔板筛选等) 存在通量较低、费时费力等缺点,获得一株具有商业价值的生产菌株往往需要花费巨额资金成本,同时消耗大量不同领域技术人员的劳动力[9],随着人们对化学品绿色制造效率的要求越来越高、种类越来越复杂,这种高投入、高成本、低效的开发策略越来越难以满足需求。
微流控芯片 (microfluidic chip) 技术,又称为芯片实验室 (Lab-on-a-chip) 或微全分析系统 (miniaturized total analysis systems) ,是指通过微加工技术将常规实验室的样品制备、反应、分离、检测等基本操作单元集成在只有几平方厘米尺寸的芯片中,从而实现微型化、自动化和集成化的技术体系。美国《Business2.0》杂志将微流控技术列为“改变世界”的七大技术之一,《Nature》杂志也称这一技术可能成为“这一世纪的技术”[10]。微流控芯片技术的发展所催生的液滴微流控 (droplet microfluidics) 筛选技术以其超高通量、高灵敏度、定量准确的功能,为合成生物学中催化元件、代谢途径乃至菌株的筛选问题的解决提供了良好的解决方案。本文综述了液滴微流控筛选技术在合成生物学领域应用的主要进展,重点关注了微反应器中目的产物分子与荧光信号偶联的策略这一核心问题,并介绍了近年来液滴微流控筛选技术在催化元件 (酶) 和代谢途径的优化改造方面的应用实例,对未来的发展趋势进行了展望。
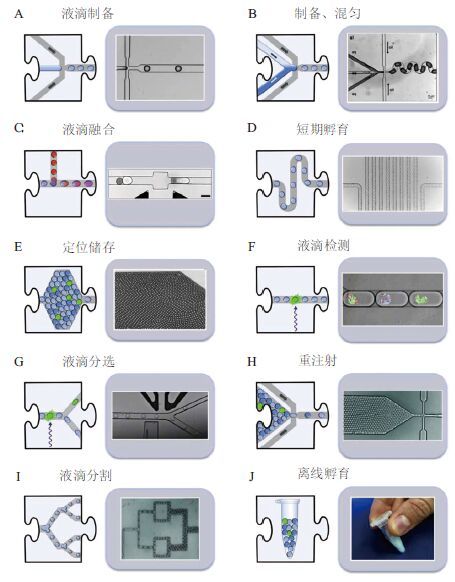
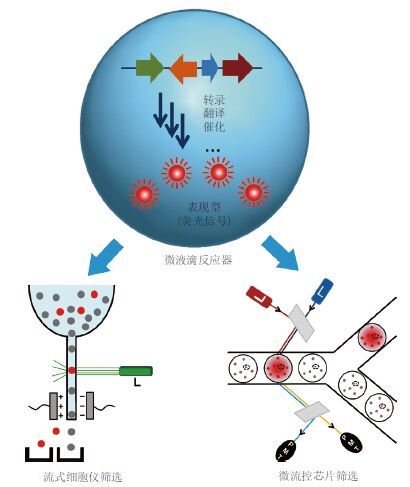
1 液滴微流控体系筛选技术原理液滴微流控技术是微流控芯片技术领域的一个重要发展方向,其原理是通过特殊的微通道设计,对微米级尺度的微液滴 (粒度CV值低于3%) 进行大量制备、混匀、融合、分割、孵育、检测、分选等操作 (图 1) ,从而完成一系列复杂的样品处理过程[11]。由于微液滴良好的生物相容性,可以作为微反应器进行细胞扩增、恒化培养、细胞裂解、DNA扩增、体外蛋白质表达、酶反应等操作,为合成生物学定向进化的筛选通量问题的解决提供了契机。在一个典型的液滴微流控的筛选体系中,科学家能利用皮升级 (pL) 体积的微液滴作为反应器,将酶基因、人工构建的代谢途径、基因表达产物等包裹在其中,利用特定的荧光探针对其进行显色 (图 2) ,再借助于商品化的流式细胞仪或微流控检测/分选芯片,对这些微液滴进行高效的分析与分选[12-14]。由于这种技术体系可以在极高的速度下工作,其筛选通量高达108/d,显著地提高了人们对生物体系进行高通量筛选的能力,为设计高效的酶、代谢途径乃至微生物菌株提供了有力的工具。
|
| 图 2 基于液滴微流控的超高通量筛选体系原理图 |
与常规分析体系相比,基于液滴微流控的高通量筛选体系具有两个独特的优势: (1) 极大提升了人们对于大规模样品的分析能力,微流控体系处理微液滴的速度可高达108/d,在合成生物学家进行催化元件、代谢途径乃至菌株的高通量筛选时,可以极大地提升效率; (2) 由于每个微液滴的体积只有皮升级,相比于常规的微升至毫升级反应体系缩小了百万倍以上,对试剂的需求量大大降低,在一些昂贵样品的分析中具有更加明显的优势。
2 微液滴筛选的荧光偶联策略现有的液滴微流控筛选体系主要使用荧光信号来对代谢产物进行定量,这主要是由于荧光检测具有很高的灵敏度,非常适合作为皮升级微反应器的检测信号。因此利用液滴微流控技术进行高通量筛选,首先需要解决的一个关键问题是将需要分析的生物活性转换为可检测的荧光信号,这是开发液滴微流控筛选系统所面临的核心问题。目前常用的荧光信号进行偶联的策略主要有以下几种 (图 3) 。

|
|
图 3 液滴微流控体系的荧光信号偶联策略
A:荧光底物酶活性检测;B:酶联荧光探针偶联代谢产物检测;C:生物传感器代谢产物检测; D:氧化酶偶联的酶联荧光检测体系;E:还原酶偶联的荧光检测体系 |
荧光底物是探测酶活性的灵敏手段,其本身无荧光或只有很低的荧光信号,而其酶促反应产物则具有很强的荧光信号,可以根据反应前后荧光强度的变化对酶活性进行定量分析 (图 3-A) 。目前已经有多种商品化荧光底物可用于酶活性检测,包括糖苷酶[15, 16]、酯酶/脂肪酶[17]、蛋白酶[18]、过氧化物酶[19]、巯基内酯酶[20]等。除此之外,也有人利用荧光共振能量转移原理 (FRET) 设计了荧光探针,利用酶反应前后底物荧光波长的强烈变化来探测酶活性,已经在蛋白酶[21-23]、酯酶/脂肪酶[24]、β-内酰胺酶[25]、神经节苷脂降解酶[26]等酶活性中得到了应用。
目前绝大多数荧光底物都是为宏观体系下比色皿中或96孔板中的酶活性检测所开发的,大都是基于荧光素、香豆素、试卤灵等疏水性较强荧光基团的衍生物。然而,在用于微液滴体系中的时候,由于微液滴的油水相界面并不是严格的物理隔离,这些疏水的荧光基团在微液滴中保留性较差,会快速扩散到油相中[27, 28]或通过临界胶束传递的方式在临近微液滴之间快速扩散[29, 30],造成不同反应体系的交叉干扰,影响酶活性检测的准确性。为解决这一问题,研究者们通过引入极性较强的磺酸基或羧基修饰,大大提高了荧光信号在微液滴中的稳定性,使香豆素在微液滴中泄漏的半衰期从2.1 s提高到2.8 d以上。目前这种策略已经成功用于氧化酶[31]、内酰胺酶[32]及葡萄糖苷酶[33]的荧光底物设计。近期,本课题组又进一步提出了液滴荧光捕获 (Fluorescence droplet entrapment,FDE) 的底物设计策略,提出自身具有较高疏水性、但在酶反应后会释放高亲水性荧光基团的底物可能更加适合微液滴体系的酶活性检测[34]。具有FDE效应的底物可以通过油相扩散到微液滴中进行酶反应,而产生的强亲水性荧光产物具有较好的液滴保留稳定性,因此能够同时增加酶反应可控性并降低荧光背景,从而实现更加准确的酶活性定量。基于FDE荧光底物设计策略,我们已经提出了包括糖苷酶、蛋白酶、酯酶、脂肪酶等重要酶活性检测的荧光底物结构,有望为一系列重要酶活性的液滴微流控高效筛选方法建立铺平道路[34]。
2.2 酶联荧光探针偶联代谢产物检测许多重要的小分子代谢产物都无法直接通过荧光进行检测,成为利用液滴微流控体系进行代谢途径和微生物菌株筛选的瓶颈。利用多酶级联反应可以间接将这些小分子物质转换为荧光信号,为解决这一问题提供有效途径。典型的策略是通过特异性识别目标代谢产物的氧化还原酶,可以催化目标化合物的氧化反应生成过氧化氢或NADH,再利用相应的偶联酶及荧光探针将过氧化氢或NADH转化为荧光信号,从而实现目标代谢产物的荧光偶联 (图 3-B) 。例如,Hammar等[35]采用乳酸脱氢酶将乳酸氧化为丙酮酸,同时将NAD+还原为NADH,再利用商品化的NADH荧光显色试剂盒 (Cayman Chemical公司生产) 将NADH转化为荧光信号 (图 3-E) ,并用于从乳酸产生军的紫外诱变库中筛选高产菌株;Wang等[36]采用乳酸氧化酶将乳酸氧化生成过氧化氢,再利用过氧化物酶 (如辣根过氧化物酶) 和过氧化氢相应的荧光探针 (如Invitrogen公司的Amplex UltraRed 探针) 将过氧化氢转化为红色荧光物质试卤灵 (resorufin) (图 3-D) ,作者利用该显色方法能够将极少量的产乳酸细胞从大量背景细胞中有效富集;类似地,Abalde-Cela等[37]采用乙醇氧化酶将乙醇氧化生成过氧化氢,再进一步将过氧化氢转化为试卤灵,利用该体系也能够将产乙醇菌株从大量背景细胞中有效富集。目前,多酶偶联策略已经可以实现对微反应器中的乳酸[35, 36]、乙醇[37]、木糖[36]、葡萄糖[38, 39]等多种产物进行定量。随着更多专一性氧化酶的开发和利用,这一体系有潜力扩展至各种糖类、氨基酸、脂类及激素类代谢产物的定量,从而为多种重要代谢途径和菌株定向进化提供有效的筛选方法。
2.3 生物传感器代谢产物检测利用微生物自身的遗传调控机制来感应代谢产物的技术,称为生物传感器 (biosensors) [9]。转录调控因子 (transcriptional regulator,TR) 是应用十分广泛的生物传感器,由一系列调控DNA序列及其编码的调控蛋白组成,DNA序列本身或其编码的蛋白质能够响应特定的代谢产物,当没有代谢产物存在时,由于调控序列本身的特殊二级结构或调控蛋白与DNA结合而阻遏了下游基因的表达;有代谢产物存在时,代谢产物与调控序列或调控蛋白结合,破坏原有的DNA二级结构或调控蛋白构象,进而解除对其下游基因表达的阻遏。例如,常见的乳糖操纵子,由该操纵子编码的阻遏蛋白能够与启动子序列结合,阻遏下游蛋白的转录翻译,当阻遏蛋白与乳糖结合后发生构象改变,释放出启动子序列,最终启动下游蛋白的表达[40]。微生物体内存在各种不同类型的转录调控因子,因而能够为不同代谢分子的荧光检测提供丰富的工具[41-43]。另一种生物传感器是基于mRNA水平调控的核糖体开关 (riboswitch) ,核糖体开关是位于RNA 5'末端的非翻译区序列,能够形成二级结构,阻碍其下游的核糖体结合位点 (ribosome-binding site,rbs) 与核糖体的结合,进而阻碍下游基因的翻译;当核糖体开关与特定的代谢分子结合后,二级结构发生变化,释放出核糖体结合位点,最终解除对下游基因翻译的抑制[44, 45]。将荧光蛋白基因置于生物传感器调控序列的下游,可以通过荧光蛋白的表达量来反映出代谢产物的浓度,从而能够实现目标产物与荧光信号的偶联 (图 3-C) ,结合流式细胞仪的FACS功能,可以分选出荧光信号强、目标产物产量高的菌株,该策略能够在催化元件性能提升[46, 47]以及高产菌株鉴定[48, 49]中发挥重要作用。例如,Cheng等[46]利用精氨酸响应的生物传感器,提高了精氨酸脱亚胺酶的活性及对底物的亲和力,突变体能够催化在生理环境下浓度极低的精氨酸,对肿瘤细胞生长具有显著的抑制作用;Siedler等[47]利用能够响应NADPH的SoxR传感器,建立了NADPH依赖型酶的筛选体系,并运用该体系提高NADPH依赖型乙醇脱氢酶的催化活性;Binder等[49]采用来源于Corynebacterium glutamicum的传感器LysG来检测赖氨酸的浓度,通过FACS筛选从宿主全基因组突变库中获得了L-赖氨酸的高产菌株并进一步鉴定出了相关的突变基因型。最近,清华大学张翀、邢新会教授课题组开发了中间产物传感器辅助的推-拉策略 (intermediate sensor-assisted push-pull strategy) ,作者采用L-色氨酸生物传感器来探测脱氧紫色杆菌素 (deoxyviolacein) 生物合成途径的重要中间体——L-色氨酸,利用该体系分别对中间体前后两个合成模块进行优化,首先通过筛选提高L-色氨酸的产量,然后筛选L-色氨酸高转化率的菌株,最终成功将脱氧紫色杆菌素的产量提高了4.4倍[50]。
2.4 其他荧光偶联策略除上述列举的荧光偶联策略,人们还针对一些特殊的应用开发了更加多样化的荧光偶联策略。Pitzler等[51]针对肌醇六磷酸酶 (phytase) 的活性设计了基于荧光凝胶的毛-核 (fur-shell) 荧光偶联方法。该方法利用酶偶联反应产生的羟基自由基引发poly (ethyleneglycol) -acrylate的聚合反应 (Fenton’s reaction) ,同时将荧光分子嵌合在聚合物中,从而以宿主细胞为核心,形成一层荧光凝胶的“毛”供检测。Larsen等[52]开发了DNA聚合酶的通用性酶活性荧光偶联策略:他们通过将单链DNA模板的5'末端共价连接荧光基团,再合成与5'端互补的寡核苷酸并连接上荧光淬灭基团,形成淬灭子 (DNA-quencher) 。在DNA复制之前,荧光基团处于淬灭状态,在聚合酶链式反应后,淬灭子与模板的结合被新合成的DNA片段取代,释放出荧光信号,荧光信号的强度反映了目标酶的催化效率。此外,Fischlechner等[53]开发了凝胶壳微球 (gel-shell beads) 的技术来实现常规微液滴的荧光信号偶联功能;Woronoff等[54]开发了活性调节翻译 (activity-fed translation,AFT) 的酶活性荧光偶联策略,酶催化产生的产物能够启动微反应器中无细胞转录翻译 (in vitro transcription and translation,IVTT) 体系,进而合成反映酶活性高低的荧光蛋白。新的荧光偶联策略的开发,将不断扩展液滴微流控技术在合成生物学领域的应用范围。
3 液滴微流控技术在催化元件筛选中的应用由各类酶分子构成催化网络为细胞提供生理活动所需的能量、合成或降解其他生物分子、传递和处理各种信号等,是构成复杂生命过程的基础[55]。利用液滴微流控技术进行高通量筛选,可以助力定向进化或宏基因组技术获得符合预期性质的新酶资源,为合成生物学提供优良的催化元件。
2010年,Agresti等[19]率先利用液滴微流控体系对辣根过氧化物酶进行了定向进化,通过对107个展示在酵母细胞表面的突变体进行筛选,成功将初始催化效率已经很高的商品化酶活性又提高了10倍。整个进化历程筛选时间累计只有10 h,比常规的孔板法提高1 000倍;消耗试剂总体积150 μL,比孔板法降低100万倍以上,充分显示出液滴微流控体系作为新一代超高通量筛选体系的巨大优势。之后,来自不同研究组的科研人员进一步对液滴微流控超高通量筛选系统进行了一系列的技术改进和体系优化,Kintses等[56]开发了微反应器中的细胞破碎技术,将单细胞和细胞裂解试剂同时包裹进微反应器,从而使该系统可以用于胞内酶的检测筛选;Hosokawa等[57]和Colin等[58]分别将该系统扩展到从宏基因组中发掘新酶资源,鉴定出了一系列新酶基因;本课题组与美国密歇根大学Katsuo Kurabayashi教授课题组合作,建立了双色荧光液滴微流控筛选体系,可以同时检测酶对两种荧光底物的催化活性,进而可以对酶的立体选择性、区位选择性、底物特异性等复杂性质进行改造。应用该体系,我们成功将嗜热酯酶对S型异丙基布洛芬底物的立体选择性提高了700倍 (Ma,Yang等,未发表) 。
目前,利用液滴微流控体系已成功对各类酶的不同性质进行了改造。表 1总结了利用液滴微流控技术进行酶性质改造或从宏基因组中发掘新酶资源的实例 (截至2016年9月) 。
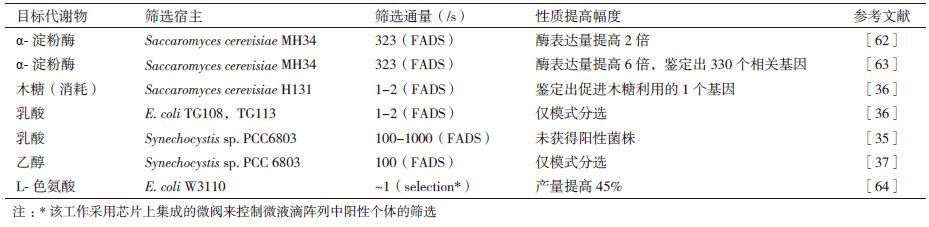
液滴微流控技术还可以对细胞所产生的特定代谢产物进行定量分析与筛选 (图 3-B) ,因此在人工代谢途径和微生物菌株的定向进化中都具有重要的应用。更重要的是,由于微反应器具有对单细胞进行区室化的功能,不同基因型的细胞可以在微反应器中独立地进行生理活动,产生的分泌型代谢产物在各自的微反应器中积累而不会相互干扰,因此液滴微流控技术特别适用于对分泌型代谢产物进行精确的定量,比常规的单细胞生物传感器具有更为广阔的应用范围。如在重要工业酶蛋白的生产菌株产量优化方面,Sjostrom等[62]对表达α-淀粉酶的酵母菌株进行了紫外诱变,并利用液滴微流控技术进行了筛选,最终获得了α-淀粉酶表达量提高两倍的稳定高产菌株。在此基础上,Huang等[63]通过进一步筛选获得了表达量提高6倍的菌株,并结合全基因组超高通量测序技术,鉴定出了突变菌株基因组中与蛋白高分泌表达相关的330个基因。这将有助于加深人们对酵母中蛋白质合成与分泌的生理生化机制的进一步理解,进而促进高效蛋白质合成途径的理性构建。在对分泌型小分子代谢产物的表达量进行定量和筛选方面,Wang等[36]分别利用液滴微流控筛选平台开发了针对酵母菌株胞外木糖消耗能力和大肠肝菌分泌生产乳酸能力的筛选方法。该技术平台首先将单细胞工程菌连同培养基包裹在微反应器中,不同的微反应器经过2 d的孵育进行细胞培养,呈现了不同的营养物质消耗及代谢产物分泌状态。通过与包含荧光探针的检测试剂进行融合,能够对微反应器中木糖、乳酸等目标检测物进行定量分析,帮助筛选得到具有符合预期生物活性的菌株。作者利用该体系,成功地鉴定出了决定木糖高消耗性状的关键基因型[36],并为进行其它小分子代谢产物的检测与筛选指明了方向。
表 2总结了利用液滴微流控技术进行代谢途径或全基因组定向进化的实例 (截至2016年9月) 。值得注意的是目前液滴微流控技术在合成生物学筛选方面的应用主要集中于方法体系的建立,而对代谢通路进行实际筛选的实例仍较少。随着该体系的进一步成熟,将会产生越来越多的合成生物学应用实例。
液滴微流控筛选体系以其超高通量、定量准确、试剂消耗低等优点,在酶催化元件、代谢途径、乃至菌株的定向进化方面体现了重要的应用潜力,推动了合成生物学的持续发展。近年来,多学科交叉技术的汇聚和融合,也使得液滴微流控筛选体系进一步向纵深发展,如目前已可以在微流控芯片上进行DNA片段组装、宿主细胞转化、细胞培养、重组蛋白表达等复杂操作,这一方面大大减少了工作量,更有利于提高实验结果的重现性,提升工作效率。另一方面,科学家们也尝试将其它非标记的检测手段整合到液滴微流控体系中,以进一步扩展筛选体系的通用性,如质谱 (mass spectrometry) [65, 66]、表面增强拉曼光谱 (surface enhanced Raman spectroscopy,SERS) [67]、小角度X射线衍射 (small angle X-ray scattering,SAXS) [68]、荧光偏振 (fluorescence anisotropy) [69]、荧光极化 (fluorescence polarization) [70]、毛细管电泳 (capillary electrophoresis) [71]等,均已在微流控芯片的检测上取得相当大的进展。可以预见,随着这些新技术体系的发展成熟,液滴微流控筛选将在合成生物学中发挥越来越大的作用,从而极大地加速生物能源、生物材料、平台化学品、天然产物等重要产品的品种创新与高效制造。
| [1] | Lartigue C, Glass JI, Alperovich N, et al. Genome transplantation in bacteria:Changing one species to another. Science, 2007, 317 (5838): 632–638. DOI:10.1126/science.1144622 |
| [2] | Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 2014, 344 (6179): 55–58. DOI:10.1126/science.1249252 |
| [3] | Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440 (7086): 940–943. DOI:10.1038/nature04640 |
| [4] | 陈大明, 刘晓, 毛开云, 等. 合成生物学应用产品开发现状与趋势. 中国生物工程杂志, 2016, 7: 117–126. |
| [5] | Manyika J, Chui M, Bughin J, et al. Disruptive technologies:Advances that will transform life, business, and the global economy[M]. McKinsey Global Institute San Francisco, CA, 2013. |
| [6] | Denard CA, Ren H, Zhao H. Improving and repurposing biocatalysts via directed evolution. Current Opinion in Chemical Biology, 2015, 25 : 55–64. DOI:10.1016/j.cbpa.2014.12.036 |
| [7] | Currin A, Swainston N, Day PJ, et al. Synthetic biology for the directed evolution of protein biocatalysts:Navigating sequence space intelligently. Chemical Society Reviews, 2014, 44 (5): 1172–239. |
| [8] | Tizei PAG, Csibra E, Torres L, et al. Selection platforms for directed evolution in synthetic biology. Biochemical Society Transactions, 2016, 44 (4): 1165–1175. DOI:10.1042/BST20160076 |
| [9] | Williams TC, Pretorius IS, Paulsen IT. Synthetic evolution of metabolic productivity using biosensors. Trends in biotechnology, 2016, 34 (5): 371–381. DOI:10.1016/j.tibtech.2016.02.002 |
| [10] | 林炳承, 秦建华. 图解微流控芯片实验室[M]. 北京: 科学出版社, 2008. |
| [11] | Kintses B, Vliet LDV, Devenish SR, et al. Microfluidic droplets:New integrated workflows for biological experiments. Current Opinion in Chemical Biology, 2010, 14 (5): 548–555. DOI:10.1016/j.cbpa.2010.08.013 |
| [12] | Wen N, Zhao Z, Fan B, et al. Development of droplet microfluidics enabling high-throughput single-cell analysis. Molecules, 2016, 21 (7): E881. DOI:10.3390/molecules21070881 |
| [13] | Kaminski TS, Scheler O, Garstecki P. Droplet microfluidics for microbiology:Techniques, applications and challenges. Lab on A Chip, 2016, 16 (12): 2168–2187. DOI:10.1039/C6LC00367B |
| [14] | Chou WL, Lee PY, Yang CL, et al. Recent advances in applications of droplet microfluidics. Micromachines, 2015, 6 (9): 1249–1271. DOI:10.3390/mi6091249 |
| [15] | Mastrobattista E, Taly V, Chanudet E, et al. High-throughput screening of enzyme libraries:In vitro evolution of a β-galactosidase by fluorescence-activated sorting of double emulsions. Chemistry & Biology, 2005, 12 (12): 1291–1300. |
| [16] | Hardiman E, Gibbs M, Reeves R, et al. Directed evolution of a thermophilic β-glucosidase for cellulosic bioethanol production. Applied Biochemistry and Biotechnology, 2010, 161 (1-8): 301–312. DOI:10.1007/s12010-009-8794-6 |
| [17] | Ma F, Xie Y, Huang C, et al. An improved single cell ultrahigh throughput screening method based on in vitro compartmentalization. PLoS One, 2014, 9 (2): e89785. DOI:10.1371/journal.pone.0089785 |
| [18] | Tu R, Martinez R, Prodanovic R, et al. A flow cytometry-based screening system for directed evolution of proteases. Journal of Biomolecular Screening, 2011, 16 (3): 285–294. DOI:10.1177/1087057110396361 |
| [19] | Agresti JJ, Antipov E, Abate AR, et al. Ultrahigh-throughput screening in drop-based microfluidics for directed evolution. Proceedings of the National Academy of Sciences, 2010, 107 (9): 4004–4009. DOI:10.1073/pnas.0910781107 |
| [20] | Aharoni A, Amitai G, Bernath K, et al. High-throughput screening of enzyme libraries:Thiolactonases evolved by fluorescence-activated sorting of single cells in emulsion compartments. Chemistry & Biology, 2005, 12 (12): 1281–1289. |
| [21] | Varadarajan N, Gam J, Olsen MJ, et al. Engineering of protease variants exhibiting high catalytic activity and exquisite substrate selectivity. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102 (19): 6855–6860. DOI:10.1073/pnas.0500063102 |
| [22] | Varadarajan N, Rodriguez S, Hwang BY, et al. Highly active and selective endopeptidases with programmed substrate specificities. Nature Chemical Biology, 2008, 4 (5): 290–294. DOI:10.1038/nchembio.80 |
| [23] | Varadarajan N, Georgiou G, Iverson BL, et al. An engineered protease that cleaves specifically after sulfated tyrosine. Angewandte Chemie, 2007, 47 (41): 7861–7863. |
| [24] | Yang Y, Babiak P, Reymond JL. Low background fret-substrates for lipases and esterases suitable for high-throughput screening under basic(pH 11)conditions. Organic & Biomolecular Chemistry, 2006, 4 (9): 1746–1754. |
| [25] | Mizukami S, Watanabe S, Hori Y, et al. Covalent protein labeling based on noncatalytic β-lactamase and a designed fret substrate. Journal of the American Chemical Society, 2009, 131 (14): 5016–5017. DOI:10.1021/ja8082285 |
| [26] | Yang GY, Li C, Fischer M, et al. A fret probe for cell-based imaging of ganglioside-processing enzyme activity and high-throughput screening. Angewandte Chemie International Edition in English, 2015, 54 (18): 5389–5893. DOI:10.1002/anie.201411747 |
| [27] | Bjerregaard S, Pedersen H, Vedstesen H, et al. Parenteral water/oil emulsions containing hydrophilic compounds with enhanced in vivo retention:Formulation, rheological characterisation and study of in vivo fate using whole body gamma-scintigraphy. International Journal of Pharmaceutics, 2001, 215 (1): 13–27. |
| [28] | Pays K, Giermanska-Kahn J, Pouligny B, et al. Double emulsions:How does release occur?. Journal of Controlled Release, 2002, 79 (1): 193–205. |
| [29] | Cheng J, Chen JF, Zhao M, et al. Transport of ions through the oil phase of w 1/o/w 2 double emulsions. Journal of Colloid and Interface Science, 2007, 305 (1): 175–182. DOI:10.1016/j.jcis.2006.09.055 |
| [30] | Wen L, Papadopoulos KD. Visualization of water transport in w 1/o/w 2 emulsions. Colloids and surfaces A:Physicochemical and Engineering Aspects, 2000, 174 (1): 159–167. |
| [31] | Prodanovic R, Ostafe R, Blanusa M, et al. Vanadium bromoperoxi-dase-coupled fluorescent assay for flow cytometry sorting of glucose oxidase gene libraries in double emulsions. Analytical and Bioanalytical Chemistry, 2012, 404 (5): 1439–1447. DOI:10.1007/s00216-012-6234-x |
| [32] | Woronoff G, El Harrak A, Mayot E, et al. New generation of amino coumarin methyl sulfonate-based fluorogenic substrates for amidase assays in droplet-based microfluidic applications. Analytical Chemistry, 2011, 83 (8): 2852–2857. DOI:10.1021/ac200373n |
| [33] | Najah M, Mayot E, Mahendra-Wijaya IP, et al. New glycosidase substrates for droplet-based microfluidic screening. Analytical Chemistry, 2013, 85 (20): 9807–9814. DOI:10.1021/ac4022709 |
| [34] | Ma F, Fischer M, Han Y, et al. Substrate engineering enabling fluorescence droplet entrapment for ivc-facs based ultrahigh-throughput screening. Analytical Chemistry, 2016, 88 (17): 8587–8595. DOI:10.1021/acs.analchem.6b01712 |
| [35] | Hammar P, Angermayr SA, Sjostrom SL, et al. Single-cell screening of photosynthetic growth and lactate production by cyanobacteria. Biotechnology for Biofuels, 2015, 8 (1): 193–201. DOI:10.1186/s13068-015-0380-2 |
| [36] | Wang BL, Ghaderi A, Zhou H, et al. Microfluidic high-throughput culturing of single cells for selection based on extracellular metabolite production or consumption. Nature Biotechnology, 2014, 32 (5): 473–478. DOI:10.1038/nbt.2857 |
| [37] | Abalde-Cela S, Gould A, Liu X, et al. High-throughput detection of ethanol-producing cyanobacteria in a microdroplet platform. Journal of The Royal Society Interface, 2015, 12 (106): 20150216. DOI:10.1098/rsif.2015.0216 |
| [38] | Ostafe R, Prodanovic R, Nazor J, et al. Ultra-high-throughput screening method for the directed evolution of glucose oxidase. Chemistry & Biology, 2014, 21 (3): 414–421. |
| [39] | Ostafe R, Prodanovic R, Ung WL, et al. A high-throughput cellulase screening system based on droplet microfluidics. Biomicrofluidics, 2014, 8 (4): 041102. DOI:10.1063/1.4886771 |
| [40] | The lac operon:A short history of a genetic paradigm[M]. Walter de Gruyter, 1996. |
| [41] | Gallegos MT, Schleif R, Bairoch A, et al. Arac/xyls family of transcriptional regulators. Microbiology & Molecular Biology Reviews Mmbr, 1997, 61 (4): 393–410. |
| [42] | Ramos JL, Martínezbueno M, Molinahenares AJ, et al. The tetr family of transcriptional repressors. Microbiology & Molecular Biology Reviews, 2005, 69 (2): 326–356. |
| [43] | Tropel D, Jr VDM. Bacterial transcriptional regulators for degradation pathways of aromatic compounds. Microbiology & Molecular Biology Reviews, 2004, 68 (3): 474–500. |
| [44] | Link KH, Breaker RR. Engineering ligand-responsive gene-control elements:Lessons learned from natural riboswitches. Gene Therapy, 2009, 16 (10): 1189–1201. DOI:10.1038/gt.2009.81 |
| [45] | Sudarsan N, Wickiser JK, Nakamura S, et al. An mrna structure in bacteria that controls gene expression by binding lysine. Genes & Development, 2003, 17 (21): 2688–2697. |
| [46] | Cheng F, Kardashliev T, Pitzler C, et al. A competitive flow cytometry screening system for directed evolution of therapeutic enzyme. Acs Synthetic Biology, 2015, 4 (7): 768–775. DOI:10.1021/sb500343g |
| [47] | Siedler S, Schendzielorz G, Binder S, et al. Soxr as a single-cell biosensor for nadph-consuming enzymes in Escherichia coli. Acs Synthetic Biology, 2014, 3 (1): 41–47. DOI:10.1021/sb400110j |
| [48] | Schallmey M, Frunzke J, Eggeling L, et al. Looking for the pick of the bunch:High-throughput screening of producing microorganisms with biosensors[J]. Current Opinion in Biotechnology, 2014, 26C(26C):148-154. |
| [49] | Binder S, Schendzielorz G, St?bler N, et al. A high-throughput approach to identify genomic variants of bacterial metabolite producers at the single-cell level. Genome Biology, 2012, 13 (5): 405–413. |
| [50] | Fang M, Wang T, Chong Z, et al. Intermediate-sensor assisted push-pull strategy and its application in heterologous deoxyviolacein production in Escherichia coli. Metabolic Engineering, 2015, 33 : 41–51. |
| [51] | Pitzler C, Wirtz G, Vojcic L, et al. A fluorescent hydrogel-based flow cytometry high-throughput screening platform for hydrolytic enzymes. Chemistry & Biology, 2014, 21 (12): 1733–1742. |
| [52] | Larsen AC, Dunn MR, Hatch A, et al. A general strategy for expanding polymerase function by droplet microfluidics. Nature Communications, 2016, 7 : 11235. DOI:10.1038/ncomms11235 |
| [53] | Fischlechner M, Schaerli Y, Mohamed MF, et al. Evolution of enzyme catalysts caged in biomimetic gel-shell beads. Nature Chemistry, 2014, 6 (9): 791–796. DOI:10.1038/nchem.1996 |
| [54] | Woronoff G, Ryckelynck M, Wessel J, et al. Activity-fed translation(aft)assay:A new high-throughput screening strategy for enzymes in droplets. Chembiochem, 2015, 16 (9): 1343–1349. DOI:10.1002/cbic.v16.9 |
| [55] | 韩云宾, 黄琛, 冯雁. 催化元件和途径的人工设计与组装. 生命科学, 2011, 23(9): 869–874. |
| [56] | Kintses B, Hein C, Mohamed MF, et al. Picoliter cell lysate assays in microfluidic droplet compartments for directed enzyme evolution. Chemistry & Biology, 2012, 19 (8): 1001–1009. |
| [57] | Hosokawa M, Hoshino Y, Nishikawa Y, et al. Droplet-based microfluidics for high-throughput screening of a metagenomic library for isolation of microbial enzymes. Biosensors and Bioelectronics, 2015, 67 : 379–385. DOI:10.1016/j.bios.2014.08.059 |
| [58] | Colin PY, Kintses B, Gielen F, et al. Ultrahigh-throughput discovery of promiscuous enzymes by picodroplet functional metagenomics. Nature Communications, 2015, 6 : 10008. DOI:10.1038/ncomms10008 |
| [59] | Stapleton JA, Swartz JR. Development of an in vitro compartmentalization screen for high-throughput directed evolution of. PLoS One, 2010, 5 (12): e15275. DOI:10.1371/journal.pone.0015275 |
| [60] | Ryckelynck M, Baudrey S, Rick C, et al. Using droplet-based microfluidics to improve the catalytic properties of rna under multiple-turnover conditions. RNA, 2015, 21 (3): 458–469. DOI:10.1261/rna.048033.114 |
| [61] | Obexer R, Pott M, Zeymer C, et al. Efficient laboratory evolution of computationally designed enzymes with low starting activities using fluorescence-activated droplet sorting. Protein Engineering Design and Selection, 2016, 29 (9): 355–366. DOI:10.1093/protein/gzw032 |
| [62] | Sjostrom SL, Bai Y, Huang M, et al. High-throughput screening for industrial enzyme production hosts by droplet microfluidics. Lab on a Chip, 2014, 14 (4): 806–813. DOI:10.1039/C3LC51202A |
| [63] | Huang M, Bai Y, Sjostrom SL, et al. Microfluidic screening and whole-genome sequencing identifies mutations associated with improved protein secretion by yeast[J]. Proceedings of the National Academy of Sciences, 2015, 112(34):E4689-E4696. |
| [64] | Jang S, Lee B, Jeong HH, et al. On-chip analysis, indexing and screening for chemical producing bacteria in microfluidic static droplet array. Lab on a Chip, 2016, 16 (10): 1909–1916. DOI:10.1039/C6LC00118A |
| [65] | Smith CA, Li X, Mize TH, et al. Sensitive, high throughput detection of proteins in individual, surfactant-stabilized picoliter droplets using nanoelectrospray ionization mass spectrometry. Analytical Chemistry, 2013, 85 (8): 3812–3816. DOI:10.1021/ac400453t |
| [66] | Sun S, Kennedy RT. Droplet electrospray ionization mass spectrometry for high throughput screening for enzyme inhibitors. Analytical Chemistry, 2014, 86 (18): 9309–9314. DOI:10.1021/ac502542z |
| [67] | Meier TA, Beulig R, Klinge E, et al. On-chip monitoring of chemical syntheses in microdroplets via surface-enhanced raman spectroscopy. Chemical Communications, 2015, 51 (41): 8588–8591. DOI:10.1039/C4CC09595B |
| [68] | Stehle R, Goerigk G, Wallacher D, et al. Small-angle x-ray scattering in droplet-based microfluidics. Lab on a Chip, 2013, 13 (8): 1529–1537. DOI:10.1039/c3lc41291a |
| [69] | Cheow LF, Viswanathan R, Chin CS, et al. Multiplexed analysis of protein-ligand interactions by fluorescence anisotropy in a microfluidic platform. Analytical Chemistry, 2014, 86 (19): 9901–9908. DOI:10.1021/ac502605f |
| [70] | Choi JW, Min KM, Hengoju S, et al. A droplet-based microfluidic immunosensor for high efficiency melamine analysis. Biosensors & Bioelectronics, 2015, 80 : 182–186. |
| [71] | Niu X, Pereira F, Edel JB, et al. Droplet-interfaced microchip and capillary electrophoretic separations. Analytical Chemistry, 2013, 85 (18): 8654–8660. DOI:10.1021/ac401383y |