2. 河南农业大学理学院, 郑州 450002
2. College of Sciences, Henan Agricultural University, Zhengzhou 450002
微生物在当今人们的生活中扮演着重要的角色,在食品、制药、医疗、石化、农业及生物修复等领域中应用十分广泛。越来越多的复杂应用需要更精巧的设计,往往需要考虑多种表型的优化,耐受性、途径流量、生长率等受多个基因及其与环境因子的相互作用的控制。缺少对复杂性状遗传基础的认知,限制了人们进行合理设计微生物的能力。操纵这些复杂性状的备选方法是在系统水平进行工程化改造[1],在不知道其机制的情况下,就可以使生物系统实现人们所期望的表型。
合成生物学是生命科学的工程化过程,合成自然界不存在的生物体系,并对现有的生物体系进行重新设计,目标是生产新的化学品、改善人类健康及解决环境问题等[2]。合成生物学工具包括生物部件、器件、模块、系统四部分,通过对这些元件逐级设计构建组合具有特定功能的生物系统。利用这些工具,人们可以构建非天然的基因调控模块来设计构建细胞生命活动的分子网络,类似于工程学中的线路设计,因此也称为基因线路 (genetic circuit) 。近些年来,科学家已构建了很多基于微生物转录和代谢复杂系统的合成生物学器件 (图 1) 。
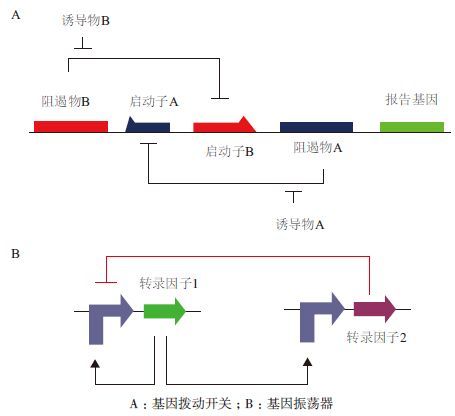
|
| 图 1 基于基因表达的合成生物学器件 A:基因拨动开关;B:基因振荡器 |
生物技术当前的发展趋势是在系统水平对微生物进行工程化改造,研究主要集中在定向进化、代谢工程和合成生物学领域。已成功应用于基因、代谢途径和基因组水平,通过设置不同阶段探索多种复杂的功能。本文讨论了一些通过微生物系统工程化实现复杂性状的策略,对当前合成生物学用于微生物系统工程化研究进展进行综述。
1 微生物系统复杂性状的定向进化复杂性状的工程化可以通过定向进化的策略来实现[3],通过多种控制设置模拟自然进化过程,达到人们所需的表型。自适应实验室进化技术 (Adaptive laboratory evolution,ALE) 就是其中一个例子,在特定选择条件下对微生物进行培养,经过数百代的培养后就可实现表型改进的进化[4]。例如,温度耐受是一个典型的复杂性状,人们对其遗传基础知之甚少,Tenaillon等[5]利用ALE方法进行了大肠杆菌的温度耐受进化研究。另外,许多利用ALE方法对大肠杆菌和酵母菌进行工程化改造的研究,成功地提高了生物燃料或化学品的产量和产率[6-8]。
定向进化策略通常依赖于人工选择压力和多代循环扩增来改进突变体,最终得到所需的复杂表型。其成功与否主要取决于两点因素,即产生功能多样化突变体的能力以及鉴定出真正表型改进突变体的筛选方法。近些年来,合成生物学领域的发展迅猛对定向进化策略做出了重要贡献,其领域的目标是设计、构建和改进自然界不存在的生物组分及系统[9]。合成生物学已发展出了很多可以加速定向进化的工具,可提高产生多样性子代的效率,同时也可以扩展筛选或选择范围。
2 遗传多样性的实现策略多样性是进化的基础,多样性程度越复杂,得到复杂的表型的可能性就越大。目前有很多种增加遗传多样性的方法被用于定向进化中。例如,易错PCR法[10, 11]和定点突变法等。易错PCR法利用低保真性的聚合酶,而定点突变法则需要预先知道目标的结构信息,在特定位点进行密码子突变以改善活性[12]。Bastian[13]利用定点突变的方法开关异丁醇生产中两个关键酶的辅因子,通过消除菌株的辅因子失调,最大限度地提高异丁醇生产理论产量。Firnberg等[14]发展出一个定点突变的改进方法,称为PFunkel。该方法允许在多个位点同时生成多个自定义文库。他们利用这个策略构建了一个完整的β-内酰胺酶基因密码子取代文库,用于识别一个与临床抑制剂抗性相关的突变。
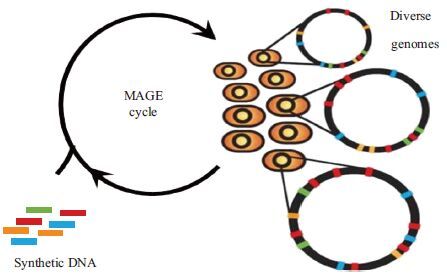
现代合成生物学工具可以有效的增加遗传多样性,向人们所期望的目标实现加速进化。使用多重自动基因组改造技术 (Multiplex automated genome engineering,MAGE) 可以在大肠杆菌靶位点增加多样性 (图 2) [15]。在单链DNA寡核苷酸上进行染色体编辑,在系统规模的多个层面上生成组合多样性。MAGE技术已成功用于大肠杆菌基因组的代谢途径优化[15-19],演化的MAGE方法也已被用于酿酒酵母的研究中[20]。
多样性也可以通过基因组改组的方法实现[21],基因组改组与经典突变筛选相比,可极大的加快进化速度,已被广泛用于各种生物体的进化[22]。合成生物学工具中最新增加的技术是成簇规律间隔短回文重复序列系统 (CRISPR) ,该技术可以对多种生物进行基因组编辑[23, 24],能够通过改变表达水平来增加多样性[25, 26],是当今研究的热点。另外,多样性也可以通过精确基因组编辑,即同源重组方法实现[27],如同饱和突变研究一样[28, 29]。CRISPR-Cas已被用来进行从点突变对整个代谢途径进行理性设计编辑[30]。现代合成生物学工具还可以利用非标准氨基酸使蛋白质进化出新的催化功能[31],或是建立完全正交核糖体-mRNA系统[32],用于核糖体功能的探索或工程化具有新的功能的核糖体。CRISPR-Cas技术还可以从目标样本或宏基因组样本中高效导入异源功能[33-35]。
3 复杂性状的筛选与选择合成生物学工具可以使人们得到更多突变体,有效的增加种群多样性。定向进化应用的关键是如何在一个群体中筛选或选择到改进的突变体。如果目标性状个体数量增加则可以通过生长选择方法得到目标,比色法或流式细胞仪细胞分选法可以用于筛选[36],但筛选方法只能用于有限的一些目标性状。合成生物学通过开发新的工具,扩大了特征谱,以便进行选择或筛选,从而解决上述问题。
合成生物学通过开发生物传感器,检测荧光蛋白触发表达或通过抗生素抗性基因进行筛选或选择过程。大多数生物传感器是基于转录因子和同源启动子对原理[17, 37],也有一些是基于RNA的检测方法[38]。新型生物传感器包含RNA传感器和蛋白质的传感器[39, 40],这些生物传感器可用于优化整个代谢通路和基因簇。例如,Raman等[17]使用MAGE技术生产全基因组靶向多样性,采用基于生物传感器的选择策略,通过该方法分别将目标产物柚皮素和葡糖酸产量增加了36倍和22倍。
生物传感器还可以与基因线路整合,以扩大输入的复杂性,同时连接所需输出[41]。更加复杂的基因线路可以通过两种方式提高筛选和选择能力:第一,一个单一的输入可以连接到多个输出 (图 3-A) ,在这种情况下目标性状可能触发级联调控网络,重新连接到有目标表型的细胞,最终进行筛选或选择。当达到足够的细胞浓度,重组细胞代谢在一个特定的输入下可以生产异丙醇[42];第二,多个输入可以通过逻辑门连接到一个单一输出上 (图 3-B) [7],导致增加的特异性表型的筛选。Hoynes-O'connor等[43]用RNA热敏元件构建3输入的基因线路,构建出可触发GFP的表达的热敏传感器。

|
| 图 3 基因线路 A:单输入多输出;B:多输入单输出 |
另一种有效的选择策略是适当增加目标性状。噬菌体辅助连续进化技术 (Phage-assisted continuous evolution,PACE) 使用修饰的M13噬菌体循环,使目标性状与感染所需的噬菌体蛋白表达相关[44]。通过让生物分子的实验室进化和噬菌体的生命周期结合在一起,让蛋白质在每24 h内进化60轮。PACE的效率是传统实验室进化方法的100倍左右,整个实验过程无需人为干预,大大节省了人力劳动成本。这个系统使用大肠杆菌的宿主细胞作为制造噬菌体的细胞工厂,利用噬菌体基因编码使生物分子繁殖,生成所需要的蛋白质,进行持续多轮的进化。最近研究人员还对PACE技术进行了改进,包括调整选择强度、对不需要的突变体进行负向选择等[45]。
4 合成生物学在系统水平的应用有了高效的多样性产生方法和筛选或选择策略,人们即便不完全了解某些微生物的遗传背景,仍然可以在系统水平上完成复杂的性状定向进化研究。新方法的产生使得生物技术将在不久的将来能够解决更多的问题。定向进化和合成生物学手段已经越来越多地应用于微生物的系统水平上。通过引入非标准氨基酸,蛋白质能够进化出自然界没有的新功能。例如,Xiao等[46]利用非标准氨基酸得到一个催化效率改进的内酰胺酶,而如果利用标准氨基酸突变方法则无法实现。利用生物系统还可以探索新的化学组成和性质,而代谢途径中的新功能则可以实现全新的应用,通过如MAGE或CRISPR-Cas等手段结合高效的筛选或选择方法进行优化[27]。另外,新近发展出的诸如ePathBrick和CasEMBLR等方法则可以进行快速通路组装并测试所期望的功能[47, 48]。因此,复杂的新功能可以使用合成生物学和定向进化工具在代谢途径水平进行工程化改造。
目前,如何处理细胞内的复杂调节是代谢途径水平工程化的一个难题,而很多情况往往是未知的。如今模块化的工程概念已被应用到合成生物学中,可以部分解决这个问题[49-51]。例如,Xu等[52]将脂肪酸生物合成分列在3个不同的模块中,可以有效优化大肠杆菌生产水平。通过优化每个模块的转录和翻译水平,通过平衡中间体的生产和消耗来消除生产瓶颈,当前这些模块化工程策略已经被大规模应用。此外,为克服天然调控机制,Temme等[53]在产酸克雷伯菌中重构了一个23.5 kb的固氮基因簇,重构的基因簇由特征显著的启动子和核糖体结合位点等合成元件所控制,重构后人们可以将一些功能基因簇转移到大肠杆菌中。如莱因衣藻光的系统II 重构,证实了种内交换的可行性[54]。重构策略的另一个十分有前景的应用是小分子组合生物合成[55],这些分子库可能成为未来新一代疗法。
基因组规模的工程化是合成和系统生物学的前瞻性应用。Ma和Isaacs等[56]开发出分级结合组装基因组工程策略——CAGE,可以将不同大肠杆菌菌株的基因组中的区域整合组装形成一个嵌合体基因组 (图 4) ,允许大规模的特异基因组区域转移,无需体外操作就能实现体内染色体的精确操控 。利用靶点编辑工具改变遗传密码,将这些密码子修饰组合并入到基因组中,通过这种方法能够测量重组频率,确认活性,并确定相关的表型[18]。
其中一个典型的例子是通过改变遗传密码来设计一种依赖于非标准氨基酸的代谢,利用合成蛋白设计来进行转基因生物控制[57]。另外,有效的DNA的组装方法加上逐渐降低DNA合成成本,使得科学家可以从头合成整个基因组[58]。探索合成重构基因组的复杂性降低将有利于人们进一步研究和操纵这些系统。2014年,Annaluru等[59]全合成了一个功能完整的酵母染色体,未来人们可以利用从头合成技术和计算机辅助设计工具重构合成染色体和基因组[60]。而这些进展为未来的理性设计和基因组订制奠定了基础,给生物技术和生物科学研究带来革命性的变革。然而,基因组设计及引导基因组进入细胞的规则尚未阐明,是目前科研人员遇到的最大障碍。
合成生物学技术还可以用于多细胞水平,工程化的微生物菌群可以通过分工扩大代谢能力,减少许多纯培养的限制因素,如代谢负担或有毒中间产物的积累等。这些可以通过代谢互养或基于群体感应信号分子的基因线路来实现有效的细胞间的通讯[61, 62]。一个标志性的研究工作是利用工程真菌细菌菌群从纤维素生物质中直接生产异丁醇[63],而最近大肠杆菌共培养的方法也被用于从葡萄糖/木糖混合物中生产复杂的对羟基苯甲酸和黏康酸[64]。一些与人类健康相关的生物技术应用也证实了预测和工程化的复杂生物之间的相互作用是可行的。肠道微生物工程菌群就是一个非常有前途的领域,未来能够治疗大范围的人类疾病。在这之前,仍然需要科学家发展多种非模式生物相关的合成生物学工具,并且对肠道微生物的相互作用机制有更深刻的了解。
5 展望设计复杂的多基因表型的能力是很多生物技术研究努力的方向,同时也正制约着这个领域的发展。将现代合成生物学工具与定向进化和代谢工程相结合,通过操纵越来越复杂的性状,可以让生物系统作为一个整体进行工程化改造。通过这些技术,人们可以对复杂的系统和性状进行工程化改造,这将会推动新生物技术在多个工业部门间的应用。利用合成生物学工具可以快速和有效的产生理性多样性,加速定向进化周期。此外,更多新型生物传感器和基因线路研发将大大提高改进突变体的筛选和选择能力,进一步加快人们对其他生物进行定向进化能力,大幅增加生物技术设计的复杂性。
| [1] | Denard CA, Ren H, Zhao H. Improving and repurposing biocatalysts via directed evolution. Curr Opin Chem Biol, 2015, 25 : 55–64. DOI:10.1016/j.cbpa.2014.12.036 |
| [2] | Way JC, Collins JJ, Keasling JD, et al. Integrating biological redesign:where synthetic biology came from and where it needs to go. Cell, 2014, 157 : 151–161. DOI:10.1016/j.cell.2014.02.039 |
| [3] | Cobb RE, Sun N, Zhao H. Directed evolution as a powerful synthetic biology tool. Methods, 2013, 60 : 81–90. DOI:10.1016/j.ymeth.2012.03.009 |
| [4] | Dragosits M, Mattanovich D. Adaptive laboratory evolution principles and applications for biotechnology. Microb Cell Fact, 2013, 12 : 64. DOI:10.1186/1475-2859-12-64 |
| [5] | Tenaillon O, Rodriguez-Verdugo A, Gaut RL, et al. The molecular diversity of adaptive convergence. Science, 2012, 335 : 457–461. DOI:10.1126/science.1212986 |
| [6] | Hu H, Wood TK. An evolved Escherichia coli strain for producing hydrogen and ethanol from glycerol. Biochem Biophys Res Commun, 2010, 391 : 1033–1038. DOI:10.1016/j.bbrc.2009.12.013 |
| [7] | Bassalo MC, Liu RM, Gill RT. Directed evolution and synthetic biology applications to microbial systems. Curr Opin Biotechnol, 2016, 39 : 126–133. DOI:10.1016/j.copbio.2016.03.016 |
| [8] | Wang Y, Manow R, Finan C, et al. Adaptive evolution of nontransgenic Escherichia coli KC01 for improved ethanol tolerance and homoethanol fermentation from xylose. J Ind Microbiol Biotechnol, 2011, 38 : 1371–1377. DOI:10.1007/s10295-010-0920-5 |
| [9] | Khalil AS, Collins JJ. Synthetic biology:applications come of age. Nat Rev Genet, 2010, 11 : 367–379. |
| [10] | Cadwell RC, Joyce GF. Randomization of genes by PCR mutagenesis. PCR Methods Appl, 1992, 2 : 28–33. DOI:10.1101/gr.2.1.28 |
| [11] | van Summeren-Wesenhagen PV, Marienhagen J. Metabolic engineering of Escherichia coli for the synthesis of the plant polyphenol pinosylvin. Appl Environ Microbiol, 2015, 81 : 840–849. DOI:10.1128/AEM.02966-14 |
| [12] | Wang W, Malcolm BA. Two-stage PCR protocol allowing introduction of multiple mutations, deletions and insertions using QuikChange site-directed mutagenesis. Biotechniques, 1999, 26 : 680–682. |
| [13] | Bastian S, Liu X, Meyerowitz JT, et al. Engineered ketol-acid reductoisomerase and alcohol dehydrogenase enable anaerobic 2-methylpropan-1-ol production at theoretical yield in Escherichia coli. Metab Eng, 2011, 13 : 345–352. DOI:10.1016/j.ymben.2011.02.004 |
| [14] | Firnberg E, Ostermeier M. PFunkel:efficient, expansive, user-defined mutagenesis. PLoS One, 2012, 7 : e52031. DOI:10.1371/journal.pone.0052031 |
| [15] | Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460 : 894–898. DOI:10.1038/nature08187 |
| [16] | Ng CY, Farasat I, Maranas CD, et al. Rational design of a synthetic Entner-Doudoroff pathway for improved and controllable NADPH regeneration. Metab Eng, 2015, 29 : 86–96. DOI:10.1016/j.ymben.2015.03.001 |
| [17] | Raman S, Rogers JK, Taylor ND, et al. Evolution-guided optimization of biosynthetic pathways. Proc Natl Acad Sci USA, 2014, 111 : 17803–17808. DOI:10.1073/pnas.1409523111 |
| [18] | Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333 : 348–353. DOI:10.1126/science.1205822 |
| [19] | Rovner AJ, Haimovich AD, Katz SR, et al. Recoded organisms engineered to depend on synthetic amino acids. Nature, 2015, 518 : 89–93. DOI:10.1038/nature14095 |
| [20] | DiCarlo JE, Conley AJ, Penttila M, et al. Yeast oligo-mediated genome engineering(YOGE). ACS Synth Biol, 2013, 2 : 741–749. DOI:10.1021/sb400117c |
| [21] | Zhang YX, Perry K, Vinci VA, et al. Genome shuffling leads to rapid phenotypic improvement in bacteria. Nature, 2002, 415 : 644–646. DOI:10.1038/415644a |
| [22] | Biot-Pelletier D, Martin VJJ. Evolutionary engineering by genome shuffling. Appl Microbiol Biotechnol, 2014, 98 : 3877–3887. DOI:10.1007/s00253-014-5616-8 |
| [23] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339 : 819–823. DOI:10.1126/science.1231143 |
| [24] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339 : 823–826. DOI:10.1126/science.1232033 |
| [25] | Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in humancells. Science, 2014, 343 : 84–87. DOI:10.1126/science.1247005 |
| [26] | Konermann S, Brigham MD, Trevino AE, et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2015, 517 : 583–588. |
| [27] | Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31 : 233–239. DOI:10.1038/nbt.2508 |
| [28] | Findlay GM, Boyle EA, Hause RJ, et al. Saturation editing of genomic regions by multiplex homology-directed repair. Nature, 2014, 513 : 120–123. DOI:10.1038/nature13695 |
| [29] | Pines G, Pines A, Garst AD, et al. Codon compression algorithms for saturation mutagenesis. ACS Synth Biol, 2015, 4 : 604–614. DOI:10.1021/sb500282v |
| [30] | Horwitz AA, Walter JM, Schubert MG, et al. Efficient multiplexed integration ofsynergistic alleles and metabolic pathways in yeasts via CRISPR-Cas. Cell Syst, 2015, 1 : 88–96. DOI:10.1016/j.cels.2015.02.001 |
| [31] | Des Soye BJ, Patel JR, Isaacs FJ, et al. Repurposing the translation apparatus for synthetic biology. Curr Opin Chem Biol, 2015, 28 : 83–90. DOI:10.1016/j.cbpa.2015.06.008 |
| [32] | Orelle C, Carlson ED, Szal T, et al. Protein synthesis by ribosomes with tethered subunits. Nature, 2015, 524 : 119–124. DOI:10.1038/nature14862 |
| [33] | Santos CNS, Regitsky DD, Yoshikuni Y. Implementation of stable and complex biological systems through recombinase-assisted genome engineering. Nat Commun, 2013, 4 : 2503. |
| [34] | Gaida SM, Sandoval NR, Nicolaou SA, et al. Expression of heterologous sigma factors enables functional screening of metagenomic and heterologous genomic libraries. Nat Commun, 2015, 6 : 7045. DOI:10.1038/ncomms8045 |
| [35] | Zingaro KA, Nicolaou SA, Yuan Y, et al. Exploring the heterologous genomic space for building, stepwise, complex, multicomponent tolerance to toxic chemicals. ACS Synth Biol, 2014, 3 : 476–486. DOI:10.1021/sb400156v |
| [36] | Xiao H, Bao Z, Zhao H. High throughput screening and selection methods for directed enzyme evolution. Ind Eng Chem Res, 2015, 54 : 4011–4020. DOI:10.1021/ie503060a |
| [37] | Liu Y, Li Q, Zheng P, et al. Developing a high-throughputscreening method for threonine overproduction based on an artificial promoter. Microb Cell Fact, 2015, 14 : 121. DOI:10.1186/s12934-015-0311-8 |
| [38] | Yang J, Seo SW, Jang S, et al. Synthetic RNA devices to expedite the evolution of metabolite-producing microbes. Nat Commun, 2013, 4 : 1413. DOI:10.1038/ncomms2404 |
| [39] | Carothers JM, Goler JA, Juminaga D, et al. Model-driven engineering of RNA devices to quantitatively program gene expression. Science, 2011, 334 : 1716–1719. DOI:10.1126/science.1212209 |
| [40] | Tinberg CE, Khare SD, Dou J, et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature, 2013, 501 : 212–216. DOI:10.1038/nature12443 |
| [41] | Brophy JAN, Voigt CA. Principles of genetic circuit design. Nat Methods, 2014, 11 : 508–520. DOI:10.1038/nmeth.2926 |
| [42] | Soma Y, Hanai T. Self-induced metabolic state switching by a tunable cell density sensor for microbial isopropanol production. Metab Eng, 2015, 30 : 7–15. DOI:10.1016/j.ymben.2015.04.005 |
| [43] | Hoynes-O'Connor A, Hinman K, Kirchner L, et al. De novo design of heat-repressible RNA thermosensors in E. coli[J]. Nucleic Acids Res, 2015, 43:6166-6179. |
| [44] | Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules. Nature, 2011, 472 : 499–503. DOI:10.1038/nature09929 |
| [45] | Carlson JC, Badran AH, Guggiana-Nilo DA, et al. Negative selection and stringency modulation in phage-assisted continuous evolution. Nat Chem Biol, 2014, 10 : 216–222. DOI:10.1038/nchembio.1453 |
| [46] | Xiao H, Nasertorabi F, Choi SH, et al. Exploring the potential impact of an expanded genetic code on protein function. Proc Natl Acad Sci USA, 2015, 112 : 6961–6966. DOI:10.1073/pnas.1507741112 |
| [47] | Xu P, Vansiri A, Bhan N, et al. ePathBrick:a synthetic biology platform for engineering metabolic pathways in E. coli[J]. ACS Synth Biol, 2012, 1:256-266. |
| [48] | Jakociunas T, Rajkumar AS, Zhang J, et al. CasEMBLR:Cas9-facilitated multiloci genomic integration of in vivo assembled DNA parts in Saccharomyces cerevisiae. ACS Synth Biol, 2015, 4 (11): 1226–1234. DOI:10.1021/acssynbio.5b00007 |
| [49] | Ajikumar PK, Xiao WH, Tyo KEJ, et al. Isoprenoid pathway optimization for taxol precursor verproduction in Escherichia coli. Science, 2010, 330 : 70–74. DOI:10.1126/science.1191652 |
| [50] | Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic protein scaffolds provide modular control over metabolic flux. Nat Biotechnol, 2009, 27 : 753–759. DOI:10.1038/nbt.1557 |
| [51] | Martin VJJ, Pitera DJ, Withers ST, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat Biotechnol, 2003, 21 : 796–802. DOI:10.1038/nbt833 |
| [52] | Xu P, Gu Q, Wang W, et al. Modular optimization of multi-gene pathways for fatty acids production in E. coli[J]. Nat Commun, 2013, 4:1409. |
| [53] | Temme K, Zhao D, Voigt CA. Refactoring the nitrogen fixation gene cluster from Klebsiella oxytoca. Proc Natl Acad Sci USA, 2012, 109 : 7085–7090. DOI:10.1073/pnas.1120788109 |
| [54] | Gimpel JA, Nour-Eldin HH, Scranton MA, et al. Refactoring the six-gene photosystem II core in the chloroplast of the green algae Chlamydomonas reinhardtii. ACS Synth Biol, 2015, 5 (7): 589–596. |
| [55] | Zhang H, Wang Y, Wu J, et al. Complete biosynthesis of erythromycin A and designed analogs using E. coli as a heterologous host[J]. Chem Biol, 2010, 17:1232-1240. |
| [56] | Ma NJ, Moonan DW, Isaacs FJ. Precise manipulation of bacterial chromosomes by conjugative assembly genome engineering. Nat Protoc, 2014, 9 (10): 2285–300. DOI:10.1038/nprot.2014.081 |
| [57] | Mandell DJ, Lajoie MJ, Mee MT, et al. Biocontainment of genetically modified organisms by synthetic protein design. Nature, 2015, 518 : 55–60. DOI:10.1038/nature14121 |
| [58] | Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemicallysynthesized genome. Science, 2010, 329 : 52–56. DOI:10.1126/science.1190719 |
| [59] | Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 2014, 344 : 55–58. DOI:10.1126/science.1249252 |
| [60] | Christen M, Deutsch S, Christen B. Genome calligrapher:a web tool for refactoring bacterial genome sequences for de novo DNA synthesis. ACS Synth Biol, 2015, 4 : 927–934. DOI:10.1021/acssynbio.5b00087 |
| [61] | Mee MT, Collins JJ, Church GM, et al. Syntrophic exchange in synthetic microbial communities. Proc Natl Acad Sci USA, 2014, 111 : E2149–E2156. DOI:10.1073/pnas.1405641111 |
| [62] | Youk H, Lim WA. Secreting and sensing the same molecule allows cells to achieve versatile social behaviors. Science, 2014, 343 : 1242782. DOI:10.1126/science.1242782 |
| [63] | Minty JJ, Singer ME, Scholz SA, et al. Design and characterization of synthetic fungal-bacterial consortia for direct production of isobutanol from cellulosic biomass. Proc Natl Acad Sci USA, 2013, 110 : 14592–14597. DOI:10.1073/pnas.1218447110 |
| [64] | Zhang H, Pereira B, Li Z, et al. Engineering Escherichia coli coculture systems for the production of biochemical products. Proc Natl Acad Sci USA, 2015, 112 : 8266–8271. DOI:10.1073/pnas.1506781112 |