代谢工程的主要目标之一就是通过基因调控增加化合物产量。随着DNA分子技术的进步,我们现在能够在基因水平对代谢网络进行操作。通过针对途径中的关键酶的编码基因,进行敲除、扩增和下调[1, 2]。基因敲除方法主要优势是增加底物向目标化合物的转化率。然而基因敲除常常导致生长率的下降,从而致使产量的减少。权衡产量和产率使冲突最小化,这样的设计和操作的优化是至关重要的[3]。在抵消基因敲除对产量的影响上[4],基因表达动态调节比静态的方法在最大化产物浓度上更有利。对于基因敲除影响产量这一问题,动态调控的解决方法就是保持动态的高度表达直到获得足够的生物量,然后关掉这些基因。合成生物学开关就是实现动态基因表达控制系统的重要元件,它能感应相关条件的变化,在适当的时间控制基因表达的“开”或“关”。目前,在代谢工程中常用的合成生物学开关有光控开关、温度诱导开关、拨动开关和核糖开关。
1 光控开关光是一个高度灵活的外界刺激,它在不改变代谢条件的前提下,实现时间空间上控制基因的精确表达。因此,在功能基因组学、系统生物学和生物技术领域光介导的基因表达调控研究进展迅速。光诱导的生物反应主要通过使用光封闭分子,或者使用带有光敏感结构域 (发色团) 的光感应器来实现光控开关的触发。光封闭化合物和有发色团分子吸收一定波长的光,然后起始一个光触发的反应。光诱导反应导致封闭基团不可逆释放或者使光感受器处于可逆的激活态。因此这两种机制都用于光依赖的基因表达控制[5]。
然而封闭化合物有一些限制,光信号到达特定细胞的过程和这种机制在大范围不同的生物体的应用都有一定障碍,而且封闭化合物光分解后不可逆转。因此,基因编码的人工光受体蛋白的发展有助于规避这些系统固有的限制。相比较而言光敏感调控系统被用于代谢工程中进行重组开关的构建[6]。人工光受体蛋白在光照射下激活其激酶活性,从而磷酸化某些启动子转录所需的关键转录因子,转录因子被激活起始转录 (图 1) 。Binder等[7]将光控开关用于谷氨酸棒状杆菌中朱栾倍半萜的生产,获得了目前为止朱栾倍半萜最高产量和滴定量。朱栾倍半萜对菌体生长有严重影响,而且Binder等使用的谷氨酸棒状杆菌细胞膜对IPTG透性较差。他们将带有封闭分子的IPTG加LAC操纵子的光控开关用于调节朱栾倍半萜合酶的表达,在生长期抑制朱栾倍半萜的合成,当菌体达到一定密度后,用光诱导朱栾倍半萜合酶基因表达。从而在不影响菌体生长情况下,获得了高产量的朱栾倍半萜。

|
| 图 1 光介导的基因表达的控制 人工光受体蛋白在光照射下激活其激酶活性,从而磷酸化某些启动子转录所需的关键转录因子,转录因子被激活起始目标基因转录 |
温度作为代谢过程的重要环境条件很早就已经应用在代谢调控中。在大肠杆菌中含有λ噬菌体的PL启动子的质粒常用来表达外源基因。通过引入λ噬菌体的CI抑制子,PL启动子下的基因表达可以被温度控制[8]。这种温度诱导需要使用一个温度敏感的抑制子,在32℃时抑制子抑制基因的表达,在42℃时诱导基因表达。Gu等[9]发现了另一种温度控制基因表达机制。这种机制通过温度控制σE因子是否与LuxR启动子 (Plux) 结合来控制基因表达。LuxR是群体感应的重要因子,将群体感应原理与温度控制开关结合可以构建出“与”门逻辑通路。当温度达到37℃并且Plux启动子被群体信号分子AHL所诱导时,Plux启动子下游的基因才能表达。温度诱导在大规模应用上来说可能是最方便的方法,温度可以通过加热和冷却来迅速切换。1990年,Shi等[10]将温度诱导开关应用于聚-3-羟基丁酸 (PHB) 的生产,在34℃下培养菌体10 h促进菌体生长,然后在40℃诱导PHB合酶表达,5 h后温度降至37℃缓解热休克,他们使用这种方法大大提高了PHB的产量和产率。2016年,Zhang等[11]将温度诱导开关用于α-酮丁酸的生产,在35℃保证细胞生长,然后切换到40℃诱导苏氨酸脱氨酶表达,使α-酮丁酸的产量在发酵26 h后达40.8 g/L,达到了工业生产要求的产量。
3 拨动开关拨动开关是一大类代谢调控开关,它们的共同特点是能够随环境 (诱导剂或其他感应底物的有无) 的改变来控制基因表达的开关。这里简单介绍几种代谢工程中使用的拨动开关。2000年,Gardner等[12]描述了一种构建在质粒上的拨动开关,它可以在两个启动子表达之间来回拨动来应答外部信号。开关由两个抑制子和两个保守的启动子组成 (图 2) 。每个启动子都会被另一个启动子表达的抑制子所抑制[13]。这样通过控制诱导剂的有无来控制开关在“开”和“关”之间切换,从而控制下游基因的表达。

|
| 图 2 双稳态拨动开关 在“关”状态,lacI从Ptet启动子转录。LacI结合到Plac启动子上,抑制tetR和gfp表达。在“开”状态,tetR和gfp从Plac启动子转录。TetR结合到Ptet启动子上,抑制lacI表达 |
随着对微生物群体感应机制研究的深入,将群体感应机制与拨动开关结合形成了能够感应菌体密度的动态调节元件。2004年,Kobayashi等[14]设计了一个由PL*启动子、Ptrc启动子、lacI基因和λcl基因组成的拨动开关。2008年,Anesiadis等[15]将群体感应应用到代谢工程中,在Kobayashi等的开关的基础上设计出了感应群体密度的拨动开关。细胞间的交流是通过一群信号分子完成的,它们是自诱导物。高丝氨酸内酯 (AHL) 就是一个这样的自诱导物,它的浓度跟环境中的细胞数量成一定比例。将AHL的表达基因设计到拨动开关中,就构建成了感应群体密度的拨动开关 (图 3) 。菌体密度决定AHl浓度,AHL浓度决定LacI表达,进而决定目的基因表达[16]。这实现了基因表达的动态调节,解决了因为基因敲除而影响细胞生长,进而影响产量的问题。

|
| 图 3 群体感应拨动开关 luxl基因表达的蛋白是AHL合成途径重要的酶,AHL可以穿过细胞膜扩散。当环境中的AHL达到一定浓度时,AHL与LuxR结合成AHL-LuxR复合物,AHL-LuxR复合物诱导lacI表达,LacI抑制cl基因及其下游要调控的基因的表达。在细胞浓度较低时,调控的基因正常表达。这样就形成了细胞低密度时开关呈“开”状态,而细胞高密度时开关呈“关”状态 |
在代谢工程生产目标产物时往往使用阻断竞争途径和过表达瓶颈酶等手段来增加产量,这种方法往往使中间体和酶积累较高水平,高水平的中间体和酶对菌体产生毒性,抑制细胞生长和降低产物产量。Liu 等在对大肠杆菌进行改造生产脂肪酸时发现改造后的菌株中乙酰辅酶A羧化酶积累很高。乙酰辅酶A羧化酶积累导致菌体生长的下降。Liu等[17]设计了一种可以感应乙酰辅酶A羧化酶的产物丙二酸单酰辅酶A浓度的拨动开关。该开关可以在丙二酸单酰辅酶A浓度低时提高乙酰辅酶A羧化酶的表达,在丙二酸单酰辅酶A浓度较高时降低乙酰辅酶A羧化酶的表达。这样既可以增加产物积累,又能降低乙酰辅酶A羧化酶积累对细胞的毒性。
Tsuruno等[18]将拨动开关应用于生产3-羟基丙酸,获得了比传统敲除方法更高的产量和产率。本实验室在对莽草酸生产的研究中,为了解决敲除莽草酸下游代谢基因莽草酸激酶而使菌株变成芳香族化合物营养缺陷型的问题,设计了一种能够调节莽草酸激酶基因表达量的开关,增加了莽草酸的产量。这种方法与直接敲除莽草酸激酶相比避免了外源添加芳香族化合物[19],从而降低了成本。
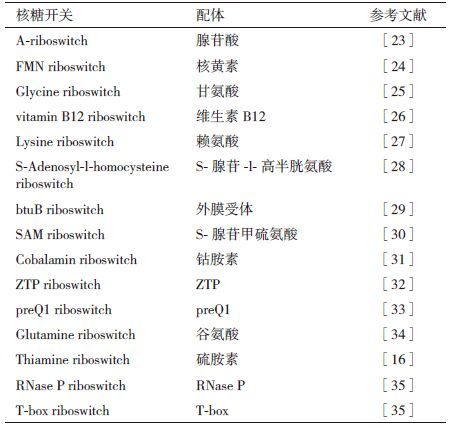
4 核糖开关近些年来,一种新的不需要蛋白转录因子参与而直接由核酸感受胞内信号分子或胞外环境变化的调控模式得到越来越多关注并被应用于一些研究领域,这种调控模式被称为核糖开关 (riboswitch) 。核糖开关结构首先由Winkler等[20]于2002年在大肠杆菌中发现并命名。迄今为止发现的核糖开关有20多种,包括Ado-cbl核糖开关、SAM核糖开关、TPP核糖开关、FMN核糖开关、赖氨酸核糖开关及glmS核糖开关等广泛应用的核糖开关 (表 1) 。因为核糖开关是由适体结构域和表达结构域的组合,一个结构域更改并不影响另一个区域功能的发挥。人们可以在不改变调控法则的情况下更改核糖开关配体结合的特异性。于是通过尽可能的绑定新的配体进而筛选出高能力的基因调控适体。核糖开关可实时感应配体的浓度,并在配体浓度低时表现出“开”的状态,基因可以正常表达;在配体浓度高时表现出“关”的状态,抑制基因表达。核糖开关通过感应底物浓度高低来实现对基因表达的动态调节。
核糖开关这种调节元件已经被用于代谢工程生产。Zhou和Zeng[21]将赖氨酸核糖开关应用于赖氨酸的生产。三羧酸循环 (TCA) 是氨基酸合成前体的重要来源,柠檬酸合酶是TCA循环的第一个酶 (图 4) 。有实验证明柠檬酸合酶活性降低有助于赖氨酸产量增加。但是柠檬酸合酶是细菌能量代谢的重要酶,直接敲除将导致菌体生长严重下降。Wang等[22]用赖氨酸核糖开关替换掉柠檬酸合酶编码基因gltA前端启动子和起始密码子之间的序列。核糖开关跟随gltA一起转录,在生产菌株产生一定的赖氨酸时,赖氨酸与核糖开关结合,下调下游基因gltA的表达。这样既避免了基因敲除导致的菌体生长下降又提高了赖氨酸的产量。本实验室在对大肠杆菌生产赖氨酸的研究中,将双重筛选基因tetA与核糖开关相结合组成核糖开关筛选系统,实现了赖氨酸生产菌株的高通量筛选,提高了赖氨酸的产量。

|
| 图 4 赖氨酸合成代谢途径 在赖氨酸浓度较低时,柠檬酸合酶基因正常表达。当赖氨酸达到一定浓度时,赖氨酸结合到核糖开关上,使核糖开关构象发生变化,核糖体不能结合在RBS上,柠檬酸合酶基因不能表达柠檬酸合酶 |
随着微生物代谢工程产物不断的研发并应用于生产中,对于代谢工程的各种工具的研究也不断深入。由于基因敲除导致生产菌株生长率下降进而影响产物产量,研究的方向也开始转移到了动态调控相关基因的表达。近年来合成生物学开关的研究已成为热点,特别是代谢工程中开关的应用相关研究具有独特的吸引力,逐步深入并取得了很大进展。预计合成生物学的开关将应用于代谢工程各种产品的生产,将极大推动相关领域的发展。
| [1] | Kim JY, Cha HJ. Down-regulation of acetate pathway through antisense strategy in Escherichia coli:improved foreign protein production. Biotechnology and Bioengineering, 2003, 83 (7): 841–853. DOI:10.1002/(ISSN)1097-0290 |
| [2] | Herring CD, Glasner JD, Blattner F. Gene replacement without selection:regulated suppression of amber mutations in Escherichia coli. Gene, 2003, 311 : 153–163. DOI:10.1016/S0378-1119(03)00585-7 |
| [3] | Schugerl K, Hubbuch J. Integrated bioprocesses. Current Opinion in Microbiology, 2005, 8 (3): 294–300. DOI:10.1016/j.mib.2005.01.002 |
| [4] | Gadkar KG, Doyle Iii FJ, Edwards JS, et al. Estimating optimal profiles of genetic alterations using constraint-based models. Biotechnology and Bioengineering, 2005, 89 (2): 243–251. DOI:10.1002/(ISSN)1097-0290 |
| [5] | Drepper T, Krauss U, Meyer zu Berstenhorst S, et al. Lights on and action! Controlling microbial gene expression by light. Appl Microbiol Biotechnol, 2011, 90 (1): 23–40. DOI:10.1007/s00253-011-3141-6 |
| [6] | Deiters A. Principles and applications of the photochemical control of cellular processes. Chembiochem:a European Journal of Chemical Biology, 2010, 11 (1): 47–53. |
| [7] | Binder D, Frohwitter J, Mahr R, et al. Light-controlled cell factories-employing photocaged isopropyl-β-d-thiogalactopyranoside for light-mediated optimization of lac promoter-based gene expression and(+)-valencene biosynthesis in Corynebacterium glutamicum. Applied and Environmental Microbiology, 2016, 82 (20): 6141–6149. DOI:10.1128/AEM.01457-16 |
| [8] | Lowman HB, Bina M. Temperature-mediated regulation and downstream inducible selection for controlling gene expression from the bacteriophage lambda pL promoter. Gene, 1990, 96 (1): 133–136. DOI:10.1016/0378-1119(90)90353-S |
| [9] | Gu D, Guo M, Yang M, et al. A σE-mediated temperature gauge con-trols a switch from LuxR-mediated virulence gene expression to the-rmal stress adaptation in Vibrio alginolyticus. PLoS Pathogens, 2016, 12 (6): e1005645. DOI:10.1371/journal.ppat.1005645 |
| [10] | Shi H, Kyuwa K, Takasu M, et al. Temperature-induced expression of phb genes in Escherichia coli and the effect of temperature patterns on the production of poly-3-hydroxybutyrate. Journal of Bioscience and Bioengineering, 2001, 91 (1): 21–26. DOI:10.1016/S1389-1723(01)80105-5 |
| [11] | Zhang C, Qi J, Li Y, et al. Production of α-ketobutyrate using engineered Escherichia coli via temperature shift. Biotechnology and Bioengineering, 2016, 113 (9): 2054–2059. DOI:10.1002/bit.v113.9 |
| [12] | Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403 (6767): 339–342. DOI:10.1038/35002131 |
| [13] | Judd EM, Laub MT, McAdams HH. Toggles and oscillators:new genetic circuit designs. McAdams, BioEssays, 2000, 22 (6): 507–509. DOI:10.1002/(SICI)1521-1878(200006)22:6<>1.0.CO;2-8 |
| [14] | Kobayashi H, Kaern M, Araki M, et al. Programmable cells:interfacing natural and engineered gene networks. Proc Natl Acad Sci U S A, 2004, 101 (22): 8414–8419. DOI:10.1073/pnas.0402940101 |
| [15] | Anesiadis N, Cluett WR, Mahadevan R. Dynamic metabolic engineering for increasing bioprocess productivity. Mahadevan, Metab Eng, 2008, 10 (5): 255–266. DOI:10.1016/j.ymben.2008.06.004 |
| [16] | Anesiadis N, Kobayashi H, Cluett WR, Mahadevan R. Analysis and design of a genetic circuit for dynamic metabolic engineering. ACS Synthetic Biology, 2013, 2 (8): 442–452. DOI:10.1021/sb300129j |
| [17] | Liu D, Xiao X, Evans BS, Zhang F. Negative feedback regulation of fatty acid production based on a malonyl-CoA sensor-actuator. ACS Synthetic Biology, 2015, 4 (2): 132–140. DOI:10.1021/sb400158w |
| [18] | Tsuruno K, Honjo H, Hanai T. Enhancement of 3-hydroxypropionic acid production from glycerol by using a metabolic toggle switch. Microbial Cell Factories, 2015, 14 : 155. DOI:10.1186/s12934-015-0342-1 |
| [19] | Gu P, Su T, Wang Q, et al. Tunable switch mediated shikimate biosynthesis in an engineered non-auxotrophic Escherichia coli. Scientific Reports, 2016, 6 : 29745. DOI:10.1038/srep29745 |
| [20] | Winkler W, Nahvi A, Breaker RR. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression. Nature, 2002, 419 (6910): 952–956. DOI:10.1038/nature01145 |
| [21] | Wang JM, Gao D, Yu X, et al. Evolution of a chimeric aspartate kinase for L-lysine production using a synthetic RNA device. Appl Microbiol Biotechnol, 2015, 99 (20): 8527–8536. DOI:10.1007/s00253-015-6615-0 |
| [22] | Li C, Lv D, Zhang L, et al. Approach to the unfolding and folding dynamics of add A-riboswitch upon adenine dissociation using a coarse-grained elastic network model. The Journal of Chemical Physics, 2016, 145 (1): 014104. DOI:10.1063/1.4954992 |
| [23] | Howe JA, Xiao L, Fischmann TO, et al. Atomic resolution mechan-istic studies of ribocil:A highly selective unnatural ligand mimic of the E. coli FMN riboswitch[J]. RNA Biol, 2016, 13(10):946-954. |
| [24] | Esquiaqui JM, Sherman EM, Ye JD, Fanucci GE. Conformational flexibility and dynamics of the internal loop and helical regions of the kink-turn motif in the glycine riboswitch by site-directed spin-labeling. Biochemistry, 2016, 55 (31): 4295–4305. DOI:10.1021/acs.biochem.6b00287 |
| [25] | Yu Y, Zhu X, Shen Y, et al. Enhancing the vitamin B12 production and growth of Propionibacterium freudenreichii in tofu wastewater via a light-induced vitamin B12 riboswitch. Applied Microbi-ology and Biotechnology, 2015, 99 (24): 10481–10488. DOI:10.1007/s00253-015-6958-6 |
| [26] | Wang J, Gao D, Yu X, et al. Evolution of a chimeric aspartate kinase for L-lysine production using a synthetic RNA device. Applied Microbiology and Biotechnology, 2015, 99 (20): 8527–8536. DOI:10.1007/s00253-015-6615-0 |
| [27] | Su Y, Hickey SF, Keyser SG, Hammond MC. In vitro and in vivo enzyme activity screening via RNA-based fluorescent biosensors for S-adenosyl-l-homocysteine(SAH). Journal of the American Chemical Society, 2016, 138 (22): 7040–7047. DOI:10.1021/jacs.6b01621 |
| [28] | Lussier A, Bastet L, Chauvier A, Lafontaine DA. A kissing loop is important for btuB riboswitch ligand sensing and regulatory control. J Biol Chem, 2015, 290 (44): 26739–26751. DOI:10.1074/jbc.M115.684134 |
| [29] | Suresh G, Srinivasan H, Nanda S, Priyakumar UD. Ligand-induced stabilization of a Duplex-like architecture is crucial for the switching mechanism of the SAM-III riboswitch. Biochemistry, 2016, 55 (24): 3349–3360. DOI:10.1021/acs.biochem.5b00973 |
| [30] | Polaski JT, Holmstrom ED, Nesbitt DJ, Batey RT. Mechanistic insights into cofactor-dependent coupling of RNA folding and mRNA transcription/translation by a cobalamin riboswitch. Cell Reports, 2016, 15 (5): 1100–1110. DOI:10.1016/j.celrep.2016.03.087 |
| [31] | Trausch JJ, Marcano-Velazquez JG, Matyjasik MM, Batey RT. Metal ion-mediated nucleobase recognition by the ZTP riboswitch. Chemistry & Biology, 2015, 22 (7): 829–837. |
| [32] | Aytenfisu AH, Liberman JA, Wedekind JE, Mathews DH. Molecular mechanism for preQ1-II riboswitch function revealed by molecular dynamics. RNA, 2015, 21 (11): 1898–1907. DOI:10.1261/rna.051367.115 |
| [33] | Ren A, Xue Y, Peselis A, et al. Structural and dynamic basis for low-affinity, high-selectivity binding of L-glutamine by the glutamine riboswitch. Cell Reports, 2015, 13 (9): 1800–1813. DOI:10.1016/j.celrep.2015.10.062 |
| [34] | Zhang J, Ferré-DAmaré AR. Trying on tRNA for size:RNase P and the T-box riboswitch as molecular rulers[J]. Biomolecules 2016, 6(2):E18. |