2. 四川大学华西医院消化内科, 成都 610041
2. Department of Gastroenterology, West China Hospital, Sichuan University, Chengdu 610041
迄今,大量来源于植物、真菌和微生物中的天然产物被广泛的应用于制药、医疗行业和工业。在过去30年制药行业的发展中,61%抗癌药物和49%的抗感染药物来源于天然产物[1]。然而传统天然产物研究的策略,即通过常规分离萃取方法获得植物中的天然产物或通过培养条件和工艺优化筛选微生物中的天然产物[2],往往具有盲目性,且耗时长、成本高,重复发现已知的化合物,已经渐渐不能满足当前人们对天然产物开发的需要。此时,合成生物学的出现与应用无疑对其产生了巨大的影响。合成生物学主要是以生物技术和工程化理念为基础,综合了系统生物学、生物化学、生物物理学、生物信息学等学科,旨在设计、改造、重构或创造生物系统,进而使这些生物系统满足人类的需求。在过去的10年中,从青蒿素的生物合成到吗啡在酵母中的表达[3, 4],从达托霉素等的生物合成途径鉴定到应用组合生物合成方法得到其类似物[5, 6],合成生物学为天然产物的研究带来了一场新的革命。随着合成生物学的发展,天然产物的研究得到了相应的促进。一方面,生物信息学的发展和完善,使天然产物研究的靶向性更明确;另一方面,合成生物学技术的蓬勃发展为设计、重构、改造天然产物生物合成系统提供了全新的思路和技术。
在植物中,天然产物主要通过植物内不同酶元件串联反应得到。对于结构复杂、高活性的植物天然产物,如抗癌药物紫杉醇,因其提取产量低、全合成效率低,其产量往往难以满足市场要求,故重构其生物合成途径并在底盘细胞中成功表达将为解决该问题提供新思路[7-10]。基于京都基因和基因组百科全书 (Kyoto Encyclopedia of Genes and Genomes,KEGG) [11]和PhytoMetaSyn[12]等数据库的代谢途径分析和相关酶的筛选,为植物天然产物合成路径在底盘细胞中异源表达的设计提供了更多信息和素材。在微生物中,有生物活性的天然产物多为微生物的次级代谢产物。常用的次级代谢产物预测工具有antiSMASH (antibiotics and Secondary Metabolites Analysis SHell) [13]和SMURF[14]等,以放线菌为例,平均每个放线菌含有20-40个次级代谢生物合成途径,其中大多数未被研究或在原始菌株生长状态下不表达 (这类基因簇即“沉默”基因簇) ,激活这些基因簇将会为新型天然产物的挖掘提供新的来源。
因此,系统的生物技术策略与高通量的操作方法对于天然产物的发现和改造有重要的意义[15]。基于有效的基因重组技术[16]和编辑手段,或异源表达策略[17-19],可快速准确的实现新天然产物的发现或已知高价值天然产物的产量提高[20]。本文主要就近几年应用于天然产物研究的基因编辑和重组表达策略做了简要介绍,讨论了这些方法用于天然产物表达簇的重组和对底盘细胞的编辑调控,最后对这些相关合成生物学技术在天然产物研究领域的应用进行简要叙述。
1 合成生物学技术的发展为实现新天然产物的发现和高价值化合物的高量表达,目标基因簇的构建与合适底盘细胞的选择十分重要。在过去的10年里,科研工作者们基于合成生物学技术开发和优化了许多基因编辑与重组的技术以用于大片段DNA的组装和编辑以及宿主细胞的改造和优化[21]。就基因重组技术而言,主要包含两大类:体内重组与体外重组。其基本原理大都是基于限制性内切酶的酶连反应或同源序列介导的重组 (表 1) 。基因编辑技术也在近几年有飞速的发展,同时促进和协助了底盘细胞的优化以及基因簇的定向改造。
这类方法主要是利用限制性内切酶剪切,暴露出黏性末端,具有互补性质的黏性末端在连接酶作用下末端识别进行重组。
2008年,Engler等[35]利用具有特殊性质的IIS型内切酶构建了一个多片段的一步克隆法,命名为Golden Gate。IIS型内切酶的特点在于,其剪切位点大都在识别位点的外侧2-4 bp,因此剪切后载体不易自连,同时由于暴露的末端与酶切位点序列无关,故可以通过设计利用同一内切酶实现多片段组装和连接。2011年,Weber等[36]基于Golden Gate发明了新的多片段重组系统模块克隆 (Modular Cloning,MoClo) ,并且通过三步克隆,成功实现了总长33 kb共11个转录单元 (共来自44个基因片段) 的基因簇构建。MoClo主要思路为:a.设计最终的载体结构及确认实验中所需的工具载体与基因片段;b.以基本的基因片段平行组装所有的转录单元 (一级载体) ;c.将上一步得到的转录单元组装得到二级载体 (由于组装效率的限制,每次最多组装6个单元) ,最后一步根据实际情况可能要多次重复才能得到最终的载体 (图 1) [37]。2012年,Werner等[22]通过在该系统一级载体与二级载体间引入过渡载体,实现了总长50 kb共17个转录单元的基因簇构建 (来自68个基因片段) 。2016年,Iverson等[38]基于MoClo建立了新的跨学科的整合设计自动化研究实验室 (Cross-disciplinary Integration of Design Automation Research lab,CIDAR) 模块克隆库,其中包含了一系列在E.coli中可通用的模块组分,包括启动子、终止子、载体和核糖体结合位点等。该系统的建立使MoClo的使用更加自动化,大大节约了设计和操作的时间。除此之外,2016年Moore等[39]基于MoClo开发了另一适用于E.coli合成生物学操作的工具包EcoFlex,并且成功实现总长31 kb、共20个基因的组装。EcoFlex系统引入了标准元件库 (与CIDAR相似) ,以及荧光报告系统 (CIDAR无该设计) ,然后基于标准元件实现用户自定义的模块组装。与CIDAR侧重于自动化模块组装相比,EcoFlex在实时检测和设计灵活上有更明显的优势。相比于传统的酶切连接,在多片段克隆上,MoClo具有设计简便、操作流程简洁、省时高效的优势。同时,CIDAR和EcoFlex系统的建立,进一步从实验设计上优化了多片段克隆。
2013年,Chen等[23]设计了一个多基因片段连接的新方法——甲基化辅助的末端剪切连接 (Methylational-ASsisted Tailorable Ends Rational ligation,MASTER ligation) ,并成功组装4个基因片段得到总长29 kb的DNA片段。MASTER主要根据同时兼具IIS与IIM性质的限制性内切酶 (restriction enzyme,RE) ,如Chen等采用MspJI,特异性识别甲基化的4-bpmCNNR (R=A/G) ,并在识别位点外侧剪切形成黏性末端,产生4 bp随意的黏性末端,从而实现多基因的按序无缝连接。与MoClo相比,由于其组装过程与序列无关 (sequence-independent) 的性质,故更适于基因片段相似性高的多片段组装。
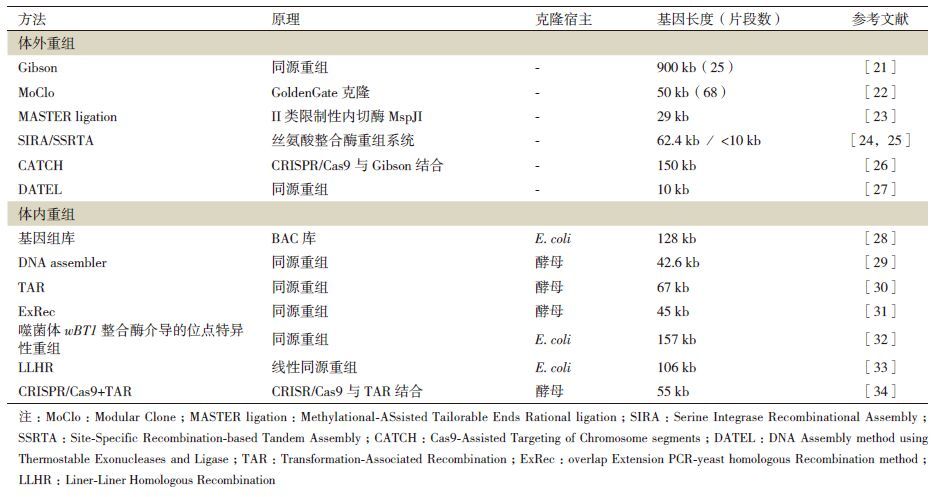
1.1.1.2 体外同源重组方法体外的同源重组方法主要有序列与连接独立型克隆 (Sequence and Ligati-on-Independent Cloning,SLIC) [40]、无缝克隆试剂盒 (Seamless Ligation Cloning Extract,SLiCE) [41]、尿嘧啶-定点切除试剂克隆 (Uracil-site Specific Excision Reagent cloning,USER) [42]等。以上方案大都仅适用于10 kb以下的片段重组,但它们可用于一些基础工具载体的构建,从而进一步用于基因编辑。2009年,Gibson等[21]报道了一种高效快捷的一步克隆法——Gibson assembly,并实现了大于50个基因片段的组装,也可实现总长约900 kb的基因序列组装,故Gibson assembly可作为大片段组装时的优先选择。该方法原理是利用T5外切酶剪切基因片段得到单链DNA臂,经Taq连接酶与聚合酶的作用,使各基因片段按序组装。2015年4月,Wang等[43]将CRISPR/Cas9系统与Gibson assembly结合实现基因簇构建或编辑,成功克隆组装出最长约29 kb的DNA片段。同年9月,Jiang等[44]发表文章,利用策略-CATCH (Cas9-Assisted Targeting of Chromosome segments) (图 2) ,成功克隆组装得到最长约150 kb的DNA片段。CRISPR/Cas9-Gibson assembly的作用原理是在CRISPR/Cas9定向剪切后,得到目的基因片段,最后采用Gibson assembly重组得到目的序列。但此方法对于大片段的操作效率仍然较低。基于CRISPR/Cas9的大片段克隆技术,由于其一步克隆得到目的片段的特点,且其片段克隆受酶切位点等的限制,操作的灵活性强,故尤适于完整大基因片段克隆。2016年,Jin等[27]发明了一个类似Gibson assembly的无缝克隆方法DATEL (DNA Assembly method using Thermostable Exonucleases and Ligase) ,验证对于2-10片段的基因组装均有效,但对于大片段的操作效率依然较低。
|
| 图 2 CATCH与CRISPR/Cas9-TAR的操作流程 |
另一种多片段组装策略基于随机组装的位点特异性重组 (Site-Specific Recombination-based Tandem Assembly,SSRTA) 由Zhang等[45]研究报道的。2010年,其课题组研究wBT1和wC31介导重组的同时,发现并鉴定了一系列attB与attP位点,次年,Zhang等[24]构建了SSRTA,完成了总长62.4 kb (10片段) 的埃博霉素生物合成途径的构建。SSRTA主要是利用wBT1整合酶的重组机制,利用wBT1特异性剪切aat位点形成末端,再经过重组酶作用形成DNA产物,该方法可以快捷高效有序的组装多片段。2014年,Colloms等[25]报道了另一种基于丝氨酸整合酶重组的方法丝氨酸整合酶重组系统 (Serine Integrase Recombinational Assembly,SIRA) ,可有效组装总长小于10 kb的4-5个基因片段。这两种方法除了可以有效的组装多片段外,且验证可以对已有的基因进行片段的增删等。
1.1.2 体内重组方法2009年,Shao等[46]基于酵母体内重组的机制构建了一个新的体内克隆方法-DNA assembler,并成功完成约19 kb,共8个片段的组装。2011年,其课题组利用该系统完成约45 kb的spectinabilin合成基因簇的构建[29]。该方案的显著优点在于:设计灵活、不受酶切位点限制、效率高以及操作简单。与DNA assembler类似的另一种方法TAR (Transformation-Associated Recombination) ,于2002年由Resnick等[47]构建,是一种基于转化偶联重组的酵母重组技术,由于其组装基因长度小于15 kb而使应用受限。通过不断优化,现已成为大片段构建的常用方法之一,如Yamanaka等[30]利用TAR组装得到总长67 kb的它纳霉素合成基因簇。2014年,Lee等[34]将CRISPR/Cas9系统与TAR联合,最终实现约55 kb的基因簇克隆。TAR-CRISPR/Cas9原理与CATCH类似 (图 2) ,即利用Cas9作为“剪刀”,从基因组中获得目的片段,再将目的片段与载体利用TAR进行组装,得到最终目的载体。DNA assembler与TAR均为利用酵母体内可自行发生同源重组的特性,二者对于组装的基因片段均需要具有相应的同源序列;虽然TAR最终形成的是环形的酵母人工染色体,而DNA assembler则可将基因片段组装到目的质粒上,也可直接整合至酵母基因组上,但是这两类方法均可有效的组装天然产物的合成基因簇。
在E. coli内的同源重组系统也有相应发展。2011年,基于全长的Rac噬菌体前蛋白RecE及其协同作用蛋白RecT的发现鉴定[32],Bian等[48]在E. coli中,建立了一个直接将DNA大片段克隆至表达载体的方法:线性-线性同源重组 (Liner-Liner Homologous Recombination,LLHR) ,并成功克隆了10个基因 (大小为10-52 kb不等) 至表达载体,且其中两个表达载体在异源宿主中表达得到相应产物luminmycin A 和luminmide A/B。LLHR的基本原理是基于E.coli中类似于重组酶系统的噬菌体前蛋白RecE在其协同蛋白RecT的协同调节下使载体与目的片段进行高效的线性-线性同源重组。2015年,Yin等[33]利用LLHR组装得到总长106 kb的salinomycin合成基因簇。另外也有研究者将CRISPR/Cas9系统引入E.coli进行重组系统设计[26, 49],并成功组装长约10 kb的丁醇表达途径[49]。虽然在E.coli中引入CRISPPR/Cas9系统对于大片段的组装不存在明显优势,但由于该方法可一步实现从基因片段到基因组的整合,故可利用其进行一些简单的生物合成途径的组装。
2014年,Du等[50]建立了一个直接克隆的策略-基于噬菌体wBT1整合酶调节的位点特异性重组。与SSTAR不同,该方法使得链霉菌基因组编辑与抗生素合成基因簇克隆可以同时进行,且整个过程在链霉菌中完成。其原理是在链霉菌中,3个导入的外源质粒中其中两个质粒分别对基因组的特定位点进行剪切,另一质粒对剪切片段进行重组并复制。Du等[50]成功利用该方法从天蓝链霉菌中克隆得到放线菌紫素的合成基因簇,从玫瑰包链霉菌NRRL15998中克隆得到纳它霉素和达托霉素的合成基因簇,且效率高达80%。相比于SSRAR,该方案在克隆大片段的基因簇操作上更简洁,同时通过外源质粒的优化,更适用于高GC含量的链霉菌基因的操作。随后2015年,Jin等[51]设计了一个新的大片段克隆系统FACs (Fungal Artificial Chromosomes) ,并成功获得56个来源于土曲霉的次级代谢产物合成途径。该方案主要是构建一个同时具有E.coli复制起始位点和曲霉复制起始位点的人工染色体,使其可在E.coli与模式的曲霉中穿梭,从而携带目的基因片段 (可达150 kb) 。真菌人工染色体能够快捷地从自然宿主基因组DNA上捕获不同大小的基因,通过对转化子末端测序来鉴定是否含有自然宿主的完整基因簇,将携带完整基因簇的真菌人工染色体在构巢曲霉中表达完成天然产物的异源生产。与基于噬菌体wBT1整合酶调节的位点特异性重组类似,FACs也是一步完成大片段基因克隆,但其目前仅限于对某些特定的真菌系统的操作。
目标基因簇的重构有时还涉及到一些优化与改造,如利用“Plug and Play”策略对目标基因插入模块化的启动子,或利用CRISPR/Cas9对调控基因进行编辑等,以实现基因簇的表达优化和调控改造[52, 53]。基于传统的基因重组策略,优化和发现新的基因重组编辑方法为不同目的的基因簇重构提供了更多的选择性。
1.2 底盘细胞的选择与改造底盘细胞往往是异源表达效率的决定因素之一,故其选择和改造也是天然产物生物合成路径构建的重要考虑方向。底盘细胞改造主要涉及到基因组的精简与优化、代谢流的控制优化和调控因子的改造等。
对于底盘细胞的精简与优化,最经典的方法为Cre-LoxP,例如2010年,Komatsu等[54]利用Cre-LoxP策略敲除了阿维链霉菌 (Streptomyces avermitilis) 全基因 (9.02 Mb) 组中约1.4 Mb的非必需基因,获得“最小基因组”阿维链霉菌底盘细胞,并成功表达外源基因簇链霉素、头孢菌素C等,且产量优于原始菌株表达量。该方法的主要原理是利用Cre重组酶可以特异性的识别LoxP位点并介导其发生重组。该系统适应性强,适用范围广,但其介导重组后会留下引入的LoxP位点,故对于需要多次非连续的基因编辑时,其应用则受到LoxP位点设计的限制。CRISPR/Cas9技术的出现则给了底盘细胞基因组精简与优化的另一种选择。CRISPR/Cas9介导的基因删除原理是 Cas9蛋白在sgRNA (single guide RNA) 的作用下,识别特异性的PAM位点,继而发生重组。例如,Cobb等[55],利用CRISPR/cas9系统敲除20 bp-30 kb的DNA时,效率可达70%-100%。相比于Cre/LoxP系统,CRISPR/cas9系统在选择靶点上更为灵活,且对于非连续性的多片段敲除上更易设计与操作。
对于代谢流的控制和调控因子的改造多通过对原始基因编辑或引入新的基因元件实现。其主要思路有: (1) 引入启动元件或高表达正调控因子。例如,Bai等[56]通过定量法鉴别了一系列可通用的合成模块调控元件 (核糖体结合位点+启动子) ,并成功将其应用于S.avermitilis中番茄红素表达基因簇的激活与过表达;为更好地在E.coli中表达异戊二烯,Chen等[57]通过协同过表达对异戊二烯合成有利的甲基赤磷酸化途径和甲戊磷酸化途径,成功将异戊二烯产量提高20倍; (2) 负调控因子的沉默或敲除。例如,已知在阿维链霉菌中阿维菌素生物合成分别受到SdrA和avaR2调控,Ulanova等[58]和Komatsu等[59]通过敲除SdrA和avaR2,不仅实现产物产量的提高;对于已改造的阿维链霉菌底盘细胞SUKA17引入ptlB,成功提高紫穗槐-1,4-二烯异源表达产量[59]。
底盘细胞作为生物合成途径表达的载体,为天然产物的合成提供了初始零件,即底盘细胞决定了生物合成底物与代谢通量,底物与代谢通量决定了天然产物合成的性质与产量。故根据实际生物合成途径选择合适的底盘细胞,并对其代谢过程的酶与调控因子加以优化,将为天然产物的异源表达锦上添花。
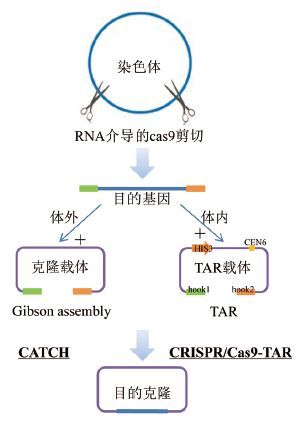
2 合成生物学在天然产物研究中的应用随着基因信息学的不断完善和编辑新方法与策略的不断发现及改造,从微生物或植物中鉴别次级代谢产物合成途径变得更加有靶向性 (图 3) 。合成生物学方法用于天然产物的研究从结果上可分为两大类:重构天然产物的合成基因簇进行异源表达,通过对原始宿主的调控实现合成基因簇的激活或过表达。
|
| 图 3 天然产物生物合成研究的整体思路 a:目的基因的获得方法;b:目的片段的体内重组方法,主要包括传统的酶切连接、MoClo、Gibson assembly、MASTER ligation、SIRA、SSRTA、DETAL等,对于小片段的线性组装也可采用OE-PCR的方法;c:目的片段的体内重组方法,主要包括DNA assembler、TAR、ExRec、LLHR等实现目的基因在宿主内组装成质粒形式或直接整合至基因组上;d:底盘细胞的改造与异源表达,将c中构建的目的载体引入优化的底盘细胞进行表达;e:基因簇的编辑与改造,在原始宿主中目的基因簇中加入启动子等调控原件,得到新的表达基因簇,或将基因簇进行体外重构,引入底盘细胞;f:对得到的新表达系统 (来自于d、e、f) 进行发酵、分析与鉴定。*标注的重构方法表示可用于有效组装基因长度大于50 kb的方法 |
异源表达作为天然产物研究的重要策略之一,主要应用可包含以下几类:第一,已知生物合成途径的高价值化合物的基因簇异源表达或调控,提高已有表达量。例如将来源于Streptomyces spheroides的新生霉素合成基因簇导入Streptomyces coelicolor M512中进行表达,使其产量提高了3.4倍[60]。第二,通过异源表达功能模糊或“沉默”的合成基因簇,实现新天然产物的发现。其主要应用的策略包含:启动子的优化,即替换原始基因簇的弱启动子或加入新的启动子;调控基因的编辑,即过表达正向调控子或沉默负调控子等。例如,Luo等[61]重构源于Streptomyces griseus中的未知基因簇,并在其上游加入启动子,成功使该基因簇在变铅青链霉菌 (Streptomyces lividans) 中表达,发现了具有重要生理活性的一类抗真菌化合物。Yamanaka等[30]通过编辑来源于Saccharomonospora sp. CNQ-490中的未知NRP基因簇,使其去除其中的LuxR型的负转录调控子,并将该基因簇转入底盘细胞天蓝链霉菌中表达,成功分离鉴定得到一个新的脂多肽化合物taromycin A。第三,对植物源天然产物的生物合成途径进行重构,实现其在模式微生物内的表达。如阿片类化合物在酵母中的成功表达[4]。
2.1.1 AnthracimycinAnthracimycin作为一类结构独特的14元大环内酯类抗生素,最先于2013年从Streptomyces sp. CNH365中分离鉴定得到其化学结构[62]。2015年,Alt等[63]从Streptomyces sp. T767中分离得到Anthracimycin,并通过异源表达分析推断得到其生物合成路径,为其进一步调控和衍生物的获得提供了依据。
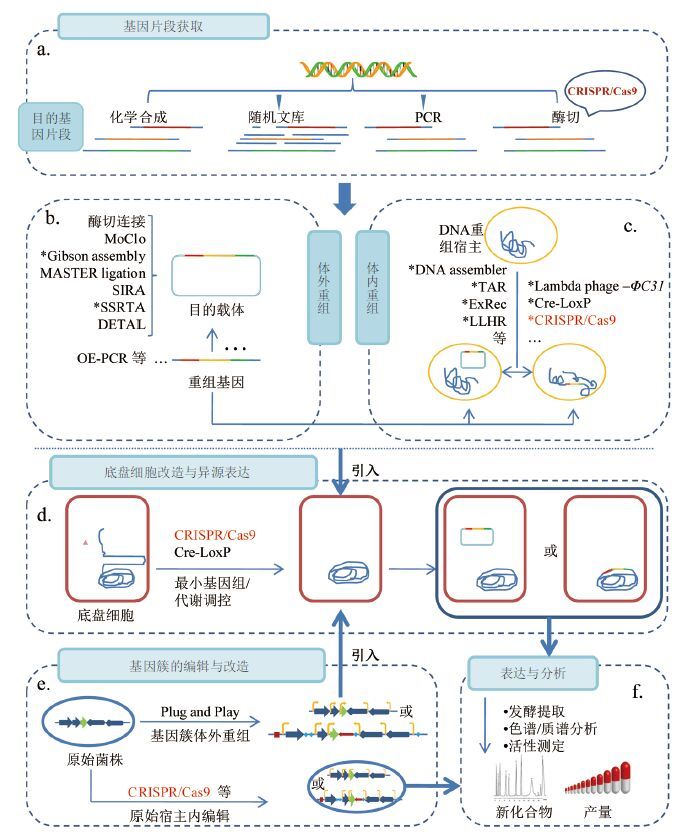
从Anthracimycin的结构分析,其主要骨架来源于PKS (polyketide synthase) 合成途径,但其萘烷部分形成类似于[4+2]的环加成,且其中3组双键的位置与典型的PKS作用结果不同。于是Silke等首先通过基因信息学的分析,对推断得到的Anthracimycin合成基因 (来源于Streptomyces sp. T767,后面以act 表示) 进行功能的判断,并通过实验验证 (同位素追踪等) 确定得到其生物合成途径 (图 4) 。Anthracimycin很可能只通过actC-F四个PKS基因作用形成,而不涉及后修饰基因作用。然后其课题组将鉴定的act 基因簇 (actC-F) 克隆并转入异源宿主S. coelicolor中表达并成功分离鉴定得到Anthracimycin。通过对act 基因簇的模型 (一类新的PKS) 构建,有利于了解这类新的大环内酯类抗生素的研究。另一方面,通过推断了解其生物合成过程,也为基因重构实现其类似物的发现奠定了基础。
|
| 图 4 Anthracimycin合成基因簇 |
从传统的发酵手段获得新的化合物依然是天然产物研究中的重要策略,但是某些生理活性较好的化合物若没有经过进一步的优化难以成为临床药物成分。另一方面,阐明高活性化合物的生物合成途径也有利于其类似物的发现。
2.1.2 nisin类似物nisin是属于羊毛硫抗生素类,由34个氨基酸组成,是一种天然抑菌剂,该类物质多具有良好的抑制革兰氏阳性菌的作用,且由于其安全性高,故也作为食品防腐剂用于食物保鲜,但该类化合物目前被分离得到的种类却不甚可观。
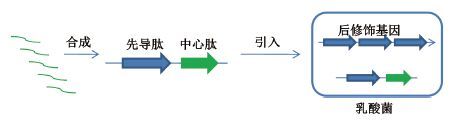
2016年,Van Heel等[64]通过BAGEL3等数据库分析,得到了72条与nisin中心肽类似的序列。随后其基于nisin合成基因的模块,保留先导肽部分从而构建了一个可适用不同核心多肽表达的元件 (图 5) 。最后将预测的序列与先导肽元件结合,引入乳酸菌中表达得到18种全新的结构差异较大的类似物,且其中5种表现出良好的药理活性。相较于传统的培养条件筛选,该系统能较快的实现未知基因的表达,同时能更具目的性的对产物进行分析。
|
| 图 5 nisin类似物研究思路 |
阿片类化合物作为西方最初使用的镇痛剂和姑息治疗药物,目前其唯一来源依然是罂粟的提取,因此其产量严重的受到气候、地域和虫害等的影响。作为重要的临床药物,不确定的产量制约了其市场需求。另外,阿片的衍生物诸如吗啡、蒂巴因等都是具有高价值的化合物,而化学合成这些结构复杂的化合物并不具有经济上的竞争性。大约30多种关于阿片类化合物的化学合成途径,但没有一种适用于大规模生产[65]。于是重构其生物合成途径在模式微生物中表达则具有突破性的意义 (图 6) 。
植物源天然产物的异源表达,首先需要筛选得到相应的酶;其次是在重构基因簇后,对底盘细胞的代谢流进行相应调控,最后完成途径的优化。
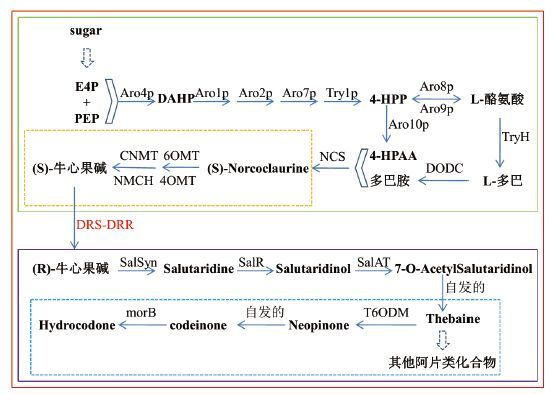
2008年,Hawkins等[66]首次通过在酵母中引入6OMT、CNMT、4OMT三种甲基转移酶,实现了BIAS合成的前体物质 (S) -牛心果碱的获得 (图 6橙色框) 。2014年,Thodey等[67]以蒂巴因为底物,在酵母中引入蒂巴因到吗啡合成途径中的脱甲基酶T6ODM和醛酮合成酶COR,最终在发酵液检测得到吗啡。此外,其在以上基础引入源于恶臭假单胞菌中的吗啡脱氢酶 (MorA) 和吗啡还原酶 (MorB) ,最终生物合成得到氢可酮、氢化吗啡酮和氧可酮 (图 6蓝色框) 。2015年,Deloache等[68]基于酶偶联法发明了一种新的生物传感器,并成功筛选得到来源于甜菜中的CYP716AD1具有酪氨酸羟化酶活性。通过PCR优化CYP716AD1,并将其引入酵母表达,成功实现从葡萄糖到阿片类化合物中间体 (S) -牛心果碱的生物合成 (图 6绿色框) 。同年,Fossati等[69]首先引入罂粟中沙罗泰里啶合成酶 (PsSAS) ,沙罗泰里啶还原酶 (PsSAR) 和salutaridinol乙酰转移酶 (PsSAT) 以及T6ODM、COR、CODM,构建得到从 (R) -牛心果碱到阿片类化合物的生物合成途径,并且在酵母中以 (R) -牛心果碱为底物时,成功分离得到吗啡 (图 6紫色框) 。诚如传统的化学反应,随着生物内反应链的加长,产物的积累变得更加困难,另外如何成功实现 (S) -reticuline到 (R) -reticuline的生物体内转化成为阿片类化合物从头合成的障碍。同年Galanie等[4]通过研究发现,DRR-DRS酶的作用,可将 (S) -reticuline成功转化为 (R) -reticuline,并通过改进底物来源的木糖辅助途径从源头提高阿片类化合物形成的代谢流,同时在实验中不断优化作用酶的表达和代谢流,从而最终实现了从头合成阿片类化合物 (图 6) 。虽然产量较低,但从无到有的合成给进一步的产量优化奠定了基础。
|
| 图 6 阿片类化合物的生物合成途径[4] E4P:4-磷酸赤藓糖;PEP:磷酸烯醇丙酮酸盐;DAHP:3-deoxy-D-arabino-2-heptulosonic acid 7-phosphate;4-HPP:4-羟基苯丙酮酸;4-HPAA:4-丙烯酸;Aro4p:DAHP合成酶;Aro1p:五功能arom酶;Aro2p:双功能变位酶与叶黄素还原酶;Aro7p:分支酸变位酶;Tyr1p:前体苯酸脱氢酶;Aro8p:芳香氨基转移酶 I;Aro9p:芳香氨基转移酶II;Aro10p:苯丙酮酸脱羧酶;TyrH:反馈抑制-酪氨酸拮抗;DODC:L-DOPA脱羧酶;NCS: (S) -norcoclaurine合成酶;6OMT:norcoclaurine 6-O-甲基转移酶;CNMT:coclaurine N-甲基转移酶;NMCH:N-methylcoclaurine羟化酶;4'OMT:3'-hydroxy-N-methylcoclaurine 4'-O-甲基转移酶;DRS-DRR:1,2-dehydroreticuline synthase-1,2-dehydroreticuline还原酶;SalSyn:salutaridine合成酶;SalR:salutaridine还原酶;SalAT:salutaridinol 7-O-酰基转移酶;T6ODM:蒂巴因 6-O-脱甲基酶;morB:吗啡还原酶 |
就阿片类化合物而言,生物合成一方面可以解决传统植物提取所面临的环境依赖性和产量不确定性;另一方面,相较于复杂冗长的化学合成,其更适合于大规模生产,且以葡萄糖为底物使其在经济上更具竞争性。
当前,利用模式微生物成功表达植物来源的天然产物的成功案例已经屡见不鲜,如在E.coli中成功表达过紫杉醇的前体紫杉二烯和紫杉酚,在酵母中成功表达过紫杉醇[9, 70, 71]、人参皂苷[72]、依托泊苷[73]等。
异源表达作为一种有效的天然产物发现与鉴定的策略,不仅成功应用于天然产物的发现,同时也为高价值化合物的高表达提供了新的策略。为成功实现目的天然产物的异源表达,需要注意以下3点:适宜的底盘细胞的选择、合理的代谢调控的优化以及待表达的基因簇的设计改造。当然,构建出多种不同高适应性的底盘细胞库与相应的调控元件库,将会加快基因簇的重构与异源表达的进程。
2.2 原始宿主调控原始宿主的调控主要包括利用操作调控子,改变代谢途径中的加工酶或转运酶或前体饲喂等实现天然产物的发现或产量提高。Wang等[74]在C-1027合成途径中,通过过表达调控子sgcR1、sgcR2和sgcR3均使目标产物的产量增加。Gottelt等[75]通过敲除天蓝链霉菌中一条PKS (Polyketide Synthase,聚酮合成酶) 生物合成途径中的转录抑制子scbR2,从而发现了新化合物coelimycin。Bai等[56]通过发明有一种基于启动子的生物感应器,成功激活并过表达了S. avermitilis中番茄红素。
Law等[76]在雷帕霉素的合成途径中引入新的甲硫氨酸腺苷转移酶,使其可识别不同甲基底物,得到多种新的16-位定点改变的雷帕霉素同系物。另外,Nguyen等[77]通过改变达托霉素合成途径中DptBC次级单位上的一个或多个模块从而改变达托霉素的环多肽中心;同时结合模块的更换、NRPS次级单位的替换、沉默后修饰的谷氨酸3-甲基转移酶等方法得到约30种达托霉素类似物,并且其中一种表现出优于达托霉素的抗E.coil imp的能力。
原始宿主内的调控主要集中于正向调控元件或生物传感器的引入、负调控因子的删除或沉默,从而实现未知基因簇的激活及过表达。另一方面,对于原始宿主内基因簇中关键的合成元件的调整将会为快速发现天然产物的类似物提供研究思路。所以建立可通用的生物传感器、发明快速准确的基因编辑手段是基于原始宿主研究天然产物的重要策略。
3 结语当前正是天然产物发现的新纪元,生物信息学为天然产物的合成基因簇鉴定与重构提供了理论参照数据,合成生物学技术的发展影响着应用的进程。在理论分析得到目的基因簇后,选择合适的基因簇改造方法,如异源表达、模块调节、组合合成等,可更准确的实现基因功能的鉴别及新化合物的发现[18, 64, 78]。同时,CRISPR/Cas9等新基因编辑技术的出现,为大片段的获得与组装编辑提供了新的策略,使基因重构表达更快速[34, 52, 79]。
虽然合成生物学方法应用于天然产物研究已经取得了一定成果,但是由于生物合成途径的重构随基因簇的增大而变得复杂,最终得到的表达系统也不一定符合预期。所以,设计构建更好的基因编辑系统和更具有优势的底盘细胞,将会进一步促进天然产物领域的发展。
| [1] | Luo Y, Enghiad B, Zhao H. New tools for reconstruction and heterologous expression of natural product biosynthetic gene clusters. Natural Product Reports, 2015, 33 : 174–182. |
| [2] | Zhang L, Yan K, Zhang Y, et al. High-throughput synergy screening identifies microbial metabolites as combination agents for the treatment of fungal infections. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104 : 4606–4611. DOI:10.1073/pnas.0609370104 |
| [3] | Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440 : 940–943. DOI:10.1038/nature04640 |
| [4] | Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349 : 1095–1100. DOI:10.1126/science.aac9373 |
| [5] | Miao V, Co?ffetlegal MF, Brian P, et al. Daptomycin biosynthesis in Streptomyces roseosporus:cloning and analysis of the gene cluster and revision of peptide stereochemistry. Microbiology, 2005, 151 : 1507–1523. DOI:10.1099/mic.0.27757-0 |
| [6] | Liao G, Shi T, Xie J. Regulation mechanisms underlying the biosynthesis of daptomycin and related lipopeptides. Journal of Cellular Biochemistry, 2012, 113 : 735–741. DOI:10.1002/jcb.v113.3 |
| [7] | Kirby J, Keasling JD. Biosynthesis of plant isoprenoids:perspectives for microbial engineering. Annual Review of Plant Biology, 2009, 60 : 335–355. DOI:10.1146/annurev.arplant.043008.091955 |
| [8] | Majumder A, Jha S. Biotechnological approaches for the production of potential anticancer leads podophyllotoxin and paclitaxel:An overview. Journal of Biological Sciences, 2009, 1 : 46–69. |
| [9] | Walker K, Croteau R. Taxol biosynthetic genes. Phytochemi-stry, 2001, 58 : 1–7. DOI:10.1016/S0031-9422(01)00160-1 |
| [10] | Guerra-Bubb J, Croteau R, Williams RM. The early stages of taxol biosynthesis:an interim report on the synthesis and identification of early pathway metabolites. Natural Product Reports, 2012, 29 : 683–696. DOI:10.1039/c2np20021j |
| [11] | Kanehisa M, Goto S. KEGG:kyoto encyclopedia of genes and genomes. Nucleic Acids Research, 2000, 27 : 29–34. |
| [12] | Misra BB. An updated snapshot of recent advances in transcriptomics and genomics of phytomedicinals. Journal of Postdoctoral Research February, 2014, 2 : 1–14. |
| [13] | Blin K, Medema MH, Kazempour D, et al. AntiSMASH 2.0-a versatile platform for genome mining of secondary metabolite producers[J]. Nucleic Acids Research, 2013, 41:204-212. |
| [14] | Khaldi N, Seifuddin FT, Turner G, et al. SMURF:Genomic mapping of fungal secondary metabolite clusters. Fungal Genetics & Biology Fg & B, 2010, 47 : 736–741. |
| [15] | Hwang KS, Kim HU, Charusanti P, et al. Systems biology and biotechnology of Streptomyces species for the production of secondary metabolites. Biotechnology Advances, 2014, 32 : 255–268. DOI:10.1016/j.biotechadv.2013.10.008 |
| [16] | Chao R, Yuan Y, Zhao H. Recent advances in DNA assembly technologies. FEMS Yeast Research, 2015, 15 : 1–9. DOI:10.1093/femsyr/fou003 |
| [17] | Luo Y, Li BZ, Liu D, et al. ChemInform abstract:Engineered biosynthesis of natural products in heterologous hosts. Cheminform, 2015, 46 : 5265–5290. |
| [18] | Weber T, Charusanti P, Musiolkroll EM, et al. Metabolic engineering of antibiotic factories:new tools for antibiotic production in actinomycetes. Trends in Biotechnology, 2014, 33 : 15–26. |
| [19] | Gaj T, Gersbach CA, Iii CFB. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 2013, 31 : 397–405. DOI:10.1016/j.tibtech.2013.04.004 |
| [20] | Luo Y, Cobb RE, Zhao H. Recent advances in natural product discovery. Current Opinion in Biotechnology, 2014, 30 : 230–237. DOI:10.1016/j.copbio.2014.09.002 |
| [21] | Gibson DG, Lei Y, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 2009, 6 : 343–345. DOI:10.1038/nmeth.1318 |
| [22] | Werner S, Engler C, Weber E, et al. Fast track assembly of multigene constructs using Golden Gate cloning and the MoClo system. Bioengineered Bugs, 2012, 3 : 38–43. DOI:10.4161/bbug.3.1.18223 |
| [23] | Chen WH, Qin ZJ, Wang J, et al. The MASTER(methylation-assisted tailorable ends rational)ligation method for seamless DNA assembly. Nucleic Acids Research, 2013, 41 : 395–408. |
| [24] | Zhang L, Zhao G, Ding X. Tandem assembly of the epothilone biosynthetic gene cluster by in vitro site-specific recombination. Scientific Reports, 2011, 1 : 40–40. |
| [25] | Colloms SD, Merrick CA, Olorunniji FJ, et al. Rapid metabolic pathway assembly and modification using serine integrase site-specific recombination. Nucleic Acids Research, 2014, 42 : 194–201. |
| [26] | Jiang Y, Chen B, Duan C, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied & Environmental Microbiology, 2015, 81 : 2506–2514. |
| [27] | Jin P, Ding W, Du G, et al. DATEL:a scarless and sequence-independent DNA assembly method using thermostable exonucleases and ligase[J]. Acs Synthetic Biology, 2016, 5(9):1028-1032. |
| [28] | Miao V, Co?ffetlegal MF, Brian P, et al. Daptomycin biosynthesis in Streptomyces roseosporus:cloning and analysis of the gene cluster and revision of peptide stereochemistry. Microbiology, 2005, 151 : 1507–1523. DOI:10.1099/mic.0.27757-0 |
| [29] | Shao Z, Luo Y, Zhao H. Rapid characterization and engineering of natural product biosynthetic pathways via DNA assembler. Molecular Biosystems, 2011, 7 : 1056–1059. DOI:10.1039/c0mb00338g |
| [30] | Yamanaka K, Reynolds KA, Kersten RD, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111 : 1957–1962. DOI:10.1073/pnas.1319584111 |
| [31] | Schimming O, Fleischhacker F, Nollmann FI, et al. Yeast homologous recombination cloning leading to the novel peptides ambactin and xenolindicin. Chembiochem, 2014, 15 : 1290–1294. DOI:10.1002/cbic.v15.9 |
| [32] | Fu J, Bian X, Hu S, et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting. Nature Biotechnology, 2012, 30 : 440–446. DOI:10.1038/nbt.2183 |
| [33] | Yin J, Hoffmann M, Bian X, et al. Direct cloning and heterologous expression of the salinomycin biosynthetic gene cluster from Streptomyces albus DSM41398 in Streptomyces coelicolor A3(2). Scientific Reports, 2015, 5 : 15081. DOI:10.1038/srep15081 |
| [34] | Lee NC, Larionov V, Kouprina N. Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J]. Nucleic Acids Research, 2015, 43(8):e55. |
| [35] | Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability[J]. PLoS One, 2008, 3:e3647. |
| [36] | Weber E, Engler C, Gruetzner R, et al. A modular cloning system for standardized assembly of multigene constructs[J]. PLoS One, 2011, 6:e16765. |
| [37] | Marillonnet S, Werner S. Assembly of multigene constructs using golden gate cloning. Methods in Molecular Biology, 2015, 1321 : 269–284. DOI:10.1007/978-1-4939-2760-9 |
| [38] | Iverson SV. Improved modular multipart DNA assembly, development of a DNA part toolkit for E. coli, and applications in traditional biology and bioelectronic systems[J]. Dissertations & Theses-Gradworks, 2016. |
| [39] | Moore SJ, Lai HE, Kelwick RJR, et al. EcoFlex:A multifunctional MoClo kit for E. coli synthetic biology[J]. Acs Synthetic Biology, 2016, 5(10):1059-1069. |
| [40] | Li M, Elledge S. Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC. Nature Methods, 2007, 4 : 251–256. DOI:10.1038/nmeth1010 |
| [41] | Zhang Y, Werling U, Edelmann W. Seamless ligation cloning extract(SLiCE)cloning method. Methods in Molecular Biology, 2014, 1116 : 235–244. DOI:10.1007/978-1-62703-764-8 |
| [42] | Nour-Eldin HH, Geu-Flores F, Halkier BA. USER cloning and USER fusion:the ideal cloning techniques for small and big laboratories. Methods in Molecular Biology, 2010, 643 : 185–200. DOI:10.1007/978-1-60761-723-5 |
| [43] | Wang JW, Wang A, Li K, et al. CRISPR/Cas9 nuclease cleavage combined with Gibson assembly for seamless cloning. Biotechniques, 2015, 58 : 161–170. |
| [44] | Jiang W, Zhao X, Gabrieli T, et al. Cas9-Assisted Targeting of CHromosome segments CATCH enables one-step targeted cloning of large gene clusters. Nature Communications, 2015, 6 : 8101. DOI:10.1038/ncomms9101 |
| [45] | Zhang L, Wang L, Wang J, et al. DNA cleavage is independent of synapsis during Streptomyces phage phiBT1 integrase-mediated site-specific recombination. Journal of Molecular Cell Biology, 2010, 2 : 264–275. DOI:10.1093/jmcb/mjq025 |
| [46] | Shao Z, Zhao H, Zhao H. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Research, 2009, 37 (2): e16. |
| [47] | Resnick MA, Larionov VL, Kouprina NY, et al. Transformation-associated recombination cloning:US, 6391642[P]. 2002-5-21. |
| [48] | Bian X, Huang F, Stewart FA, et al. Direct cloning, genetic engineering, and heterologous expression of the syringolin biosynthetic gene cluster in E. coli through Red/ET recombineering[J]. Chembiochem, 2012, 13:1946-1952. |
| [49] | Bassalo MC, Garst AD, Halweg-Edwards AL, et al. Rapid and efficient one-step metabolic pathway integration in E. coli[J]. Acs Synthetic Biology, 2016, 5(7):561-568. |
| [50] | Du D, Wang L, Tian Y, et al. Genome engineering and direct cloning of antibiotic gene clusters via phage ?BT1 integrase-mediated site-specific recombination in Streptomyces. Scientific Reports, 2015, 5 : 8740. DOI:10.1038/srep08740 |
| [51] | Jin WB, Ye R, Clevenger KD, et al. Fungal artificial chromosomes for mining of the fungal secondary metabolome. Bmc Genomics, 2015, 16 : 1–10. DOI:10.1186/1471-2164-16-1 |
| [52] | Kang HS, Charloppowers Z, Brady SF. Multiplexed CRISPR/Cas9 and TAR-mediated promoter engineering of natural product biosynthetic gene clusters in yeast. Acs Synthetic Biology, 2016, 5 (9): 1002–1010. DOI:10.1021/acssynbio.6b00080 |
| [53] | Liu X. Generate a bioactive natural product library by mining bacterial cytochrome P450 patterns. Synthetic & Systems Biotechnology, 2016, 1 : 95–108. |
| [54] | Komatsu M, Uchiyama T, Omura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107 : 2646–2651. DOI:10.1073/pnas.0914833107 |
| [55] | Cobb RE, Wang Y, Zhao H. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system. Acs Synthetic Biology, 2015, 4 (6): 273–728. |
| [56] | Bai C, Zhang Y, Zhao X, et al. Exploiting a precise design of universal synthetic modular regulatory elements to unlock the microbial natural products in Streptomyces. Proceedings of the National Academy of Sciences, 2015, 112 : 12181–12186. DOI:10.1073/pnas.1511027112 |
| [57] | Chen Y, Xiang G, Yu J, et al. Synergy between methylerythritol phosphate pathway and mevalonate pathway for isoprene production in Escherichia coli. Metabolic Engineering, 2016, 37 : 79–91. DOI:10.1016/j.ymben.2016.05.003 |
| [58] | Ulanova D, Kitani S, Fukusaki E, et al. SdrA, a new DeoR family regulator involved in streptomyces avermitilis morphological development and antibiotic production. Applied & Environmental Microbiology, 2013, 79 : 7916–7921. |
| [59] | Komatsu M, Komatsu K, Koiwai H, et al. Engineered streptomyces avermitilis host for heterologous expression of biosynthetic gene cluster for secondary metabolites. Acs Synthetic Biology, 2013, 2 : 384–396. DOI:10.1021/sb3001003 |
| [60] | Dangel V, Westrich L, Smith MCM, et al. Use of an inducible promoter for antibiotic production in a heterologous host. Applied Microbiology & Biotechnology, 2010, 87 : 261–269. |
| [61] | Luo Y, Huang H, Liang J, et al. Activation and characterization of a cryptic polycyclic tetramate macrolactam biosynthetic gene cluster. Nature Communications, 2013, 4 : 94–105. |
| [62] | Jang KH, Nam SJ, Locke JB, et al. Anthracimycin, a potent anthrax antibiotic from a marine-derived actinomycete. Angewandte Chemie International Edition, 2013, 52 : 7822–7824. DOI:10.1002/anie.v52.30 |
| [63] | Alt S, Wilkinson B. Biosynthesis of the novel macrolide antibiotic anthracimycin. Acs Chemical Biology, 2015, 10 (11): 2468–2479. DOI:10.1021/acschembio.5b00525 |
| [64] | Van Heel AJ, Kloosterman TG, Montalbanlopez M, et al. Discovery, production and modification of 5 novel lantibiotics using the promiscuous nisin modification machinery. Acs Synthetic Biology, 2016, 5 (10): 1146–1154. DOI:10.1021/acssynbio.6b00033 |
| [65] | Reed JW, Hudlicky T. The quest for a practical synthesis of morphine alkaloids and their derivatives by chemoenzymatic methods. Accounts of Chemical Research, 2015, 48 : 674–687. DOI:10.1021/ar500427k |
| [66] | Hawkins KM, Smolke CD. Production of benzylisoquinoline alkaloids in Saccharomyces cerevisiae. Nature Chemical Biology, 2008, 4 : 564–573. DOI:10.1038/nchembio.105 |
| [67] | Thodey K, Galanie S, Smolke CD. A microbial biomanufacturing platform for natural and semi-synthetic opiates. Nature Chemical Biology, 2014, 10 : 837–844. DOI:10.1038/nchembio.1613 |
| [68] | Deloache WC, Russ ZN, Narcross L, et al. An enzyme-coupled biosensor enables(S)-reticuline production in yeast from glucose. Nature Chemical Biology, 2015, 11 : 465–471. DOI:10.1038/nchembio.1816 |
| [69] | Fossati E, Narcross L, Ekins A, et al. Synthesis of morphinan alkaloids in Saccharomyces cerevisiae. PLoS One, 2015, 10 : e0124459. DOI:10.1371/journal.pone.0124459 |
| [70] | Kirby J, Keasling JD. Biosynthesis of plant isoprenoids:perspectives for microbial engineering. Annual Review of Plant Biology, 2009, 60 : 335–355. DOI:10.1146/annurev.arplant.043008.091955 |
| [71] | Guerra-Bubb J, Croteau R, Williams RM. The early stages of taxol biosynthesis:an interim report on the synthesis and identification of early pathway metabolites. Natural Product Reports, 2012, 29 : 683–696. DOI:10.1039/c2np20021j |
| [72] | Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast. Cell Research, 2014, 24 : 770–773. DOI:10.1038/cr.2014.28 |
| [73] | Lau W, Sattely ES. Six enzymes from mayapple that complete the biosynthetic pathway to the etoposide aglycone. Science, 2015, 349 : 1224–1228. DOI:10.1126/science.aac7202 |
| [74] | Wang L, Hu Y, Zhang Y, et al. Role of sgcR3 in positive regulation of enediyne antibiotic C-1027 production of Streptomyces globisporus C-1027. Bmc Microbiology, 2009, 9 : 12683–12690. |
| [75] | Gottelt M, Kol S, Gomezescribano JP, et al. Deletion of a regulatory gene within the cpk gene cluster reveals novel antibacterial activity in streptomyces coelicolor A3(2). Microbiology, 2010, 156 : 2343–2353. DOI:10.1099/mic.0.038281-0 |
| [76] | Law BJC, Struck AW, Bennett MR, et al. Site-specific bioalkylation of rapamycin by the RapM 16-O-methyltransferase. Chemical Science, 2015, 6 : 2885–2892. DOI:10.1039/C5SC00164A |
| [77] | Nguyen KT, Ritz D, Gu JQ, et al. Combinatorial biosynthesis of novel antibiotics related to daptomycin. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103 : 17462–17467. DOI:10.1073/pnas.0608589103 |
| [78] | Zarinstutt JS, Barberi TT, Gao H, et al. Prospecting for new bacterial metabolites:a glossary of approaches for inducing, activating and upregulating the biosynthesis of bacterial cryptic or silent natural products. Natural Product Reports, 2015, 33 : 54–72. |
| [79] | Liu Y, Tao W, Wen S, et al. In vitro CRISPR/Cas9 system for efficient targeted DNA editing. Mbio, 2015, 6 (6): e01714–15. |