2. 山东省医药生物技术研究中心,济南 250062;
3. 山东大学 微生物技术国家重点实验室,济南 250100
2. Shandong Medicinal Biotechnology Center,Ji’nan 250062;
3. The State Key Laboratory of Microbial Technology,Shandong University,Ji’nan 250100
链霉菌能产生大量具有重要生物活性的次级代谢产物,其中许多已被广泛应用于医学、农业和畜牧业等多个领域[1]。目前已经应用于临床的抗生素有一半以上分离自链霉菌,如氯霉素、四环素、红霉素、利福霉素等;链霉菌在发酵过程中产生的阿维菌素可以应用于畜牧业或农业生产中,作为杀螨剂、杀虫剂使用。链霉菌次级代谢物的合成由营养和环境因素敏感的多个并行调控网络共同调控[2, 3]。
天蓝色链霉菌[Streptomyces coelicolor A3(2) ]作为链霉菌的模式菌株,已于2002年完成测序[4]。天蓝色链霉菌生长繁殖过程极其复杂,具体过程为:首先孢子萌发长出丝状体并延伸至培养基中形成基内菌丝,生长到一定阶段后基内菌丝向培养基外部生长形成气生菌丝,并合成一系列的次级代谢产物[5],随后气生菌丝顶端分化为孢子丝,成熟断裂形成孢子。研究表明其可以产生至少4种次级代谢产物:放线紫红素(Actinorhodin,ACT)、十一烷基灵菌红素(Undecylprodigiosin,RED)、次甲基霉素(Methylenomycin,Mmy)、钙依赖抗生素(Calcium-dependent antibiotic,CDA),还有一种隐性代谢产物黄色色素coelimycin P1[6, 7]。
双组份信号转导系统(TCS)是原核生物一种普遍且重要的信号转导系统,TCS由组氨酸激酶(Histidine kinase,HK)和应答调节蛋白(Response regulator,RR)组成,通过蛋白磷酸化形式进行信号传递和转换[8]。天蓝色链霉菌中有67对典型的TCS,部分TCS的功能已被鉴定,如PhoR-P、AbsA1/A2和AfsQ1/Q2等。非典型的TCS包括不成对的HK和孤立的RR,目前对它们的调控研究并不深入,本研究中的sco1135为链霉菌中的孤立调控蛋白,为进一步研究sco1135参与次级代谢的复杂调控网络,我们对sco1135在参与形态发育及次级代谢物合成方面的分子调控机制进行研究。
本研究在S. coelicolor M145的基础上,运用分子生物学方法构建sco1135的缺失突变菌株△sco1135和回补菌株△sco1135com,通过表型分析、抗生素定量观察以及qRT-PCR的方法,对△sco1135和△sco1135com的孢子生长、抗生素合成以及相应的分子机制进行研究,以期阐明sco1135对SS. coelicolor M145的生长发育和次级代谢物合成的调控机制。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒本研究所用菌株及质粒,见表 1。
本研究所用的引物,见表 2。
LB培养基用于大肠杆菌培养,培养温度30℃或37℃;MS(SFM)、YBP、R2、R2YE、MM、MYM培养基用于链霉菌培养,培养温度30℃;培养基配制过程见链霉菌操作手册[9]。
1.1.4 试剂限制性内切酶及用于扩增的高保真酶Fastpfu DNA聚合酶购自Thermo Scientific公司,用于qRT-PCR的SYBR Premix Ex Taq(Tli RNaseH Plus)购于TaKaRa公司。
1.2 方法 1.2.1 天蓝色链霉菌缺失突变菌株和回补菌株的构建为了确定天蓝色链霉菌基因组中sco1135的功能,本研究对sco1135进行了同框缺失突变(in-frame deletion)。突变株的构建流程为:(1) 扩增带有sco1135基因上下45 bp序列的Apr抗性片段,大小为1 459 bp;(2) 将cosmid7B11转入BW25113/pIJ790,作为PCR-targeting[12]的宿主细胞;(3) 抗性片段上sco1135的同源序列与7B11上的序列发生同源重组,得到突变cosmid 7B11∷aac(3) IV;(4) 将突变cosmid 7B11∷aac(3) IV转入ET12567/pUZ8002,与M145进行属间接合转移;(5) 接合转移在MS培养基上培养,待长出接合转移子后接种到MS(Apr)培养基上进行两轮筛选培养,得到突变株△sco1135;为了确定△sco1135的表型变化是否由sco1135基因缺失引起的,本研究以pMS82为载体,对△sco1135进行基因回补,将带启动子的sco1135整合到突变株中得到回补菌株△sco1135com,回补序列包括上游含启动子的328 bp序列、目的基因和下游的94 bp序列。同时,为排除载体pMS82对链霉菌形态分化和抗生素合成的影响,用含有pMS82的M145菌株作对照。本研究所涉及的DNA相关的分子生物学实验操作具体方法参见分子克隆实验指南[13],链霉菌基因组的提取等具体方法参见链霉菌手册[9]。
1.2.2 菌株平板表型观察将野生型M145、△sco-1135、△sco1135com和M145/pMS82菌株的孢子分别划线于YBP、MS、R2、R2YE、MM和MYM固体培养基上,30℃倒置培养,从24 h开始每隔12 h观察记录各个菌株的生长状态和表型差异。
1.2.3 菌体生长曲线、放线紫红素和十一烷基灵菌红素产量的测定利用YBP固体培养基对不同时间点M145、△sco1135和△sco1135com的菌体生长量及天蓝色链霉菌中ACT和RED的产量进行测定,具体参见Yu等[14]方法。
1.2.4 M145野生型菌株和突变菌株的部分基因转录水平分析利用试剂盒进行天蓝色链霉菌野生型和突变型菌株总RNA的提取,得到的RNA与六核苷酸随机引物在65℃下进行退火,在反转录酶M-MLV作用下,将总RNA样品中的mRNA反转录成cDNA。选取部分与孢子形成及产素有关的基因,根据序列设计特异性引物,以M145基因组中的hrdB基因为内参,以cDNA为模板,对M145野生型菌株和突变菌株中部分基因进行qRT-PCR分析,定量测定基因表达水平的差异。
2 结果 2.1 sco1135突变菌株和回补菌株的构建及验证我们通过同框缺失突变的方法获得了突变菌株△sco1135和回补菌株△sco1135com,利用表 2中引物1135-Verify-Forward和1135-Verify-Reverse对△sco1135和△sco1135com进行PCR验证。理论上,发生双交换的突变株会扩增出一条1 591 bp的条带,由于回补DNA片段整合到突变株基因组上的位置并不是整合到sco1135的原先位置,所以扩增可以得到两条带:1 591 bp的回补片段扩增条带和679 bp的sco1135基因原位缺失后的扩增条带。
如图 1-D所示,野生型M145扩增出一条约679 bp的条带(泳道1) ;突变菌株(泳道2) 对应的扩增条带1 591 bp,回补菌株扩增出一条野生型条带和一条突变型条带,与理论(图 1)相一致。

|
| 图 1 △sco1135和△sco1135com构建原理图及菌株验证 A-C:野生菌株、突变菌株和回补菌株的构建原理图;D:野生菌株和突 变菌株的验证图。M:DNA Marker;1:野生菌株;2:突变菌株;3:回补菌株 |
本研究分别在MS、R2、R2YE、MM、YBP和MYM培养基上对M145野生型、△sco1135、△sco1135com及M145/pMS82对照菌株的孢子生长过程进行表型观察。发现四者在MS、R2、R2YE、MM和MYM固体培养基上的孢子形成过程基本相同。如图 2所示,在YBP培养基上,M145在60 h时开始生长出气生菌丝,随着培养时间的延长,ACT扩散到培养基中,气生菌丝分化成孢子,120 h M145已产生大量孢子,并且孢子丰满,颜色为灰色;而△sco1135产孢明显延迟于野生型M145;pMS82空载对照菌株形态发育基本同步于突变菌株。

|
| 图 2 M145、△sco1135、△sco1135com和M145/pMS82在YBP培养基上产孢对比 |
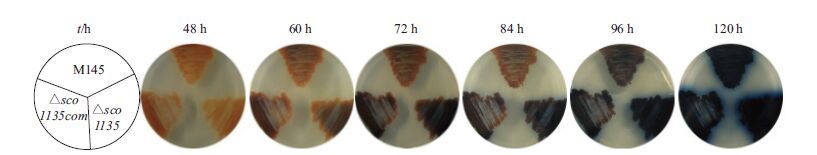
为了研究sco1135 对M145次级代谢产物的影响,我们在YBP培养基上分别培养M145、△sco1135及△sco1135com进行抗生素定量观察。如图 3所示,野生型M145生长至60 h时开始产生ACT,ACT产量随着培养时间的延长不断累积,120 h培养基整体呈现深蓝色;72 h后,△sco1135培养基颜色深于同期的M145。
|
| 图 3 M145、△sco1135和△sco1135com在YBP培养基上次级代谢差异 |
检测不同发酵时间点YBP固体培养基和菌体的上清液吸光度值OD640。定量ACT的产生水平,如图 4-A和B显示,后期培养基和菌体中突变菌株的ACT产量是野生菌株的2-3倍,测定结果经统计学分析发现,72 h后同一时间点野生菌株和突变菌株中的ACT产量均具有显著差异。

|
| 图 4 M145、△sco1135和△sco1135com的ACT、RED产量和生长曲线对比 |
检测不同发酵时间点的菌体沉淀甲醇盐酸浸出液的、吸光度值OD530定量RED的产生水平,结果(图 4-C)显示,△sco1135在各个时间点的RED产生水平跟野生型并没有明显差异,与培养基观察结果(图 3)基本一致。
同时对M145野生型、突变型及回补菌株的菌体干重进行了定量分析,结果(图 4-D)显示,突变菌株和回补菌株的菌体干重接近同期野生型。
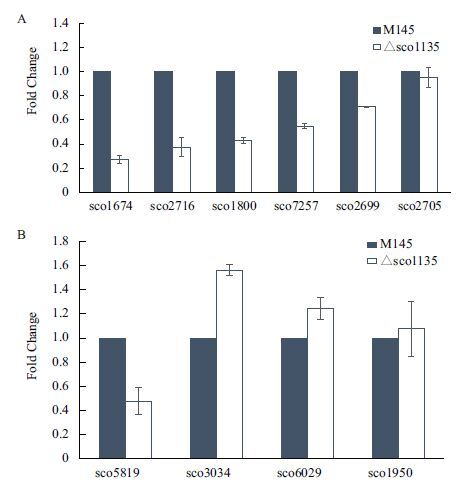
2.4 孢子形成相关基因的转录水平分析我们选取部分与形态发育相关的基因:whi(sco3034、sco6029、sco1950、sco5819) 、ram(sco6681、sco6682) 、bld(sco1489、sco5723) 、chp(sco1674、sco1800、sco2699、sco2716、sco7257、sco2699) ,对它们在M145和△sco1135中的表达水平进行定量分析。表型观察(图 2)显示,M145在60 h时已经生长出气生菌丝,说明在60 h前M145和△sco1135生长情况已经开始发生变化,所以我们提取M145和△sco1135在YBP培养基上生长48、60和72 h的RNA,利用qRT-PCR对whi(sco3034、sco6029、sco1950、sco5819) 、ram(sco6681、sco6682) 、bld(sco1489、sco5723) 和chp(sco1674、sco1800、sco-2699、sco2716、sco7257、sco2705) 基因的表达水平进行了定量分析。
实验结果表明48 h时产孢相关基因在突变株和野生型中的表达量有明显差异,我们将每个基因在M145中48 h的表达值设定为1,突变株的数值为与M145中相应表达量的比值。chp基因分别在M145和△sco1135中48 h的表达水平比较(图 5-A)显示,sco1800、sco2699、sco7257和sco2705在M145和△sco1135中的表达水平并没有明显的差异,而sco2716(chpA)和sco1674(chpC)在M145中的表达量分别是在△sco1135中表达量的2.5倍和5倍;whi基因分别在M145和△sco1135中48 h的表达水平比较(图 5-B)表明,M145中sco5819(whiH)的表达水平约是在△sco1135中表达水平的2倍,而sco3034、sco6029和sco1950在M145和△sco1135中的表达水平并没有明显差异;检测的其余部分ram、bld基因的表达量在M145和△sco1135中基本持平。以上结果说明sco1135能够调控chpA、chpC和whiH基因的表达来调控孢子的形成。
|
| 图 5 qRT-PCR检测M145和△sco1135中chp(A)和whi(B)系列部分基因转录水平 |
选取部分与次级代谢产物ACT合成相关的基因act(sco5085、sco5072、sco5086) ,对它们在M145和△sco1135中的表达水平进行定量分析。结果发现,72 h时基因在M145和△sco1135中的表达水平有明显差异,将每个基因在M145中72 h的表达值设定为1,突变株的数值为与M145中相应表达量的比值。结果(图 6)显示,sco5085、sco5072和sco5086在△sco1135中的表达水平约是在M145中表达水平的13-20倍,说明sco1135可通过调控sco5085、sco5072和sco5086的表达调控ACT的合成。

|
| 图 6 qRT-PCR检测M145和△sco1135中act系列部分基因转录水平 |
Brandan Hillerich教授课题组研究的天蓝色链霉菌M145中sco1135基因的缺失导致突变株在培养基MYM上不产孢,与孢子产生相关的基因whiG(sco6021) 、whiB(sco3034) 和whiH(sco5819) 在突变菌株中均不表达;该课题组的另一研究表明,sco1135基因产物是一种DNA结合蛋白,能够直接与sco1135和sco1134间的基因间隔区反应抑制转录,增强黄嘌呤脱氢酶的活性[15]。与该研究不同的是,本研究构建了sco1135的突变菌株△sco1135和回补菌株△sco1135com,通过表型分析、抗生素定量观察发现△sco1135在YBP培养基上出现产孢延迟,ACT产量提高的现象,因此我们通过qRT-PCR对sco1135部分产孢相关基因(whi、ram、bld、chp)和产ACT相关基因(act)进行了分析。其中whi基因是M145产孢所必需的基因[16],sco3034、sco6029、sco1950、sco5819基因在气生菌丝和孢子发育过程中起到关键作用;ram基因簇(sco6681-sco6685) 是M145孢子发育过程中的关键基因[17];bld基因簇包括bldA、sco1489、sco5723、sco3323等13个基因,sco1489(bldD)是M145孢子发育过程中起中心作用的调节子,负调控其它bld基因[18];chp基因簇包含M145气生菌丝脱离基质菌丝周围水性环境生长到空气中所必需的基因[19],包括sco1674、sco1675、sco1800、sco2699、sco2716、sco2717、sco2718、sco7257八个基因。通过数据分析发现,48 h时chpA、chpC和whiH基因在突变菌株中的表达水平较野生型中降低了50%-75%,与表型分析结果一致,说明sco1135能够调控chpA、chpC和whiH基因的表达来调控孢子的形成。其次我们检测了部分act基因(sco5085、sco5072、sco5086) 在M145和△sco1135中表达水平的差异,act能够特异性调控天蓝色链霉菌中ACT的合成,act基因簇包括sco5072-sco5092,sco5085是调控基因,它可以正调控M145中放线菌素的合成,其基因缺失将导致放线菌素合成基因簇中部分基因不转录[20, 21];sco5072和sco5086是结构基因,分别编码羟脂酰-CoA脱氢酶和酮脂酰还原酶。转录水平分析结果发现72 h时sco5085、sco5072、sco5086在突变株中的转录水平较野生型提高13-20倍,因此sco1135可以通过调控sco5085、sco5072和sco5086的表达来调控ACT的合成。
4 结论本研究构建了sco1135的缺失突变菌株△sco-1135和回补菌株△sco1135com发现,△sco1135在MYM培养基上能够正常生长,而在YBP培养基上出现产孢延迟和ACT产量提高的现象,△sco1135com的孢子生长和抗生素ACT产量基本与同期野生菌株一样。此外,转录水平分析发现,sco1135能够调控基因sco2716、sco1674、sco5819、sco5085、sco5072和sco5086的表达,从而间接参与调控M145的孢子形成及次级代谢产物ACT的产生。
| [1] | Challis GL, Hopwood DA. Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Str-eptomyces species. Proc Natl Acad Sci USA, 2003, 100 (Suppl 2): 14555–14561. |
| [2] | Hopwood DA. Therapeutic treasures from the deep. Nat Chem Biol, 2007, 3 (8): 457–458. DOI:10.1038/nchembio0807-457 |
| [3] | Nodwell JR. Novel links between antibiotic resistance and antibiotic production. J Bacteriol, 2007, 189 (10): 3683–3685. DOI:10.1128/JB.00356-07 |
| [4] | Bentley SD, Chater KF, Cerde?o-Tárraga AM, et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature, 2002, 417 (6885): 141–147. DOI:10.1038/417141a |
| [5] | Hwang KS, Kim HU, Charusanti P, et al. Systems biology and biotechnology of Streptomyces species for the production of secondary metabolites. Biotechnol Adv, 2014, 32 (2): 255–268. DOI:10.1016/j.biotechadv.2013.10.008 |
| [6] | Gomez-Escribano JP, Song L, Fox DJ, et al. Structure and biosynthesis of the unusual polyketide alkaloid coelimycin P1, a metabolic product of the cpk gene cluster of Streptomyces coelicolor M145. Chemical Science, 2012, 3 : 2716–2720. DOI:10.1039/c2sc20410j |
| [7] | Liu G, Chandra G, Tan H, et al. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiol Mol Biol Rev, 2013, 77 (1): 112–143. DOI:10.1128/MMBR.00054-12 |
| [8] | Wang W, Shu D, Lu Y, et al. Cross-talk between an orphan response regulator and a noncognate histidine kinase in Streptomyces coelicolor. FEMS Microbiol Lett, 2009, 294 (2): 150–156. DOI:10.1111/fml.2009.294.issue-2 |
| [9] | Kieser T, Bibb M, Buttner M, et al. Practical Streptomyces Genetics:a laboratory Manual[M]. Norwich: John Innes Foundation, 2000. |
| [10] | Gust B, Challis GL, Fowler K, et al. PCR-targeted Streptomyces gene replacement identifies a protein domain needed for biosynthesis of the sesquiterpene soil odor geosmin. Proc Natl Acad Sci USA, 2003, 100 : 1541–1546. DOI:10.1073/pnas.0337542100 |
| [11] | Gregory MA, Till R, Smith MC. Integration site for Streptomyces phage phiBT1 and development of site-specific integrating vectors. Journal of Bacteriol, 2003, 185 (17): 5320–5323. DOI:10.1128/JB.185.17.5320-5323.2003 |
| [12] | Gust B, Kieser T, Chater KF. Redireccted technology:PCR-targeting sysrem in Streptomyces coelicolor A3(2):a laboratory manual[M]. Norwich: John Innes Foundation, 2002. |
| [13] | Sambrook J, Russell D. Molecular Cloning:A Laboratory Manual[M]. New York: Cold Spring Harbor Laboratory Press, 2001. |
| [14] | Yu Z, Zhu H, Dang F, et al. Differential regulation of antibiotic biosynthesis by DraR-K, a novel two-component system in Strepto-myces coelicolor. Mol Microbiol, 2012, 85 (3): 535–556. DOI:10.1111/j.1365-2958.2012.08126.x |
| [15] | Hillerich B, Westpheling J. A new TetR family transcriptional regulator required for morphogenesis in Streptomyces coelicolor. Journal of Bacteriol, 2008, 190 (1): 61–67. DOI:10.1128/JB.01316-07 |
| [16] | Chater KF. Regulation of sporulation in Streptomyces coelicolor A3(2):A checkpoint multiplex?. Current Opinion in Microbiology, 2001 (4): 667–673. |
| [17] | Keijser BJ, van Wezel GP, Canters GW, et al. The Ram-dependence of Streptomyces lividans difference is by bypassed by copper. J Molecul Microbiol Biotechnol, 2014, 82 (6): 1093–1098. |
| [18] | Kim JM, Won HS, Kang SO. The C-terminal domain of the transcriptional regulator BldD from Streptomyces coelicolor A3(2)constitutes a novel fold of winged-helix domains. Proteins, 2014, 82 (6): 1093–1098. DOI:10.1002/prot.v82.6 |
| [19] | Bibb MJ, Domonkos A, Chandra G, et al. Expression of the chaplin and rodlin hydrophobic sheath proteins in Streptomyces venezuelae is controlled by σBldN and a cognate anti-sigma factor, RsbN. Molecular Microbiology, 2012, 84 (6): 1033–1049. DOI:10.1111/j.1365-2958.2012.08070.x |
| [20] | Taguchi T, Itou K, Ebizuka Y, et al. Chemical characterisation of disruptants of the Streptomyces coelicolor A3(2)actVI genes involved in actinorhodin biosynthesis. Journal of Antibiotics, 2000, 53 (2): 144–152. DOI:10.7164/antibiotics.53.144 |
| [21] | Iqbal M, Mast Y, Amin R, et al. Extracting regulator activity profiles by integration of de novo motifs and expression data:characterizing key regulators of nutrient depletion responses in Streptomyces coelicolor. Nucleic Acids Res, 2012, 40 (12): 5227–5239. DOI:10.1093/nar/gks205 |






