合成生物学 ( synthetic biology ) 是以系统生物学的研究为基础,使用工程化设计思路,串联构建标准化的元器件和模块,以获得具有新的生物功能的生命体系,或者从头合成全新的人工生物体系,其示意图如图 1 所示。其研究内容可以大体分为3个层次:一是利用已知功能的天然生物模体 ( motif ) 或模块 ( module ) 构建成新型调控网络并表现出新功能;二是采用从头合成 ( de novo synthesis ) 的方法,人工合成基因组 DNA 并重构生命体;第三个层次则是在前两个研究领域得到充分发展之后,创建完整的全新生物系统乃至人工生命体 ( artificial life ) 。
|
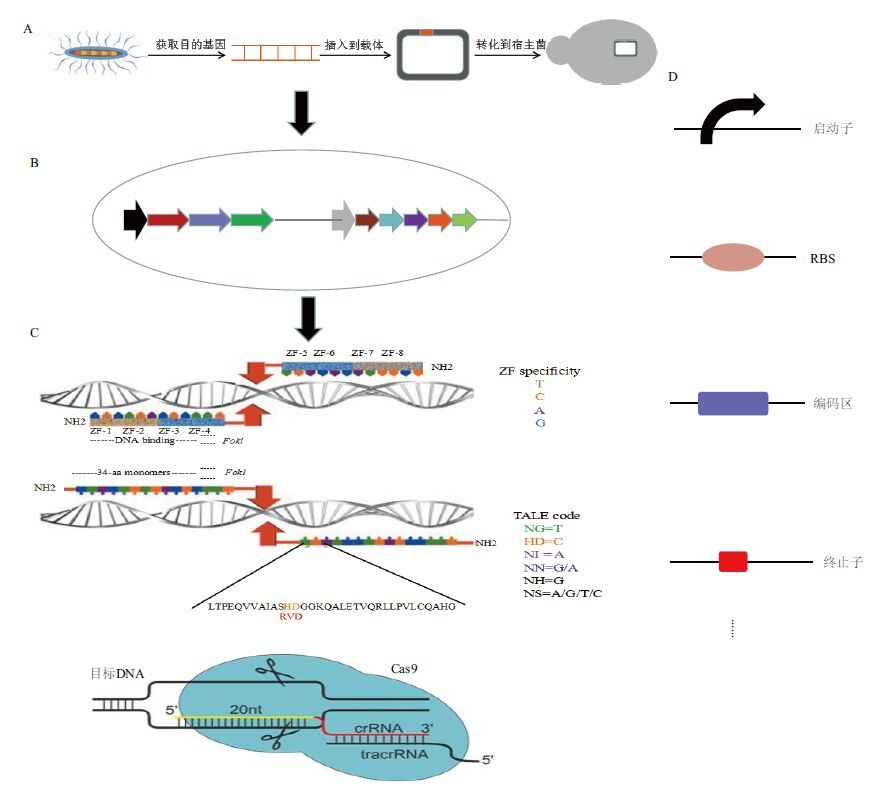
| 图 1 合成生物学的原理 A:基因重组的过程;B:模块化不同的基因;C:基因编辑的3种方法;D:基因重组的基本元件 |
合成生物学发展到今天已有上百年的历史,但直到1953年,沃森和克里克发现了DNA 双螺旋结构,开启了分子生物学时代,使遗传学研究深入到分子水平才真正为合成生物学奠定了实际的基础。此后,合成生物学开始以迅猛的速度发展,1972年,Jackson和Berg[1]博士等通过将细菌病毒的DNA拼接到猴子病毒 SV40.1中,创建了首例重组DNA分子;1973 年,Cohen[2]首次将DNA片段与质粒连接,并转化入大肠杆菌 ( Escherichia coli ) ,这为后来构建异源代谢途径奠定了基础;1978年,诺贝尔生理或医学奖颁给发现DNA 限制酶的纳森斯 ( Daniel Nathans ) 、亚伯 ( Werner Arber ) 和史密斯 ( Hamilton Smith ) ,当时斯吉巴尔斯基在《基因》杂志中写了一段评论:限制酶将带领我们进入合成生物学的新时代[3];1980年,Hobom[4]开始用合成生物学的概念来表述基因重组技术。此后,聚合酶链式反应 ( PCR ) 技术快速发展,成为生物学研究中的极为重要的工程技术,而基因测序技术也由此得以进步。在基因组学获得巨大成功的基础上,2000年,E. Kool在美国化学年会上重新定义“合成生物学”为基于系统生物学的遗传工程,用基因片段、人工碱基 DNA 分子、基因调控网络与信号转导路径人为的设计与构建具有新的生物功能的生命体系。这是一门涉及微生物学、分子生物学、系统生物学、遗传工程、材料科学以及计算科学等多个领域的综合性交叉学科。
1 基本元件与基因重组技术 1.1 基本元件启动子、基因编码序列、终止子、转录调控蛋白因子、核糖体结合位点 ( RBS ) 、调控小 RNA 分子等都是合成生物学的基本元件。人们开始系统定量表征自然界的天然元件,并开发了许多基于天然元件的突变人工元件。除了上述基础的元件外,人们还开发了一些新型的元件,如基因间序列元件、DNA 位点标签元件、RNA 适配子元件等,并着力不断扩大这些人工元件的种类和数量。这些元件共同构成合成生物学的元件库。
1.2 基因重组技术基因重组技术又称重组DNA技术 ( recombinant DNA technique ) ,按照人们的意愿,利用重组DNA的工具在体外重新组合脱氧核糖核酸 ( DNA ) 分子,再将重组分子导入到受体细胞中,并使它们在相应的细胞中增殖的遗传操作。这种操作可把特定的基因组合到载体上,并使之在受体细胞中增殖和表达,因此不受亲缘关系限制。
1972年,斯坦福大学的生物化学家Berg[1]博士通过将细菌病毒的DNA拼接到猴子病毒SV40.1 中,创建了首例重组DNA分子;1973年,Cohen[2]首次将DNA片段与质粒连接,并转化入大肠杆菌 ( Escherichia coli ) ;1978年,诺贝尔奖颁给了发现核酸限制性酶的Daniel Nathans等[3],限制酶的发现带我们进入合成生物学的新时代。此后,聚合酶链式反应 ( PCR ) 技术快速发展,成为生物学研究中的极为重要的工具。科学家们运用PCR技术获取DNA片段,然后利用酶切、酶连、转化等重组DNA的手段,构建了一系列的代谢途径,大大的促进了合成生物学的发展。2009年,Gibson[5]博士和他的同事开发了一种组装多条线性DNA片段的方法,即Gibson assembly,运用该方法重组代谢途径,可以省去酶切、酶连等步骤,大大节省了实验时间,并且重组效率也有明显提高。
合成生物学的目标就是简化设计和构建生命体系的过程,使生物合成更简单快捷[6]。Knight[7]建立了一套新的名为“BioBricks”的克隆策略,使生物组件的标准化装配成为可能,就像传统的机械制造那样,各种组件具备一定的标准的接口,它们之间能以标准的方式连接装配形成更大的组件。每个BioBrick都是一段DNA,它包含特定的信息以及编码相对应的特定功能。例如,启动子、核糖体结合位点、蛋白质编码序列、终止子,或是它们的组合。每个Biobrick 的上下游包含特定的酶切位点,只需要通过酶切连接反应,就可以将任意一个标准化后的BioBrick组件插入到其他的BioBrick组件的上游或者下游,并且,新的组合序列仍然是标准化的BioBrick组件。迭代这样的操作,可以利用简单的手段,从简单的组件出发,构建大规模的复杂系统。Shetty等[6]利用BioBrick组件顺利的构建了7个BioBrick载体,并利用这些载体成功组装了其他的标准化的BioBrick组件。
2 代谢途径的模块化表达越来越多的研究表明,利用合成生物学工具,模块化表达不同功能的异源基因,能够有效的协调大量异源基因的高表达,优化代谢途径,避免过量代谢中产物的积累,提高目的产物的积累效率,另外,还可以促进人工代谢途径的构建[8-12]。在代谢工程中,为了得到有价值的目的产物,科学家们需要克服一些基因的过表达、不匹配的辅因子间的氧化还原不平衡、有害中间产物的富集等障碍,这就需要借助分子生物学的手段模块化表达不同功能的基因,然后选择最适合的模块组合导入相应的代谢途径中,以得到最优的代谢途径 ( 图 2-A ) 。1,4-丙二醇 ( 1,4-Butanediol,BDO ) 是一种合成高附加值纤维材料的重要前体。由于BDO在生物体内并没有发现现成的代谢途径,所有的代谢途径都需要从异源生物中获得[8]。Genomatica公司利用Biopathway prediction algorithms的热力学模型[13]研究了符合热力学规律的潜在BDO合成途径,筛选出7个最优的反应步骤[8]。随后,以4-羟基丁酸 ( 4-hydroxybutyrate ) 为分界点,将7个异源基因分为上游和下游两个模块,每个模块分别有 4个和 3个异源基因。上游模块和下游模块基因表达经过优化后,被转入大肠杆菌中表达,从无到有实现了 18 g/L BDO的产量。
对于不同模块的调控,则通过调整控制模块转录的启动子的强弱组合实现。现阶段最成熟的方法是通过建立功能启动子库,然后筛选不同强弱的启动子,通过组合控制模块转录的不同强度的启动子,最后获得兼顾目的产物积累和细胞生长的启动子组合策略。例如,Zhang等[14]通过理性调节3个模块,创建了生产脂肪酸乙酯 ( fatty acid ethyl ester,FAEE ) 的代谢网络。3个模块中A模块的产物是脂肪酸,B模块的产物是乙醇,C模块的产物是生物柴油,为了提高FAEE的产量,调节3个模块的启动子强弱的不同组合,最后使FAEE的产量达到1.5 g/L ( 图 2-B ) [14]。
3 基因组编辑在合成生物学的应用中,基因组编辑 ( Genome editing ) 是异源合成途径的重构及模块优化的基本操作手段。所谓基因组编辑是在基因组水平上对DNA 序列进行定点改造修饰的遗传操作技术[15]。该技术被誉为“后基因组时代生命科学研究的助力器”。其原理是通过构建一个人工内切酶,在靶位点切断DNA,产生 DNA双链断裂 ( Double-strand break,DSB ) ,进而诱导细胞内的 DNA修复系统进行非同源末端连接 ( Nonhomologous end joining,NHEJ ) 和同源重组修复 ( Homologous recombination,HR ) 。通过这两种修复途径,基因组编辑技术可以实现定点基因敲除、特异突变引入和定点修饰。基因组编辑可分为4类,即锌指核酸酶 ( Zinc-finger nuclease,ZFN ) 技术、类转录激活因子核酸酶 ( Transcription activator-like effector nuclease,TALEN ) 技术、成簇规律间隔短回文重复 ( Clustered regulatory interspac-ed short palindromic repeat,CRISPR ) 技术以及多重自动基因组改造 ( multiplex automated genome engine-ering,MAGE ) /接合组装基因组改造 ( conjugative assembly genome engineering,CAGE ) 。前3种技术的联系与区别,见表 1。
第一个使用定制DNA核酸内切酶的基因组编辑策略就是锌指核酸酶 ( 简称ZFN ) 。ZFN是异源二聚体,其中每个亚基含有一个锌指结构域和一个FokⅠ核酸内切酶结构域,ZFN技术是通过这两部分发挥作用的。锌指蛋白 ( 简称ZFP ) 通过与特定的靶序列结合发挥重要的转录调控作用,不同的ZFP具有类似的Cys2、His2或Cys4结构框架,ZFP结合DNA的特异性与框架外特定氨基酸的变异有关。FokⅠ是一种非特异性的核酸内切酶,FokⅠ结构域必须二聚化才具备核酸酶活性[16]。将人工构建的锌指蛋白与改造后的FokⅠ限制性内切酶融合,就得到ZFN,它能靶向切割特定序列,产生DSB。每对ZFNs的结合序列的间隔区域通常为5-7 bp,以确保FokⅠ二聚体的形成[17]。一般采用模块组装法和寡聚体库工程化筛选构建法来构建ZFN,ZFNs的构建需要花费的时间长,工作量也较大,目前只有少数实验室在运用这一技术平台。
3.2 类转录激活因子核酸酶技术 ( TALEN技术 )TALEN技术是基于TALE ( transcription activat-or-like effector ) 结构域的基因打靶技术,TALE是植物病原体黄单胞菌分泌的一类效应蛋白因子,能够特异的结合DNA,此结合域有一段高度保守的重复单元,该单元中仅第12和13位的氨基酸不同。TALEN中通常使用的切割结构域来自于无序列特异性的FokⅠ核酸内切酶,FokⅠ的使用与优化主要得益于ZFN的研究成果[18-20]。需要说明的是,由于每个TALE单体只靶向1个核苷酸,针对每一个靶位点的上下游各设计一个TALEN,FokⅠ通常需要以二聚体的形式发挥其切割DNA序列的功能[21]。TALENS技术很好地解决了ZFNS技术存在的构建困难、成本高及周期长等问题。但是TALEN技术也并非完美无缺,由于针对不同靶点,每次都需构建新的TALE array,工作程序繁琐。
3.3 CRISPR/Cas9CRISPR/Cas9是继“锌指核酸内切酶 ( ZFN ) ”、“类转录激活因子效应物核酸酶 ( TALEN ) ”之后出现的第3代“基因组定点编辑技术”。与前两代技术相比,成本低、制作简便、快捷高效的优点,使其迅速风靡于世界各地的实验室,成为科研、医疗等领域的有效工具。
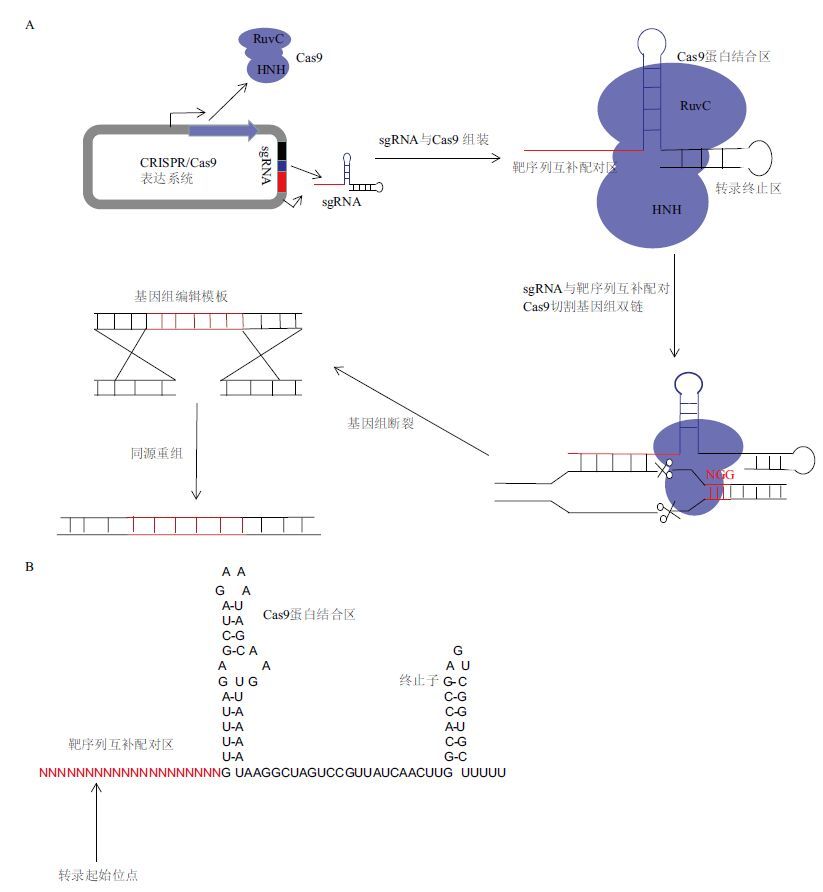
CRISPR是一种来自细菌降解入侵的病毒DNA或其他外源DNA的免疫机制[22]。在细菌及古细菌中,CRISPR系统共分成3类,其中Ⅰ类和Ⅲ类需要多种CRISPR相关蛋白 ( Cas蛋白 ) 共同发挥作用,而Ⅱ类系统只需要一种Cas蛋白即可,这为其能够广泛应用提供了便利条件[23]。CRISPR/Cas9通过将入侵噬菌体和质粒DNA的片段整合到CRISPR中,并利用相应的CRISPR RNAs ( crRNAs ) 来指导同源序列的降解,从而提供免疫性[24]。目前,在基因组定向编辑方面,运用最广泛的是源自于生脓链球菌 ( Streptococcus hyogenes ) 的Type II CRISPR/Cas9系统。该系统的工作原理是crRNA ( CRISPR-derived RNA ) 通过碱基配对与 tracrRNA ( Trans-activating RNA ) 结合形成 tracrRNA/crRNA复合物,此复合物引导核酸酶 Cas9蛋白在与crRNA配对的基因组序列靶位点剪切双链DNA,形成双链缺口 ( Double strand break,DSB ) 。然后细菌通过非同源重组 ( non-homologous end joining,NHEJ ) 或同源重组 ( homo-logous recombination,HR ) 两种方式修复DSB[25]。近来,研究表明,通过基因工程手段对crRNA和tracrRNA进行改造,将其连接在一起得到sgRNA ( single-guide RNA ) 。融合的RNA具有与野生型RNA类似的活力,但由于结构得到了简化,故更方便研究者使用。通过将表达sgRNA的原件与表达Cas9的原件相连接,得到可以同时表达两者的质粒,将其转染细胞,便能够对目的基因进行定点切割 ( 图 3-A ) [26]。基因工程手段得到的sgRNA一般包括3个部分 ( 图 3-B ) :靶序列互补配对区 ( Base-pairing region,20 nt ) 、Cas9蛋白结合区 ( Cas9 handle Region,42 nt ) 、转录终止区 ( terminator,40 nt ) 。sgRNA引导Cas9蛋白识别基因组上的PAM ( Protospacer adjacent motif ) 序列 ( S. hyogenes Cas9识别序列为NGG ) ,并与PAM序列的5'端20个核苷酸序列互补配对,然后Cas9蛋白在PAM序列上游 5'端第3个碱基处切割形成 DSB[27]。由于PAM序列结构简单 ( 5'-NGG-3' ) ,几乎可以在所有的基因中找到大量靶点,并且该技术具有准确、高效、简便的特点,因此得到广泛的使用。CRISPR-Cas9系统已经成功应用于植物、细菌,如大肠杆菌[28]、酵母、果蝇[29]、鱼类及哺乳动物如人类、小鼠、斑马细胞等[30]物种的基因组编辑,是目前最高效的基因组编辑系统。
|
| 图 3 II型CRISPR/Cas9 A:II型CRISPR/Cas9的原理图;B:sgRNA的结构图 |
基因组的自由编辑一直是生物学家的梦想,但大部分DNA编辑工具均作用缓慢、价格昂贵且效率低下。为了解决这个问题,哈佛医学院等机构开发出快速且简单的基因组规模编辑工具,能够利用“查找和替换”改写活细胞的基因组。TAG终止密码子是大肠杆菌基因组中最稀有的,只有314个,这也让它成为替换的首要目标。研究人员利用多重自动基因组改造 ( MAGE ) 这种方法,用同义的TAA密码子位点来特异性地替换32种大肠杆菌菌株中的TAG终止密码子[31]。Wang等[32]曾于2009年在《自然》杂志上介绍过这种方法。他们利用这种方法能够测定单个的重组频率,证实每次修饰的可行性,并鉴定出相关的表型。 MAGE这种小规模的改造方法替换了部分但不是全部的TAG密码子。之后,利用细菌天然的接合能力,研究人员诱导细胞以更大规模转移包含TAA密码子的基因。这种新方法称为接合组装基因组改造 ( CAGE ) 。这使其能够从根本上改造基因组,从几个到几兆核苷酸[31]。基因工程发展至今,人们已能精确的操作微生物的基因型,但是在发展上受到设计要求的限制。在MAGE发展的基础上,Quintin等[33]利用Merlin ( http://merlincad.org ) 通过计算和设计,与OptMAGE相比消除了E. coli基因组中7个Avr II的限制位点,不但提高了重组效率,而且减少了脱靶效应。
4 在各领域中的应用合成生物学发展的原动力是人们对可再生性生物燃料的追求,人们期望利用合成生物学的方法,将自然界中大量存在的可再生的糖类和纤维素物质转化燃料,以取代石油等不可再生能源。而现今合成生物学的应用已经扩展至很多领域,如生物燃料生产、天然产物合成、生物医药、合成新物种等。
4.1 在生物能源领域的应用能源问题已不是单纯的能源问题,它已经延伸到了政治、经济、军事等各个方面,早在20 世纪,能源问题就引起了各个国家的高度重视,如何开发清洁、经济、可再生的能源,成为各国科学家们关注的热点。随着基因重组、分子克隆、基因编辑等合成生物学技术的发展,通过生物手段来解决能源问题也成为一种可能和必然趋势。目前已有许多科学家利用合成生物学手段,在生产生物能源的领域取得了一定的进展。
4.1.1 合成生物学在氢气能源开发上的应用氢气作为燃料是一种非常清洁的能源,“零排放”的特点使其成为应用潜力巨大的能源之一。随着合成生物学的迅速发展,使用生物的手段生产氢,也成为生物科学家们关注的热点。美国弗吉尼亚理工大学的 Zhang等[34]运用合成生物学技术,将13种已知的酶组成一个非天然的新的催化体系,使淀粉和水在温和条件下生产氢成为现实,实现了氢的生物制造。
4.1.2 合成生物学在有机燃料合成上的应用Sonderegger等[35]在啤酒酵母导入异源的磷酸转乙酰酶和乙醛脱氢酶,发酵得到乙醇,使得用醋酸为底物发酵乙醇产量提高20%,用木糖发酵乙醇产量提高了40%。Trinh等[36]通过定向基因编辑,去除大肠杆菌中乙醇代谢的竞争途径,使碳流朝乙醇的方向富集,提高乙醇的产量。由于乙醇燃料具有腐蚀性、吸水性及易挥发性,其作为燃料的大规模使用具有一些潜在的隐患,目前很多科学家将目光更多地集中于一些高碳醇燃料上,如异丙醇、1-丁醇、异丁醇和高级脂肪醇等。Hanai等[37]通过将异源的丙酮丁醇梭菌中的异丙醇代谢通路中的关键基因整合入大肠杆菌,实现了异丙醇在大肠杆菌中的高效表达,产量达到了4.9 g/L。Atsumi等[38]通过将编码丙酮丁醇梭菌的1-丁醇代谢通路中的关键酶基因插入大肠杆菌,同时阻断大肠杆菌本身存在的与1-丁醇生产竞争乙酰辅酶A和NADH的代谢通路,与野生型相比,1-丁醇的产量提高了约3倍,达到373 mg/L。该课题组通过整合枯草芽孢杆菌中的硫解酶 ( thiolase ) 基因thl、大肠杆菌中的乙酰辅酶A乙酰转移酶 ( acetyltransferase ) 的基因atoB,实现了1-丁醇在大肠杆菌中的生产提高3倍以上。Steen 等[39]在现有单糖到脂肪酸的合成途径的基础上,在大肠杆菌中引入异源的脂肪酸到脂肪醇的代谢相关酶,其中包括fadD ( 来自大肠杆菌的酰基辅酶 A 合成酶 ) 、acrI ( 来自不动杆菌ADPI的acyl-CoA 还原酶 ) 等,成功构建了由单糖到脂肪醇的整体代谢途径。通过表达acrI和fadD,同时阻断了脂肪酸的其他代谢通路,实现了脂肪醇在大肠杆菌中的高效表达。同时,该研究小组还利用合成生物学方法对大肠杆菌和酿酒酵母进行改造,实现了一种药烷型倍半萜烯的高效表达,这种药烷型倍半萜烯加氢反应后可作为一种新型的绿色燃料[40]。Deng和Fong[41]将异源的adhE2基因整合入褐色嗜热裂孢菌的基因组内,实现了利用该菌直接发酵生物质底物生产丙醇,产量达 0.48 g/L 。
4.2 合成生物学在化工产品上的应用利用合成生物学技术重组的工程菌,不仅可以用于生产绿色燃料,在工业化工产品领域也发挥着巨大作用。目前,已有科学家利用合成生物学技术改造细胞来实现生产塑料和纺织品的化工前体。己二酸 ( adipic acid或者adipate ) 又称肥酸,是一种重要的二元羧酸,它能够发生成盐反应、酯化反应、酰胺化反应等,并能与二元胺或二元醇缩聚成高分子聚合物[42-44]。当前己二酸最重要的应用领域是合成尼龙类纤维 ( 如尼龙6,6 ) [45, 46]。Niu等[47]通过在大肠杆菌中过量表达人工合成的β-酮己二酸途径,直接发酵葡萄糖生产顺式-己二烯二酸,然后直接利用上述发酵液化学脱氢得到己二酸。Jung和Yang等[48, 49]利用合成生物学技术对菌株进行改造,成功构建了非天然生产环保型可降解性塑料聚羟基脂肪酸 ( PHA ) 的生产和代谢模块,并优化了能够用糖稳定生产PHA 的工艺。美国杜邦公司实现了利用大肠杆菌合成重要的工业原料1,3-丙二醇等[50]。Deng等[51]将谷氨酸棒状杆菌ATCC 13032中的丙酮酸羧化酶 ( pyruvate carboxylase ) 基因插入褐色嗜热裂孢菌的基因组中表达,使苹果酸的产率提高了47.9%。
4.3 合成生物学在医药领域的应用 4.3.1 萜类及生物碱类药物的生物合成萜类 ( terpenoid ) ,是一种在天然宿主中表达量低、结构复杂并具有多个手性中心的小分子物质,其中很多萜类化合物具有较高的药用价值,如具有抗癌作用的紫杉醇和抗疟疾作用的青蒿素。目前萜类的生产主要依赖于植物栽培、组织和细胞培养、生物转化等,尽管已经取得了很大的研究进展,但是仍然面临着产量低、培养过程难等一些问题。通过生物科技手段提高该类化合物的产量具有极高的商业价值和社会意义。目前科研工作者已利用合成生物学手段实现了该类化合物产量的提高。
在自然界中,青蒿素是青蒿产生的倍半萜烯酯的内过氧化物,被WTO推荐为治疗疟疾的首选药物,但在自然界中产量很低。化学方法合成青蒿素十分困难,且成本高昂,使得青蒿素的供应短缺,许多患者无法得到及时治疗。2006年,Ro等[52]利用酿酒酵母 ( Saccharomyces cerevisiae ) 实现了青蒿素的前体物质青蒿酸的生物合成。Keasling 研究组[53]以酿酒酵母S288C为底盘细胞构建表达青蒿素前体物青蒿二烯的酿酒酵母工程菌株,其根据大肠杆菌的密码子偏好性,高表达青蒿二烯合成酶 ( amorphadi-ene synthase,ADS ) 基因,过表达Erg20和tHmgr,以及Upc2转录因子的突变体基因Upc2-1;用Met3启动子弱化鲨烯合成酶的表达来普遍提高甲羟戊酸途径的表达;在此基础上表达青蒿二烯氧化酶 ( AMO ) 、负责青蒿二烯三步氧化的细胞色素 P450 和青蒿细胞色素还原酶 ( AaCPR ) 功能模块,并在酵母中对FPP合成途径进行了5次改造,改造后酵母青蒿酸的合成能力大大提高,合成量达到153 mg/L,是之前报道的最大合成水平的500倍。通过对代谢途径不断改造和优化,目前青蒿素的产量已经得到了若干数量级的提高,最终将青蒿素的合成成本降为原来的1/10。此研究成果有望实现抗疟疾药物的低成本生产,对全世界疟疾治疗的发展起到了巨大的促进作用。为此,Keasling教授被美国《发现》杂志评为2006年最有影响的科学家,并获得高达 4 000万美元的Bill and Melinda Gates基金资助,用于研究青蒿素的产业化生产。
紫杉醇 ( paclitaxel ) 及其衍生物具有抗恶性肿瘤、防治移植硬化动脉、抗瘢痕形成和抗血管生成等作用,目前主要被用于治疗恶性肿瘤。紫杉醇是紫杉次级代谢产生的双萜类物质,但是紫杉醇与青蒿素一样,在自然界中产量不高,且化学合成困难,随着合成生物学技术的发展,紫杉醇的生物合成也成为了生物学家关注的热点。紫杉醇的自然合成是一个复杂的酶促反应,涉及近 20种酶。大致途径为由萜类化合物生物合成的共同前体双 ( 牻牛儿基 ) 二磷酸盐 ( geranylgeranyl diphosphate,GGPP ) 通过紫杉二烯合成酶的作用生成关键中间产物紫杉二烯,其经过酶促反应转为紫杉烷,紫杉烷经过一系列的还原、加羟、酰基转移等修饰反应,形成紫杉醇的最终前体巴卡亭III。2008年,Engels等[54]将紫杉二烯的合成途径转入啤酒酵母中,通过过表达tHmgr1、Upc2-1,并用嗜酸热硫化叶菌牻牛儿基焦磷酸合成酶 ( GGPPS ) 替换红豆杉 GGPPS,并依照酿酒酵母的密码子偏好性对紫杉二烯合成酶基因进行密码子优化,紫杉二烯的产量提高到了 8.7 mg/L。同时酿酒酵母细胞中 GGPP 的含量为 33.1 mg/L,这表明紫杉二烯的产量还有提高的空间。
吗啡是从罂粟未成熟果实的乳汁 ( 阿片 ) 中提取而得,是罂粟的次级代谢产物,属于阿片类生物碱,早在几千年前人们就发现了它的药用价值,有强烈的麻醉、镇痛作用,其镇痛范围广泛,几乎适用于各种严重疼痛包括晚期癌变的剧痛,并且镇痛时能保持意识及其他感觉不受影响。此外,还有明显的镇静作用,能消除疼痛所引起的焦虑、紧张、恐惧等情绪反应,还能引起某种程度的惬意和欣快感,目前仍是不可替代的镇痛药物之一。但目前主要还是依靠从罂粟中提取,其生产受到天气、季节等因素制约,随着合成生物学的发展及广泛应用,很多科学家也把关注点转移到吗啡的生物合成上。合成阿片类生物碱的完整通路中包括20多个步骤,涉及20多种酶,要在酵母中构建这个完整的通路,是个不小的挑战。2015年,这个困难被攻克,2015年的8月13日,Science刊登了美国斯坦福大学 Smolke团队的新工作成果:其在酵母中构建出阿片类生物碱生物合成的完整通路,并成功生产蒂巴因和氢可待因[55]。Smolke团队研究酵母生产阿片类生物碱已有近10年的时间,他们于2014年在酵母中实现了由蒂巴因到吗啡的转化,他们研究发现由蒂巴因 ( thebaine ) 合成吗啡的过程中T6ODM ( thebaine-6-O-demethylase ) ,COR ( co-deinonere ductase ) ,CODM ( codeine-O-demethylase ) 这3种酶的基因拷贝数之比为2:1:3时,吗啡的产量提升约2倍[56]。
4.3.2 合成生物学在疾病治疗上的应用自古以来人们就饱受各种健康问题的困扰,解决人们的健康问题始终是医学及生物学研究的核心热点。随着合成生物学的快速发展和广泛应用,用合成生物学的手段解决健康问题也引起了科学家们广泛的兴趣,除了上述的一些关于药物研发上的贡献,在疾病的疗法上,合成生物学通过构建模块化的生物元件、疾病治疗通路等,创造了许多新的疾病治疗方法。利用合成生物学构建的基因工程菌在缺氧环境中表达的特性,可让其在肿瘤缺氧的组织环境中,制造某种细胞毒性蛋白而启动癌细胞死亡程序,进而杀死癌细胞。还有某些合成的生物药物可能更容易接近肿瘤组织,有选择性地杀死癌细胞。美国加州大学的Voigt课题组设计了一种细菌,可以侵入肿瘤细胞并将其杀死[57]。他们向细菌中引入了多个模块,将细菌对癌细胞的入侵与特定环境信号连接起来。利用数字电路门控思想,使细菌实现了对外界环境的探测。当细菌处于低氧环境并且密度超过一定阈值时,细菌将表达来自假结核耶尔森菌的透明质酸酶 ( invasin ) ,从而杀死癌细胞。我们还可以根据肿瘤特异性和治疗的需要,将其中的特异性模块进行更换,可以实现其对不同种类肿瘤细胞的治疗作用,很好地体现了合成生物学技术在应用上的灵活性。乙硫异烟胺是一种治疗肺结核的药物,主要是通过结合分枝杆菌酶EthA激活药效。但由于体内的EthR抑制EthA基因转录,因此在治疗上乙硫异烟胺总是无法达到预期的效果。Weber等[58]利用合成生物学技术设计了一个哺乳动物的基因线路,构建了一个连有 EthR反式作用因子的报告基因来筛选鉴定 EthR抑制子,进而消除对乙硫异烟胺的拮抗作用,极大地提高了乙硫异烟胺在体内的生物利用率从而大大提高了其抗结核疗效。Kemmer等[59]利用构建的基因回路,将其植入细胞,实现了对体内的尿酸浓度的检测与控制。该回路中,当尿酸浓度低于阈值时,细菌转录抑制物HucR与特异结合的DNA模体HucO抑制下游报告基因与重组黄曲霉菌尿酸酶的表达,当尿酸浓度高于阈值时,HucR的转录抑制作用解除,通过激活报告基因与黄曲霉菌尿酸酶的表达,实现了对体内尿酸浓度的监测与稳控。
4.4 合成生物学在工业污染物检测中的应用通过合成生物学技术,可以利用电路设计思路同生物细胞相结合,设计改造基因线路,提高生物传感性,实现一些化学物质的生物检测。西班牙的Lorenzo教授课题组[60]一直致力于合成生物学框架内的甲苯及其衍生化合物的生物监测研究。该课题组构建了XylR-Pu-Lux 的甲苯类化合物的检测模块,将其整合到恶臭假单胞菌 KT2440 基因组,并通过二步转座策略,剔除抗性基因,实现了符合环境监测要求的甲苯检测工程菌株的构建。密歇根大学的iGEM团队更是在此基础上,在此模块中加入自杀机制,提高了工程菌株的应用价值,降低了其对环境的副作用。该检测平台的建立,体现了合成生物学技术在化合物检测上的应用,该技术思想不仅可用于工业污染物的检测,还具有一定的军事意义,目前已有国家将其应用于地雷生物武器的检测。
5 结语近年来随着合成生物学的发展,很多领域的科学家们对合成生物学投入了高度的热情,积极的利用合成生物学的手段来解决相应的难题,并已在多个领域取得了突破性的进展。然而,其在造福人类的同时,也带来了新一轮的科学伦理的争论。为了方便筛选,很多细菌在改造的同时都导入了抗生素抗性基因,所以理论上讲,合成生物学的某些产物可能会给人类和环境带来一些威胁。但是,我们若能趋利避害,做到一方面要鼓励支持合成生物学研究的发展,充分发挥其对解决环境、能源、健康等问题以及认识生命本质中发挥的重要作用;另一方面要加强对合成生物学的监管,建立必要的准入和审批制度,合成生物学定能给人类和环境带来巨大的积极影响,并成为解决环境、能源、健康等问题的重要手段。
| [1] | Jackson D, Symons RH, Berg P, et al. Biochemical method for inserting new genetic information into DNA of Simian Virus 40:circular SV40 DNA molecules containing lambda phage genes and the galactose operon of Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 1972, 69 (10): 2904–2909. DOI:10.1073/pnas.69.10.2904 |
| [2] | Cohen SN, Chang AC, Boyer HW, et al. Construction of biologically functional bacterial plasmids in vitro. Proceedings of the National Academy of Sciences of the United States of America, 1973, 70 (11): 3240–3244. DOI:10.1073/pnas.70.11.3240 |
| [3] | Fuchs C, Rosenvold EC, Honigman A, et al. A simple method for identifying the palindromic sequences recognized by restriction endonucleases:the nucleotide sequence of the AvaII site. Gene, 1978, 4 (1): 1–23. DOI:10.1016/0378-1119(78)90011-2 |
| [4] | Hobom B. Gene surgery:on the threshold of synthetic biology. Med Klin, 1980, 75 (24): 834–841. |
| [5] | Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 2009, 6 (5): 343–345. DOI:10.1038/nmeth.1318 |
| [6] | Shetty RP, Endy D, Knight TF. Engineering BioBrick vectors from BioBrick parts. Journal of Biological Engineering, 2008, 2 (1): 5. DOI:10.1186/1754-1611-2-5 |
| [7] | Knight T. Idempotent vector design for standard assembly of biobricks[J]. MIT Artificial Intelligence Laboratory;MIT Synthetic Biology Working Group, 2003. |
| [8] | Moon TS, Yoon S, Lanza AM, et al. Production of glucaric acid from a synthetic pathway in recombinant Escherichia coli. Applied and Environmental Microbiology, 2009, 75 (3): 589–595. DOI:10.1128/AEM.00973-08 |
| [9] | Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol. Nature Chemical Biology, 2011, 7 (7): 445–452. DOI:10.1038/nchembio.580 |
| [10] | Martin CH, Dhamankar H, Tseng HC, et al. A platform pathway for production of 3-hydroxyacids provides a biosynthetic route to 3-hydroxy-y-butyrolactone. Nature Communications, 2013, 4 : 1414. DOI:10.1038/ncomms2418 |
| [11] | Paddon CJ, Keasling JD. Semi-synthetic artemisinin:a model for the use of synthetic biology in pharmaceutical development. Nature Reviews Microbiology, 2014, 12 (5): 355–367. DOI:10.1038/nrmicro3240 |
| [12] | Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496 (7446): 528–532. DOI:10.1038/nature12051 |
| [13] | Cho A, Yun H, Park JH, et al. Prediction of novel synthetic pathways for the production of desired chemicals. BMC Systems Biology, 2010, 4 : 35. DOI:10.1186/1752-0509-4-35 |
| [14] | Zhang F, Carothers JM, Keasling JD. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids. Nature Biotechnology, 2012, 30 (4): 354–359. DOI:10.1038/nbt.2149 |
| [15] | 谢科, 饶力群, 李红伟, 等. 基因组编辑技术在植物中的研究进展与应用前景. 中国生物工程杂志, 2013, 33(6): 99–104. |
| [16] | Kim SC, Skowron PM, Szybalski W, et al. Structural requirements forFokI-DNA interaction and oligodeoxyribonucleoti 319de-instructed cleavage. Journal of Molecular Biology, 1996, 258 (4): 638–649. DOI:10.1006/jmbi.1996.0275 |
| [17] | 程曦, 王文义, 邱金龙. 基因组编辑:植物生物技术的机遇与挑战. 生物技术通报, 2015, 31(4): 25–33. |
| [18] | Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci USA, 1996, 93 (3): 1156–1160. DOI:10.1073/pnas.93.3.1156 |
| [19] | Smith J, Bibikova M, Whitby FG, et al. Requirements for double-strand cleavage by chimeric restriction enzymes with zinc finger DNA-recognition domains. Nucleic Acids Res, 2000, 28 (17): 3361–3369. DOI:10.1093/nar/28.17.3361 |
| [20] | Bibikova M, Carroll D, Segal DJ, et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases. Mol Cell Biol, 2001, 21 (1): 289–297. DOI:10.1128/MCB.21.1.289-297.2001 |
| [21] | Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors. Science, 2009, 326 (5959): 1501. DOI:10.1126/science.1178817 |
| [22] | Garneau JE, Dupuis M, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature, 2010, 468 (7320): 67–71. DOI:10.1038/nature09523 |
| [23] | Jansen R, Embden JD, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol, 2002, 43 (6): 1565–1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [24] | Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation. Annual Review of Genetics, 2011, 45 : 273297. |
| [25] | Gasiunas G, Siksnys V. RNA-dependent DNA endonuclease Cas9 of the CRISPR system:Holy Grail of genome editing?. Trends in Microbiology, 2013, 21 (11): 562–567. DOI:10.1016/j.tim.2013.09.001 |
| [26] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNAguided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337 (6096): 816–821. DOI:10.1126/science.1225829 |
| [27] | Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 2013, 8 (11): 2281–2308. DOI:10.1038/nprot.2013.143 |
| [28] | Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31 (3): 233–239. DOI:10.1038/nbt.2508 |
| [29] | Yu Z, Ren M, Wang Z, et al. Highly efficient genome modifications mediated by CRISPR/Cas9 in Drosophila. Genetics, 2013, 195 (1): 289–291. DOI:10.1534/genetics.113.153825 |
| [30] | Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154 (2): 442–451. DOI:10.1016/j.cell.2013.06.044 |
| [31] | Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333 (6040): 348–353. DOI:10.1126/science.1205822 |
| [32] | Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460 (7257): 894–898. DOI:10.1038/nature08187 |
| [33] | Quintin MJ, Ma NJ, Ahmed S, et al. Merlin:Computer-aided oligonucleotide design for large scale genome engineering with MAGE. Acs Synthetic Biology, 2016, 5 (6): 452–458. DOI:10.1021/acssynbio.5b00219 |
| [34] | Zhang YH, Evans BR, Mielenz JR, et al. High-yield hydrogen production from starch and water by a synthetic enzymatic pathway. PLos One, 2007, 2 (5): 1–6. |
| [35] | Sonderegger M, Schümperli M, Sauer U. Metabolic engineering of a phosphoketolase pathway for pentose catabolism in Saccharomyces cerevisiae. Appl Environ Microbiol, 2004, 70 (5): 2892–2897. DOI:10.1128/AEM.70.5.2892-2897.2004 |
| [36] | Trinh CT, Unrean P, Srienc F. Minimal Escherichia coli cell for the most efficient production of ethanol from hexoses and pentoses. Appl Environ Microbiol, 2008, 74 (12): 3634–3643. DOI:10.1128/AEM.02708-07 |
| [37] | Hanai T, Atsumi S, Liao JC. Engineered synthetic pathway for isopropanol production in Escherichia coli. Appl Environ Microbiol, 2007, 73 (24): 7814–7818. DOI:10.1128/AEM.01140-07 |
| [38] | Atsumi S, Cann AF, Connor MR, et al. Metabolic engineering of Escherichia coli for 1-butanol production. Metab Eng, 2008, 10 (6): 305–311. DOI:10.1016/j.ymben.2007.08.003 |
| [39] | Steen EJ, Kang Y, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass. Nature, 2010, 463 (7280): 559–562. DOI:10.1038/nature08721 |
| [40] | Peralta-Yahya PP, Ouellet M, Chan R, et al. Identification and microbial production of a terpene-based advanced biofuel. Nat Commun, 2011, 2 : 483. DOI:10.1038/ncomms1494 |
| [41] | Deng Y, Fong SS. Metabolic engineering of Thermobifida fusca for direct aerobic bioconversion of untreated lignocellulosic biomass to 1-propanol. Metabolic Engineering, 2011, 13 (5): 570–577. DOI:10.1016/j.ymben.2011.06.007 |
| [42] | Van Duuren J, Brehmer B, Mars AE, et al. A limited LCA of bio-adipic acid:Manufacturing the nylon‐6, 6 precursor adipic acid using the benzoic acid degradation pathway from different feedstocks. Biotechnology and Bioengineering, 2011, 108 (6): 1298–1306. DOI:10.1002/bit.23074 |
| [43] | Sato K, Aoki M, Noyori R. A” green” route to adipic acid:Direct oxidation of cyclohexenes with 30 percent hydrogen peroxide. Science, 1998, 281 (5383): 1646–1647. DOI:10.1126/science.281.5383.1646 |
| [44] | Picataggio S, Beardslee T. Biological methods for preparing adipic acid:U. S. Patent 8, 241, 879[P]. 2012-8-14. |
| [45] | Noack H, Georgiev V, Blomberg MRA, et al. Theoretical insights into heme-catalyzed oxidation of cyclohexane to adipic acid. Inorganic Chemistry, 2011, 50 (4): 1194–1202. DOI:10.1021/ic101405u |
| [46] | Li X, Wu D, Lu T, et al. Highly efficient chemical process to convert mucic acid into adipic acid and DFT studies of the mechanism of the rhenium-catalyzed deoxydehydration. Angewandte Chemie International Edition, 2014, 53 (16): 4200–4204. DOI:10.1002/anie.201310991 |
| [47] | Niu W, Draths KM, Frost JW. Benzene-free synthesis of adipic acid. Biotechnology Progress, 2002, 18 (2): 201–211. DOI:10.1021/bp010179x |
| [48] | Jung YK, Kim TY, Park SJ, et al. Metabolic engineering of Escheri-chia coli for the production of polylactic acid and its copolymers. Biotechnol Bioeng, 2010, 105 (1): 161–171. DOI:10.1002/bit.v105:1 |
| [49] | Yang TH, Kim TW, Kang HO, et al. Biosynthesis of polylactic acid and its copolymers using evolved propionate CoA transferase and PHA synthase. Biotechnol Bioeng, 2010, 105 (1): 150–160. DOI:10.1002/bit.v105:1 |
| [50] | Celińska E. Debottlenecking the 1, 3-propanediol pathway by metabolic engineering. Biotechnol Adv, 2010, 28 (4): 519–530. DOI:10.1016/j.biotechadv.2010.03.003 |
| [51] | Deng Y, Mao Y, Zhang XJ. Metabolic engineering of a laboratory-evolved Thermobifida fusca muC strain for malic acid production on cellulose and minimal treated lignocellulosic biomass. Biotechnology Progress, 2016, 32 (1): 14–20. DOI:10.1002/btpr.2180 |
| [52] | Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440 (7086): 940–943. DOI:10.1038/nature04640 |
| [53] | Hale V, Keasling JD, Renninger N, et al. Microbially derived artemisinin:A biotechnology solution to the global problem of access to affordable antimalarial drugs. Am J Trop Med Hyg, 2007, 77 : 198–202. |
| [54] | Engels B, Dahm P, Jennewein S. Metabolic engineering of taxadiene biosynthesis in yeast as a first step towards Taxol(Paclitaxel)production. Metab Eng, 2008, 10 (3-4): 201–206. DOI:10.1016/j.ymben.2008.03.001 |
| [55] | Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349 : 1095–1100. DOI:10.1126/science.aac9373 |
| [56] | Thodey K, Galanie S, Smolke CD. A microbial biomanufacturing platform for natural and semisynthetic opioids. Nat Chem Biol, 2014, 10 : 837–844. DOI:10.1038/nchembio.1613 |
| [57] | Anderson JC, Clarke EJ, Arkin AP, et al. Environmentally controlled invasion of cancer cells by engineered bacteria. Journal of Molecular Biology, 2006, 355 (4): 619–627. DOI:10.1016/j.jmb.2005.10.076 |
| [58] | Weber W, Schoenmakers R, Keller B, et al. A synthetic mammalian gene circuit reveals antituberculosis compounds. Proceedings of the National Academy of Sciences, 2008, 105 (29): 9994–9998. DOI:10.1073/pnas.0800663105 |
| [59] | Kemmer C, Gitzinger M, Daoud-El Baba M, et al. Self-sufficient control of urate homeostasis in mice by a synthetic circuit. Nature Biotechnology, 2010, 28 (4): 355–360. DOI:10.1038/nbt.1617 |
| [60] | De Las Heras A, Carre?o CA, De Lorenzo V. Stable implantation of orthogonal sensor circuits in Gram-negative bacteria for environmental release. Environmental Microbiology, 2008, 10 (12): 3305–3316. DOI:10.1111/emi.2008.10.issue-12 |






