2. 湘西州森林生态研究实验站,湘西 416000
2. Xiangxi Forest Ecological Research Station,Xiangxi 416000
产丁醇梭菌作为ABE(acetone-butanol-ethanol)发酵[1]的主要菌株,是一类严格厌氧的革兰氏阳性菌[2]。它们可以利用多种碳源(葡萄糖、木糖、阿拉伯糖、纤维二糖等)[3]通过代谢转化为丙酮、丁醇和乙醇,是一类重要的产溶剂工业微生物[4]。丁醇作为一种新型的可替代汽油燃料的可再生能源,可直接应用于未加改造的汽油发动机[5],在新能源利用上具有重大意义。目前,用于丁醇生产的梭菌类型主要可分为以下4种类型:丙酮丁醇梭菌(Clostridium acetobutylicum)、拜氏梭菌(Clostridium beijerickii)、糖丁酸梭菌(Clostridium saccharobutylicum)及糖乙酸多丁醇梭菌(Clostridium saccharoperbutylacetonicum)。近年来,在石油化工合成技术[6]的竞争下,开发高产丁醇梭菌已刻不容缓。现有的用于提高丁醇产量的方法主要有发酵条件优化[7-10]和改造新的菌株[11],在菌株改造方面有不定向的诱变育种[12, 13]和定向的基因改造[14, 15]。但受限于分子操作技术和产丁醇梭菌本身的特点[16],基因改造技术在过去10年里进展缓慢,其基本策略有基因失活、重组和依赖于质粒的基因表达系统等。本文就近10年所进行的对产丁醇梭菌的基因改造方面做一个概括和总结。
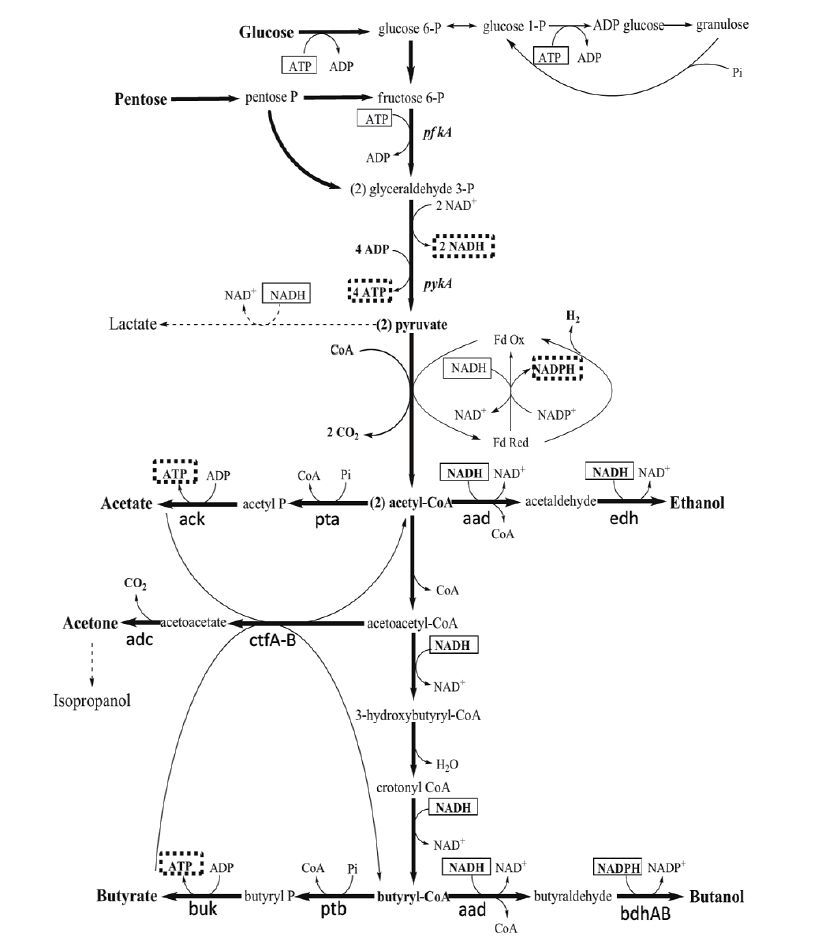
1 丁醇合成代谢的过程ABE(acetone-butanol-ethanol)发酵是生物法产丁醇的最主要方法,其发酵过程是一个复杂的生物反应过程,包括2个不同的阶段:产酸阶段和产溶剂阶段[17]。在溶剂的转变过程中至少涉及到245个基因的差异表达[18],两阶段之间的转换涉及到生物化学反应、基因调控和环境之间的相互作用[19]。其发酵的过程为:细菌利用葡萄糖(或者其他多糖)作为底物,通过EMP(糖酵解)途径生成2分子丙酮酸,在铁氧还蛋白氧化还原酶作用下生成乙酰-CoA,并产生氢气,主要代谢反应由6-磷酸果糖激酶和丙酮酸激酶催化。由乙酰-CoA生成丁醇的过程分为产酸阶段和产溶剂阶段。在细胞生长初期和指数生长期,所需能量由产酸过程提供,随着乙酸(由磷酸乙酰转移酶和乙酸激酶催化)和丁酸(由磷酸丁酰转移酶和丁酸激酶催化)的增加,发酵液pH降低,细胞生长受到抑制。当pH下降到一定值(pH达到5左右),细胞进入稳定阶段,并开始产溶剂。产酸阶段产生的乙酸和丁酸被重新利用吸收,在CoA 转移酶的作用下转化为乙酰-CoA和丁酰-CoA。乙酰-CoA和丁酰-CoA在醛脱氢酶和醇脱氢酶的作用下生成乙醇和丁醇。随着发酵的进行,底物的耗尽和产物丁醇的毒害作用,菌体开始自溶或生成孢子,发酵活动逐渐降低直至终止。具体代谢过程见图 1[20]。
|
| 图 1 产丁醇梭菌代谢图 ack:乙酸激酶基因;aad:乙醛脱氢酶基因;adc:乙酰乙酸脱羧酶基因;buk:丁酸激酶基因;ctfA-B:乙酰乙酰-CoA转移酶基因;edh:乙醇脱氢酶基因;pfkA:6-磷酸果糖激酶基因;pykA:丙酮酸激酶基因;pta:磷酸转乙酰激酶基因;ptb:磷酸丁酰转移酶基因;bdhAB:丁醇脱氢酶基因 |
丙酮在20世纪50年代作为ABE发酵的首要产物,被用于战争物资,使得ABE发酵得到快速发展。但是,在丁醇作为发酵的主要产物之后,丙酮的合成路线被研究者们作为首要的剔除代谢路径来增加丁醇的产量[21, 22]。丙酮的合成涉及到乙酰乙酰-CoA转移酶(COAT)和乙酰乙酰脱羧酶(AADC)。Tummala等[22]利用asRNA对丙酮丁醇梭菌adc进行抑制发现,构建载有抑制adc的质粒和控制质粒(无抑制adc的元件)所产生的丙酮并没有明显的变化。随后,为了验证adc对丙酮的合成是否是必需的关键基因,Tummala将质粒pFNK7(载有ctfA和ctfB基因:乙酰乙酰-CoA转移酶基因亚基)和质粒pFNK6(含有ctfA、ctfB和adc)转化突变菌株(缺乏所有溶剂生成的突变菌),发酵结果表明,丙酮只在含有pFNK6质粒中产生,说明adc基因编码的乙酰乙酰脱羧酶是丙酮合成所必需的关键酶。随后,Tummala对ctfA-B的两亚基进行失活,发现丙酮产量明显减少,说明ctfA-B基因编码的乙酰乙酰-CoA转移酶是丙酮合成的限速酶。同样,在Hönicke等[23]的实验中也证明了adc基因的缺失会导致丙酮的合成受限,其所构建的adc基因突变菌仅产生2 mmol/L丙酮(野生菌20 mmol/L),进一步证明了adc基因对丙酮合成的重要性。
2.2 乙酸合成乙酸合成的代谢酶是磷酸乙酰转移酶(PTA)和乙酸激酶(AK),乙酸作为产酸阶段的主要代谢物之一,当酸的积累达到一定程度(pH<5)时,宿主菌会进入产溶剂阶段。因此,乙酸合成的多少将会影响宿主菌进入产溶剂阶段的时间和丁醇的产量。Cooksley等[24]通过ClosTron技术失活丙酮丁醇梭菌ack基因发现,乙酸含量明显减少,乙醇和丁醇的产量却显著提高,其所构建的ptb(磷酸丁酰转移酶基因)突变菌株的乙酸产量却比野生菌高,说明减少乙酸合成的代谢流可以提高乙醇的生成,而阻断丁酸合成的路径可以提高乙酸的产量。而Kuit等[25]利用同样的技术对ack基因进行失活发现,乙酸的含量只是稍微减少,但是丁醇和乙醇的产量相比于野生菌分别提高了16%和59%。Jang等[26]发现,单独对ack、pta基因进行敲除并不会完全消除乙酸的形成,而且经过对其酶活的测定,发现被敲除的酶依然具有活性,只是降低到原来的16%。Lehmann[27]的实验结果也证明,对pta进行敲除,乙酸的合成相比于野生菌并没有明显降低。而Wang等[28]对拜氏梭菌pta基因进行失活,发现乙酸的产量明显下降,而丁酸显著增加。从这些研究结果可以看出,阻断丁酸的合成路径可以提高乙酸的产量,说明两者在维持环境pH值是相辅相成的两个因素。
2.3 丁酸合成丁酸合成比乙酸的合成的代谢网络更加复杂,其代谢过程中直接相关的2个关键酶为丁酰转移酶(PTB)和丁酸激酶(BK)。Shao等[29]利用Ⅱ型内含子对丙酮丁醇梭菌进行buk的失活,发现丁醇的产量比野生菌株提高了44%,与前人的研究结果相一致[30, 31]。Wang[28]等对拜氏梭菌利用II型内含子对buk进行失活,发现乙酸和丁酸的产量没有明显变化,但是可以将溶剂的产量提高20%-30%,并且提高了葡萄糖的利用速率。Lehmann等[32]实验对丙酮丁醇梭菌ptb基因进行敲除,PTB的酶活只有野生菌株的1%,丁酸的合成路径基本被抑制,说明ptb基因所表达的酶为丁酸合成过程中的限速酶。
2.4 生物乙醇/生物丁醇合成生物醇合成的代谢酶为醇/醛脱氢酶(由基因adhE1、adhE2、bdhA和bdhB编码)。为了探讨这些代谢酶在醇合成中的作用,Harris等[30]构建含有aad(醇/醛脱氢酶)基因的质粒pTAAD导入丙酮丁醇梭菌的突变菌PJC4BK(buk基因失活)发现,其乙醇的产量从2.6 g/L增加到4.5 g/L,而丁醇和丙酮的产量与突变菌PJC4BK相近,说明aad并不是丁醇合成过程中的限速酶。随后,Cooksley等[24]利用ClosTron技术构建了醛/醇脱氢酶的突变丙酮丁醇梭菌菌株adhE1-、adhE2-、bdhA-和bdhB-发现,adhE-突变菌只产生少许的溶剂(丁醇2.5、丙酮1.7、乙醇2.5 mmol/L),但其酸产量有明显增加。而且,突变菌株adhE2-、bdhA-和bdhB-对比于野生菌株,其产物产量基本无变化,说明adhE1是负责溶剂产生的主要基因。
2.5 EMP代谢途径改造目前对EMP(糖酵解)途径代谢基因改造的研究还比较少,重点还是集中在研究下游代谢酶对丁醇产量的影响。2013年,Ventura等[20]对丙酮丁醇梭菌进行基因改造,通过对6-磷酸果糖激酶和丙酮酸激酶的过表达,发现丁醇和乙醇的产量分别提高了29.4%和85.5%,丙酮并无明显增加,丁醇和总溶剂的产量分别提高到19.12和28.02 g/L。Ventura对实验结果作出了合理的分析,得出了溶剂产量的增加是因为这两种酶的过表达使得细胞产生了更多的ATP和NADH。最新研究发现[33],通过失活拜氏梭菌基因Cbei_4110(编码NADH醌氧化还原酶)来阻碍NAD(P)H消耗,发现葡萄糖利用率和丁醇产量都显著提高。ATP作为细胞的能量提供者和酸代谢途径的生成物,可以使细胞更快的生长,同时又能抑制产酸代谢途径(负反馈调节作用)。而NADH作为产溶剂过程中的反应物,可以有效地促进代谢向产溶剂方向进行。受此研究内容的启发,本实验课题研究组正在构建载有pyk(丙酮酸激酶基因)和pfk(6-磷酸果糖激酶基因)基因的目标菌(Clostridium Saccharobutylicum BAA-117),希望能够进一步验证EMP代谢途径对丁醇产量的提高有重要影响,并完善其理论依据。
3 其他代谢酶基因改造 3.1 丁醇耐受性基因改造丁醇的毒害作用对丁醇产量的提高是一个非常重要的限制因素,其限制作用主要体现在对细胞膜流动性的改变。因此,在主要代谢途径之外,研究者们通过研究其他相关基因,发现有些基因可以提高丁醇的耐受性。例如,Xu等[14]通过对拜氏梭菌的基因cac3319(编码组氨酸激酶)进行敲除,发现突变菌能在含有20 g/L丁醇下正常生长,且丁醇的产量由12.6 g/L增加到21 g/L。表明基因cac3319对于溶剂生成和丁醇耐受性的提高有着重要的作用。Tomas等[34]将含有热激蛋白基因(groES和 groEL)的质粒转入丙酮丁醇梭菌 ATCC824中,构建了新的菌株,发现其总溶剂浓度与野生菌株、质粒对照菌株相比分别提高40%和33%,说明热激蛋白可以提高细胞的耐受性,间接提高溶剂的产量。但是,提高丁醇的耐受性并不一定可以提高丁醇的产量,在Mann等[35]的实验中发现,过表达热激蛋白基因grpE和htpG可以提高丁醇的耐受性,但丁醇产量并没有提高。此外,Jia等[36]研究发现,由基因SMB_G1518和基因SMB_G1519编码的两种未知的功能性蛋白对菌种耐丁醇有很大的影响,其结果表明当破环基因SMB_G1518的表达或者阻断下游表达能明显的增加丁醇的耐受性,而当过表达SMB_G1519基因则降低菌株的丁醇耐受性能力。表明这两基因所构成的促进/阻遏系统将在丙酮丁醇梭菌耐丁醇能力中扮演重要的调控作用。
3.2 产溶剂抑制基因改造ABE发酵是一个复杂的代谢过程,因此,影响丁醇生成的因素比较复杂。除了提高细菌对丁醇的耐受性,研究者发现通过阻断产溶剂抑制基因也能大幅度提高丁醇产量。Nair等[37]通过同源重组沉默了丙酮丁醇梭菌溶剂抑制基因solR,构建了基因工程菌SolRB和SolRH,2株工程菌产生了较高浓度的ABE,其中SolRB工程菌的ABE产量分别为8.1 g/L、17.8 g/L和1.0 g/L,总溶剂能够达到26.9 g/L,表明通过解除抑制基因作用能够明显提高溶剂的 产量。
3.3 孢子生成基因改造在发酵后期由于丁醇浓度的升高,将会抑制细菌的生长,使细菌提前进入孢子期。因此,有大量研究通过对孢子生成的基因进行改造。Harris等[38]通过将控制孢子形成的基因spo0A在丙酮丁醇梭菌ATCC824中失活和高效表达,分别构建了SKO1和pMPSOA工程菌株,结果表明SKO1基本不产丙酮、丁醇,发酵后期细胞呈杆状,不形成梭状孢子,而pMPSOA产生较多的丁醇,并且能够形成梭状孢子,说明spo0A是控制溶剂产生和孢子形成的一个转录调控因子。Scotcher等[39]构建了工程菌株pMSpo和pASspo,分别高效表达spoIIE和降低spoIIE的表达,结果显示spoIIE过量表达不能提高溶剂产量;相反,spoIIE的下调使丙酮、丁醇和乙醇的产量分别提高了43%、110%和225%,并且使孢子延期形成,形态也发生了改变,这表明spoIIE并不直接影响溶剂产生,而是通过使孢子延期形成,使细菌的产溶剂期延长,进而提高丁醇产量。
4 基因改造过程中的问题产溶剂梭菌作为丁醇的天然生产菌株,丁醇产量比其他类型的基因改造菌要高,但是由于其严格的厌氧培养条件,在实际培养及分子操作中会带来很多困难。例如,CaCl2法转化率低且必须在厌氧环境中进行,而且,在转化过程中常出现转化效率低或者转化子被产溶剂菌的Cac824I二型修饰酶系统降解[40]。应对这些问题,Truffaut等[41]在含有蔗糖的培养基中培养细菌,通过添加溶菌酶和青霉素,用PEG完成转化,但此制备方法步骤繁杂且周期长。此外,为了避免质粒被宿主菌降解和提高转化效率。Mermelstein等[42]利用大肠杆菌中含有的质粒PAN1对所转化的质粒进行甲基化修饰,可以避免梭菌的降解系统,然后通过电转化并成功转化。Dong等[40]利用二型内含子技术破坏丙酮丁醇梭菌DSM1731的二型限制内切酶的基因,使得改造后的突变菌能直接转化而不需要甲基化DNA。随后这种方法被许多研究者广为使用[15, 43]。但是梭菌的基因失活策略如自杀质粒基因敲除[44]、反义RNA抑制[45, 46]、复制型质粒同源重组[47, 48]、II型内含子基因敲除技 术[49, 50]等实验要求较高,不利于产溶剂菌的研究。
5 提高丁醇产量的改造策略近年来,研究者们为了提高丁醇的产量,将策略都集中在优化丁醇的发酵条件和构建新的改造菌,虽然都取得了很好的效果,但是丁醇的产量始终不能大幅度提高。一方面受限于产丁醇梭菌分子遗传操作的困难;另一方面缺乏对产丁醇梭菌整个ABE代谢过程(包括与环境的互作)的充分理解。随着系统代谢工程[51]、转录组学[52]和蛋白质组学[53]的发展,产丁醇梭菌的代谢研究已经进入快速发展阶段,Liao等[54]采用模型构建的策略将ABE发酵过程的动态整合成一个模型,希望通过对基因调控、环境互作和代谢反应3个影响因素的整合能够更好的解释ABE发酵的动态变化,为高产丁醇菌株的发酵条件优化和基因改造提供了方向性指导。对于如何提高丁醇的产量,其所构建的模型可以为广大研究者对发酵条件优化以及基因改造策略提供理论依据。笔者认为,提高丁醇产量的改造策略有:(1)加强EMP代谢途径,为细菌提供更多能量,减少产酸代谢流,同时加强溶剂产生基因spo0A的表达;(2)失活产酸基因ack和buk,同时加强spo0A的表达;(3)加强热激蛋白的表达,提高丁醇的耐受性并过表达溶剂生产基因(aad和bdhAB)。尽管对于产丁醇梭菌的代谢路径研究已经非常透彻,但是,对于细菌在不同环境的应对机制还了解不深。产丁醇梭菌作为天然的丁醇生产菌株,其优势及缺点都非常明显。所以,依然需要深入开展对于构建新的高产丁醇菌株和发现非厌氧产丁醇菌株[55]的研究。
| [1] | Moon HG, Jang Y, Cho C, et al. One hundred years of clostridial butanol fermentation. Fems Microbiology Letters, 2016, 363 : 3–18. |
| [2] | Berezina OV, Zakharova NV, Yarotsky CV, et al. Microbial producers of butanol. Applied Biochemistry And Microbiology, 2012, 48 (7): 625–638. DOI:10.1134/S0003683812070022 |
| [3] | Cho C, Jang YS, Moon HG, et al. Metabolic engineering of clostridia for the production of chemicals. Biofuels, Bioproducts and Biorefining, 2015, 9 : 211–225. DOI:10.1002/bbb.2015.9.issue-2 |
| [4] | Ding J, Xu G, Han R, et al. Biobutanol production from corn stover hydrolysate pretreated with recycled ionic liquid by Clostridium saccharobutylicum DSM 13864. Bioresource Technology, 2016, 199 : 228–234. DOI:10.1016/j.biortech.2015.07.119 |
| [5] | Jin C, Yao M, Liu H, et al. Progress in the production and application of n-butanol as a biofuel. Renewable and Sustainable Energy Reviews, 2011, 15 (8): 4080–4106. DOI:10.1016/j.rser.2011.06.001 |
| [6] | Uyttebroek M, Van Hecke W, Vanbroekhoven K. Sustainability metrics of 1-butanol. Catalysis Today, 2015, 239 : 7–10. DOI:10.1016/j.cattod.2013.10.094 |
| [7] | Wang QY, Zhang C, Yao R, et al. Butanol Fermentation by Clostridium saccharobutylicum based on poplar wood. Biores-ources, 2015, 10 (3): 5395–5406. |
| [8] | 钟洁, 王义强, 华涟滩, 等. 基于杨木纤维发酵产丁醇工艺条件的研究. 中南林业科技大学学报, 2015(7): 125–130. |
| [9] | 彭牡丹, 王义强, 王启业, 等. 糖丁酸梭状芽孢杆菌BAA-117利用不同碳源发酵产燃料丁醇的研究. 可再生能源, 2014(11): 1703–1709. |
| [10] | Li H, Zhang Q, Yu X, et al. Enhancement of butanol production in Clostridium acetobutylicum SE25 through accelerating phase shift by different phases pH regulation from cassava flour. Bioresource Technology, 2016, 201 : 148–155. DOI:10.1016/j.biortech.2015.11.027 |
| [11] | Zheng J, Tashiro Y, Wang Q, et al. Recent advances to improve fermentative butanol production:Genetic engineering and fermentation technology. Journal of Bioscience and Bioengineering, 2015, 119 (1): 1–9. DOI:10.1016/j.jbiosc.2014.05.023 |
| [12] | 毛绍名, 章怀云. 丙酮丁醇梭菌高耐丁醇突变株的选育及其生理特性的研究. 中南林业科技大学学报, 2012(8): 103–107. |
| [13] | 王义强, 王启业, 华连滩, 等. 高产丁醇菌株诱变选育及发酵研究. 中南林业科技大学学报, 2015(10): 120–126. |
| [14] | Xu M, Zhao J, Yu L, et al. Engineering Clostridium acetobutylicum with a histidine kinase knockout for enhanced n-butanol tolerance and production. Applied Microbiology and Biotechnol, 2015, 99 (2): 1011–1022. DOI:10.1007/s00253-014-6249-7 |
| [15] | Ehsaan M, Kuit W, Zhang Y, et al. Mutant generation by allelic exchange and genome resequencing of the biobutanol organism Clostridium acetobutylicum ATCC 824. Biotechnology for Biofuels, 2016, 9 (1): 4–24. DOI:10.1186/s13068-015-0410-0 |
| [16] | 顾阳, 杨晟, 姜卫红. 产溶剂梭菌分子遗传操作技术研究进展. 生物工程学报, 2013(8): 1133–1145. |
| [17] | Lutke-Eversloh T, Bahl H. Metabolic engineering of Clostridium acetobutylicum:recent advances to improve butanol production. Current Opinion in Biotechnology, 2011, 22 (5): 634–647. DOI:10.1016/j.copbio.2011.01.011 |
| [18] | Grimmler C, Janssen H, Krauβe D, et al. Genome-wide gene expression analysis of the switch between acidogenesis and solventogenesis in continuous cultures of Clostridium acetobutylicum. Journal of Molecular Microbiology and Biotechnology, 2011, 20 (1): 1–15. DOI:10.1159/000320973 |
| [19] | Lee JW, Na D, Park JM, et al. Systems metabolic engineering of microorganisms for natural and non-natural chemicals. Nature Chemical Biology, 2012, 8 (6): 536–546. DOI:10.1038/nchembio.970 |
| [20] | Ventura JS, Hu H, Jahng D. Enhanced butanol production in Clostridium acetobutylicum ATCC 824 by double overexpression of 6-phosphofructokinase and pyruvate kinase genes. Applied Microbiology and Biotechnology, 2013, 97 (16): 7505–7516. DOI:10.1007/s00253-013-5075-7 |
| [21] | Jiang Y, Xu C, Dong F, et al. Disruption of the acetoacetate decarboxylase gene in solvent-producing Clostridium acetobutylicum increases the butanol ratio. Metabolic Engineering, 2009, 11 (4-5): 284–291. DOI:10.1016/j.ymben.2009.06.002 |
| [22] | Tummala SB, Welker NE, Papoutsakis ET. Design of antisense RNA constructs for downregulation of the acetone formation pathway of Clostridium acetobutylicum. Journal of Bacteriology, 2003, 185 (6): 1923–1934. DOI:10.1128/JB.185.6.1923-1934.2003 |
| [23] | H?nicke D, Lütke-Eversloh T, Liu Z, et al. Chemostat cultivation and transcriptional analyses of Clostridium acetobutylicum mutants with defects in the acid and acetone biosynthetic pathways. Applied Microbiology and Biotechnology, 2014, 98 (23): 9777–9794. DOI:10.1007/s00253-014-6040-9 |
| [24] | Cooksley CM, Zhang Y, Wang H, et al. Targeted mutagenesis of the Clostridium acetobutylicum acetone-butanol-ethanol fermentation pathway. Metabolic Engineering, 2012, 14 (6): 630–641. DOI:10.1016/j.ymben.2012.09.001 |
| [25] | Kuit W, Minton NP, López-Contreras AM, et al. Disruption of the acetate kinase(ack)gene of Clostridium acetobutylicum results in delayed acetate production. Applied Microbiology and Biotechnology, 2012, 94 (3): 729–741. DOI:10.1007/s00253-011-3848-4 |
| [26] | Jang YS, Lee JY, Lee J, et al. Enhanced butanol production obtained by reinforcing the direct butanol-forming route in Clostridium acetobutylicum. Mcrobiology, 2012, 3 (5): e312–e314. |
| [27] | Lehmann D, H?nicke D, Ehrenreich A, et al. Modifying the product pattern of Clostridium acetobutylicum. Applied Microbiology and Biotechnology, 2012, 94 (3): 743–754. DOI:10.1007/s00253-011-3852-8 |
| [28] | Wang Y, Li X, Milne CB, et al. Development of a gene knockout system using mobile group II introns(targetron)and genetic disruption of acid production pathways in Clostridium beijerinckii. Applied and Environmental Microbiology, 2013, 79 (19): 5853–5863. DOI:10.1128/AEM.00971-13 |
| [29] | Shao L, Hu S, Yang Y, et al. Targeted gene disruption by use of a group II intron(targetron)vector in Clostridium acetobutylicum. Cell Res, 2007, 17 (11): 963–965. DOI:10.1038/cr.2007.91 |
| [30] | Harris LM, Desai RP, Welker NE, et al. Characterization of recombinant strains of the Clostridium acetobutylicum butyrate kinase inactivation mutant:need for new phenomenological models for solventogenesis and butanol inhibition?. Biotechnology and Bioengineering, 2000, 67 (1): 1–11. DOI:10.1002/(ISSN)1097-0290 |
| [31] | Harris LM, Blank L, Desai RP, et al. Fermentation characterization and flux analysis of recombinant strains of Clostridium acetobutylicum with an inactivated solR gene. Journal of Industrial Microbiology and Biotechnology, 2001, 27 (5): 322–328. DOI:10.1038/sj.jim.7000191 |
| [32] | Lehmann D, Radomski N, Lütke-Eversloh T. New insights into the butyric acid metabolism of Clostridium acetobutylicum. Applied Microbiology and Biotechnology, 2012, 96 (5): 1325–1339. DOI:10.1007/s00253-012-4109-x |
| [33] | Liu J, Guo T, Wang D, et al. Enhanced butanol production by increasing NADH and ATP levels in Clostridium beijerinckii NCIMB 8052 by insertional inactivation of Cbei_4110. Applied Microbiology and Biotechnology, 2016, 100 (11): 4985–4996. DOI:10.1007/s00253-016-7299-9 |
| [34] | Tomas CA, Beamish J, Papoutsakis ET. Transcriptional analysis of butanol stress and tolerance in Clostridium acetobutylicum. Journal of Bacteriology, 2004, 186 (7): 2006–2018. DOI:10.1128/JB.186.7.2006-2018.2004 |
| [35] | Mann MS, Dragovic Z, Schirrmacher G, et al. Over-expression of stress protein-encoding genes helps Clostridium acetobutylicum to rapidly adapt to butanol stress. Biotechnology Letters, 2012, 34 (9): 1643–1649. DOI:10.1007/s10529-012-0951-2 |
| [36] | Jia K, Zhang Y, Li Y. Identification and characterization of two functionally unknown genes involved in butanol tolerance of Clostridium acetobutylicum. PLoS One, 2012, 7 (6): e38815. DOI:10.1371/journal.pone.0038815 |
| [37] | Nair RV, Green EM, Watson DE, et al. Regulation of the sol locus genes for butanol and acetone formation in Clostridium acetobutylicum ATCC 824 by a putative transcriptional repressor. Journal of Bacteriology, 1999, 181 (1): 319–330. |
| [38] | Harris LM, Welker NE, Papoutsakis ET. Northern, morphological, and fermentation analysis of spo0A inactivation and overexpression in Clostridium acetobutylicum ATCC 824. Journal of Bacteriology, 2002, 184 (13): 3586–3597. DOI:10.1128/JB.184.13.3586-3597.2002 |
| [39] | Scotcher MC, Bennett GN. SpoIIE regulates sporulation but does not directly affect solventogenesis in Clostridium acetobutylicum ATCC 824. Journal of Bacteriology, 2005, 187 (6): 1930–1936. DOI:10.1128/JB.187.6.1930-1936.2005 |
| [40] | Dong H, Zhang Y, Dai Z, et al. Engineering Clostridium Strain to Accept Unmethylated DNA. PLoS One, 2010, 5 : e90382. |
| [41] | Truffaut N, Hubert J, Reysset G. Construction of shuttle vectors useful for transforming Clostridium acetobutylicum. Fems Microbiol Letters, 1989, 49 (1): 15–20. |
| [42] | Mermelstein LD, Papoutsakis ET. In vivo methylation in Escherichia coli by the Bacillus subtilis phage phi 3T I methyltransferase to protect plasmids from restriction upon transformation of Clostridium acetobutylicum ATCC 824. Applied and Environmental Microbiology, 1993, 59 (4): 1077–1081. |
| [43] | Croux C, Nguyen N, Lee J, et al. Construction of a restriction-less, marker-less mutant useful for functional genomic and metabolic engineering of the biofuel producer Clostridium acetobutylicum. Biotechnology for Biofuels, 2016, 9 (1): 23. DOI:10.1186/s13068-016-0432-2 |
| [44] | Green EM, Bennett GN. Genetic manipulation of acid and solvent formation in clostridium acetobutylicum ATCC 824. Biotechnology and Bioengineering, 1998, 58 (2-3): 215–221. DOI:10.1002/(ISSN)1097-0290 |
| [45] | Desai RP, Papoutsakis ET. Antisense RNA strategies for metabolic engineering of Clostridium acetobutylicum. Applied and Environmental Microbiology, 1999, 65 (3): 936–945. |
| [46] | Tummala SB, Junne SG, Papoutsakis ET. Antisense RNA downregulation of coenzyme A transferase combined with alcohol-aldehyde dehydrogenase overexpression leads to predominantly alcohologenic Clostridium acetobutylicum fermentations. Journal of Bacteriology, 2003, 185 (12): 3644–3653. DOI:10.1128/JB.185.12.3644-3653.2003 |
| [47] | Al-Hinai MA, Fast AG, Papoutsakis ET. Novel system for efficient isolation of Clostridium double-crossover allelic exchange mutants enabling markerless chromosomal gene deletions and DNA integration. Applied And Environmental Microbiology, 2012, 78 (22): 8112–8121. DOI:10.1128/AEM.02214-12 |
| [48] | Soucaille P, Figge R, Croux C. Process for chromosomal integration and DNA sequence replacement in Clostridia[Z] . Google Patents, 2014. http://cn.bing.com/academic/profile?id=e6660abae7a6dfd5eb7be1bc7c7da501&encoded=0&v=paper_preview&mkt=zh-cn |
| [49] | Shao L, Hu S, Yang Y, et al. Targeted gene disruption by use of a group II intron(targetron)vector in Clostridium acetobutylicum. Cell Research, 2007, 17 (11): 963–965. DOI:10.1038/cr.2007.91 |
| [50] | Heap JT, Pennington OJ, Cartman ST, et al. The ClosTron:a universal gene knock-out system for the genus Clostridium. Journal of Microbiological Methods, 2007, 70 (3): 452–464. DOI:10.1016/j.mimet.2007.05.021 |
| [51] | Liao C, Seo S, Lu T. System-level modeling of acetone-butanol-ethanol fermentation. Fems Microbiology Letters, 2016, 363 (9): w74. |
| [52] | Wang Y, Li X, Blaschek HP. Effects of supplementary butyrate on butanol production and the metabolic switch in Clostridium beijerinckii NCIMB 8052:genome-wide transcriptional analysis with RNA-Seq. Biotechnology for Biofuels, 2013, 6 (1): 138. DOI:10.1186/1754-6834-6-138 |
| [53] | Mao S, Luo Y, Zhang T, et al. Proteome reference map and comparative proteomic analysis between a wild type Clostridium acetobutylicum DSM 1731 and its mutant with enhanced butanol tolerance and butanol yield. Journal of Proteome Research, 2010, 9 (6): 3046–3061. DOI:10.1021/pr9012078 |
| [54] | Liao C, Seo S, Celik V, et al. Integrated, systems metabolic picture of acetone-butanol-ethanol fermentation by Clostridium acetobutylicum. Proceedings of the National Academy of Sciences, 2015, 112 (27): 8505–8510. DOI:10.1073/pnas.1423143112 |
| [55] | Ng CY, Takahashi K, Liu Z. Isolation, characterization, and optimization of an aerobic butanol-producing bacterium from Singapore. Biotechnology And Applied Biochemistry, 2016, 63 (1): 86–91. DOI:10.1002/bab.2016.63.issue-1 |




