生物体内广泛存在一类基因编码的蛋白质或RNA元件 ( 例如转录因子、核糖体开关 ) ,它们能够响应细胞内特定代谢物效应物的浓度,通过改变自身结构调控下游基因元件特定信号的输出。代谢物生物传感器就是基于这一原理,通过特定的生物识别元件响应目标代谢物化合物,并将其转化为目的输出信号 ( 如荧光、细胞的存活、特定代谢通路的开闭 ) [1]。
代谢物生物传感器作为一种重要的合成生物学工具,可以视作微生物中的仪表,将代谢物浓度转化为特定信号的输出,因而能够在线的感应细胞体内代谢物浓度及其变化,为代谢途径设计及优化提供工具。近年来,研究者逐渐尝试将代谢物生物传感器应用到微生物细胞工厂的构建领域,由于其能在单细胞层次实现胞内代谢产物的浓度响应,在微生物细胞工厂的改造和优化中日益显现出巨大的应用潜力。目前已经有相关综述论述了代谢物生物传感器的原理和应用[2-6],本文将着重综述近年来代谢物生物传感器在微生物细胞工厂构建中的应用实例及其存在的挑战。
1 代谢物生物传感器构建原理代谢物生物传感器的主要组成部分包括生物识别元件和信号输出元件,前者能够响应特定的代谢物造成自身结构变化,后者将响应元件结构变化信号转化为目的输出信号。自然界中存在丰富的响应机理不同的生物识别元件,其中转录因子 ( Transcription factors,TFs ) 和核糖开关是目前最常见的生物识别元件。
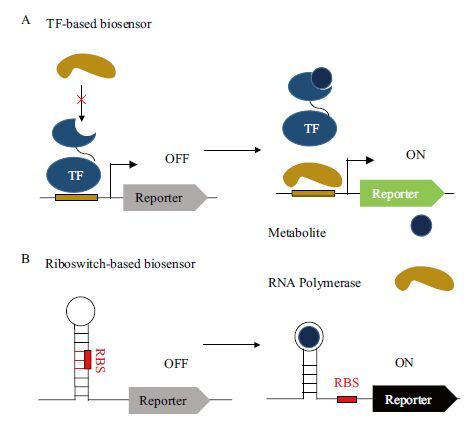
1.1 基于转录 ( 激活、抑制 ) 因子的代谢物生物传感器转录因子能够结合在调控基因DNA的增强子或启动子区域,独自或与其它蛋白组成复合体,通过促进或阻止RNA聚合酶对于特定基因的结合,进而调控DNA到信使RNA的转录速率[7]。转录因子特异的与目标代谢物结合,发生别构作用,对基因的调控作用发生变化,如转录抑制因子结合代谢物后对RNA聚合酶的空间位阻效应消失。利用转录因子的这一特性,将目标代谢物浓度转化为荧光蛋白、细胞存活等输出信号,从而构建出响应目标代谢物的生物传感器 ( 图 1-A ) 。
转录因子在生物体内广泛存在,以大肠杆菌 ( Escherichia coli,E.coli ) 为例,目前已经发现了超过230种转录因子,响应的代谢物包括糖类、糖磷酸、氨基酸和脂类等[8]。随着生物信息学技术的发展,已经有一些调控相互作用和转录因子结合位点的数据库,如Regulon DB、RegTransBase等。其中RegTransBase数据库提供了原核生物转录因子,其结合位点以及小分子效应物的信息,涵盖了875个效应物,1 488个转录因子,为转录因子的应用奠定了坚实的基础。目前研究者利用天然转录因子,已开发了不同的生物传感器,可以响应木糖[9]、1-丁醇[10]、支链氨基酸[11, 12]、碱性氨基酸[8, 13]、酪氨酸[14]、葡糖糖酸、柚皮素[15]以及乙酰辅酶A[16]、丙二酰辅酶A[17]、NADPH[18]等代谢物。
尽管天然存在的转录因子能够响应许多不同种类的小分子代谢物,但是在实际应用中,目标代谢物往往不存在天然响应或者响应性能不理想的转录因子,通过工程化改造的手段能够改变转录因子响应的特异性。例如,来自于E.coli的能够天然响应L-阿拉伯糖的转录因子AraC已经被改造为能够特异性响应D-阿拉伯糖[19]、甲羟戊酸[20]、三乙酸内酯 ( TAL ) [21, 22]和四氢嘧啶[23],为基于转录因子的代谢物生物传感器的应用提供了更多的潜力。此外,通过对传感器元件的改造,还可实现传感器的跨种属应用。例如,来源于E.coli的甲羟戊酸传感器通过改造已经成功应用于扭脱甲基杆菌 ( Methylobacterium extorquens,M. extorquens ) AM1中,实现对甲羟戊酸的响应[24]。
1.2 基于核糖开关的代谢物生物传感器核糖开关是一类在mRNA的前导区中被发现的cis-编码的调控RNAs,它们能够在对应的效应物存在或不存在的情况下发生形变[25]。一般地,核糖开关通过在对应配体存在时,采用能够诱导转录终止或者抑制翻译起始结构构象,在对应配体不存在时,采用能够使得转录延长或翻译开始的替代构象来控制下游基因的表达[26]。利用这种机制,将下游基因设计为易于检测的输出信号,同样能够构建代谢物生物传感器 ( 图 1-B ) 。
|
| 图 1 代谢物生物传感器响应机制 ( A ) 基于转录因子 ( Transcription factor,TF ) 的生物传感器:转录抑制因子与目标代谢物结合后,发生别构作用,不再与DNA结合,开始转录表达; ( B ) 基于核糖开关的生物传感器:在目标代谢物存在时,核糖开关不再隔绝核糖体结合位点 ( Ribosomal binding site,RBS ) ,开始下游基因表达 |
目前为止,已经有至少19个不同的效应物-结合核糖开关种类被鉴定[27],核糖开关能够结合的效应物包括一些重要的代谢物,如葡糖-6-磷酸[28]、赖氨酸[29]和甘氨酸[30],辅酶因子[31-35]如维生素B12,S-腺苷甲硫氨酸 ( S-Adenosyl-L-methionine,SAM ) 、硫胺素焦磷酸、黄素单核苷酸 ( Flavin mononucleotide,FMN ) ,以及镁离子[36]、氟离子[37]等。基于核糖开关的特性,许多研究组致力于发展基于核糖开关的代谢物生物传感器[38-40],例如通过将维生素B12的核糖开关与报告基因β-半乳糖苷酶,荧光素酶或者红色荧光蛋白相结合,研究维生素B12的代谢转运机理[41]。此外,还有基于适体酶核糖开关设计的代谢物生物传感器[42],适体酶能够利用配体来控制锤头状核酶,进而控制mRNA转录的稳定性或翻译的可行性。
尽管基于核糖开关的代谢物生物传感器潜力巨大,但是在代谢工程中的应用仍旧受到限制[43],主要原因是能够响应目标代谢物的核酸适配体缺乏以及核酸适配体与驱动区域的连接研究不充分[5]。近年来随着合成生物学的迅速发展,已经出现了一些能够辅助设计核糖开关的工具[44],将有可能极大促进基于核糖开关的代谢物生物传感器在代谢工程中的应用。
1.3 其他类型的代谢物生物传感器除了转录因子与核糖开关外,其他的能够响应特定代谢物的生物识别元件也被应用于代谢物生物传感器的设计。例如,压力响应的启动子[45-47],核糖体干扰肽[48],能够发生荧光共振能量转移 ( Fluorescence resonance energy transfer,FRET ) 的代谢物-感应蛋白[49, 50]等。这些生物识别元件的挖掘将有力地推动代谢物生物传感器在代谢工程中的应用。
2 代谢物生物传感器的性能评价代谢物生物传感器的响应性能决定了其能否在微生物细胞工厂构建中实际发挥作用。其性能评价的主要依据是输入输出响应曲线 ( 图 2 ) ,代谢物浓度与生物传感器响应输出 ( Arbitrary reporter units,AU ) 之间的关系刻画了生物传感器的转换函数,提供了生物传感器的性能信息,包括灵敏度 ( Sensitivity ) 、响应范围、线性检测范围、检测阈值等信息,为实际应用提供参考[51]。其中灵敏度表示响应曲线的斜率,描述了在两个样品输入产物浓度之间的最小的可微变化。响应范围表示代谢物生物传感器输出信号在最高上限和背景状态之间的差异。线性检测范围表示代谢物浓度能够与输出表达信号线性相关联的范围。转化函数表示代谢物生物传感器输入和输出之间的数学关系。

|
| 图 2 代谢物生物传感器响应曲线 |
代谢物生物传感器响应效应物的灵敏度和特异性是其在应用过程中重点关注的性能,因而在实际应用中,需要首先测定生物传感器的响应曲线,以确定其能否满足应用需求。实际操作中,往往通过外源添加目标代谢物的方法来测定响应曲线,但是由于细胞膜具有选择透过性,代谢物可能通过主动运输进入细胞,使得胞内与胞外代谢物浓度并不一定线性相关,因而需要测定胞内代谢物的浓度。
3 代谢物生物传感器在微生物细胞工厂中的应用代谢物生物传感器在微生物细胞工厂中典型的应用包括高产目标化合物菌株的高通量筛选和微生物胞内代谢动态调控 ( Dynamic control ) 等[5]。例如代谢物生物传感器可以在单细胞水平上将代谢物化合物浓度转化为荧光输出或者细胞生长优势,进而通过高通量的流式细胞仪 ( Fluorescence activated cell sorting,FACS ) 或者细胞优势表型压力选择,实现对特定高产菌株的高通量筛选 ( Screen ) 或者选择 ( Selection ) 。在代谢动态调控方面,利用人工构建的代谢物生物传感器模拟微生物原有的精细调控体系,实现目的代谢途径根据效应物的浓度动态开闭,可以帮助微生物体内的代谢流实时响应宿主细胞的代谢状态或者宿主所处的外部环境的变化,使代谢网络有更强的鲁棒性和更高的生产效率。此外,近期有报道利用代谢物生物传感器实现非遗传细胞间异质性选择 ( Nongenetic selection ) ,提高代谢途径的生物合成。
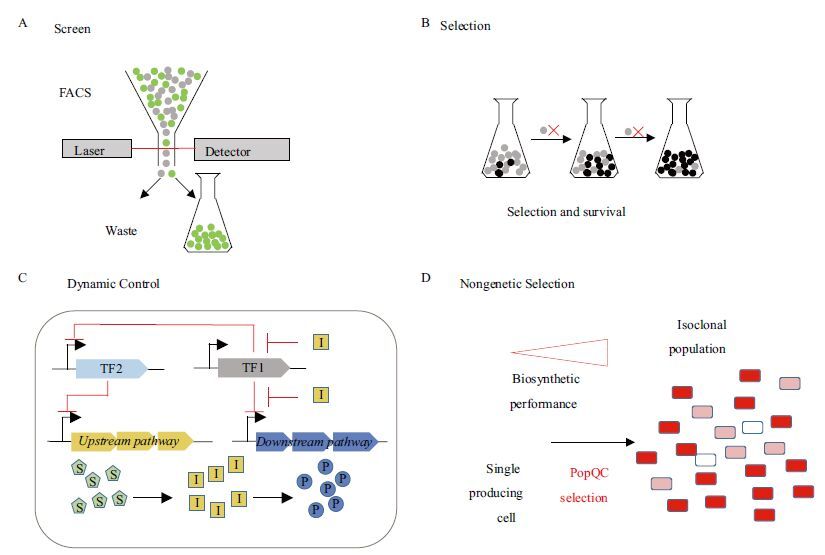
3.1 高通量筛选生物体系的复杂性使得工程化改造菌株往往需要通过多轮设计-构造-检验循环 ( Design-Build-Test cycle,DBT循环 ) 才能够找到基因元件的最优组合。DBT循环的通量决定了菌株优化所需要的时间。近年来,随着合成生物技术的发展,设计和构建的通量迅速提高,已经能够构建库容超过109的多样性库,但是目前检验的通量仍旧限制在102/d或103/d的水平,极大地限制了DBT循环的通量[52]。代谢物生物传感器能够在单细胞水平将效应物浓度转化为荧光输出,结合FACS技术 ( 图 3-A ) ,筛选的通量能够达到109细胞/d,使得DBT循环的通量提高106倍。
代谢物传感器在目的产物高通量筛选领域的应用最早报道于2012年,基于来源于谷氨酸棒杆菌 ( Corynebacterium glutamicum,C. glutamicum ) 的能够响应精氨酸、组氨酸和赖氨酸的转录因子LysG,以及其对应的启动子lysE,以黄色荧光蛋白 ( Yellow fluorescent protein,YFP ) 作为输出信号,构建的代谢物生物传感器被应用于在化学诱变微生物突变库中筛选赖氨酸高产菌株[8]。这一传感器随后也被用于筛选合成途径中酶的突变体,消除了原有的反馈抑制作用,提高了对应氨基酸的积累量[13]。另一个同样来源于C. glutamicum的转录因子Lrp能够响应包括缬氨酸、亮氨酸、异亮氨酸在内的支链氨基酸,也被应用于优化C. glutamicum中氨基酸的生产[11, 12]。高通量筛选系统同样可以应用于对酶的进化。基于酚类特异性响应的转录激活子DmpR在E.coli中构建的高通量筛选有机磷酸酯降解酶活性的筛选系统,在3轮突变和FACS筛选后,分离出了发生3个氨基酸替换的突变体MPH,kcat/Km提高100倍[53, 54]。
代谢物生物传感器结合高通量筛选的技术能够辅助微生物调控网络的工程化改造 ( Sensor-assisted transcriptional regulator engineering,SATRE ) 。本文作者团队在M. extorquens AM1中成功应用这一策略,利用工程化改造后能够响应甲羟戊酸的生物传感器,结合FACS手段,实现了M. extorquens AM1中代谢流的再分配,将来源于甲醇的乙酰辅酶A更多的导向甲羟戊酸合成,使得其产量提高60%[24]。
来源于细菌的转录因子通过改造也能够应用于真核细胞中,例如来源于细菌的响应木糖的转录因子XylR被改造为能够在酿酒酵母 ( Saccharomyces cerevisiae,S. cerevisiae ) 中响应木糖的基因元件[9]。研究者优化了多种来源的XylR抑制子和操纵子在报告基因上游区域的插入位点,利用优化后的生物传感器进行木糖转运蛋白的改造,将该蛋白的转运效率提高了6.5倍。
除了可以应用目标化合物直接作为效应物应用于基因表达调控,目标化合物的前体或者指示剂也能够作为效应物。还原反应是细胞代谢过程的核心,大约1/4的已知酶为氧化还原酶,其中大多数为NADPH依赖型[55]。在E.coli内能够间接响应胞内NADPH/NADP+的比例的转录因子SoxR被应用于NADPH依赖的酶的高通量改造[16]。将色氨酸作为紫色杆菌素合成途径中的重要节点,本文作者团队开发了基于色氨酸新生肽tnaC的新型生物传感器,利用正向荧光信号筛选色氨酸高产菌株,进一步利用负向荧光信号筛选消耗色氨酸的高效下游途径,进而成功构建了从葡萄糖合成色氨酸及高产下游衍生物的E.coli工程菌[48]。来源于枯草芽孢杆菌 ( Bacillus subtilis,B. subtilis ) 的特异性响应丙二酰辅酶A的转录因子FapR和操纵子fapO已经成为途径工程中的重要工具,在E.coli[56, 57]与S. cerevisiae[17]中都得到了成功应用。此外通过报告菌株将目标产物转化为可检测的信号的双菌筛选系统为高通量检测工业上全细胞催化菌株分泌代谢物的产量提供了可行性[58]。作为概念验证,研究者利用E.coli将B. subtilis生产的维生素B2转化为FMN,利用能够特异性响应FMN的核糖开关调控输出信号绿色荧光蛋白 ( Green fluorescent protein,GFP ) 的表达,提高了在B. subtilis中维生素B2的合成。
3.2 选择通过人为设计条件,模拟自然进化的机制,使得不同基因型的菌株之间存在生长差异,使得具有生长优势的突变体在培养过程中富集,进而得到理想表型的过程称为选择。若代谢物生物传感器的输出信号为与生长相关的基因,能够实现代谢物产量与菌株生长优势相关联 ( 图 3-B ) ,实现通量1010细胞/轮的筛选。
2013年报道了在E.coli中开发的1-丁醇生物传感器/生长选择体系[10]。四环素外排泵TetA的表达受到来源于丁香假单胞菌 ( Pseudomonas butanovora,P. butanovora ) 特异性响应乙醇的转录因子BmoR调控,从而使的1-丁醇的产量与菌株在四环素中的生长情况相关联。该体系应用于筛选丁醇合成途径上游基因的RBS序列优化的突变体,成功提高了丁醇的产量。另一个成功的案例是2014年报道的结合多重自动基因工程 ( Multiplex genome engineering,MAGE ) 和代谢物生物传感器进行定向进化。来源于恶臭假单胞菌 ( Pseudomonas putida,P. putida ) 的抑制蛋白TtgR在结合包含柚皮素在内的黄酮类化合物时产生去除抑制效果,利用该机制可构建柚皮素生物传感器。研究者结合该生物传感器,并利用TolC外膜孔蛋白作为选择标记,实现在十二烷基硫酸钠存在时正向筛选,在E.coli毒素E1存在时反向筛选[15]。在4轮MAGE靶向突变和筛选后,获得了柚皮素产量提高36倍的菌株。相同的策略应用于葡萄糖二酸合成途径中,基于特异性响应的转录因子CdaR,葡萄糖二酸的产量提高22倍。核糖开关同样能够应用于菌株进化。在E. coli 中,应用赖氨酸-OFF核糖开关将赖氨酸生产与菌株生长相关联。在赖氨酸存在时,该核糖开关能够抑制下游tetA基因的表达,而TetA对Ni2+敏感,使得在Ni2+存在时,高产赖氨酸的突变菌株具有生长优势,进而产生富集[59]。
|
| 图 3 代谢物生物传感器在微生物细胞工厂中的应用 ( A ) 高通量筛选:在单细胞水平上将代谢物化合物浓度转化为荧光输出,通过高通量的流式细胞仪 ( Fluorescence activated cell sorting,FACS ) 筛选高产菌株; ( B ) 生长/选择:将代谢物浓度转化为生长优势,进行细胞优势表型压力选择,富集高产菌株; ( C ) 动态调控:实现目的代谢途径根据效应物的浓度动态开闭,实现微生物体内的代谢流实时响应宿主细胞的代谢状态或者宿主所处的外部环境的变化,‘S’:Substrate,‘I’:Intermediate ,‘P’:Product; ( D ) 非遗传细胞间异质性选择:富集细胞群体中的高产细胞,提高代谢途径的生物合成效率 |
除了E.coli,将代谢物生物传感器与影响微生物生长的基因相耦合的策略同样能够应用于S. cerevisiae中。结合来源于E.coli的转录因子MetJ和其对应的操纵子metO,在S. cerevisiae中构建了SAM的生物传感器[60]。通过融合MetJ和转录激活区域B42,并将metO放置在报告基因上游,MetJ-B42混合体在S. cerevisiae中成为一个SAM诱导的转录激活子。为了更好地调制报告基因,研究者构建了双输入元件,报告基因受到TetR的调控。选用his3和leu2作为缺陷型报告信号,筛选了S. cerevisiae基因组突变库,鉴定出目标基因GAL11能够将SAM产量提高7倍。
3.3 动态调控代谢工程中,最大程度的表达生物合成途径中相应的酶通常不能够获得最高产量,这是由于酶和途径的过表达会给细胞带来代谢压力,导致途径中的有毒中间产物积累,或者使得重要代谢物的缺乏影响细胞生长。虽然目前已经有一些有效的优化途径基因表达的策略,如优化启动子强度,核糖体结合位点强度,酶的降解速率,以及近期发展迅速的能够实现多目标基因的调控dCas9[61]和反义-RNA[62]技术,但是这些策略大多是将表达水平控制在固定水平。利用代谢物生物传感器响应胞内代谢状态动态调控细胞内基因的代谢水平,模拟微生物天然存在的代谢调控网络,能够避免有毒代谢中间产物的过量积累,平衡细胞生长所需的前提供应与目标代谢产物的合成 ( 图 3-C ) 。
早在2000年,已经实现通过能够响应胞内乙酰磷酸的水平的调节因子Ntr,控制E.coli中番茄红素合成途径中的两个关键酶基因的表达水平,使得番茄红素产量提高约18倍[63]。另一个经典的案例是利用脂肪酰辅酶-A的生物传感器FadR动态调控脂肪酸乙酯 ( Fatty acid ethyl ester,FAEE ) 合成基因的表达[47]。FAEE合成包含乙醇和脂肪酰基辅酶A的合成反应,而这两个的前体物质对于生长有毒害或抑制作用。转录抑制因子FadR与酰基辅酶A和脂肪酸都有相对较弱的亲和作用。这一效应物的偏好性顺序,使得脂肪酸的积累诱导酰基辅酶A合酶的表达,随后脂肪酰基辅酶A合成反过来诱导从丙酮酸到乙醇的合成以及蜡质合成酶表达。这一动态调控元件避免了乙醇的过度累积,同时平衡了细胞生长和FAEE合成所需要的脂肪酸代谢流,在进一步优化启动子强度后,FAEE产量达到1.5 g/L,相比于没有动态调控的对照菌株提高了3.5倍。
丙二酰辅酶A是脂肪酸合成、黄酮类化合物合成途径中的重要前体,但是过表达丙二酰辅酶A的合成途径乙酰辅酶A羧化酶 ( accABCD编码 ) 对细胞生长有害。基于响应丙二酰辅酶A的转录因子FapR构建的负反馈元件,使脂肪酸合成产量提高了34%[56]。转录因子FapR通过结合在fapO上抑制基因表达,FapR与丙二酰辅酶A结合后,从结合位点脱离下来。随后的研究工作进一步揭示了FapR介导的E.coli启动子PGAP表达激活作用,和取决于丙二酰辅酶A的抑制作用,并采用了类似的动态调控设计,使得脂肪酸的产量相比于静态表达提高了2.1倍[64]。在S. cerevisiae中应用FapR和葡萄糖浓度响应的启动子PHXT1,围绕关键中间产物丙二酰辅酶A设计得分层动态调控系统,使得3-羟基丙酸的产量提高10倍[65]。
核糖开关同样被应用于动态调控系统。作为概念验证,来源于E.coli和B. subtilis的赖氨酸-OFF的核糖开关被整合在C. glutamicum染色体中gltA基因 ( 编码柠檬酸合酶 ) 上游,使得赖氨酸产量分别提高63%和38%[66]。为了进一步提高C. glutamicum中赖氨酸的产量,研究者开发了赖氨酸-ON核糖开关[67],动态上调赖氨酸转运蛋白 ( gltA ) 表达量。为了构造新的核糖开关,简并碱基被插入到赖氨酸核酸适配体与双重选择标记tetA上游的RBS区域。3轮正-反筛选后,成功构建了能够在赖氨酸存在时激活上游基因表达的核糖开关。利用这一赖氨酸-ON的核糖开关动态控制gltA表达量,赖氨酸产量提高了21%。
实现动态调控的另一重要策略在于实现细胞生长和目标产物生产过程的解耦。例如,利用良好表征的lux群体相应系统,当E.coli细胞密度达到理想水平,动态调控代谢流从TCA循环到异丙醇合成,异丙醇产量提高了3倍[68]。当酰基同型丝氨酸内酯 ( N- ( β-ketocaproyl ) -L-Homoserine lac- tone,AHL ) 浓度达到一定水平时,调节因子LuxR激活启动子Plux的表达。异丙醇生产途径和TetR抑制蛋白表达受到AHL-LuxR响应系统调控,而编码柠檬酸合酶的gltA基因受到TetR控制。同时能够通过控制添加的IPTG浓度和LacI蛋白表达调控LuxR开关发生时细胞浓度和AHL水平。
除了以上提到的利用转录因子和核糖开关构建的代谢物生物传感器实现动态调控外,压力响应的启动子实现代谢途径的动态调控也是重要的策略。能够特异性响应特定效应物的启动子也可以归为代谢物生物传感。通过转录组分析鉴定出了对于法尼焦磷酸 ( Farnesyl pyrophosphate,FPP ) 表现出压力响应启动的特性,利用FPP-抑制的启动子PgadE调节FPP合成,FPP-激活的启动子PrstA调节下游紫穗槐二烯的合成,相比于静态调控转化率提高了两倍[69]。相似的策略应用在S. cerevisiae中,通过下调竞争性麦角固醇合成途径,提高了紫穗槐二烯的产量[46]。通过结合修饰的GAL调控系统和葡萄糖浓度响应的启动子PHXT1分别调控的鲨烯合成途径和β-胡萝卜素途径,使甲羟戊酸途径和竞争性的鲨烯途径顺序响应葡萄糖浓度实现动态调控,使类胡萝卜素产量达到1 156 mg/L[45]。
3.4 非遗传细胞间异质性选择在蛋白和代谢物浓度水平上,细胞间天然存在非遗传因素导致的异质性。由于细胞内蛋白表达机器在不同细胞间存在随机分配现象,在任意培养体系中均存细胞间差异,针对目的产物的生产,也就是存在非遗传因素造成的高效生产和低效生产细胞[70]。由于低效的细胞消耗了营养却没有有效合成目标产物,非遗传细胞异质性的存在影响了目标代谢物的合成效率。基于转录因子的细胞群体控制系统 ( Population quality control,PopQC ) 能够持续富集高效生产的细胞,降低低效生产细胞的比例 ( 图 3-D ) ,进而提高合成效率[18]。PoPQC的概念在游离脂肪酸 ( Free fatty acid,FFA ) 生产中得到验证。高FFA浓度水平对应于细胞内高浓度的酰基辅酶A,酰基辅酶A与转录因子FadR结合后,激活下游TetA外排泵的表达,使细胞具有四环素抗性。通过PopQC富集培养的高产FFA的亚群体细胞,使得FFA的产量提高3倍。类似的策略应用于E.coli,利用特异性响应酪氨酸的TyrR转录因子,提高了酪氨酸生产。
以往的提高生物合成效率的策略大多忽视了非遗传细胞异质性差异的影响。但是,低效生产细胞的大量存在,特别是在工业生产中,不同时间尺度上微环境的差异会放大细胞异质性的影响,确实极大地限制了合成效率。因而易于设计和应用的PopQC若能与传统优化的策略相结合,有望成为提高合成效率,实现低成本合成的通用策略。
4 展望随着代谢工程、系统生物学、合成生物学的迅速发展,使得微生物能够生产一系列有价值的化合物。为了实现包括生物燃料、药物、大宗化学品等在内的化合物高效生产,需要在细胞中构建高效的合成途径,提高微生物细胞工厂的产量 ( Titer ) 、转化率 ( Yield ) 和生产速率 ( Productivity ) 。代谢物生物传感器作为合成生物学提供的有力工具,能够大大加快微生物细胞工厂的构建。高通量筛选和选择将大大提高代谢途径设计及优化过程中DBT循环的通量,加快构建的速度。而利用代谢物生物传感器实现宿主内代谢途径的动态调控将使得微生物具有更高的生产效率。基于代谢物生物传感器的减少由于非遗传差异导致的低效生产细胞比例的方法有望应用于工业微生物发酵,提高合成效率。但是对于代谢物生物传感器的生物元件的挖掘,对响应机理的进一步认识以及如何将其更好地应用于代谢工程仍旧是亟需研究的重点。
虽然目前已经报道了许多成功应用代谢物生物传感器的案例,但是想要实现广泛应用,仍旧需要进一步挖掘生物体内广泛存在的调控元件。生物信息学的发展为调控元件的挖掘奠定了基础,但是传统的基于全细胞挖掘代谢物生物传感器的方法,由于存在细胞膜与细胞壁的束缚,同时细胞内代谢调控网络复杂程度高,以及细胞培养过程使得设计-构建-检验的流程长,难以快速高通量定量评价代谢物生物传感器的性能。近年来,利用细胞提取物来完成转录翻译过程的无细胞体系迅速发展[71, 72]。基于无细胞合成体系进行代谢物生物传感器挖掘和构建工作,有望大大加快设计-构建-检验的进程。另一方面随着合成生物学技术的发展,有望为来源不同生物的调控元件移植到宿主中进行应用提供通用的策略,如将来源于细菌的转录激活子应用于酵母细胞中[73]。
在实际应用中,代谢物生物传感器的响应特性制约了应用需求。对于目标菌株的高通量筛选和选择而言,生物传感器的响应特性决定了其筛选和选择所得菌株的阈值和分辨率。对于动态调控而言,生物传感器的响应特性也尤为重要,研究表明不同响应特性的胞内代谢物生物传感器对回路动态调控的实际效果影响很大,目标产物的最终产量有时甚至相差数10倍[69]。如果要使代谢物生物传感器真正广泛应用到代谢工程研究和实际应用中,如何能够工程化传感器使其具备满足应用需求的响应特性成为亟需解决的难题。目前已经有一些经验性方法改造生物传感器的特性,如在转录水平改变相关基因的启动子强度[74-77],或在转录后水平RBS序列优化[78]、使用正交核糖体[79]、优化密码子[80]等策略,然而由于目前对蛋白质或者mRNA序列-结构-功能的认识仍旧处于非常初级的阶段[81],这些方法仍旧属于假设-试错循环的范畴,导致生物传感器改造周期漫长,很难对最终的改造结果进行预测。这使得代谢物生物传感器的应用受到重大挑战,急需解决如何从分子层次认识响应机理,配体识别机制,解析序列-结构-功能之间的关系等关键的科学问题。结合近年来快速发展的高通量分选技术和深度测序技术,有望开发快速、易于工程化应用的生物传感器改造方法。
随着生物元件的不断挖掘,对生物识别元件响应机理认识的深入,工程化改造方法的成熟,将会有更多的代谢物生物传感器应用于微生物细胞工厂的构建,结合快速发展的高通量测序技术、自动化技术等手段,有望实现微生物细胞工厂高通量、智能化构建。
| [1] | Michener JK, Thodey K, Liang JC, et al. Applications of genetically-encoded biosensors for the construction and control of biosynthetic pathways. Metab Eng, Elsevier, 2012, 14 (3): 212–222. DOI:10.1016/j.ymben.2011.09.004 |
| [2] | Eggeling L, Bott M, Marienhagen J. Novel screening methods-biosensors. Curr Opin Biotechnol, 2015, 35 : 30–36. |
| [3] | Qian S, Cirino PC. Using metabolite-responsive gene regulators to improve microbial biosynthesis. Current Opinion in Chemical Engineering, 2016, 14 : 93–102. DOI:10.1016/j.coche.2016.08.020 |
| [4] | Libis V, Delépine B, Faulon JL. Sensing new chemicals with bacterial transcription factors. Curr Opin Microbiol, 2016, 33 : 105–112. DOI:10.1016/j.mib.2016.07.006 |
| [5] | Liu D, Evans T, Zhang F. Applications and advances of metabolite biosensors for metabolic engineering. Metabolic Engineering, 2015, 31 : 15–22. |
| [6] | Williams TC, Pretorius IS, Paulsen IT. Synthetic evolution of metabolic productivity using biosensors. Trends in Biotechnology, 2016, 34 (5): 371–381. DOI:10.1016/j.tibtech.2016.02.002 |
| [7] | Latchman DS. Transcription factors:An overview. Int J Biochem Cell Biol, 1997, 29 (12): 1305–1312. DOI:10.1016/S1357-2725(97)00085-X |
| [8] | Binder S, Schendzielorz G, Stabler N, et al. A high-throughput approach to identify genomic variants of bacterial metabolite producers at the single-cell level. Genome Biology, 2012, 13 (5): 1–12. |
| [9] | Wang M, Li S, Zhao H. Design and engineering of intracellular-metabolite-sensingregulation gene circuits in Saccharomyces cerevisiae. Biotechnol Bioeng, 2016, 113 (1): 206–215. DOI:10.1002/bit.v113.1 |
| [10] | Dietrich JA, Shis DL, Alikhani A, et al. Transcription factor-based screens and synthetic selections for microbial small-molecule biosynthesis. ACS Synthetic Biology, 2013, 2 (1): 47–58. DOI:10.1021/sb300091d |
| [11] | Mustafi N, Grünberger A, Kohlheyer D, et al. The development and application of a single-cell biosensor for the detection of l-methionine and branched-chain amino acids. Metab Eng, 2012, 14 (4): 449–457. DOI:10.1016/j.ymben.2012.02.002 |
| [12] | Mahr R, G?tgens C, Gaetgens J, et al. Biosensor-driven adaptive laboratory evolution of l-valine production in Corynebacterium glutamicum. Metab Eng, 2015, 32 : 184–194. DOI:10.1016/j.ymben.2015.09.017 |
| [13] | Schendzielorz G, Dippong M, Grünberger A, et al. Taking control over control:Use of product sensing in single cells to remove flux control at key enzymes in biosynthesis pathways. ACS Synthetic Biology, 2014, 3 (1): 21–29. DOI:10.1021/sb400059y |
| [14] | Chou HH, Keasling JD. Programming adaptive control to evolve increased metabolite production. Nature Communications, 2013, 4 (2595): 1–8. |
| [15] | Raman S, Rogers JK, Taylor ND, et al. Evolution-guided optimization of biosynthetic pathways. Proceedings of the National Academy of Sciences, 2014, 111 (50): 17803–17808. DOI:10.1073/pnas.1409523111 |
| [16] | Siedler S, Schendzielorz G, Binder S, et al. SoxR as a single-cell biosensor for NADPH-consuming enzymes in Escherichia coli. ACS Synthetic Biology, 2014, 3 (1): 41–47. DOI:10.1021/sb400110j |
| [17] | Li S, Si T, Wang M, et al. Development of a synthetic Malonyl-CoA sensor in Saccharomyces cerevisiae for intracellular metabolite monitoring and genetic screening. ACS Synthetic Biology, 2015, 4 (12): 1308–1315. DOI:10.1021/acssynbio.5b00069 |
| [18] | Xiao Y, Bowen CH, Liu D, et al. Exploiting non-genetic, cell-to-cell variation for enhanced biosynthesis. Nature Chemical Biology, 2016, 12 (5): 339–344. DOI:10.1038/nchembio.2046 |
| [19] | Tang SY, Fazelinia H, Cirino PC. AraC regulatory protein mutants with altered effector specificity. Journal of the American Chemical Society, 2008, 130 (15): 5267–5271. DOI:10.1021/ja7109053 |
| [20] | Tang SY, Cirino PC. Design and application of a mevalonate-responsive regulatory protein. Angewandte Chemie-International Edition, 2011, 50 (5): 1084–1086. DOI:10.1002/anie.v50.5 |
| [21] | Frei CS, Wang Z, Qian S, et al. Analysis of amino acid substitutions in AraC variants that respond to triacetic acid lactone. Protein Science, 2016, 25 (4): 804–814. DOI:10.1002/pro.2873 |
| [22] | Tang SY, Qian S, Akinterinwa O, et al. Screening for enhanced triacetic acid lactone production by recombinant Escherichia coli expressing a designed triacetic acid lactone reporter. J Am Chem Soc, 2013, 135 (27): 10099–10103. DOI:10.1021/ja402654z |
| [23] | Chen W, Zhang S, Jiang P, et al. Design of an ectoine-responsive AraC mutant and its application in metabolic engineering of ectoine biosynthesis. Metab Eng, 2015, 30 : 149–155. DOI:10.1016/j.ymben.2015.05.004 |
| [24] | Liang WF, Cui LY, Cui JY, et al. Biosensor-assisted transcriptional regulator engineering for Methylobacterium extorquens AM1 to improve mevalonate synthesis by increasing the acetyl-CoA supply[J]. Metabolic Engineering, 2016 doi:10.1016/j.ymben.2016.11.010. |
| [25] | Epshtein V, Mironov AS, Nudler E. The riboswitch-mediated control of sulfur metabolism in bacteria. Proc Natl Acad Sci USA, 2003, 100 (9): 5052–5056. DOI:10.1073/pnas.0531307100 |
| [26] | Mellin JR, Cossart P. Unexpected versatility in bacterial riboswi-tches. Trends in Genetics, 2015, 31 (3): 150–156. DOI:10.1016/j.tig.2015.01.005 |
| [27] | Breaker RR. Prospects for riboswitch discovery and analysis. Molecular Cell, 2011, 43 (6): 867–879. DOI:10.1016/j.molcel.2011.08.024 |
| [28] | Winkler WC, Nahvi A, Roth A, et al. Control of gene by a natural metabolite-responsive ribozyme. Nature, 2004, 428 (6980): 281–286. DOI:10.1038/nature02362 |
| [29] | Grundy FJ, Lehman SC, Henkin TM. The L box regulon:lysinesensing by leader RNAs of bacterial lysine biosynthesis genes. Proc Natl Acad Sci USA, 2003, 100 (21): 12057–62. DOI:10.1073/pnas.2133705100 |
| [30] | Mandal M. A glycine-dependent riboswitch that uses cooperative binding to control gene expression. Science, 2004, 306 (5694): 275–279. DOI:10.1126/science.1100829 |
| [31] | Winkler W, Nahvi A, Breaker RR. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene. Nature, 2002, 419 (6910): 952–956. DOI:10.1038/nature01145 |
| [32] | Nahvi A, Sudarsan N, Ebert MS, et al. Genetic control by a metabo-lite binding mRNA. Chem Biol, 2002, 9 (9): 1043–1049. DOI:10.1016/S1074-5521(02)00224-7 |
| [33] | Sudarsan N, Wickiser JK, Nakamura S, et al. An mRNA structure in bacteria that controls gene by binding lysine. Genes and Development, 2003, 17 (21): 2688–2697. DOI:10.1101/gad.1140003 |
| [34] | Winkler WC. An mRNA structure that controls gene by binding FMN. Proc Natl Acad Sci USA, 2002, 99 (25): 15908–15913. DOI:10.1073/pnas.212628899 |
| [35] | Mironov AS, Gusarov I, Rafikov R, et al. Sensing small molecules by nascent RNA:A mechanism to control transcription in bacteria. Cell, 2002, 111 (5): 747–756. DOI:10.1016/S0092-8674(02)01134-0 |
| [36] | Dann CE, Wakeman CA, Sieling CL, et al. Structure and mechanism of a metal-sensing regulatory RNA. Cell, 2007, 130 (5): 878–892. DOI:10.1016/j.cell.2007.06.051 |
| [37] | Baker JL, Sudarsan N, Weinberg Z, et al. Widespread genetic switches and toxicity resistance proteins for fluoride. Science, 2012, 335 (6065): 233–235. DOI:10.1126/science.1215063 |
| [38] | Cho EJ, Lee JW, Ellington AD. Applications of aptamers as sensors. Annu Rev Anal Chem, 2009, 2 : 241–264. DOI:10.1146/annurev.anchem.1.031207.112851 |
| [39] | Strack RL, Jaffrey SR. New approaches for sensing metabolites and proteins in live cells using RNA. Current Opinion in Chemical Biology, 2013, 17 (4): 651–655. DOI:10.1016/j.cbpa.2013.05.014 |
| [40] | Henkin TM. Riboswitch RNAs:using RNA to sense cellular metabolism. Genes Dev, 2008, 22 (24): 3383–3390. DOI:10.1101/gad.1747308 |
| [41] | Fowler CC, Brown ED, Li Y. Using a riboswitch sensor to examine coenzyme B12 metabolism and transport in E. coli[J]. Chemistry and Biology, 2010, 17(7):756-765. |
| [42] | Michener JK, Smolke CD. High-throughput enzyme evolution in Saccharomyces cerevisiae using a synthetic RNA switch. Metabolic Engineering, 2012, 14 (4): 306–316. DOI:10.1016/j.ymben.2012.04.004 |
| [43] | Gredell JA, Frei CS, Cirino PC. Protein and RNA engineering to customize microbial molecular reporting. Biotechnology Journal, 2012, 7 (4): 477–499. DOI:10.1002/biot.v7.4 |
| [44] | Espah Borujeni A, Mishler DM, Wang J, et al. Riboswitch calculator. Design of synthetic riboswitches from diverse RNA aptamers. [EB/OL]. 2014. https://salis. psu. edu/software/RiboswitchCalculator_EvaluateMode. |
| [45] | Xie W, Ye L, Lv X, et al. Sequential control of biosynthetic pathways for balanced utilization of metabolic intermediates in Saccharomyces cerevisiae. Metab Eng, 2015, 28 : 8–18. DOI:10.1016/j.ymben.2014.11.007 |
| [46] | Yuan J, Ching CB. Dynamic control of ERG9 for improved amorpha-4, 11-diene production in Saccharomyces cerevisiae. Microbial Cell Factories, 2015, 14 (38): 1–10. |
| [47] | Zhang F, Carothers JM, Keasling JD. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids. Nat Biotechnol, 2012, 30 (4): 354–359. DOI:10.1038/nbt.2149 |
| [48] | Fang M, Wang T, Zhang C, et al. Intermediate-sensor assisted push-pull strategy and its application in heterologous deoxyviolacein production in Escherichia coli. Metab Eng, 2016, 33 : 41–51. DOI:10.1016/j.ymben.2015.10.006 |
| [49] | Peroza EA, Ewald JC, Parakkal G, et al. A genetically encoded F?rster resonance energy transfer sensor for monitoring in vivo trehalose-6-phosphate dynamics. Analytical Biochemistry, 2015, 474 : 1–7. DOI:10.1016/j.ab.2014.12.019 |
| [50] | Behjousiar A, Kontoravdi C, Polizzi KM. In situ monitoring of intracellular glucose and glutamine in CHO cell culture. PLoS One, 2012, 7 (4): 1–9. |
| [51] | Dietrich JA, Mckee AE, Keasling JD. High-throughput metabolic engineering:Advances in small-molecule screening and selection. Annu Rev Biochem, 2010, 79 (1): 563–590. DOI:10.1146/annurev-biochem-062608-095938 |
| [52] | Rogers JK, Church GM. Multiplexed engineering in biology. Trends in Biotechnology, 2016, 34 (3): 198–206. DOI:10.1016/j.tibtech.2015.12.004 |
| [53] | Jeong YS, Choi SL, Kyeong HH, et al. High-throughput screening system based on phenolics-responsive transcription activator for directed evolution of organophosphate-degrading enzymes. Protein Eng Des Sel, 2012, 25 (11): 725–731. DOI:10.1093/protein/gzs071 |
| [54] | Choi SL, Rha E, Lee SJ, et al. Toward a generalized and high-throughput enzyme screening system based on artificial genetic circuits. ACS Synthetic Biology, 2014, 3 (3): 163–171. DOI:10.1021/sb400112u |
| [55] | Mcdonald AG, Boyce S, Tipton KF. ExplorEnz:The primary source of the IUBMB enzyme list[J]. Nucleic Acids Research, 2009, 37(suppl. 1):593-597. |
| [56] | Liu D, Xiao Y, Evans BS, et al. Negative feedback regulation of fatty acid production based on a malonyl-CoA sensor-actuator. ACS Synthetic Biology, 2015, 4 (2): 132–140. DOI:10.1021/sb400158w |
| [57] | Xu P, Wang W, Li L, et al. Design and kinetic analysis of a hybrid promoter-regulator system for malonyl-CoA sensing in Escherichia coli. ACS Chemical Biology, 2014, 9 (2): 451–458. DOI:10.1021/cb400623m |
| [58] | Meyer A, Pellaux R, Potot S, et al. Optimization of a whole-cell biocatalyst by employing genetically encoded product sensors inside nanolitre reactors. Nature Chemistry, 2015, 7 (8): 673–678. DOI:10.1038/nchem.2301 |
| [59] | Yang J, Seo SW, Jang S, et al. Synthetic RNA devices to expedite the evolution of metabolite-producing microbes. Nature Communications, 2013, 4 : 1–7. |
| [60] | Umeyama T, Okada S, Ito T. Synthetic gene circuit-mediated monitoring of endogenous metabolites:Identification of GAL11 as a novel multicopy enhancer of S-adenosylmethionine level in yeast. ACS Synthetic Biology, 2013, 2 (8): 425–430. DOI:10.1021/sb300115n |
| [61] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene. Cell, 2013, 152 (5): 1173–1183. DOI:10.1016/j.cell.2013.02.022 |
| [62] | Nakashima N, Ohno S, Yoshikawa K, et al. A vector library for silencing central carbon metabolism genes with antisense RNAs in Escherichia coli. Appl Environ Microbiol, 2014, 80 (2): 564–573. DOI:10.1128/AEM.02376-13 |
| [63] | Farmer WR, Liao JC. Improving lycopene production in Escherichia coli by engineering metabolic control. Nature Biotechnology, 2000, 18 (5): 533–537. DOI:10.1038/75398 |
| [64] | Xu P, Li L, Zhang F, et al. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control. Proc Natl Acad Sci USA, 2014, 111 (31): 11299–11304. DOI:10.1073/pnas.1406401111 |
| [65] | David F, Nielsen J, Siewers V. Flux control at the Malonyl-CoA node through hierarchical dynamic pathway regulation in Saccharomyces cerevisiae. ACS Synth Biol, 2016, 5 (3): 224–233. DOI:10.1021/acssynbio.5b00161 |
| [66] | Zhou LB, Zeng AP. Exploring lysine riboswitch for metabolic flux control and improvement of l-lysine synthesis in Corynebacterium glutamicum. ACS Synthetic Biology, 2015, 4 (6): 729–734. DOI:10.1021/sb500332c |
| [67] | Zhou LB, Zeng AP. Engineering a lysine-ON riboswitch for metabolic control of lysine production in Corynebacterium glutamicum. ACS Synth Biol, 2015, 4 (12): 1335–1340. DOI:10.1021/acssynbio.5b00075 |
| [68] | Soma Y, Hanai T. Self-induced metabolic state switching by a tunable cell density sensor for microbial isopropanol production. Metab Eng, 2015, 30 : 7–15. DOI:10.1016/j.ymben.2015.04.005 |
| [69] | Dahl RH, Zhang F, Alonso-Gutierrez J, et al. Engineering dynamic pathway regulation using stress-response promoters. Nature Biotechnology, 2013, 31 (11): 1039–1046. DOI:10.1038/nbt.2689 |
| [70] | Lidstrom ME, Konopka MC. The role of physiological heterogeneity in microbial population behavior. Nature Chemical Biology, Nature Publishing Group, 2010, 6 (10): 705–712. |
| [71] | Carlson ED, Gan R, Hodgman CE, et al. Cell-free protein synthesis:Applications come of age. Biotechnology Advances, 2012, 30 (5): 1185–1194. DOI:10.1016/j.biotechadv.2011.09.016 |
| [72] | Hodgman CE, Jewett MC. Cell-free synthetic biology:Thinking outside the cell. Metab Eng, 2012, 14 (3): 261–269. DOI:10.1016/j.ymben.2011.09.002 |
| [73] | Skjoedt M, Snoek T, Kildegaard K, et al. Engineering prokaryotic transcriptional activators as metabolite biosensors in yeast. Nature Chemical Biology, 2016, 12 (11): 951–958. DOI:10.1038/nchembio.2177 |
| [74] | De Mey M, Maertens J, Lequeux GJ, et al. Construction and model-based analysis of a promoter library for E. coli:an indispensable tool for metabolic engineering[J]. BMC Biotechnology, 2007, 7(1):1-14. |
| [75] | Block DHS, Hussein R, Liang LW, et al. Regulatory consequences of gene translocation in bacteria. Nucleic Acids Research, 2012, 40 (18): 8979–8992. DOI:10.1093/nar/gks694 |
| [76] | Murphy KF, Balázsi G, Collins JJ. Combinatorial promoter design for engineering noisy gene. Proc Natl Acad Sci USA, 2007, 104 (31): 12726–12731. DOI:10.1073/pnas.0608451104 |
| [77] | Shis DL, Bennett MR. Library of synthetic transcriptional and gates built with split T7 RNA polymerase mutants. Proc Natl Acad Sci USA, 2013, 110 (13): 5028–5033. DOI:10.1073/pnas.1220157110 |
| [78] | Salis HM, Mirsky EA, Voigt CA. Automated design of synthetic ribosome binding sites to control protein. Nature Biotechnology, 2009, 27 (10): 946–950. DOI:10.1038/nbt.1568 |
| [79] | Chubiz LM, Rao CV. Computational design of orthogonal ribosomes. Nucleic Acids Res, 2008, 36 (12): 4038–4046. DOI:10.1093/nar/gkn354 |
| [80] | Welch M, Govindarajan S, Ness JE, et al. Design parameters to control synthetic gene in Eschorichia coli. PLoS One, 2009, 4 (9): 1–10. |
| [81] | Raman S, Taylor N, Genuth N, et al. Engineering allostery. Trends in Genetics, 2014, 30 (12): 521–528. DOI:10.1016/j.tig.2014.09.004 |




