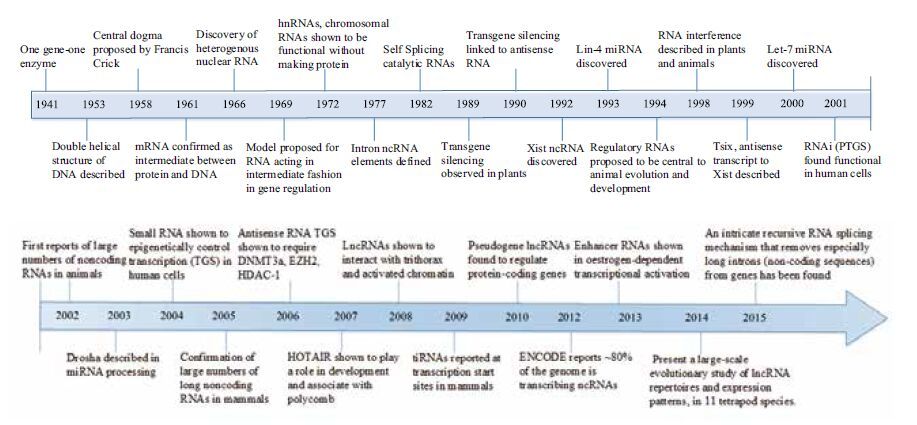
非编码序列、非编码基因以及非编码RNA的研究已成为生命科学领域研究的热点,生命起源于RNA分子这一话题重新唤起了人们对RNA世界的重视。非编码RNA(non-coding RNA,ncRNA)曾一度被认为是无功能的“垃圾序列(junk sequence)”、“转录噪声(transcriptional noise)”,但现在大量的研究发现它能够调控基因的转录、翻译等重要生命过程。具有基因特异性调控作用的长链非编码RNA(long non-coding RNA,lncRNA)在20世纪90年代早期被发现。由于基因组微阵列技术的开发和基因组、转录组测序技术的发展使研究非编码RNA成为可能[1],越来越多的非编码RNA被发现,为人们展示一个丰富的非编码RNA的世界[2-4],见图 1。
1 长链非编码RNA的概述 1.1 非编码RNA的种类非编码RNA主要是分为短链非编码RNA和长链非编码RNA,包括转运RNA(tRNA),核糖体RNA(rRNA),小核RNA(snoRNA),微RNA(microRNA),小干扰RNA(siRNA),PIWI蛋白相互作用的RNA(piRNA)和不同类型的长链非编码RNA(lncRNA),见表 1。
长链非编码RNA是指长度大于200 nt的非编码RNA,该概念在2002年由日本科学家首次提出[10],但并未得到研究人员的关注。直到2007年,Rinn等[11]发现了在乳腺癌组织中表达增加的HOTAIR—一条2.2 kb的功能性长链非编码RNA基因,研究发现HOTAIR RNA可以与蛋白复合体 polycomb2相互作用,修饰染色质,抑制HOX基因的转录,并进而调节生物体的生长发育。全基因组技术的发展、“普遍转录”的概念的提出,尤其是伴随着芯片和下一代测序技术的发展,越来越多功能性非编码RNA相继被发现,如经典的长链非编码RNA,Xist[12]、H19[13]和JPX[14]等。
长链非编码RNA具有5′端帽子结构和3′端聚腺苷酸尾巴,其基因结构类似mRNA[15],长链非编码RNA和miRNA可相互作用,可以充当天然“miRNA海绵”降低miRNA水平[16],而miRNA能够介导长链非编码RNA降解,长链非编码RNA和miRNA二者又可竞争结合mRNAs。长链非编码RNA还可与其他生物大分子(DNA、RNA及蛋白质)发生相互作用,从多个层次上调控其生物功能[17]。许多已确定的长链非编码RNA是由RNA聚合酶Ⅱ(RNAPⅡ)转录和可变剪切。长链非编码RNA与癌症、肿瘤等许多复杂疾病的发生和发展的过程相关[18, 19]。长链非编码RNA也是肿瘤抑制或癌基因表达的调节剂。Wang等[20]阐述了长链非编码RNA的4种不同的效应机制,认为这些分子可以作为信号分子、诱饵分子及导向、功能支架等。长非编码 RNA 具有复杂多样的生物功能,Guttman等[21]分析探讨了其生物功能潜在的分子机制,提出长非编码 RNA与其他生物分子相互作用形成某种模块结构发挥其生物功能。Batista等[22]提出长非编码 RNA 是细胞地址码关键组分的假说。长链非编码RNA调控表达的分子机制是多样的,分为顺式调控和反式调控,能够参与mRNA的拼接、组蛋白的修饰,还能够结合转录因子激活或抑制下游基因的表达等,关于长链非编码RNA的复杂生物学机制仍然不是很清楚,需进一步研究。
长链非编码RNA主要分为正义lncRNA(sense ln-cRNA)、反义lncRNA(antisence RNA)、双向lncRNA((bidirectional lncRNA)、基因内lncRNA(intronic ln-cRNA)及基因间lncRNA(intergenic lncRNA)5种类型。基因间长链非编码RNA(lincRNAs)是新发现lncRNAs。这种RNA的长度范围从几百到数万个碱基,数千碱基它们的基因组中位于两个基因之间的间隔。超过3 000个人类的lincRNAs已被确定,不同的lincRNAs参与多种生物过程。相对传统的长链非编码RNA,近年来又发现几种类型的长链非编码RNA,包括增强子RNA(eRNA)、竞争性内源RNA(ceRNAs)、环形RNA及反向的长链非编码RNA[23-25]。
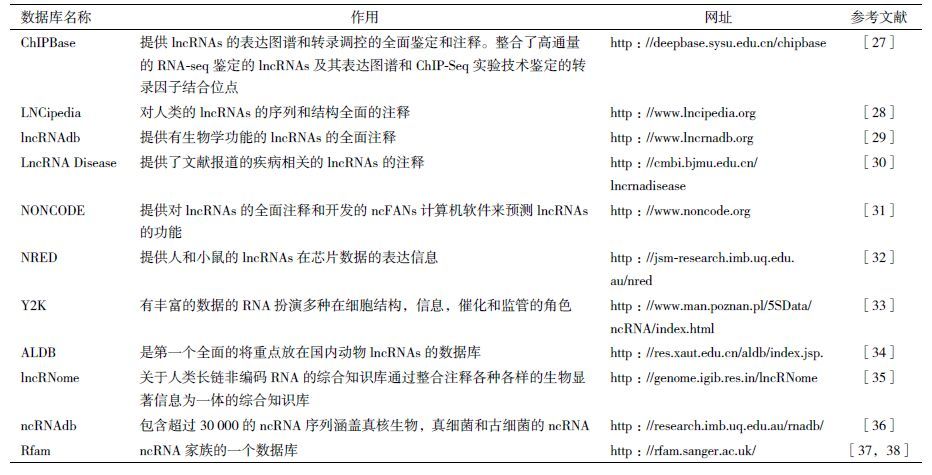
2 长链非编码RNA有关的数据库现在越来越多的长链非编码RNA被鉴定出来,相应的数据库也逐渐的建立,NONCODE是ncRNA权威的综合的知识数据库之一,专用于非编码RNA(不含tRNA和rRNA)。还有其他的一些辅助工具包括PLEK[26],PLEK是一个高效校准、免费的计算工具来区分mRNA和长链非编码RNA。在物种缺乏参考基因组RNA的转录序列的,PLEK特别适合PacBio或454测序数据和大规模转录数据(网址:Https://sourceforge.net/projects/plek/files/.)还有一些其他的常用的数据库,见表 2。
长链非编码RNA在真核生物中履行许多基本的调节功能,其失调与人类疾病有密切相关,包括癌症、肿瘤等。研究表明长链非编码RNA参与不同的生理过程,能够维持细胞的稳态,是基因表达,细胞分化,增殖,迁移,细胞凋亡的重要调节剂,也对干细胞有维护的作用。长链非编码RNA改变和它们的表达失调及活性破坏与癌细胞的病理变化相关联,这些长链非编码RNA分子可作为诊断标志物和治疗靶标。
与癌症相关的非编码RNA是19世纪20年代伴随多重PCR技术的使用而首次被发现,H19是第一个被发现与癌症相关的长链非编码RNA,之后Xist、mirRNA lin-4相继被发现,SAGE、全基因组转录平铺阵列、转录测序等技术的发展,进一步促使相关的非编码RAN的发现。关于反义RNA Tist能够调剂Xist、与癌症相关的miRNA、肺癌转移相关的转录物MALAT1,与癌症中调剂表达相关的TUCRs都相继报道。HULC是首个在肝癌中发现的过表达的长链非编码RNA[39]。PCGEM1在胰腺癌早期的发生和发展中起重要作用[40],表明长链非编码RNA在癌症领域的研究已经逐渐开始,下面介绍几个常见且研究机制比较清楚的与癌症相关的长链非编码RNA。
3.1 H19H19是一个长2.3 kb的长链非编码RNA基因,在脊椎动物胚胎发育过程中高表达,但之后的发育过程中除骨骼组织和软骨组织外的大多数组织中表达下调[41-43]。研究发现在肝癌、膀胱癌和乳腺癌中发现H19被上调,这意味着H19可能与癌症的发生有关[44, 45]。在结肠癌症中证明其是直接由致癌转录因子c-Myc活化,这表明H19可能是在c-Myc和下游基因表达之间起介导作用[46]。相反,在肿瘤抑制基因和转录激活p53基因被证明下调H19表达[47, 48]。H19转录作为前体的miR-675(参与发育调控)[49]。H19还能与血管生成素和成纤维细胞生长因子相互作用,可改变其表达而诱发肿瘤[50, 51]。
3.2 HOTAIRHOTAIR是一个长度为2.2 kb的基因,位于哺乳动物12q13.13上[52]。HOTAIR基因发展在原发性和转移性乳腺肿瘤上表达高度上调,高度上调,能够增加正常乳腺组织的转录,可达正常转录水平的2 000倍[53]。此外,研究证明,如果表达HOTAIR基因的细胞被移植到小鼠乳腺组织,会增加原发肿瘤的生长速率[53]。HOTAIR RNA 在剪接和聚腺苷酸化时不编码任何蛋白质,但证明与蛋白质抑制复合体(polycomb repressive complex2,PCR2)有关联,PCR2由H3K-27甲基化酶EZH2和EED组成[53-55]。蛋白家族(PcG)能够调节成千上万个基因的转录抑制和控制分化的途径,在多能干细胞和人类癌症上起作用。HOXD座位在第2号染色体上的基因位点是PRC2靶标,HOTAIR作为一个分子支架出现,至少与2个不同的组蛋白修饰复合物的结合。RNA的5′区结合PRC2复合体负责H3K27甲基化,而3′的HOTAIR区域结合LSD1,组蛋白赖氨酸甲基化酶介导H3K4me2[54, 56]脱甲基酶。虽然,HOTAIR的精确机制还不是很清楚,但很显然HOTAIR能促进肿瘤染色质转移,与癌症、肿瘤密切相关。
3.3 CCAT2CCAT2是一种新发现的长度为0.34 kb的长链非编码RNA,位于哺乳动物8q24.21上,与结肠直肠癌、肺癌和乳腺癌有关。2013年Ling等[57]发现,CCAT2与和癌症相关的rs6983267 SNP有关联,有研究表明此SNP影响CCAT2表达。CCAT2在CRC(大肠癌)肿瘤组织高水平表达,但在正常结肠组织中以较低的水平表达[57]。经过全基因组关联研究确定了SNP rs6983267和癌症风险之间的关联,得出CCAT2能促进肿瘤的生长和转移、诱导染色体不稳定[57]。Redis等[58]证明CCAT2在乳腺肿瘤中表达,通过实时荧光定量PCR和原位杂交的方法,综合其在正常乳腺组织和患病者组织表达情况,并分析病患在临床、组织学、病理学及其他生物学因素下CCAT2的表达情况,最终得出,CCAT2的表达影响SNP位点基因型对细胞迁移和耐药。
4 非编码RNA生物信息学分析考虑到长链非编码RNA的作用,有必要对长链非编码RNA有一个系统的了解。但是,现有的资源仅仅描述长链非编码RNA的基因结构。为了找到非编码RNA基因,要有意义的搜索RNA(辅助)结构,而不是一级序列。这是比搜索蛋白质编码空间结构更有挑战性和苛刻的问题,因为RNA的结构化序列的不正常的信号,如开放阅读框。现在预测长链非编码RNA的方法主要是通过RNA二级结构的保守性实现。利用二代测序技术Illumina、Solexa技术、高通量RNA测序(RNA-Seq)及计算方法,已从各种生物体内识别越来越多的非编码RNA。
长链非编码RNA尽管缺乏编码蛋白能力,但在很多方面类似mRNA,没有开放阅读框架但具有mRNA结构特征(帽式结构和PolyA尾巴)的RNA[59]。参与剪接模式也类似的mRNA[60],在基因的位置上与mRNA有重叠,而且表达量相比mRNA较低。此外,大多数长链非编码RNA的拼接具有类似的外显子/内含子的长度[60, 61]。因此可以从区别mRNA和长链非编码RNA角度来鉴别长链非编码RNA。
4.1 预测长链非编码RNA的方法为识别长链非编码RNA,首先要获得所有转录本,包括在细胞中的ncRNAs和mRNA,然后区分mRNA和其他类型的长链非编码RNA。传统的技术,如芯片,专注于鉴定的蛋白质编码RNA转录物。新的技术,如RNA测序,不限于蛋白质编码RNA转录物的鉴定,通过该技术已发现许多新的非编码RNA转录本。
4.1.1 芯片技术是传统的微阵列技术使用探针来确定mRNA的表达水平的转录本来确定长链非编码RNA。
4.1.2 SAGE和EST(SAGE基因表达系列分析)技术产生大量短序列标签并且能够识别已知和未知的转录本,SAGE被证明是研究长链非编码RNA一种有效的方法。Gibb等[62]编制了272个SAGE库,超过240万个标签,它们能够在人体正常组织和癌组织中识别长链非编码RNA的表达。Lee等[63]用SAGE在雄性生殖细胞识别潜在的长链非编码RNA。然而,在大规模研究中,SAGE与芯片相比昂贵的多,因此没有被广泛采用。EST(表达序列标签)是一种短序列的cDNA,是从cDNA克隆的单稳测序产生的。现在的公共数据库包含超过7 260万EST(2011基因库),使得它可能发现新的转录本。
4.1.3 RNA-Seq是随着下一代测序(NGS)技术的发展而出现的,RNA测序已被广泛地用于新的转录本发现和基因表达分析。在研究基因表达时有很多优点,能够检测到低峰度的转录本,并且能确定新的剪接异构体。RNA-seq是目前使用最广泛的鉴定长链非编码RNA的技术。Nam等[64]整合了RNA-seq、poly(A)位点、染色体信息,获得长链非编码RNA。Prensner等[65]使用RNA-seq在人类前列腺癌组织和细胞上研究长链非编码RNA,并得出结论其可用于癌症亚型分类。
4.1.4 RNA的免疫共沉淀(RNA-IP)技术是一种新型开发出鉴别长链非编码RNA与特定的蛋白质。首先使用蛋白质的抗体分离长链非编码RNA蛋白质复合物。然后,利用RNA-IP构建cDNA文库随后进行交互的深度测序长链非编码RNA。Zhao等[66]利用该技术发现了一个1.6 kb的Xist,Xist在一定范围内与PRC2相互作用。
4.1.5 计算机方法来预测长链非编码RNA,包括ORF长度策略、二级序列的保守型、机器学习的方法,如CONC利用一系列蛋白质的功能,如氨基酸组成,二级结构和肽长度,利用SVM模型区分长链非编码RNA。从mRNA[67]CPC(编码潜在的计算器)也使用SVM进行建模、利用提取序列特征和比较基因组学的特性来评估转录和编码潜力[68, 69]。Lu等[70]发展机器学习的方法,集成CG含量,DNA保护和表达信息来预测线虫长链非编码RNA。
4.2 非编码RNA生物信息学分析对于特定类型的ncRNA,可以使用现成的特定数据库和预测方法。例如,预测miRNA的miRBase、预测tRNA的tRNA-SE和GtRNAdb、预测snoRNA的snoSeeker等。预测全基因组的ncRNA还可以通过二级结构预测算法寻找新的非编码RNA,包括热力学模型、同源比对模型、统计学习模型等。二级结构还可以与各种芯片数据、RAN测序数据及蛋白质序列数据整合,通过机器学习手段,在全基因上对非编码RNA进行预测。
序列的相似性构建的模型,最基本的形式是使用BLAST寻找序列,如基因组和EST注释[71, 72],或通过构建一些模型来预测RNA结构,模型的数据可以从RNAseP RNA和SRP RNAs[73, 74]获得,或从数据库Rfam获得。还可以对RNA二级结构做了协方差模型,例如INFERNAL和RaveNnA[75-77]。这些使RNA结构连同相应的序列变异构成的一个更具体的概率模型。由于RNA的结构是变化的,所以预测RNA结构的模型具有专一性。典型的例子就是RNAse PRNA[78]和telomerase RNA[79]。这些依靠结构来寻找非编码RNA的整体思路属于比较基因组学,并且同时需考虑RNA二级结构折叠自由能,以及二级结构的保守性和稳定性。虽然部分发挥功能的RNA往往具有稳定的结构,但仅通过最低折叠自由能的计算不足以在全基因组范围内将ncRNA鉴别出来。
5 展望长链非编码RNA一直被视为转录过程的“暗物质”物(dark matter)而未受重视[80]。近年来通过对长链非编码RNA的生物信息学预测及在癌症方面的研究,逐步揭示非编码 RNA 的重要生物学功能。长链非编码RNA作为一类特殊的RNA分子,已成为遗传学和分子生物学关注的新热点。长链非编码RNA与癌症的关系及如何预测新的长链非编码RNA还是未来研究的重点。
| [1] | ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature , 2012, 489 (7414) : 57–74. DOI:10.1038/nature11247 |
| [2] | Morris KV, Mattick JS. The rise of regulatory RNA. Nature Reviews Genetics , 2014, 15 (6) : 423–437. DOI:10.1038/nrg3722 |
| [3] | Necsulea A, Soumillon M, Warnefors M, et al. The evolution of lncRNA repertoires and expression patterns in tetrapods. Nature , 2014, 505 (7485) : 635–640. DOI:10.1038/nature12943 |
| [4] | Scotti MM, Swanson MS. RNA mis-splicing in disease. Nature Reviews Genetics , 2016, 17 (1) : 19–32. |
| [5] | 陈润生. 关于非编码 RNA 研究的一些思考[C]. 第四届全国生物信息学与系统生物学学术大会论文集, 2010. http://cpfd.cnki.com.cn/article/cpfdtotal-zgxk201010001002.htm |
| [6] | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell , 1993, 75 (5) : 843–854. DOI:10.1016/0092-8674(93)90529-Y |
| [7] | Fire A, Xu SQ, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature , 1998, 391 (6669) : 806–811. DOI:10.1038/35888 |
| [8] | Elbashir SM, Harborth J, Lendeckel W, et al. Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. Nature , 2001, 411 (6836) : 494–498. DOI:10.1038/35078107 |
| [9] | Kung JTY, Colognori D, Lee JT. Long noncoding RNAs:past, present, and future. Genetics , 2013, 193 (3) : 651–669. DOI:10.1534/genetics.112.146704 |
| [10] | Okazaki Y, Furuno M, Kasukawa T, et al. Analysis of the mouse transcriptome based on functional annotation of 60, 770 full-length cDNAs. Nature , 2002, 420 (6915) : 563–573. DOI:10.1038/nature01266 |
| [11] | Rinn JL, Kertesz M, Wang JK, et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell , 2007, 129 (7) : 1311–1323. DOI:10.1016/j.cell.2007.05.022 |
| [12] | Bartolomei MS, Zemel S, Tilghman SM. Parental imprinting of the mouse H19 gene. Nature , 1991, 351 (6322) : 153–155. DOI:10.1038/351153a0 |
| [13] | Brockdorff N, Ashworth A, Kay GF, et al. Conservation of position and exclusive expression of mouse Xist from the inactive X chromosome. Nature , 1991, 351 (6324) : 329–331. DOI:10.1038/351329a0 |
| [14] | Tian D, Sun S, Lee JT. The long noncoding RNA, Jpx, is a molecular switch for X chromosome inactivation. Cell , 2010, 143 (3) : 390–403. DOI:10.1016/j.cell.2010.09.049 |
| [15] | Guttman M, Amit I, Garber M, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature , 2009, 458 : 223–227. DOI:10.1038/nature07672 |
| [16] | Yoon JH, Abdelmohsen K, Srikantan S, et al. LincRNA-p21 suppresses target mRNA translation. Molecular Cell , 2012, 47 (4) : 648–655. DOI:10.1016/j.molcel.2012.06.027 |
| [17] | Mercer TR, Mattick JS. Structure and function of long noncoding RNAs in epigenetic regulation. Nature Structural & Molecular Biology , 2013, 20 (3) : 300–307. |
| [18] | Tsai MC, Spitale RC, Chang HY. Long intergenic noncoding RNAs:New links in cancer progression. Cancer Res , 2011, 71 : 3–7. |
| [19] | Wapinski O, Chang HY. Long noncoding RNAs and human disease. Trends Cell Biol , 2011, 21 : 354–361. DOI:10.1016/j.tcb.2011.04.001 |
| [20] | Wang KC, Chang HY. Molecular mechanisms of long noncoding RNAs. Molecular Cell , 2011, 43 (6) : 904–914. DOI:10.1016/j.molcel.2011.08.018 |
| [21] | Guttman M, Rinn JL. Modular regulatory principles of large non-coding RNAs. Nature , 2012, 482 (7385) : 339–346. DOI:10.1038/nature10887 |
| [22] | Batista PJ, Chang HY. Long noncoding RNAs:cellular address codes in development and disease. Cell , 2013, 152 (6) : 1298–1307. DOI:10.1016/j.cell.2013.02.012 |
| [23] | Kim TK, Hemberg M, Gray JM, et al. Widespread transcription at neuronal activity-regulated enhancers. Nature , 2010, 465 (7295) : 182–187. DOI:10.1038/nature09033 |
| [24] | Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis:the Rosetta Stone of a hidden RNA language?. Cell , 2011, 146 (3) : 353–358. DOI:10.1016/j.cell.2011.07.014 |
| [25] | Djebali S, Davis CA, Merkel A, et al. Landscape of transcription in human cells. Nature , 2012, 489 (7414) : 101–108. DOI:10.1038/nature11233 |
| [26] | Li A, Zhang J, Zhou Z. PLEK:a tool for predicting long non-coding RNAs and messenger RNAs based on an improved k-mer scheme. BMC Bioinformatics , 2014, 15 (1) : 311. DOI:10.1186/1471-2105-15-311 |
| [27] | Yang JH, Li JH, Jiang S, et al. ChIPBase:a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data. Nucleic Acids Research , 2013, 41 (D1) : D177–D187. DOI:10.1093/nar/gks1060 |
| [28] | Volders PJ, Verheggen K, Menschaert G, et al. An update on LNCipedia:a database for annotated human lncRNA sequences. Nucleic Acids Research , 2015, 43 (D1) : D174–D180. DOI:10.1093/nar/gku1060 |
| [29] | Amaral PP, Clark MB, Gascoigne DK, et al. lncRNAdb:a reference database for long noncoding RNAs. Nucleic Acids Research , 2011, 39 (Suppl 1) : D146–D151. |
| [30] | Chen G, Wang Z, Wang D, et al. LncRNADisease:a database for long-non-coding RNA-associated diseases. Nucleic Acids Research , 2013, 41 (D1) : D983–D986. DOI:10.1093/nar/gks1099 |
| [31] | Xie C, Yuan J, Li H, et al. NONCODEv4:exploring the world of long non-coding RNA genes. Nucleic Acids Research , 2014, 42 (D1) : D98–D103. DOI:10.1093/nar/gkt1222 |
| [32] | Dinger ME, Pang KC, Mercer TR, et al. NRED:a database of long noncoding RNA expression. Nucleic Acids Research , 2009, 37 (suppl 1) : D122–D126. |
| [33] | Erdmann VA, Szymanski M, Hochberg A, et al. Non-coding, mRNA-like RNAs database Y2K. Nucleic Acids Research , 2000, 28 (1) : 197–200. DOI:10.1093/nar/28.1.197 |
| [34] | Li A, Zhang J, Zhou Z, et al. ALDB:a domestic-animal long noncoding RNA database. PLoS One , 2015, 10 (4) : e0124003. DOI:10.1371/journal.pone.0124003 |
| [35] | Bhartiya D, Pal K, Ghosh S, et al. lncRNome:a comprehensive knowledgebase of human long noncoding RNAs. Database , 2013, 2013 : bat034. |
| [36] | Szymanski M, Erdmann VA, Barciszewski J. Noncoding RNAs database(ncRNAdb). Nucleic Acids Research , 2007, 35 (Suppl 1) : D162–D164. |
| [37] | Gardner PP, Daub J, Tate JG, et al. Rfam:updates to the RNA families database. Nucleic Acids Research , 2009, 37 (Suppl 1) : D136–D140. |
| [38] | Nawrocki EP, Burge SW, Bateman A, et al. Rfam 12. 0:updates to the RNA families database. Nucleic Acids Research , 2014 : gku1063. |
| [39] | Panzitt K, Tschernatsch MMO, Guelly C, et al. Characterization of HULC, a novel gene with striking up-regulation in hepatocellular carcinoma, as noncoding RNA. Gastroenterology , 2007, 132 (1) : 330–342. DOI:10.1053/j.gastro.2006.08.026 |
| [40] | Romanuik TL, Wang G, Morozova O, et al. LNCaP Atlas:gene expression associated with in vivo progression to castration-recurrent prostate cancer. BMC Medical Genomics , 2010, 3 (1) : 43. DOI:10.1186/1755-8794-3-43 |
| [41] | Castle JC, Armour CD, L?wer M, et al. Digital genome-wide ncRNA expression, including SnoRNAs, across 11 human tissues using polyA-neutral amplification[J]. 2010, 5(7):e11779. http://cn.bing.com/academic/profile?id=1976658794&encoded=0&v=paper_preview&mkt=zh-cn |
| [42] | Poirier F, Chan CT, Timmons PM, et al. The murine H19 gene is activated during embryonic stem cell differentiation in vitro and at the time of implantation in the developing embryo. Development , 1991, 113 (4) : 1105–1114. |
| [43] | Lustig O, Ariel I, Ilan J, et al. Expression of the imprinted gene H19 in the human fetus. Molecular Reproduction and Development , 1994, 38 (3) : 239–246. DOI:10.1002/(ISSN)1098-2795 |
| [44] | Matouk I, DeGroot N, Mezan S, et al. The H19 non-coding RNA is essential for human tumor growth and may serve as an anti-tumor target[C]//Human Gene Therapy. 140 Huguenot Street, 3rd FL, New Rochelle, NY 10801 USA:Mary Ann Liebert INC, 2007, 18(10):1034-1034. http://cn.bing.com/academic/profile?id=2049536473&encoded=0&v=paper_preview&mkt=zh-cn |
| [45] | Barsyte-Lovejoy D, Lau SK, Boutros PC, et al. The c-Myc oncogene directly induces the H19 noncoding RNA by allele-specific binding to potentiate tumorigenesis. Cancer Research , 2006, 66 (10) : 5330–5337. DOI:10.1158/0008-5472.CAN-06-0037 |
| [46] | Berteaux N, Lottin S, Monté D, et al. H19 mRNA-like noncoding RNA promotes breast cancer cell proliferation through positive control by E2F1. Journal of Biological Chemistry , 2005, 280 (33) : 29625–29636. DOI:10.1074/jbc.M504033200 |
| [47] | Dugimont T, Montpellier C, Adriaenssens E, et al. The H19 TATA-less promoter is efficiently repressed by wild-type tumor suppressor gene product p53. Oncogene , 1998, 16 (18) : 2395–2401. DOI:10.1038/sj.onc.1201742 |
| [48] | Farnebo M, Bykov VJN, Wiman KG. The p53 tumor suppressor:a master regulator of diverse cellular processes and therapeutic target in cancer. Biochemical and Biophysical Research Communications , 2010, 396 (1) : 85–89. DOI:10.1016/j.bbrc.2010.02.152 |
| [49] | Cai X, Cullen BR. The imprinted H19 noncoding RNA is a primary microRNA precursor. RNA , 2007, 13 (3) : 313–316. DOI:10.1261/rna.351707 |
| [50] | Tsang WP, Ng EKO, Ng SSM, et al. Oncofetal H19-derived miR-675 regulates tumor suppressor RB in human colorectal cancer. Carcinogenesis , 2010, 31 (3) : 350–358. DOI:10.1093/carcin/bgp181 |
| [51] | Baniol M, Hagege H, Petit JS, et al. Long-range chromatin interactions at the mouse Igf2/H19 locus reveal a novel paternally expressed long non-coding RNA. Nucleic Acids Research , 2011, 39 (14) : 5893–5906. DOI:10.1093/nar/gkr209 |
| [52] | Rinn JL, Kertesz M, Wang JK, et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell , 2007, 129 (7) : 1311–1323. DOI:10.1016/j.cell.2007.05.022 |
| [53] | Gupta RA, Shah N, Wang KC, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature , 2010, 464 (7291) : 1071–1076. DOI:10.1038/nature08975 |
| [54] | Tsai MC, Manor O, Wan Y, et al. Long noncoding RNA as modular scaffold of histone modification complexes. Science , 2010, 329 (5992) : 689–693. DOI:10.1126/science.1192002 |
| [55] | Simon JA, Kingston RE. Occupying chromatin:Polycomb mechanisms for getting to genomic targets, stopping transcriptional traffic, and staying put. Molecular Cell , 2013, 49 (5) : 808–824. DOI:10.1016/j.molcel.2013.02.013 |
| [56] | Hayami S, Kelly JD, Cho HS, et al. Overexpression of LSD1 contributes to human carcinogenesis through chromatin regulation in various cancers. International Journal of Cancer , 2011, 128 (3) : 574–586. DOI:10.1002/ijc.v128.3 |
| [57] | Ling H, Spizzo R, Atlasi Y, et al. CCAT2, a novel noncoding RNA mapping to 8q24, underlies metastatic progression and chromosomal instability in colon cancer. Genome Research , 2013, 23 (9) : 1446–1461. DOI:10.1101/gr.152942.112 |
| [58] | Redis RS, Sieuwerts AM, Look MP, et al. CCAT2, a novel long non-coding RNA in breast cancer:expression study and clinical correlations. Oncotarget , 2013, 4 (10) : 1748. DOI:10.18632/oncotarget |
| [59] | Hung T, Chang HY. Long noncoding RNA in genome regulation:prospects and mechanisms. RNA Biology , 2010, 7 (5) : 582–585. DOI:10.4161/rna.7.5.13216 |
| [60] | Derrien T, Johnson R, Bussotti G, et al. The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure, evolution, and expression. Genome Research , 2012, 22 (9) : 1775–1789. DOI:10.1101/gr.132159.111 |
| [61] | Guttman M, Amit I, Garber M, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature , 2009, 458 (7235) : 223–227. DOI:10.1038/nature07672 |
| [62] | Gibb EA, Vucic EA, Enfield KS, et al. Human cancer long non-coding RNA transcriptomes. PLoS One , 2011, 6 (10) : e25915. DOI:10.1371/journal.pone.0025915 |
| [63] | Lee TL, Xiao A, Rennert OM. Identification of novel long noncoding RNA transcripts in male germ cells[M]. New York: Springer, 2012 : 105-114. |
| [64] | Nam JW, Bartel DP. Long noncoding RNAs in C. elegans. Genome Research , 2012, 22 (12) : 2529–2540. DOI:10.1101/gr.140475.112 |
| [65] | Prensner JR, Iyer MK, Balbin OA, et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression. Nature Biotechnology , 2011, 29 (8) : 742–749. DOI:10.1038/nbt.1914 |
| [66] | Zhao J, Sun BK, Erwin JA, et al. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science , 2008, 322 (5902) : 750–756. DOI:10.1126/science.1163045 |
| [67] | Liu J, Gough J, Rost B. Distinguishing protein-coding from non-coding RNAs through support vector machines. PLoS Genet , 2006, 2 (4) : e29. DOI:10.1371/journal.pgen.0020029 |
| [68] | Altschul SF, Madden TL, Schäffer AA, et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs. Nucleic Acids Research , 1997, 25 (17) : 3389–3402. DOI:10.1093/nar/25.17.3389 |
| [69] | Kong L, Zhang Y, Ye ZQ, et al. CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine. Nucleic Acids Research , 2007, 35 (Suppl 2) : W345–W349. |
| [70] | Lu ZJ, Yip KY, Wang G, et al. Prediction and characterization of noncoding RNAs in C. elegans by integrating conservation, secondary structure, and high-throughput sequencing and array data. Genome Research , 2011, 21 (2) : 276–285. DOI:10.1101/gr.110189.110 |
| [71] | Chinwalla AT, Cook LL, Delehaunty KD, et al. Initial sequencing and comparative analysis of the mouse genome. Nature , 2002, 420 (6915) : 520–562. DOI:10.1038/nature01262 |
| [72] | Gorodkin J, Cirera S, Hedegaard J, et al. Porcine transcriptome analysis based on 97 non-normalized cDNA libraries and assembly of 1, 021, 891 expressed sequence tags. Genome Biology , 2007, 8 (4) : R45. DOI:10.1186/gb-2007-8-4-r45 |
| [73] | Andersen ES, Rosenblad MA, Larsen N, et al. The tmRDB and SRPDB resources. Nucleic Acids Research , 2006, 34 (Suppl 1) : D163–D168. |
| [74] | Brown JW. The ribonuclease P database. Nucleic Acids Research , 1999, 27 (1) : 314–314. DOI:10.1093/nar/27.1.314 |
| [75] | Weinberg Z, Ruzzo WL. Faster genome annotation of non-coding RNA families without loss of accuracy[C]//Proceedings of the eighth annual international conference on Resaerch in computational molecular biology. ACM, 2004:243-251. http://cn.bing.com/academic/profile?id=2102066220&encoded=0&v=paper_preview&mkt=zh-cn |
| [76] | Nawrocki EP, Eddy SR. Query-dependent banding(QDB)for faster RNA similarity searches. PLoS Comput Biol , 2007, 3 : e56. DOI:10.1371/journal.pcbi.0030056 |
| [77] | Nawrocki EP, Kolbe DL, Eddy SR. Infernal 1. 0:inference of RNA alignments. Bioinformatics , 2009, 25 (10) : 1335–1337. DOI:10.1093/bioinformatics/btp157 |
| [78] | Ellis JC, Brown JW. The evolution of RNase P and its RNA[M]. New York: Springer, 2010 : 17-40. |
| [79] | Xie M, Mosig A, Qi X, et al. Structure and function of the smallest vertebrate telomerase RNA from teleost fish. Journal of Biological Chemistry , 2008, 283 (4) : 2049–2059. DOI:10.1074/jbc.M708032200 |
| [80] | Yang L, Duff MO, Graveley BR, et al. Genomewide characterization of non-polyadenylated RNAs. Genome Biol , 2011, 2 : R16. |