微生物多样性的变化能较早地反映土壤质量变化过程,是评价自然或人为干扰引起的土壤变化的重要指示因子。同时微生物的分布并不是随机的,存在“微生物生物地理地图”[1]。古菌作为一类区别于真细菌和真核生物的第三域生命形式存在[2],随着分子生物学技术的不断发展,人们发现它们在自然界中的分布范围十分广泛。Cytryn与Ochsenreiter等[3, 4]发现在盐湖、天然盐场和一些高盐环境中存在着大量古菌,其中广古菌门(Euryarchaeota)的盐杆菌科(Halobacteriaceae)为优势菌群,它们生存的适宜pH范围为 6-1 1。
艾比湖(Ebinur Lake)是新疆第一大咸水湖,它不仅影响着新疆北部地区的生态环境,同时在当地的经济发展中发挥着重要的作用[5]。随着人类活动的日益频繁,艾比湖的生态环境正在逐渐恶化[6-9]。了解并恢复艾比湖湿地生态环境已经迫在眉睫。目前,对艾比湖湿地土壤古菌多样性的研究主要是通过可培养方法[10, 11],但是此方法由于技术条件的限制,分离得到的古菌在种类和数量上会产生一定的误差。因此,本研究首次通过构建古菌16S rDAN克隆文库,分析艾比湖湿地博乐河入口土壤古菌多样性与群落结构,以及微生物群落结构组成对环境的响应,旨为当地政府的湿地生态保护与改善湿地环境提供基础理论依据。
1 材料与方法 1.1 材料 1.1.1 材料的采集2014年7月,于艾比湖湿地博乐河入口处与艾比湖呈水平方向设置3个湿地采样点(表 1)。每个采样点随机选择3个位点,去除表层土壤,垂直方向在 0-3 5 cm土层采集等量土样,混合均匀后作为一个采样点的样本。然后再将3个湿地采样点的样本去除草根与石块后充分混匀作为艾比湖湿地博乐河入湖口的样本用于试验研究。用手提式冰箱迅速带回实验室分成两份,一份保存于-20 ℃ 冰箱用于DNA提取,一份自然风干过100目筛用于理化性质分析。
PCR仪(Eppendorf),高速冷冻离心机。The PowerSoil® DNA Isolation Kit(MO-BIO,USA),胶回收试剂盒,PCR产物纯化试剂盒,限制性内切酶AfaⅠ和MspⅠ(TaKaRa)。
1.2 方法 1.2.1 土壤理化性质的测定土壤含水量测定采用105 ℃ 烘干比重法;pH测定采用电位法,使用PHS-3C型酸度计测定,土壤浸提液制备水土比例5 ∶ 1;盐度测定采用电导法测定,使用DJ-320电导仪测定;有机质含量测定用重铬酸钾容量法(外加热法);速效磷含量测定采用碳酸氢钠浸提(钼锑抗比色法测定); Na+与K+的测定采用火焰光度法。样品设置3个重复,取其平均值。
1.2.2 古菌16S rDNA克隆文库的构建 1.2.2.1 土壤总DNA的提取土壤总DNA的提取用 PowerSoil® DNA Isolation Kit(MOBIO,USA)按操作步骤进行。
1.2.2.2 古菌16S rRNA基因的扩增采用古菌通用引物[1]:Arch21F:5′-TTCCGGTTGATCCYGCCGGA-3′和Arch958R:5′-YCCGG CGTTGAMTCCAATT- 3′(Y代表C/T;M代表A/C)。25 µL的PCR反应体系包括:10 µL 2×PCR mix(GENEray Biotechnology),0.4 µL(10 mmol/L)上下游引物,65 ng 土壤总DNA,12 µL ddH2O。反应程序为:94 ℃ 预变性5 min;94 ℃ 变性30 s,59 ℃ 退火30 s,72 ℃ 延伸90 s,循环数为35;72 ℃ 延伸10 min。PCR产物经1%的琼脂糖凝胶检测,用琼脂糖凝胶DNA回收试剂盒进行切胶回收。
1.2.2.3 古菌16S rDNA克隆文库的构建将纯化后的PCR产物与pMD18-T载体连接并转入大肠杆菌DH5α中。通过蓝白斑筛选共随机挑取850个阳性克隆子于96孔板中,-80 ℃ 保存备用。
1.2.2.4 ARDRA酶切分型所有阳性克隆子的PCR产物分别用限制性内切酶AfaⅠ和MspⅠ进行酶切分型。10 µL酶切体系:BSA 1 µL,10×Buffer 2 µL,10 U/µL AfaⅠ(或MspⅠ)1 µL,PCR纯化产物6 µL。37 ℃ 酶切4 h后,1 µL 10× Loading Buffer终止反应。酶切产物用4%的琼脂糖凝胶电泳检测。综合双酶切图谱谱型,选取不同谱型的阳性 克隆子若干进行测序。
1.2.2.5 克隆文库与系统发育分析使用Mothur软件和在线软件Bellerophon,去除测序完成的古菌16S rDNA序列中可能存在的嵌合体。将相似度≥97%的序列划分为一个OTU,并通过在线比对下载相似度最高的序列,使用Clustalx 1.83和Phylip-3.68软件构建进化树。用文库覆盖度Coverage C评价构建文库对环境微生物多样性的呈现程度(C=1-n1/N,N为所分析的克隆总数,n1为仅有一个克隆的操作分类单位数);使用origin86和aRarefactwin软件绘制文库的稀疏曲线判定文库的丰富度;文库的多样性通过Shannon、Simpson、Chao指数进行评价。
1.3 核酸序列登录号使用sequin软件将已经去除嵌合体的古菌16S rDNA序列提交至GenBank。共获得117条古菌16S rDNA序列,核酸序列登录号为:KT216078 - KT216195。
2 结果 2.1 艾比湖湿地博乐河入口土壤理化性质本研究测定的3个样点土壤理化性质如表 2所示。Na+的含量与2012年7月的测定结果[12]无太大变化,且Na+的含量较酸性和中性土壤高[13, 14]。样地中3个样点的pH平均值为8.75,说明该样地的土壤呈碱性。博乐河的断流导致土壤含水量相较于2012年7月的19.93%下降至12.49%。可溶性总盐含量为3.28 ms/cm,根据可溶性总盐含量划分标准将样地土壤划分为中度盐土[15]。可溶性总盐含量比2012年7月的7.07 ms/cm有大幅下降。

|
| 图 1 土壤总DNA M:DSTM2000; 1-3 :为同一个样地的3次平行实验 |
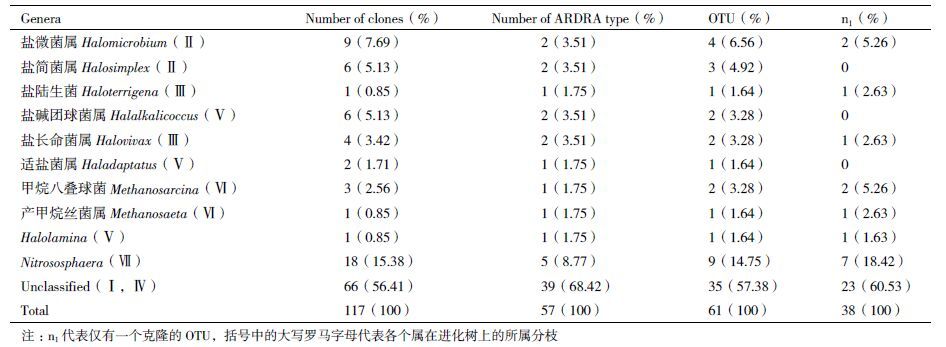
对艾比湖湿地博乐河入湖口土壤古菌16S rDNA克隆文库中的阳性克隆进行菌落PCR,验证插入片段大小在920 bp左右(图 2)。经过Afa I与Msp I限制性内切酶分型(图 3,图 4)后,得到57种不同的酶切谱型,每种不同的ARDRA谱型随机选取若干阳性克隆子进行测序,得到117条序列。

|
| 图 2 古菌16S rDNA克隆文库阳性克隆部分检测结果 M:DSTM2000; 1-2 2:不同阳性克隆的PCR验证结果;P:代表阳性对照;N:代表阴性对照 |
|
| 图 3 古菌16S rDNA克隆文库部分Afa I酶切图谱 M:DSTM2000; 1-2 4:分别代表部分阳性克隆PCR产物的Afa I酶切结果 |

|
| 图 4 古菌16S rDNA克隆文库部分Msp I酶切图谱 M:DSTM2000; 1-2 4:分别代表部分阳性克隆PCR产物的Msp I酶切结果 |
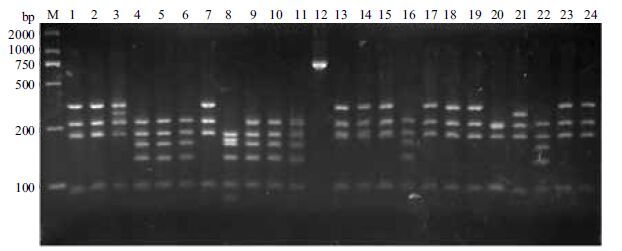
利用Mothur软件将117条序列划分为61个OTU。计算得出文库的覆盖度为69.84%,用Rarefaction曲线反映的湿地古菌的丰富度见图 5。文库的多样性指数H(3.87)、1/D(55.28)、Chao(161.43)反映出样地的古菌群落多样性丰富。
|
| 图 5 古菌16S rDNA克隆文库稀疏曲线 |
文库中所有古菌16S rDNA序列划分为61个OTUs,所属类群分布见表 3。选取其中具有代表性的序列与已知环境样品中的已知菌株序列共同构建系统发育树,所有序列共划分为七大类群(图 6),Cluster Ⅰ - Cluster Ⅵ隶属于广古菌门(Euryarchaeota),Cluster Ⅶ隶属于奇古菌门(Thaumarchaeota)。

|
| 图 6 古菌16S rDNA克隆文库系统发育树 系统发育树通过Phylip-3.68软件采用邻位法构建,所有的参考序列均来自于GenBank,分支上的数值表示经1 000次计算后的置信度值,文库中的序列用黑色菱形标注,*代表不能确定到具体属的序列 |
ClusterⅠ包含8个OTU(13.1%),共13条序列(11.11%)。代表序列B13(KT216083)、B30(KT216091)、B209(KT216169)隶属于盐杆菌科(Halobacteriaceae),与分离于中国东部沿海天然盐场的已知菌株序列之间相似度为95% - 98%。Cluster Ⅳ包含8个OTU(13.1%),共14条序列(11.97%)。代表序列B35(KT216092)、B226(KT216174)、B169(KT216155)、B118(KT216131)、B168(KT216154)、B233(KT216176)、B119(KT216132)、B23(KT216087)与分离于中国西北部天然湖泊、云南天然盐湖以及从死海分离得到的已知菌株序列之间的相似度为93% - 95%,隶属于盐杆菌科。
ClusterⅡ包含5个OTU(8.2%),共13条序列(11.11%)。代表序列B107(216125)、B167(KT216153)、B147(KT216146)、B111(KT216127)与日本东洋大学提交的已知菌株序列之间的相似度为95% - 99%,隶属于盐微菌属(Halomicrobium)。代表序列B140(KT216143)、B144(KT216145)与GenBank中的已知菌株序列之间的相似度均为93%,隶属于Halosimplex。
Cluster Ⅲ 包含13个OTU(21.3%),共23条序列(19.66%)。代表序列B113(KT216129)与分离于天然盐湖盐场的已知菌株序列(KM496557)之间的相似度为97%,隶属于盐杆菌科;代表序列B177(KT216156)与已知菌株序列(NR113516)之间的相似度为97%,隶属于Halovivax;代表序列B148(KT216147)与分离于罗布泊的已知菌株序列(JF449421)之间的相似度为99%,隶属于盐陆生菌属(Haloterrigena)。
ClusterⅤ包含7个OTU(11.5%),共18条序列(15.38%)。主要包括Halalkalicoccus、Halolamina、Haladaptatus。代表序列B81(KT216114)、B74(KT216110)与已知菌株序列之间的相似度为97% - 99%,隶属于Haladaptatus。代表序列B54(KT216-099)、B44(KT216096)与已知菌株序列之间的相似度为92% - 96%,隶属于盐杆菌科。
ClusterⅥ包含11个OTU(18.03%),共22条序列(18.80%)。其中主要包括Methanosarcina、Methanosaeta、Methanomasillcoccus,与已知菌株序列之间的相似度为92% - 99%。代表序列B217(KT216172)与已知菌株序列(CP002565)之间的相似度为98%,隶属于Methanosaeta。代表序列B101(KT216122)与已知菌株序列(CP009515)之间的相似度为99%,隶属于甲烷八叠球菌属。
ClusterⅦ包含9个OTU(14.8%),共18条序列(15.38%)。主要包括Nitrososphaera,与已知菌株序列之间的相似度为96% - 99%。代表序列B105(KT216124)与未分类的已知奇古菌门菌株(CP102850)之间的相似度为93%。代表序列B163(KT216152)、B82(KT216119)与已知菌株序列之间的相似度为分别为97%和98%,都隶属于Nitrososphaera。代表序列B20(KT216086)与源于土壤的未分类氨氧化古菌序列(CP012850)之间的相似度为96%。
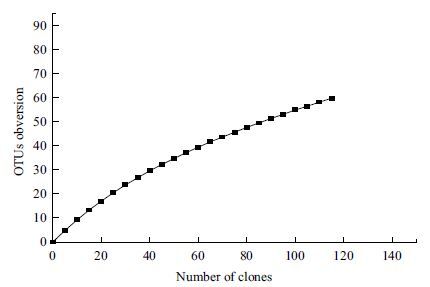
2.5 土壤理化因子与古菌之间相关性分析(RDA)使用canoco 4.5软件对土壤理化性质与古菌类群之间的相关性进行分析。结果(图 7)显示,Axis轴的累积方差为99.7%,在Axis轴上各理化因子的贡献率大小为pH>Na+>SOM>MC>Salinity。奇古菌门(Thaumarchaeota)中的Nitrosophaera与土壤中Na+含量、有机质含量(SOM)、含水量(MC)、含盐量之间呈极大地负相关关系,与土壤的pH呈正相关关系;盐杆菌科的Halovivax、Halosimplex与土壤中Na+含量、有机质含量(SOM)、含水量(MC)、含盐量、pH之间呈负相关关系;盐杆菌科中的Haloterrigena、Halolamina、Haladaptaus以及Methanosaeta、Methanosarcina与土壤中Na+含量、有机质含量(SOM)、含水量(MC)、含盐量呈正相关关系,与土壤pH呈负相关关系;盐杆菌科的Halalkalicoccus与土壤理化因子之间相关性不明显。
|
| 图 7 土壤理化因子与古菌之间的RDA分析 实心原点代表不同的古菌类群,黑色箭头表示不同的理化因子 |
通过对艾比湖湿地土壤古菌群落结构多样性分析发现,广古菌门中的盐杆菌科为土壤中古菌的优势菌群。在Ochsenreiter等[4]对3个不同高盐环境中古菌的研究,Dave等[16]对Bhavnagar海岸盐田古菌的多样性研究,以及Zafrilla等[17]对西班牙Vinalopo山谷中的两个内陆盐田生态系统中古菌多样性的研究中,发现在多数陆地高盐环境中盐杆菌科都作为优势菌群存在。盐杆菌科的古菌通过在胞内积累大量的K+,与环境中高浓度的Na+保持动态平衡,从而生活在高盐环境中,并作为优势菌群存在[16, 18]。本样地土壤溶液呈碱性,且Na+含量较高,此适应机制为盐杆菌科作为优势菌群在本样地中存在提供了合理的解释。
在属的水平上,盐杆菌科的Halomicrobium、Halalkalicoccus、Halosimplex为优势菌群,共7个OTU(11.48%),17个(14.53%)阳性克隆子。此结果与崔恒林和许学伟等[10,11]对艾比湖湿地可培养古菌多样性的研究结果存在差异,他们的研究结果显示Haloterrigena为盐杆菌科的优势菌群。表明艾比湖湿地土壤中主要的优势菌群可能为非培养类群。Ochsenreiter等[4]通过非培养和可培养两种方式对西班牙天然结晶盐场(FC)、美国碱性湖泊(EMF)、德国钾矿废水(DIE)3个高盐环境中的古菌多样性进行比较,发现盐杆菌科在古菌群落组成中为优势菌群,但通过非培养方式对环境中的古菌多样性进行预测的结果与采用可培养方式得到的结果之间存在很大的差异。对于这一结果的差异,Ahmad[19]在对盐场沉积物中古菌多样性的研究中认为,大部分能够被分离的嗜盐古菌对NaCl的最低要求为9%(W/V),而用分子手段检测到的嗜盐古菌对NaCl的最低要求为2.5%(W/V)。对本样地土壤中的古菌与土壤理化因子进行RDA分析发现,多数古菌与土壤溶液的pH呈负相关关系,它们只能存活于弱碱性的环境下,随着pH的增加,其数量就会不断减少。
与韩晶[12]2012年7月以本样地土壤为样本,构建的细菌16S rDNA克隆文库比较发现,细菌克隆文库的覆盖度为64.5%,古菌文库的覆盖度为69.84%,在一定程度上说明细菌的多样性大于古菌的多样性。He等[20]对人工湿地中古菌和细菌群落结构多样性进行研究的结果显示,古菌群落的丰富度和多样性都小于细菌。Aller等[21]对文库覆盖度足够大的143个古菌和45个细菌的16S rDNA克隆文库进行比较研究发现,在多数环境中古菌的多样性小于细菌的多样性。他们认为古菌种属类型的多少与所构建文库的覆盖度大小无关,覆盖度大并不能说明其中包含的古菌多样性丰富。引物的偏好性、随机选取的阳性克隆子的数量以及不同微生物的基因组浓度等原因也会对文库的多样性产生影响。同时他们认为古菌有着比细菌更加严格的生存环境要求。
在本研究中出现了少量的奇古菌门[22]古菌,它们是一类广泛存在于中温环境中的非培养古菌[23],奇古菌门中的氨氧化古菌(Ammonia-Oxidi-zing Archaea,AOA)能够编码氨单加氧酶(amoA),并通过催化氨氧化获取能量进行自养生长,这一发现改变了人们对“细菌是氨氧化过程的主要驱动”的观点。已有的证据表明,AOA在海洋氨氧化过程中发挥了重要作用,而氨氧化细菌(Ammonia-Oxidizing Bacteria,AOB)则是土壤氨氧化过程的主要驱动者[24]。由于AOA在氮循环中的重要作用,AOA已经成为了地理学、生物学、生态学和环境科学等交叉领域的研究对象[25]。
4 结论通过以艾比湖湿地土壤为环境样本,构建古菌16S rDNA克隆文库,最终得到古菌序列117条。通过GenBank在线比对,117条古菌16S rDNA序列主要隶属于广古菌门与奇古菌门,其中广古菌门盐杆菌科古菌为优势菌群,占所有序列的63.6%。117条古菌16S rDNA序列与GenBank中已知古菌序列之间的同源性介于85% - 99%。其中有17条古菌16S rDNA序列与GenBank中的已知序列之间同源性≥97%,能够确定到具体的属,占所有序列的14.53%;剩余的100条古菌16S rDNA序列与GenBank中的已知序列之间的同源性均<97%,未确定到具体的属。因此,本研究认为艾比湖湿地土壤中仍存在大量潜在的古菌新种。
| [1] | 阎冰. 红树林土壤细菌和古菌的16S rDNA多样性研究[D]. 武汉: 华中农业大学, 2007. http://cdmd.cnki.com.cn/article/cdmd-10504-2007210101.htm |
| [2] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms. Proceeding of the National Academy of Sciences of the United States of America , 1977, 74 (11) : 5088–5090. DOI:10.1073/pnas.74.11.5088 |
| [3] | Cytryn E, Minz D, Oremland RS, Cohen Y. Distribution and diversity of archaea corresponding to the limnological cycle of a hypersaline stratified lake(Solar lake, Sinai, Egypt). Applied and Environmental Microbiology , 2000, 66 (8) : 3269–3276. DOI:10.1128/AEM.66.8.3269-3276.2000 |
| [4] | Ochsenreiter T, Pfeifer F, Schleper C. Diversity of Archaea in hyper-saline environments characterized by molecular-phylogenetic and cultivation studies. Extremophiles , 2002, 6 (4) : 267–274. DOI:10.1007/s00792-001-0253-4 |
| [5] | Zhang F, Tiyip T, Johnson VC, et al. The influence of natural and human factors in the shrinking of the Ebinur Lake, Xinjiang, China, during the 1972-2013 period. Environmental Monitoring and Assessment , 2015, 187 (1) : 4128. DOI:10.1007/s10661-014-4128-4 |
| [6] | 李磊, 李艳红, 陈成贺日. 1972~2011年期间艾比湖面积变化研究. 湿地科学 , 2014, 12 (2) : 263–267. |
| [7] | 刘文军, 张鹏, 李丽华, 等. 新疆艾比湖湖面面积变化分析. 干旱区研究 , 2010, 27 (1) : 64–68. |
| [8] | 孙丽, 高亚琪. 新疆艾比湖流域耕地面积变化对艾比湖湖面面积的影响分析. 广西农业科学 , 2010, 41 (8) : 848–852. |
| [9] | 王宏, 塔西甫拉提·特依拜, 谢霞. 艾比湖区域土壤盐渍化时空演变研究. 干旱区资源与环境 , 2011, 25 (12) : 137–144. |
| [10] | 崔恒林, 杨勇, 迪丽拜尔·托乎提. 新疆两盐湖可培养嗜盐古菌多样性研究. 微生物学报 , 2006, 46 (2) : 171–176. |
| [11] | 许学伟, 吴敏, 迪丽拜尔·托乎提, 等. 新疆艾比湖和伊吾湖可培养嗜盐古菌多样性. 生物多样性 , 2006, 14 (4) : 359–362. |
| [12] | 韩晶, 胡文革, 王艳萍, 等. 新疆艾比湖湿地博乐河入口处土壤细菌多样性分析. 微生物学通报 , 2014, 41 (11) : 2244–2253. |
| [13] | Cat?o E, Castro AP, Barreto CC, et al. Diversity of Archaea in Brazilian savanna soils. Archives of Microbiology , 2013, 195 (7) : 507–512. DOI:10.1007/s00203-013-0882-x |
| [14] | 王雪, 樊贵盛. Na+含量对土壤入渗能力影响的试验研究. 太原理工大学学报 , 2009, 40 (4) : 391–394. |
| [15] | 鲍士旦. 土壤农化分析[M]. 北京: 中国农业出版社, 2007 : 178. |
| [16] | Dave BP, Soni A. Diversity of halophilic archaea at salt pans around Bhavnagar Coast, Gujarat. Proceedings of the National Academy of Sciences, India Section B: Biological Sciences , 2013, 83 (2) : 225–232. DOI:10.1007/s40011-012-0124-z |
| [17] | Zafrilla B, Martínez-Espinosa RM, Alonso MA, Bonete MJ. Biodiversity of Archaea and floral of two inland saltern ecosystems in the Alto Vinalopó Valley, Spain. Saline Systems , 2010, 6 (1) : 10–22. DOI:10.1186/1746-1448-6-10 |
| [18] | de Costa MS, Santos H, Galinski EA. An overview of the role and diversity of compatible solutes in Bacteria and Archaea. Advances in Biochemical Engineering/Biotechnology , 1998, 61 : 117–153. DOI:10.1007/BFb0102286 |
| [19] | Ahmad N, Johri S, Sultan P, et al. Phylogenetic characterization of archaea in saltpan sediments. Indian Journal of Microbiology , 2012, 51 (2) : 132–137. |
| [20] | He T, Guan W, Luan ZY. Spatiotemporal variation of bacterial and archaeal communities in a pilot-scale constructed wetland for surface water treatment. Applied Microbiology and Biotechnology , 2016, 100 (3) : 1479–1488. DOI:10.1007/s00253-015-7072-5 |
| [21] | Aller JY, Kemp PF. Are Archaea inherently less diverse than Bacteria in the same environments?. FEMS Microbiology Ecology , 2008, 65 (1) : 74–87. DOI:10.1111/fem.2008.65.issue-1 |
| [22] | Brochier-Armanet C, Boussau B, Gribaldo S, Forterre P. Mesophilic Crenarchaeota: proposal for a third archaeal phylum, the Thauma-rchaeota. Nature Reviews Microbiology , 2008, 6 (3) : 245–252. DOI:10.1038/nrmicro1852 |
| [23] | 张丽梅, 贺纪正. 一个新的古菌类群—奇古菌门(Thaumarchaeota). 微生物学报 , 2012, 52 (4) : 411–421. |
| [24] | 贾仲君, 翁佳华, 林先贵. 氨氧化古菌的生态学研究进展. 微生物学报 , 2010, 50 (4) : 431–437. |
| [25] | 刘正辉, 李德豪. 氨氧化古菌及其对氮循环贡献的研究进展. 微生物学通报 , 2015, 42 (4) : 774–782. |